OpenMMをステップバイステップで 〜 Part 4 ColabでOpenMM シミュレーションを実行 〜
OpenMMの使い方を順番にたどる記事のPart 4です。過去の記事で、タンパク質構造の前処理(Part 1、Part 2)とシミュレーション実行のためのスクリプト(Part 3)を手に入れるところまでが終わりました。
あとは計算を実行するだけ!でも大変そうだからColabでやってしまおう!ってなお話です。*1
1. 準備
1-1. 使用ファイル
今回使用するファイルは以下の2つです。
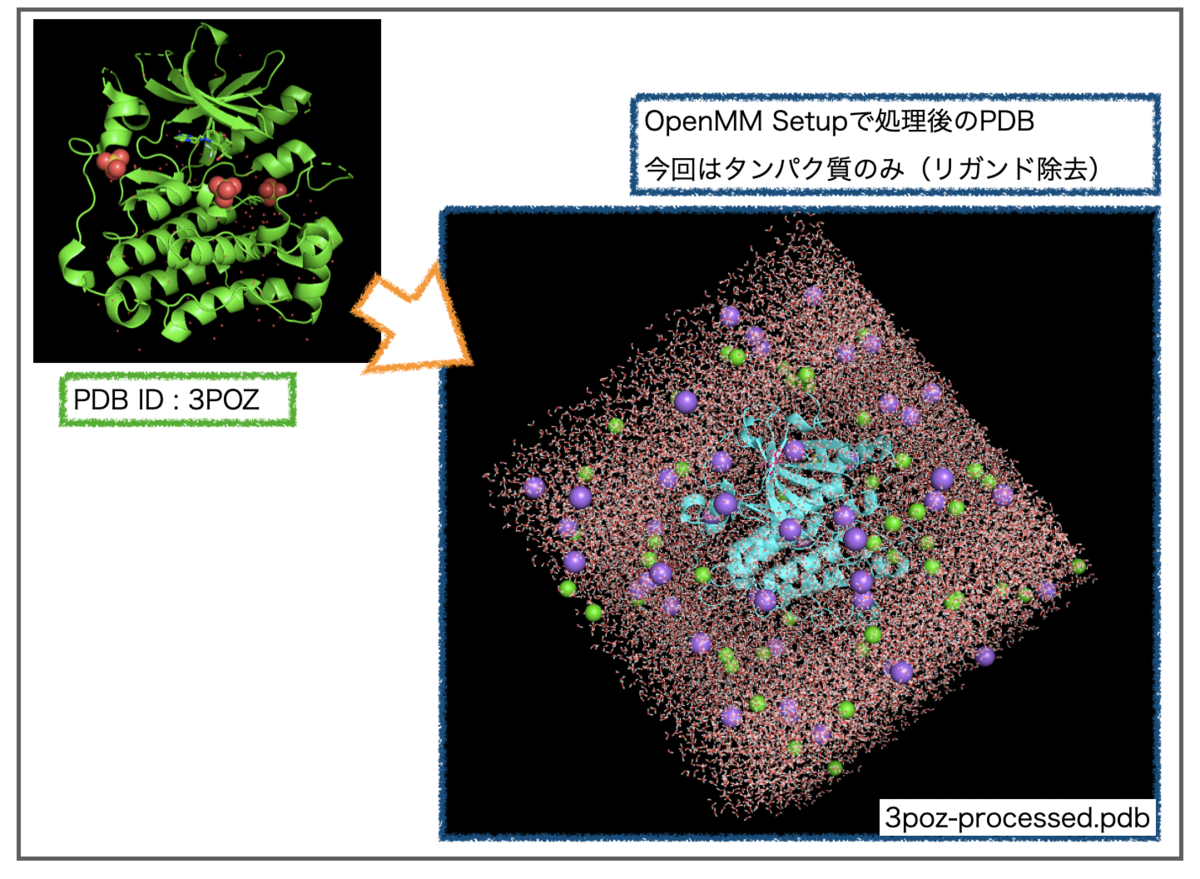
- ① PDB ID: 3POZをMDシミュレーション用に処理したファイル(
3poz-processed.pdb) - ② OpenMMシミュレーションを実行するスクリプト(
run_openmm_simulation.py)
それぞれの作成手順は過去の記事をご覧いただければと思います。
処理済みのPDBファイルはこんな見た目です。
1-2. スクリプトの修正
Google Colabでシミュレーションを実行する前に、前回作成したスクリプトを少し修正しました。
修正内容は2点です。
- ① シミュレーションの途中経過(残り時間 etc.)をColabノートブック上に表示させる
- ② シミュレーション後の最終状態をPDBx/mmCIFフォーマットでも出力する
1-2-1. 途中経過の出力
MDシミュレーションのトラジェクトリ生成段階(プロダクション)は計算時間がかかるので、「計算そのものが進んでいるか?」「どれくらい時間がかかるのか?」心配になります。
計算のログ自体はログファイル(log.txt)に書き出すようにスクリプトで設定されていますが、計算途中でも確認できた方が安心できそうです。
そこでColabノートブックのセルの出力にも同様の内容を書き出すことにしました。

上図のように、プロダクションを行う「simulation.step(steps)」の直前にもう一つStateDataReporter()を追加しました。標準出力(stdout)に進捗情報を表示するためのコードです。
from sys import stdout simulation.reporters.append(StateDataReporter(stdout, 1000, step=True, progress=True, remainingTime=True, speed=True, totalSteps=steps, separator="\t"))
1-2-2. 最終状態のPDBx/mmCIF出力
前回のスクリプト作成では、シミュレーション後の出力ファイルをXMLにしていました。
# Write file with final simulation state simulation.saveState("final_state.xml")
最終状態の系を可視化するならPyMolで読み込めた方が楽だよね!と、いうことで構造を出力するようにしました。

OpenMM SetupのOutputタブで「Save final simulation state:PDBx/mmCIF (no velocities)」とすると上記のようになります。
この状態のスクリプトを使っても良いですし、シミュレーション終了後に同じコードを実行してもファイルを書き出すことができます。
state = simulation.context.getState(getPositions=True, enforcePeriodicBox=system.usesPeriodicBoundaryConditions()) with open("final_state.pdbx", mode="w") as file: PDBxFile.writeFile(simulation.topology, state.getPositions(), file)
2. Google Colabでシミュレーション
2-1. 手順
それではGoogle Colabに計算してもらいましょう!
手順は以下です。*2

くどくど書いてますが、インストールしてスクリプト(.py)を実行するだけです。
2-2. 実行
では実際に計算を進めていきましょう!
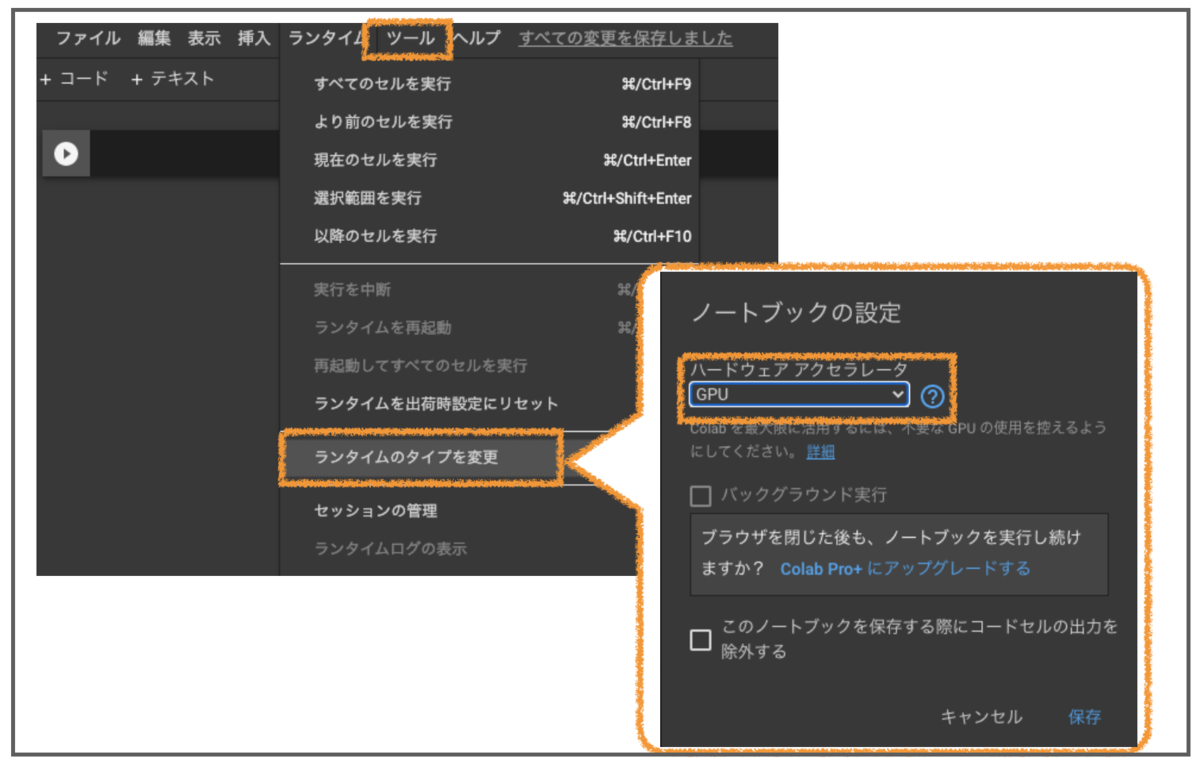
2-2-1. GPUに設定
Colabにログインして新しいノートブックをつくったら忘れずにハードウェアをGPUにしておきます。
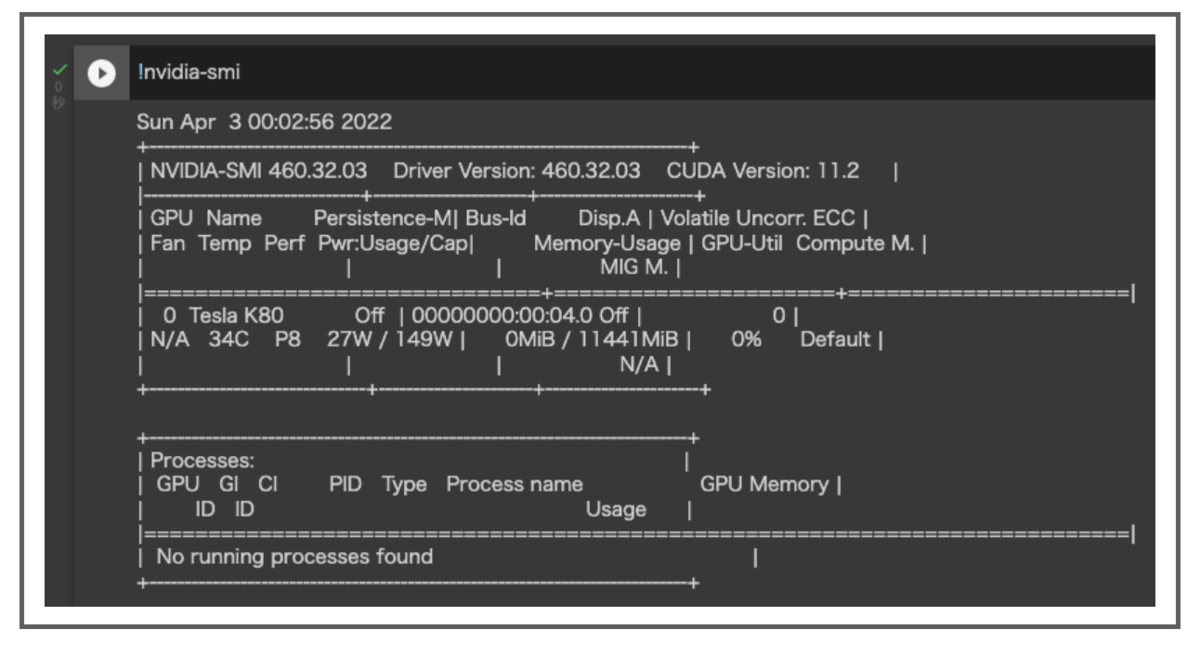
以下をセルで実行して、GPUに切り替わったことを確認することもできます。
!nvidia-smi
2-2-2. OpenMMのインストール
次に、OpenMMをインストールします。
OpenMMはcondaがあれば簡単にインストールできるので、最初にminicondaをインストールします。
!wget -qnc https://repo.anaconda.com/miniconda/Miniconda3-latest-Linux-x86_64.sh !bash Miniconda3-latest-Linux-x86_64.sh -bfp /usr/local !rm Miniconda3-latest-Linux-x86_64.sh
wgetでLinux用のminicondaを取ってきて、「/usr/local」にインストールしています。終わったら不要なファイルをrmで削除してます。
次に、OpenMMをconda-forgeチャンネルからインストールします。
!conda install -c conda-forge openmm=7.6 python=3.7 -y
インストールできたらOpenMMが使えるようにパスを通しておきます。
import sys sys.path.append('/usr/local/lib/python3.7/site-packages/')
忘れててModuleNotFoundErrorで怒られました。
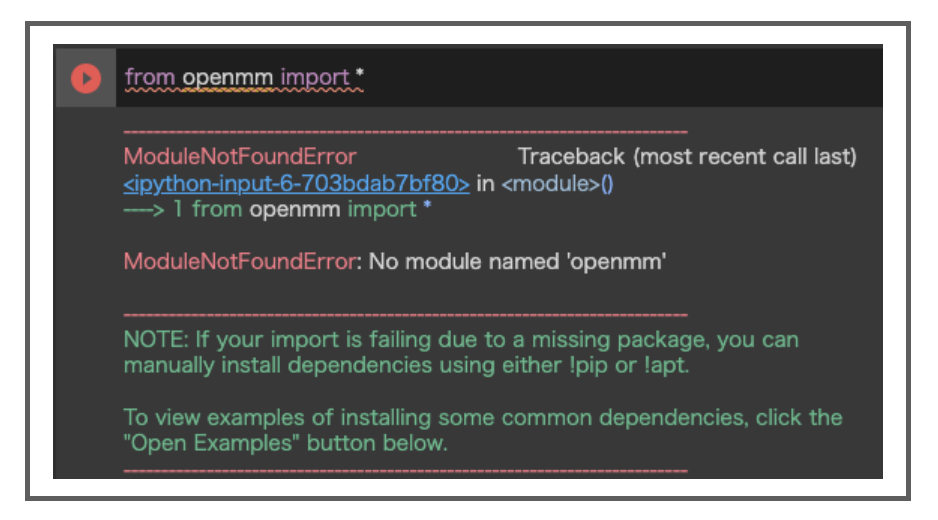
2-2-3. ファイルアップロード
Colabに計算で使用するPDBファイル(3poz-processed.pdb)とスクリプト(run_openmm_simulation.py)をアップロードします。
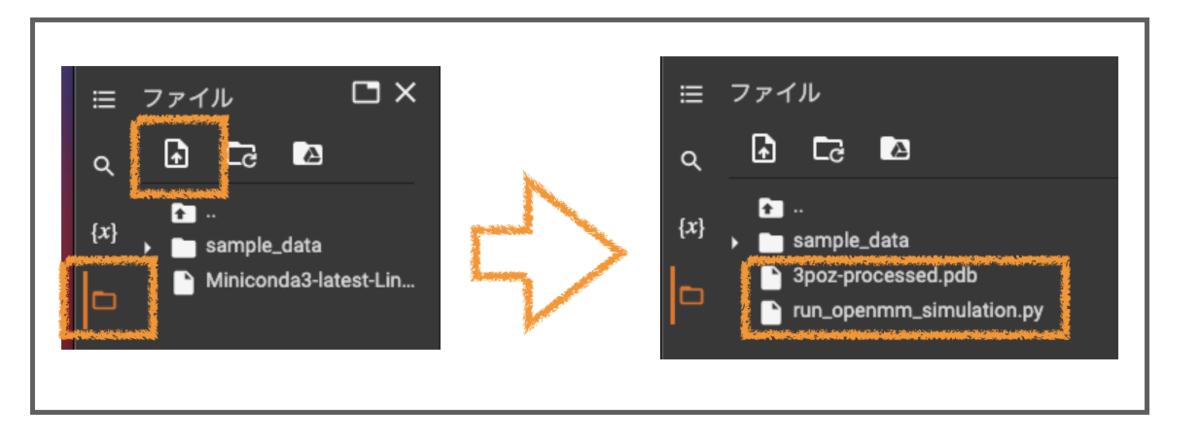
Colabの左側でクリックするだけ。前からこんなに簡単でしたっけ?
2-2-4. スクリプトの実行
あとはスクリプトを実行するだけ!
%run run_openmm_simulation.py
標準出力に経過を出すように書き換えたので進捗が確認できます。
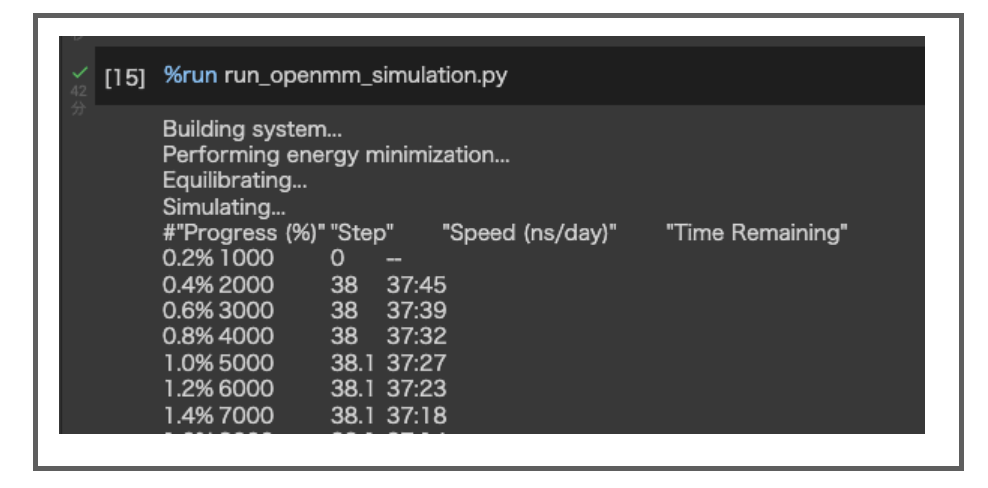
今回はトラジェクトリの長さを1nsとしていますが、プロダクションだけでおおよそ40分程度かかりました(平衡化などを含めると小一時間くらい)。
2-2-4. 結果のダウンロード
計算が終わると左側ファイルに色々と新しいファイルができています。
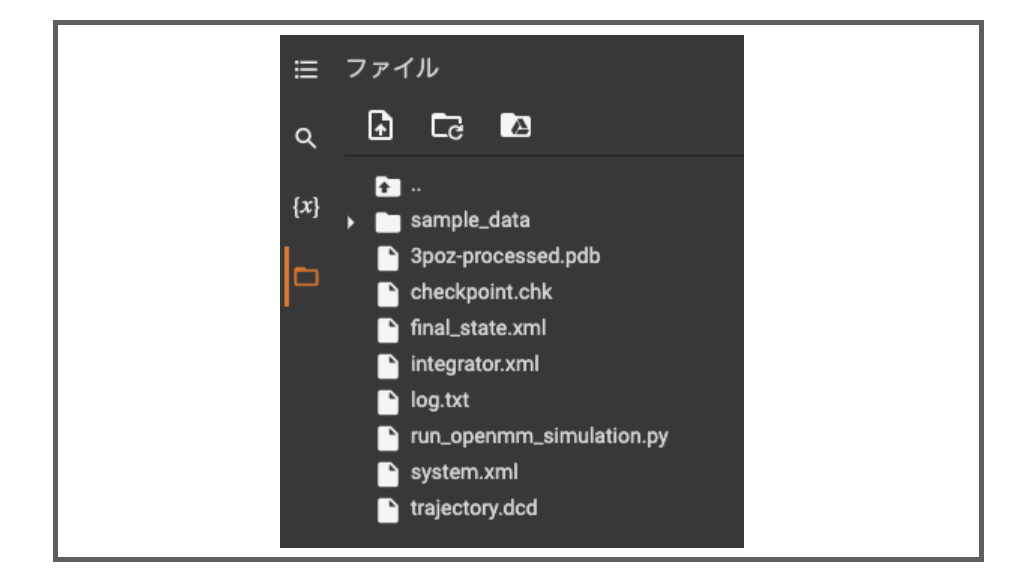
google.colabのfilesを使って、簡単にローカルPCにダウンロードできます。*3
from google.colab import files files.download("trajectory.dcd") files.download("log.txt") files.download("checkpoint.chk") files.download("system.xml") files.download("final_state.xml") files.download("final_state.pdbx")
Colabはランタイムが切断されるとファイルが全て消えてしまうので、とりあえず目につくものをダウンロードしておきました。
容量が大きいファイルはブラウザが「本当にダウンロードして良いの?」と聞いてくる場合があるので許可してください。*4
PCではなくGoogle Driveに保存したい時はマウントしてコピーすれば良いそうです。
例えば以下でGooge Driveのマイドライブディレクトリに「trajectory.dcd」が保存されます。
from google.colab import drive drive.mount('/content/drive') !cp trajectory.dcd /content/drive/MyDrive/trajectory.dcd
Colabの左側に出ているデフォルトのディレクトリ名はcontentとなっているようです。上記でcontent/driveにGoogle Driveがマウントされます。
パスに困った時は左側に表示されるディレクトリで「パスをコピー」すればOKです。

3. PyMolで可視化
結果のファイルが手に入りました!取り急ぎPyMolで可視化してみましょう。
シミュレーションの最終時点のファイルは「final_state.pdbx」です。OpenMM Setupのデフォルトのファイル名では拡張子が「pdbx」となっているので「.cif」に名前を変えてからPyMolに入れました。

境界からタンパク質がはみ出したぞ????なんでや???
よくわからないですが動画にしました。構造を読んだ状態でトラジェクトリ(trajectory.dcd)をPyMolに読み込ませれば良さそうです。
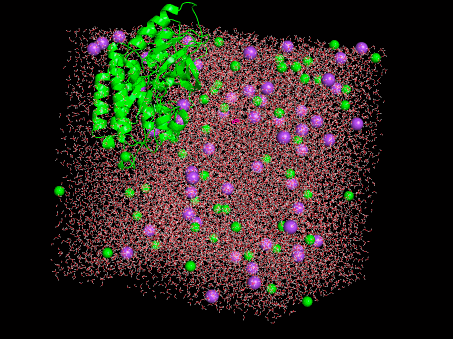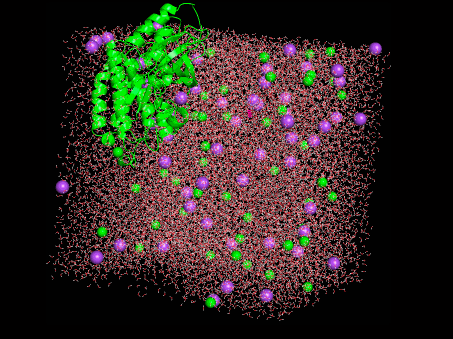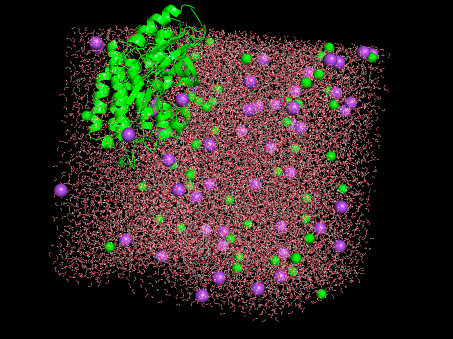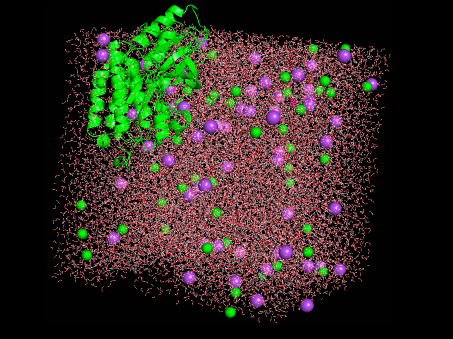
かわいい!!・・・けど、はみ出してる。。。
4. Center of Mass Motion?
境界からタンパク質が出てしまっている原因がよくわからないので検索するとCenter of Mass Motionという用語に行き当たりました。OpenMM User Guideに3.6.12 Removing Center of Mass Motionという項目があります。
曰く、デフォルトではOpenMMアプリケーションは「CMMotionRemover」を系に付与しており、「各ステップ毎に重心の動きをすべて取り除くことで、時間発展に伴い系全体がドリフトしてしまうことを防ぐ」のだそう、、、。
ドリフトが何かよく分かりませんが、タンパク質が境界外に出るのはかなり「drift(漂流)」感のある動きです。
上記項目では、「removeCMMotion=Falseを設定すればドリフトを許容することもできるけど、かなりレアなシミュレーションだよねー」と書いてありました。
デフォルトから変えていないので重心は動かない設定のはずなのですが・・・。心当たりのある方、ご教示いただけると嬉しいです。
5. おわりに
以上、今回はGoogle Colab上でOpenMMの計算を実行する方法について見てみました。手順がくどくて読みにくい記事になってしまいましたが、記憶力の悪い私の備忘録ということでお許しください。
得られた結果を可視化するとタンパク質が境界外に出ているというなんとも気持ちが悪いものでした。何はともあれ、一応は動きのある結果が得られたということで、Colab上での計算手順は確認できたのでヨシ!
・・・困った。
以上、今回もかなり間違いが多そうです。お詳しい方ご教示いただけると嬉しいです。
ではでは!
6. 追記
境界からタンパク質がはみ出ることについてよくわからないと書きましたが、OpenMMのGitHub Frequently Asked Questionsにそのものズバリ書いてありました。
「Why does my protein stick out past the edge of the water box?」
「問題ないよ!周期境界条件(periodic boundary conditions)をきちんと理解していないだけだよ!」ってな感じみたいです。
シミュレーション本番においても、計算後のトラジェクトリ描画においても、「境界をどのようにとるか?」「分子をどのように描画するか?」によって見え方が異なってくるようです。
GitHubのIssue #2688 Simple example of using SystemBuilder to simulate protein-ligand complexの議論中でも、タンパク質が境界から飛び出た図が引用されていますが、特におかしい挙動ではない(The trajectory produced looks just fine)と書かれています。
以上、同様の描画が得られて気になる方はご参照いただければと思います。
*1:イラストはいらすとやさんより
*2:Making it rainの手順を参考にさせていただいています。
*3:参考:Google Colabのファイルのアップロードとダウンロード
*4:ダウンロードが始まらないと思ってたら、ポップアップが出てました。気づかなかった。。。