静電ポテンシャルからタンパク質-リガンド複合体を眺めよう 〜パート②:Tyk2阻害剤への適用
リガンド-タンパク質複合体を静電ポテンシャル図で眺める記事の後編です。前編で使い方をお勉強したESP-DNNを実際のより複雑な例に適用してみましょう!
計算対象としては②TYK2キナーゼ阻害剤 (ΔG_expデータセット)を用います。

1. 計算対象
1-1. Tyk2キナーゼ阻害剤データセット
今回取り上げる例はTYK2キナーゼ阻害剤(ΔG_expデータセット)です。
自由エネルギーとリガンド結合親和性との関係に焦点をあてたデータセットで、もともとは以下の文献に由来するもののようです。
Accurate and Reliable Prediction of Relative Ligand Binding Potency in Prospective Drug Discovery by Way of a Modern Free-Energy Calculation Protocol and Force Field
J. Am. Chem. Soc. 2015, 137, 7, 2695–2703
自由エネルギー計算の分野では有名なものらしく、Open Force Fieldのベンチマーク(protein-ligand-benchmark)にも含まれています。
また、機械学習を自由エネルギー計算に応用した下記プレプリントでも対象として取り上げられています。(bioRxiv, CC-BY 4.0)
- 日本語解説:Preferred Netowrks Blog Neural Network Potentialを用いたタンパク-薬剤結合自由エネルギー計算の改善
具体的には以下のようなキナーゼ阻害剤化合物とデータです。*1
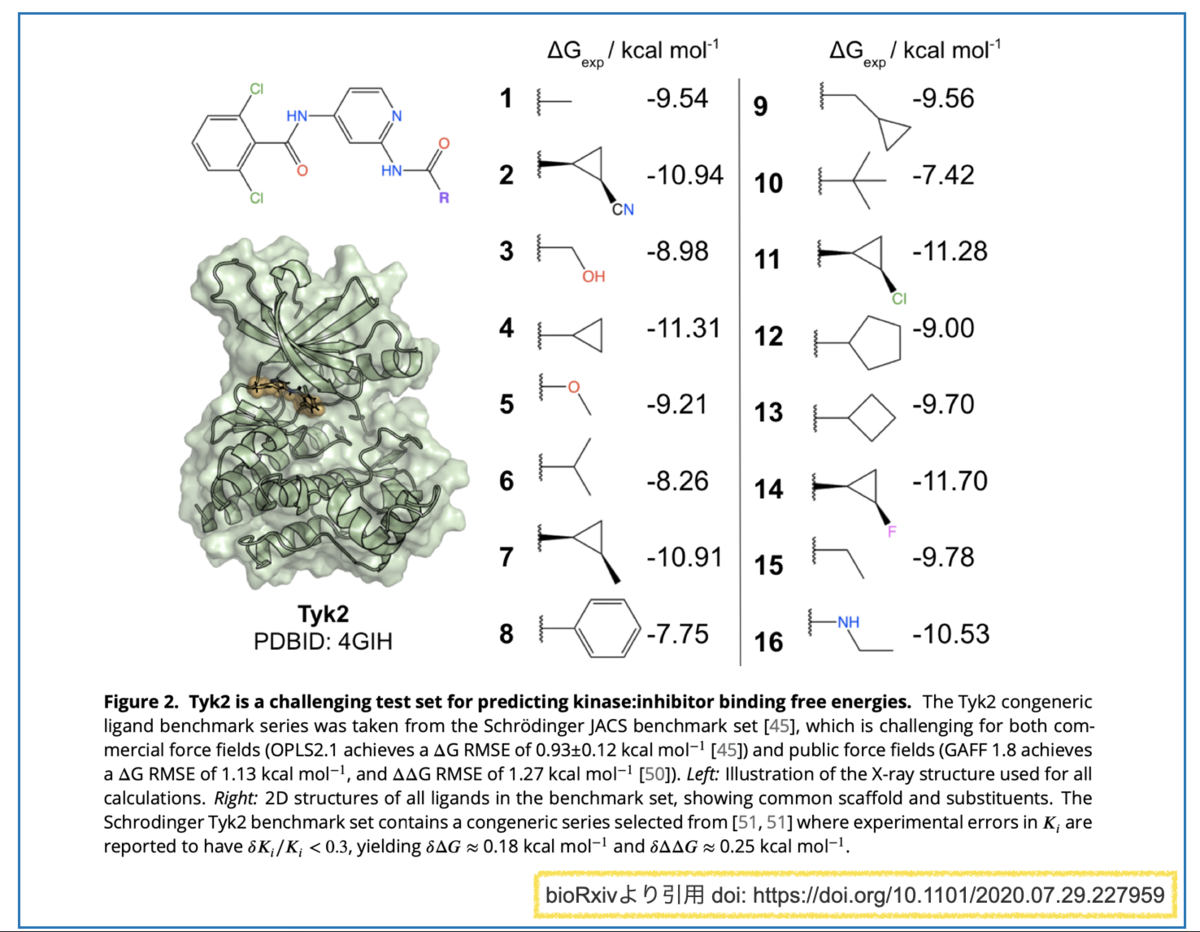
共通の骨格を有し、Rグループが異なる化合物群です。それぞれについて結合親和性のエネルギー(ΔG_exp)が求められています。
置換基の構造で「-7.4 kcal/mol ~ -11.7 kcal/mol」と広い幅で親和性が変化していてとても興味深いですね。
この活性の差を静電ポテンシャルで考察できたら面白そう!!!
1-2. データセットの取得元と変換
さて、Tyk2キナーゼ阻害剤データセットの化合物構造ですが、今回はOpen Force FieldのGitHubに格納されているものを利用させていただくことにしました。*2
上記にタンパク質(pdb)およびリガンドの構造データ(sdf)が含まれています。
注意点として、ESP-DNNの対応入力ファイルはPDBです。そこでリガンドデータをRDKitでそれぞれPDBに変換して用いました。
from rdkit import Chem m = Chem.SDMolSupplier("lig_ejm_54.sdf")[0] mH = Chem.AddHs(m, addCoords=True) Chem.MolToPDBFile(mH, "lig_ejm_54.pdb")
リガンドのsdfファイルは一つずつ別々のディレクトリに入っています。以下の図に構造とGitHubのディレクトリ名との対応を書いたのでご参考までに。。。
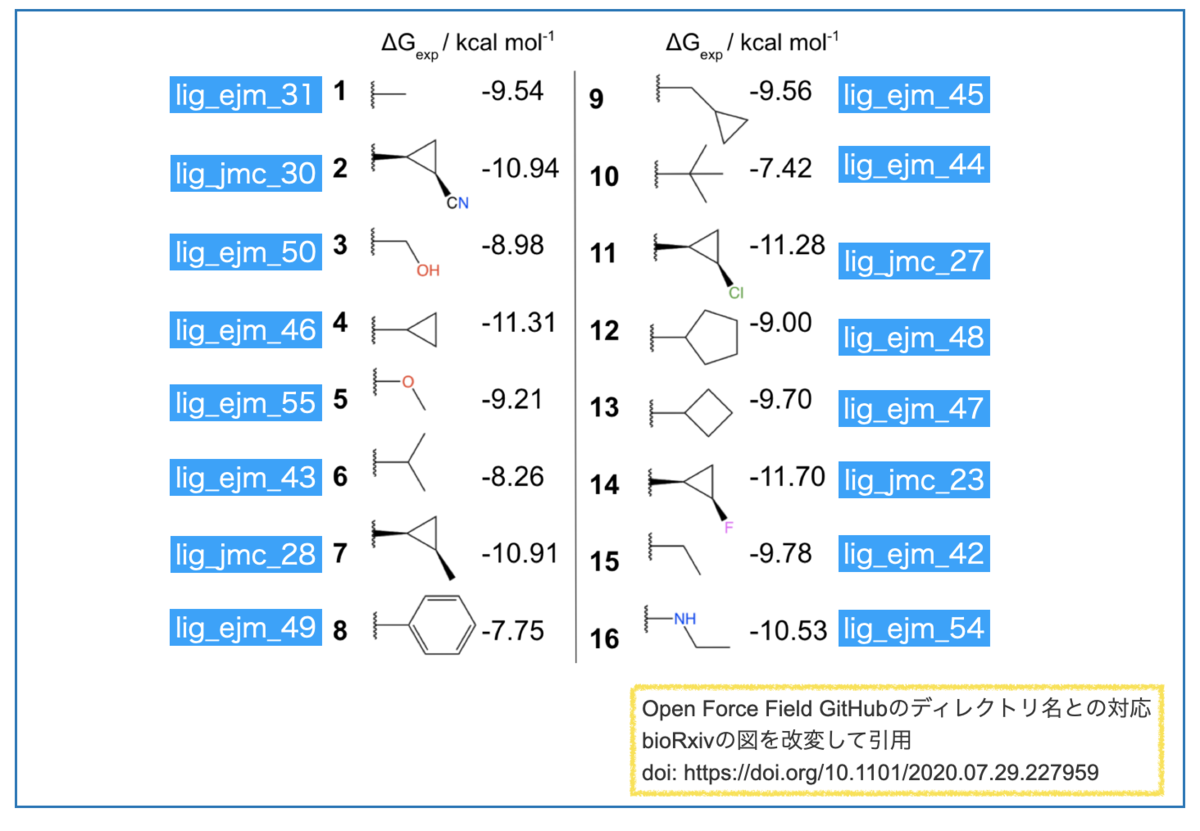
以上が今回の計算対象とデータの取得元です。
2. ESPを計算して活性差を考察しよう
2-1. 計算対象の絞り込みと傾向
「早速計算してみよう!」といきたいところですが、テーブルの化合物数がそこそこ多いです(16個)。
そこで、データセットの中でも構造が似通ったシクロプロピル基を持つもののみに注目してみます(下図、青枠)。
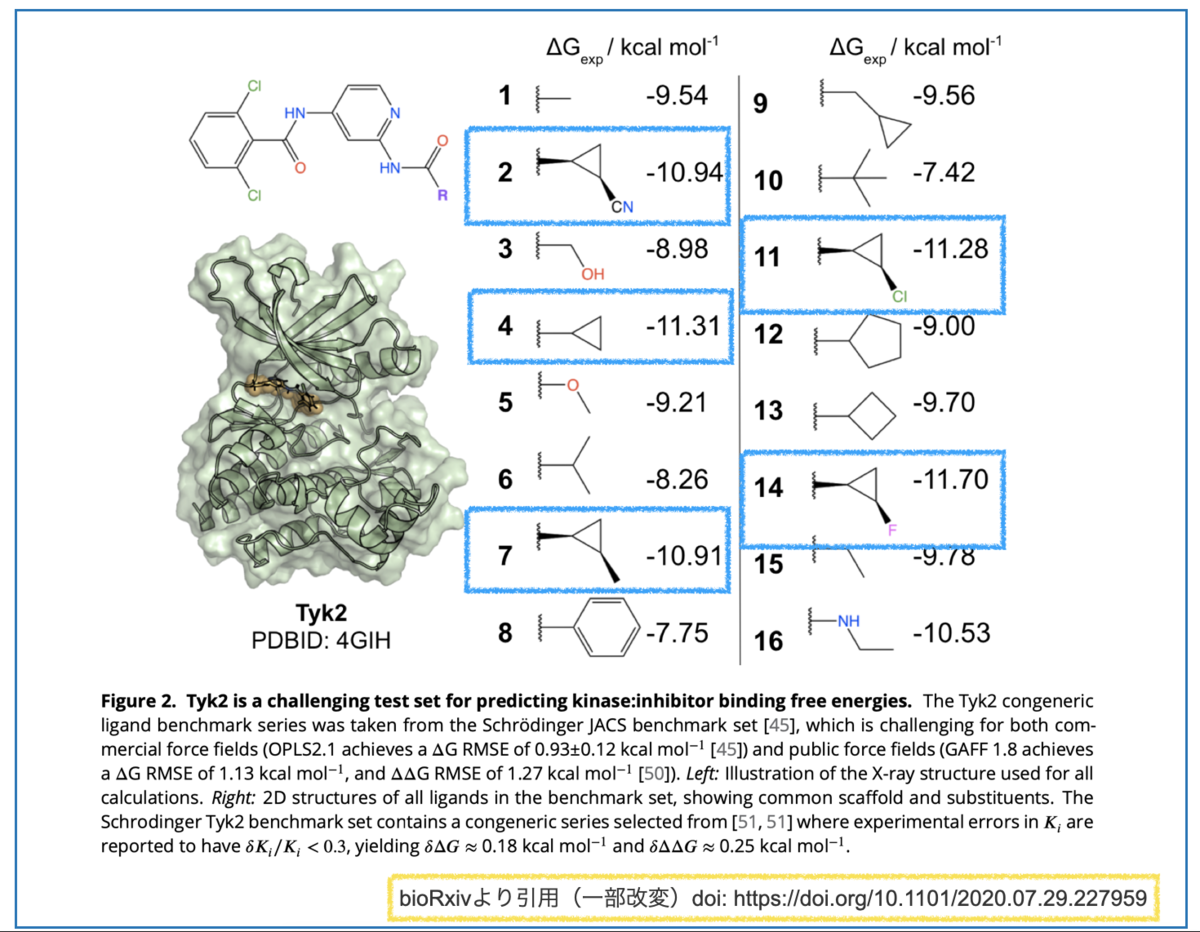
F置換(14)> 無置換(4)~ Cl置換(11) > CN置換(2)~ メチル置換(7)の順に活性が下がっています。
電子求引基の方が活性が良さそう(F vs. H, Cl > Me)ですが、大きくなると活性に悪そうです(H > CN, Me)。
2-2. PyMolで眺める
2次元の構造式で見た傾向が3次元ではどうなるか?PyMolで描画してみました。

複合体図の下半分はシクロプロピル基周辺の拡大です。近接するアミノ酸残基「Tyr、Arg、Leu」をSpheres表示にしています。
拡大してみると、シクロプロピル基上の置換基を側鎖が取り囲んでいます。「大きい置換基は好まれない」というのは複合体構造でも納得できそうです。
また、親水性のArgがあるので、疎水性のメチル基よりも「分極した電子求引基の方が好まれる」といった考察も可能かもしれません。
・・・このままではよくわからないので、上の構造を静電ポテンシャル図で描画してみましょう!
2-3. ESP-DNNの結果で親和性を考察しよう
タンパク質(Tyk2)と上記3つのリガンド構造をESP-DNNで計算してみました。Google Colab上で計算し、NGL viewerで描画しています。具体的な計算と描画の方法は前回の記事をご参照ください。
2-3-1. タンパク質
まずタンパク質の静電ポテンシャル図はこんな感じです。
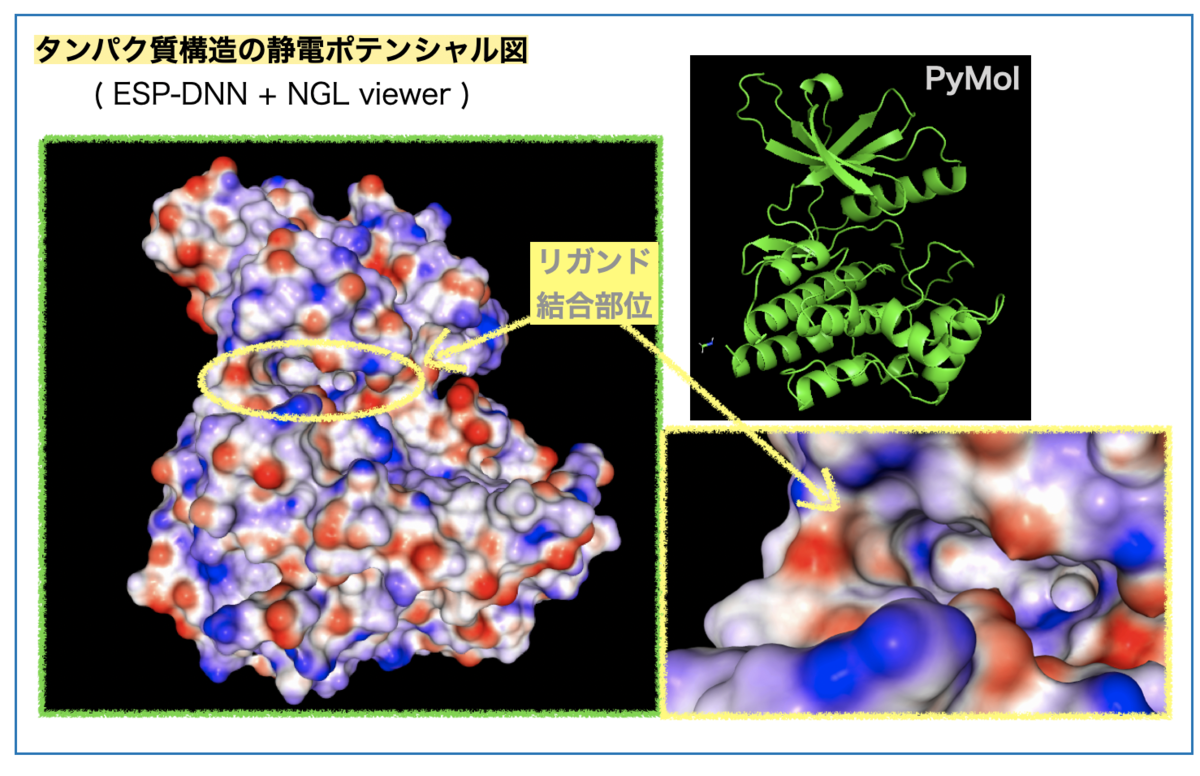
ESP図の赤い部分の原子は負の(部分)電荷をもち、青い部分の原子は正の(部分)電荷をもちます。
真ん中左寄りのくぼみがリガンドの結合するポケットです(黄色丸枠)。ポケットを拡大(右下)すると右奥が青色、溶媒に面した左部分に青と赤色が混在しています。
リガンドのシクロプロピル基周辺は溶媒に面した向かって左に相当します。この付近のESPの色がどの残基に対応するか?、眺めてみましょう。
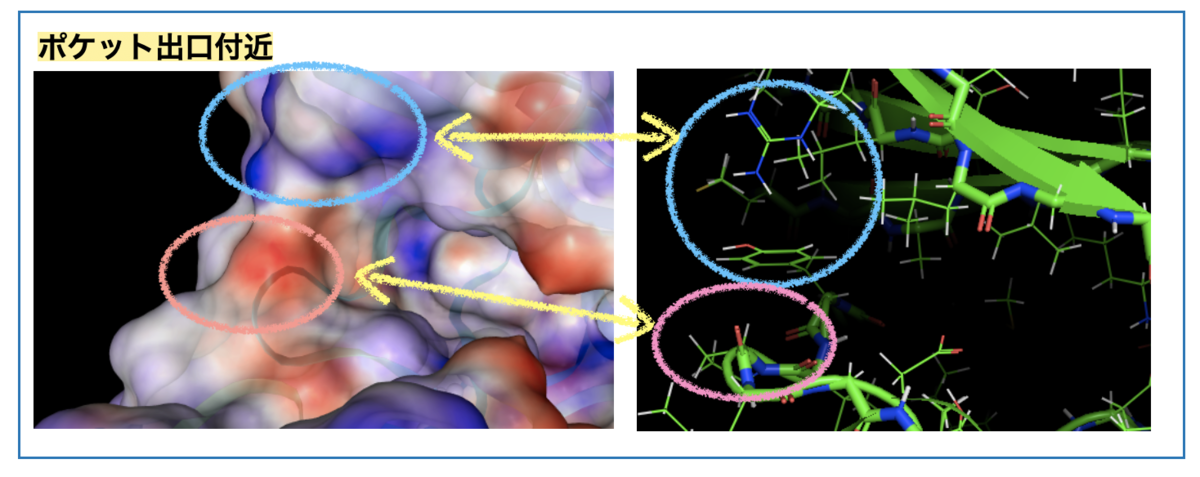
凡そですが、青い箇所は先に見たArgやTyrの側鎖に対応し、赤い箇所は主鎖(Pro)のカルボニル酸素に対応していそうです。*3
水素結合のドナーになりそうな水素原子近辺が青(+)、アクセプターになりそうな箇所が赤(-) と解釈することができるかもしれません。
2-3-2. リガンドの比較
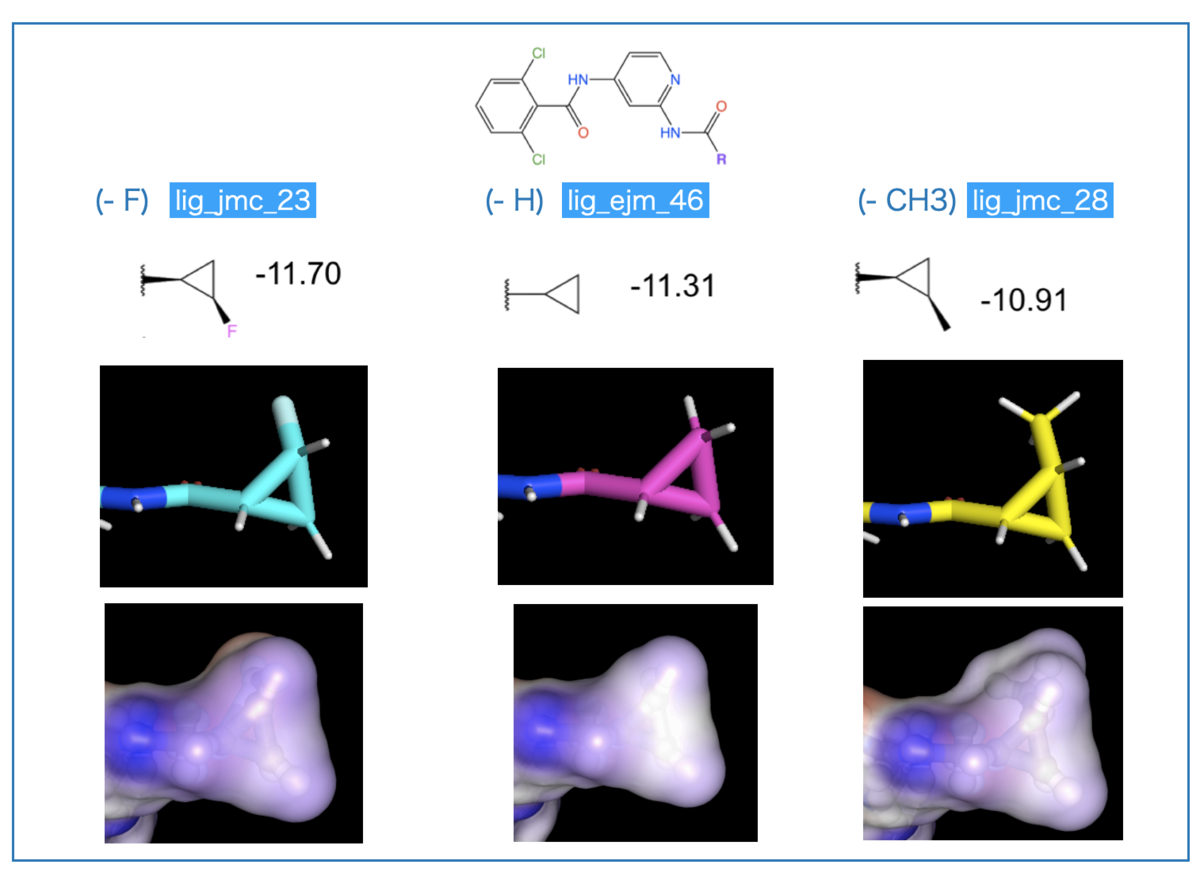
つぎにリガンドのESP図を見てみましょう。こんな感じです。

リガンドの右側、ジクロロベンゼンをみるとCl基以外の水素原子が青色(+) になっています。ベンゼン環上で置換基導入が他の位置に及ぼす電子的な影響が視覚的でわかりやすいですね!
シクロプロピル基は左側、黄色の丸枠で囲んだ部分です。シクロプロピル基の置換基について以下の特徴が見えます。
- フッ素置換(
lig_jmc_23)ではFが赤色
- フッ素置換(
- 無置換(
lig_ejm_46)では青みがかった白
- 無置換(
- メチル置換(
lig_jmc_28)では青みがかった白の大きな構造
- メチル置換(
F以外ではシクロプロピル基全体がプラスっぽく、Fではマイナスっぽさが追加されている感じでしょうか?
置換基とは反対側の面からシクロプロピル基を眺めてみましょう。
こんな感じ。
面白いことに、F置換は、無置換やメチル置換と比べて青色が濃くなっています。強い電子求引基で引っ張られた逆側にプラス性が強く現れるというのはわかりやすいですね!
2-3-3. リガンドとタンパク質の比較
タンパク質とリガンドをそれぞれ確認したので、組み合わせるとどうなるか?重ねてみましょう。
まずは一番シンプルな無置換の構造です。
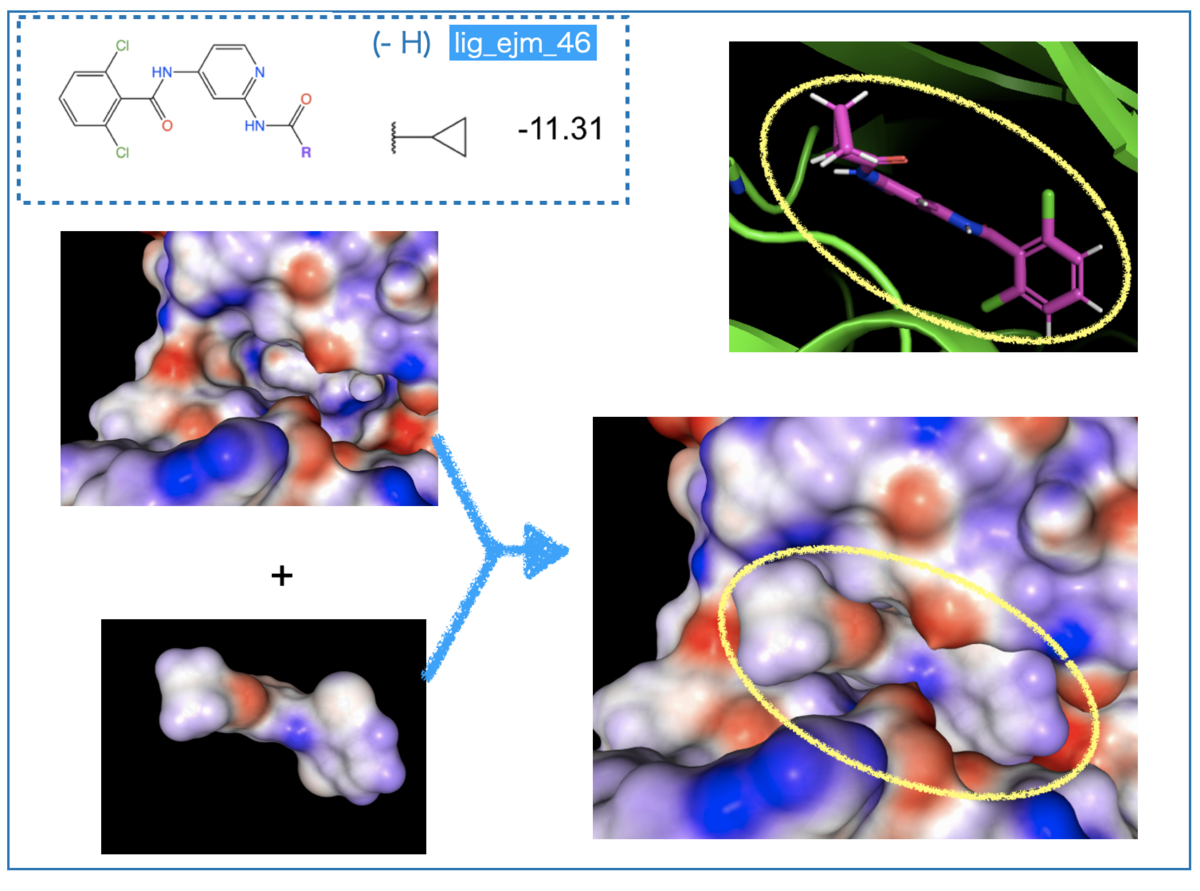
リガンド右側、ジクロロベンゼンの青い箇所がタンパク質側の赤い静電ポテンシャル面と接しているのが見えます。相補的な赤と青がタンパク-リガンドで接しており、相互作用に適していると考察できそうです。
また、リガンド中央の赤い箇所はシクロプロピル基に隣接したアミド結合のカルボニル酸素です。こちらもタンパク質側の青みがかった部分と接しており、相補的な組み合わせになっています。
データセットで「共通の骨格部分がタンパク質との相互作用に適した構造で一定の親和性を担保している」と考えると興味深いですね。
ついで活性の良いフッ素置換です。
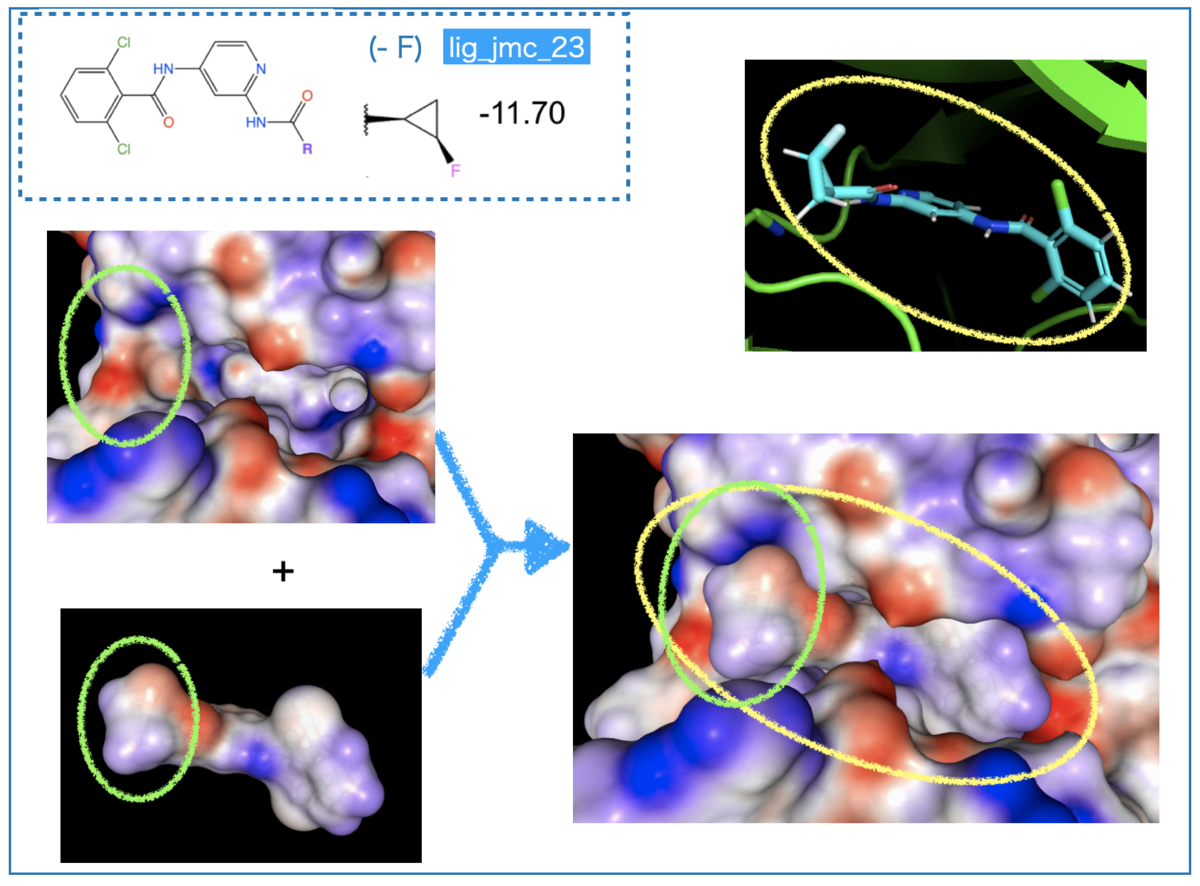
共通骨格は無置換と同じなので、シクロプロピル基(左側)に着目します。
F置換により赤色の箇所が増えています。ここと接するタンパク質の表面は青色となっています(先に見たArg側鎖 etc.の領域に相当)。
つまり、F置換で相補的な静電ポテンシャル面が増加しており、これによって親和性が向上したと考察できそうです。
またリガンド上で、F置換は置換基と反対側のシクロプロピル基表面をより濃い青色に変化させていました。タンパク質ポケットを見ると該当箇所は赤色となっています(上図左の緑丸)。
重ねた図では見えなくなっていますが、「赤色(タンパク)と濃い青色(リガンド)が接している」ため、この箇所でも相補性が良くなっていると推測されます。
次に、活性の落ちるメチル基置換です。
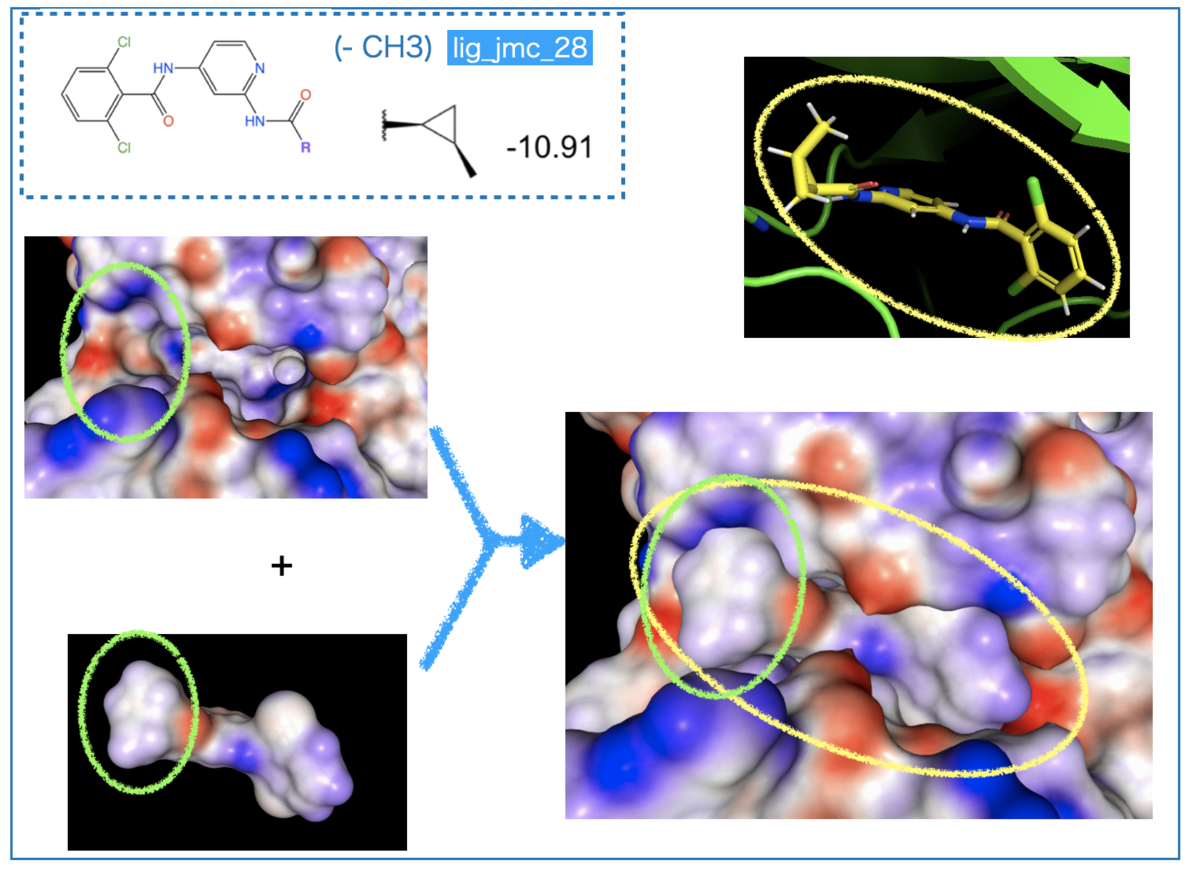
導入されたメチル基は静電ポテンシャルがほぼニュートラル(白〜薄い青色)です。複合体の左側、緑色の丸で囲んだ箇所を見ると、タンパク質の青色表面とメチル基の表面が接しています。
「(部分)電荷を帯びている箇所にニュートラルなものが当たっている」という状態で、相補性が良いとは言い難くなっています。
つまり、「置換基の導入によりタンパクとぶつかる面が増えたが、静電ポテンシャルの相性でメリットがないため、立体障害により親和性が落ちた」と考察できそうです。
「立体障害が親和性にネガティブに働きそう」というのは(2次元構造式の)構造活性相関やPyMolの描画で見た内容とも合いそうですね。
以上をまとめると、小さな置換基でタンパク質と相補的な静電ポテンシャル表面をつくることができる電子求引性のフッ素導入は親和性の向上に最適だった、と言えそうです。
3. おわりに
以上、今回はESP-DNNで計算した静電ポテンシャル図をもとに、Tyk2キナーゼ阻害剤の親和性の差について考察してみました。
定性的な議論ではありますが、「タンパク質とリガンドの表面の色が相補的(赤 - 青)となることと、親和性が良い(ΔG_expが小さい)」ことに関係性がありそうなことが見えました。
(2D/3Dの)構造式や複合体を普通に描画して眺めるだけではわからない観点(ESP)をとりいれることで、一歩踏み込んで活性について議論できそうなことがわかってよかったです。
ESPをまともに計算しようとすると量子力学計算が必要でどうして良いかわかりませんが、ESP-DNNを使うことで簡単かつ高速で計算ができました。
論文のタイトル通り、まさしく実用的(Practical)なツールですね。
Practical High-Quality Electrostatic Potential Surfaces for Drug Discovery Using a Graph-Convolutional Deep Neural Network J. Med. Chem. 2020, 63, 16, 8778–8790
すばらしいツールを公開してくださっている研究者の皆様に感謝です!
さあ、ESP-DNNで観察眼を鍛えて、創薬人格付けチェックに正解できる能力者(エスパー)に君もなろう!
おしまい。
(おまけ)ダジャレ師匠の素晴らしい記事
「私のキャリアパス紹介 〜アカデミアから民間企業に転職して」 小寺正明(Preferred Networks)