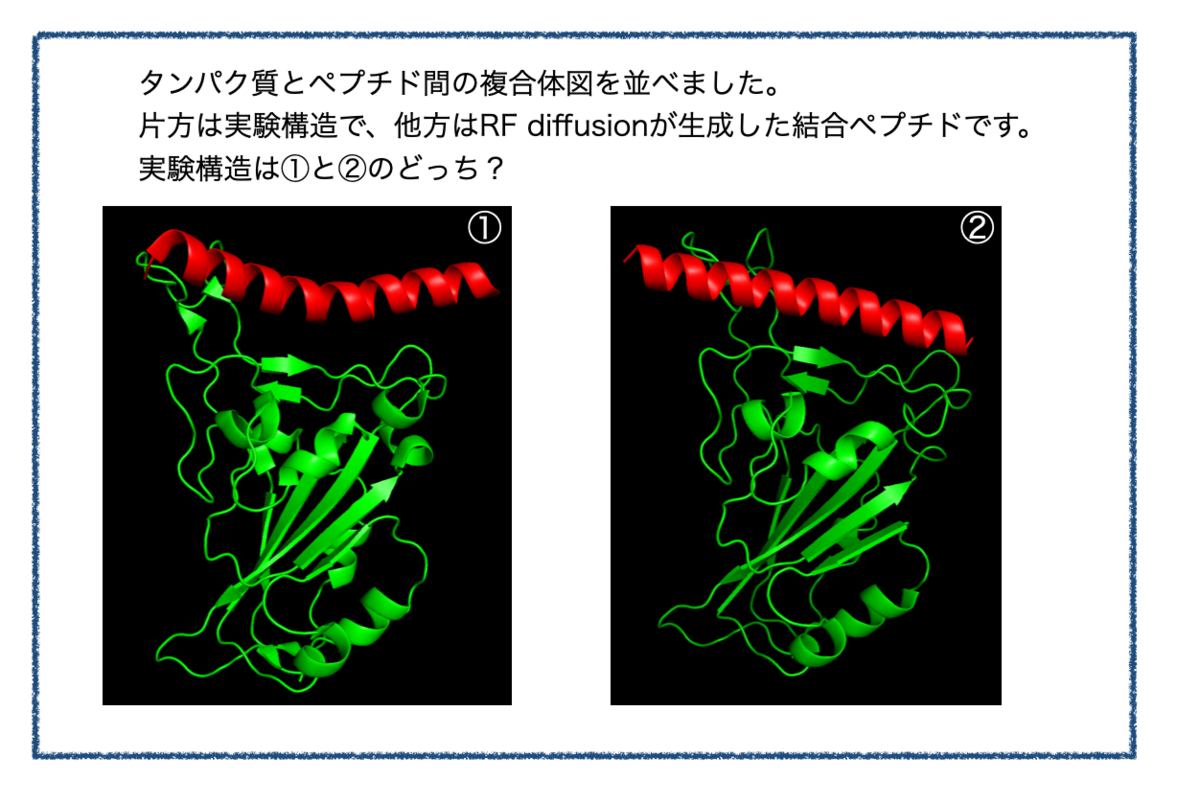
クイズ:どっちが自然の構造? 〜RF diffusionで人工ペプチドを生成〜
毎年恒例、お正月の創薬ちゃんの創薬人格付けチェック。今年はタンパク質の複合体がお題でしたね。
元旦恒例 #創薬人格付けチェック!
— 叢雲くすり (創薬ちゃん) 💊LLM創薬チャレンジ開催中 (@souyakuchan) 2023年1月1日
今回はタンパク質–ペプチド間相互作用。動画(平行法ステレオ図)に示す2つの結合構造のうち、片方が正解構造、もう片方はAlphaFold2が生成したまことしやかな幻だ。
正しいと思うのはどちらだろうか? 次のツイートで投票を取る。#souyakuchan_quiz#格付けチェック pic.twitter.com/AtAHShY9rG
アンケート結果を見てAlphaFold2の幻を見破った方が多いのにびっくりしました。創薬ちゃんフォロワーのレベル高い。。。
さらなる高みをめざし、続問をつくってみました!
- ムービー
- きっかけ
- お題のタンパク質
- Google ColabでRF diffusion(hotspot指定なし)
- Google ColabでRF diffusion(hotspot指定あり)
- 答え
- ProteinMPNNによる残基の生成とAlphaFoldでの検証
- まとめ
ムービー
GIFも貼っときます。
① 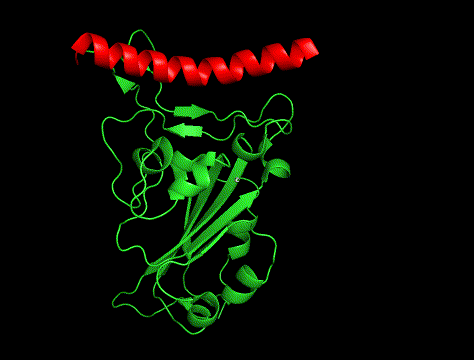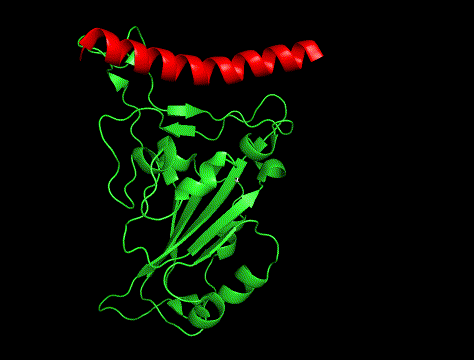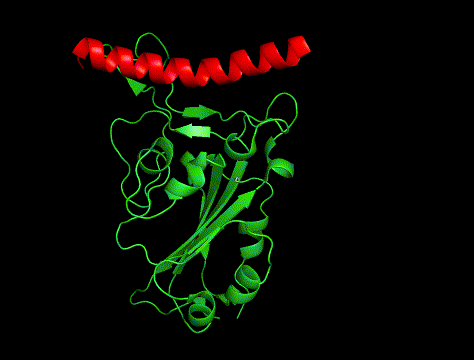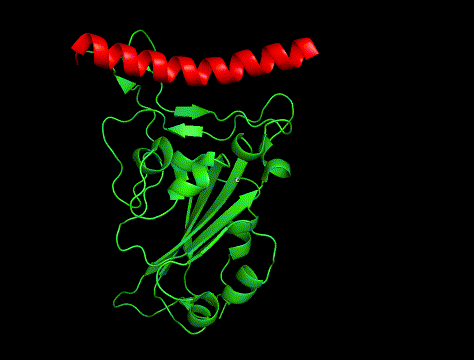
② 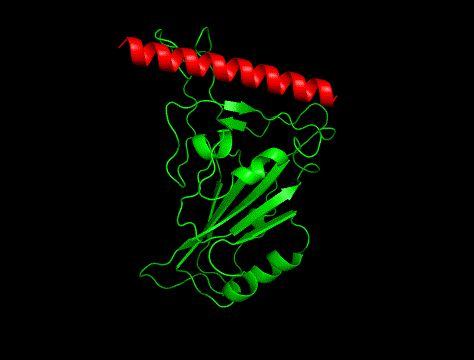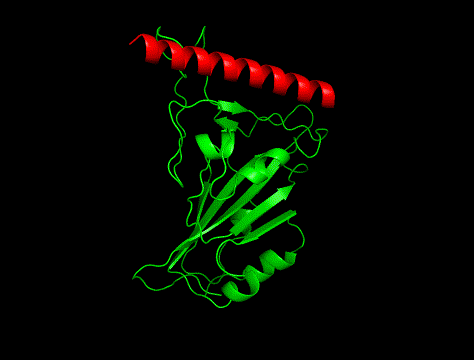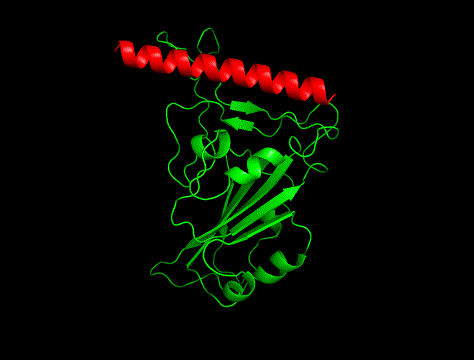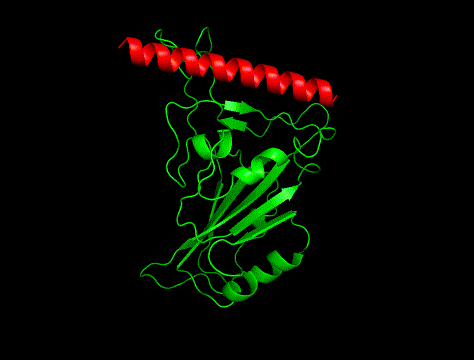
きっかけ
LabCodeさんのブログ記事でRF Diffusionというのを知りました。
Baker Labで開発された技術で、標的のタンパク質構造に対して結合するペプチドをつくってくれるそうです。すごい。
これで新しいペプチドを作ったら、自然界の構造と見分けられるのだろうか?
Twitterのプロたちは見分けるに違いない!
お題のタンパク質
標的タンパク質は格付けチェックにあわせてSARS-CoV-2 Spikeタンパク質の受容体結合ドメイン(RBD)としました。
進化的に獲得され保存されている相互作用様式に該当しないような MSA の薄いものにはまだ AF2 も弱いということかもしれない。今回の出題は SARS-CoV-2 Spike RBD と ACE2 の RBD 結合モチーフ部分であり、つまり自然界で最近たまたま出会ってたまたま結合したものに相当する。 pic.twitter.com/hRxwCg2TQp
— 叢雲くすり (創薬ちゃん) 💊LLM創薬チャレンジ開催中 (@souyakuchan) 2023年1月22日
自然界の結合相手としてACE2のRBD結合モチーフが知られているそうです。
複合体のX線結晶構造解析としては「PDB ID: 6M0J」などがありました。

論文はこちら
相互作用がある残基はExtended Data Table 2にまとめられています。
PyMolで描画だ!

チロシンが複数あって目にとまりますが、疎水性相互作用というよりも水素結合を結構作っているみたいです。ほー。
Google ColabでRF diffusion(hotspot指定なし)
ではRBDに結合するペプチドをつくってみましょう!
LabCodeさんの先のブログ記事に丁寧に解説してくださっていますので、手順はそちらをご参照ください。
Google Colabでノートブックにアクセスしてボタンを押すだけです。
元の構造には「PDB="6M0J"」を使いました。
このうち結合対象は「E鎖」で、生成するペプチドの長さを「30」としました。これらはcontigsという設定項目に記載するそうなので「contigs="E:30"」としました。
ペプチド生成の様子をアニメーションにすることができます。

おー!!すごい。
・・・でも結合して欲しい箇所そこじゃない。上の方のループと結合して欲しいのに。。。
Google ColabでRF diffusion(hotspot指定あり)
RF diffusionにはhotspotという設定項目もあり、ここに結合して欲しい箇所の残基を入力することで、結合ペプチドの生成対象箇所を絞り込めるようです。
さきの文献のTable 1を参考にスパイクタンパク RBDの残基を指定してみます。少し数が多いですがE鎖の残基番号をつかって「hotspot = "E417,E446,E449,E487,E489,E493,E500,E501,E502,E505"」 としました。
もう一回ペプチドを生成だ!

今回はちゃんと図の上側を対象に生成されているようです。
さきほどのアニメーションよりもすぐに構造ができているので構造生成感は低めですが、ヘリックス構造が瞬時に構築されているのは驚きです。
答え
冒頭の画像比較につかっているのは「hotspot指定あり」で生成した構造でした。
「①と②のどちらが正解か?」をみるまえに、まず実験構造(PDB : 6M0J)から結合位置のみを切り取った構造を描画してみます。
こんな感じ。

ACE2のRBD結合箇所もヘリックス(赤色)で、ポケットに合わせてか、少し湾曲しています。
一方、RF Diffusionで出てきたペプチド骨格はこんな感じ。
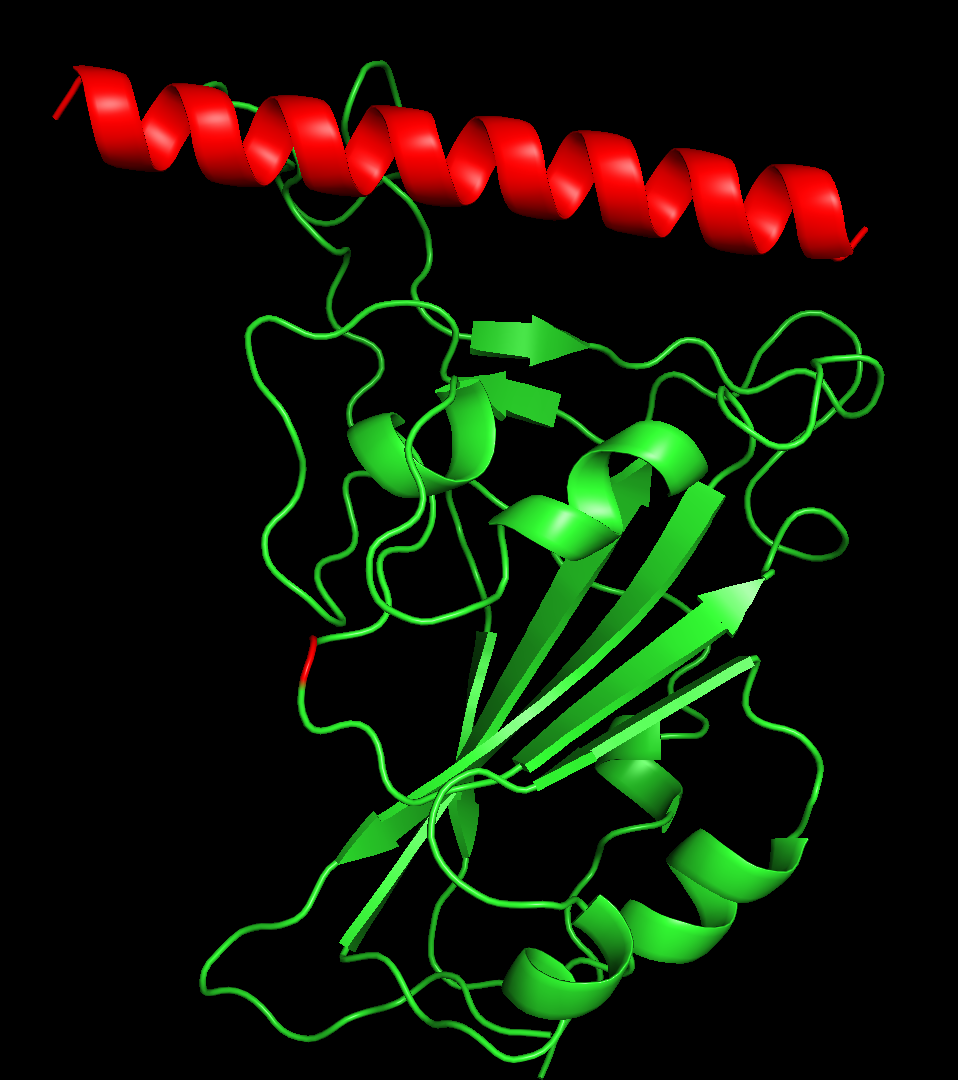
ヘリックスが真っ直ぐ!
というわけで、実験構造は①でした。
ProteinMPNNによる残基の生成とAlphaFoldでの検証
クイズはおしまいですが、ペプチド生成には続きがあります。
RF diffusionで出てくるのは骨格のみです。実際に、先のRF diffusionで出てくる構造では残基がすべてグリシン(G)となっていました。
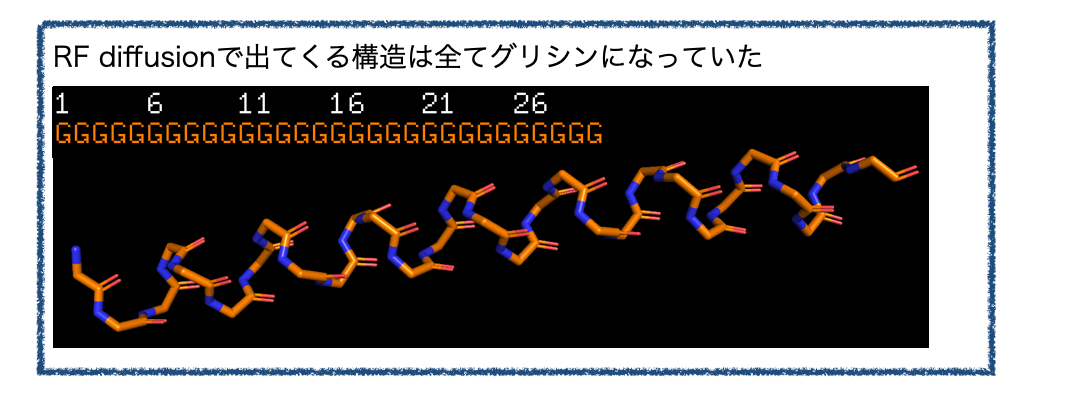
もっと具体的なペプチドにするにはProteinMPNNというソフトで骨格に合わせた残基を生成し、生成した残基から再度AlphaFold2で立体構造を予測して望みの構造になっているか検証する必要があるそうです。
これらの工程も同じGoogle Colabのノートブックに用意されているのでそのまま実行してみましょう!
生成した構造はPDBファイルとしてダウンロードできます。一押しの構造は「best.pdb」というファイル名になっていました。
ベストはこれだ!

いろいろな残基をくみあわせたヘリックス構造になっています。リシン(K)とグルタミン酸(E)が多めでしょうか?
複合体として描画してみましょう。

さっきと結合位置がかわってる???
残念ながらAlphaFoldによる検証では、予測された複合体の構造が望みのものとは異なりました。・・・残念。
まとめ
というわけで、今回はRF diffusionで創薬ちゃん格付けチェックの続編を(勝手に)つくってみました。
すんなりとヘリックス構造がでてきてびっくりしました。構造生成のアニメーションも格好いいです。
面白かった点として、「hotspotを指定しない」場合にはRBDのループと反対側(図下側)を対象にペプチドが生成されたこと、「hotspotを指定して」骨格を作った場合でもProteinMPNNを経たAlphaFoldの検証では異なる結合位置となっていたことがあります。
創薬ちゃんのクイズを見返してみると、AlphaFoldによる幻はRBDのループと反対側に対して結合するように予測されているように見えます。「hotspotを指定しない」場合に似ていますね。
スパイクタンパク質全体の一部(RBDのみ)を切り取った構造を対象にしているから生じた問題かもしれませんが、AlphaFoldによる複合体予測の癖(?)が垣間見えるようで面白いですね。
ではでは!