化学構造式のOCRソフト Img2Molで画像をSMILESに変換しよう!
少し前になりますがKaggleでBristol-Myers Squibb - Molecular Tranlationというコンペティションが開催されていました。
化学構造式を含む画像をその化学構造のInchi(International Chemical Idendifier)文字列に変換する精度を競うものでした。*1
巨大製薬企業のブリストル・マイヤーズスクイブがこのようなコンペティションを行うくらいなので、化学構造式のOCR(Optical Charactier Recognition)、「画像から化学構造式情報をコンピュータ処理できる形で抜き出すタスク」がニーズがありかつ難しいものであることが分かります。
さて今年のRDKit UGM 2021でも同様のタスクについての発表がありました。
その名もImg2Mol‼︎
- 発表の動画: YouTube : Djork-Arné Clevert: Img2Mol – SMILES Recognition from Depictions ofChemical Structure
- 発表のスライド:Clevert_Img2Mol.pdf
- 文献 : Img2Mol – accurate SMILES recognition from molecular graphical depictions Chem. Sci., 2021,12, 14174-14181
- GitHub : bayer-science-for-a-better-life/Img2Mol
そんな難しいタスクのためのプログラムが公開されている??
それなら遊ぶしかない!やってみましょう!
文献を流し読み
Img2Molはバイエルの機械学習チームが開発したプログラムで、化学構造式の画像をSMILESに変換してくれます。文献がオープンアクセスになっているのでざっと眺めてみます*2。
化学構造式OCRのモチベーション
そもそもなぜ化学構造式を画像から機械処理できる形で取り出すタスクに需要があるのでしょうか?
それは創薬化学の重要な情報源である文献と特許がともにコンピューター処理に適した形で情報を提供していないからです。
最近ではJournal of Medicinal ChemistryなどSupplementary Informationで化学構造のSMILESを列挙したファイルを提供するものも出てきましたが、まだまだ多くの文献で化学構造式は画像でのみ示されています。また特許では公開されている書類がPDFや画像形式であり、さらに産業上の理由から敢えて曖昧に書かれていることもあります。
このような状況なので、最新の情報を効率的に2次利用できる形でとりだしアップデートしていくのはとても大変です。というわけで画像から構造式をとりだすことにニーズがあるそうです。
ところで生理活性物質の公開データベースとしてみんな大好きなChEMBLも、専門家による手作業のキュレーションで情報更新されているようです。維持・運用のコストを考えると感謝しながら使わないといけませんね。
Img2Molのアプローチ
だいたいモチベーションがわかりました。では実際にタスクをこなすにはどうすれば良いでしょうか?
課題としてはこんな感じです。
1. 画像中の構造式を認識する
2. 認識した構造式をコンピューター処理できる形に取り出す
3. 取り出した構造は化学的に妥当なものである必要がある
1.と2.は全てのOCRに共通で、3.は化学ならではの課題です。
で、この化学ならではの部分をどうするか?
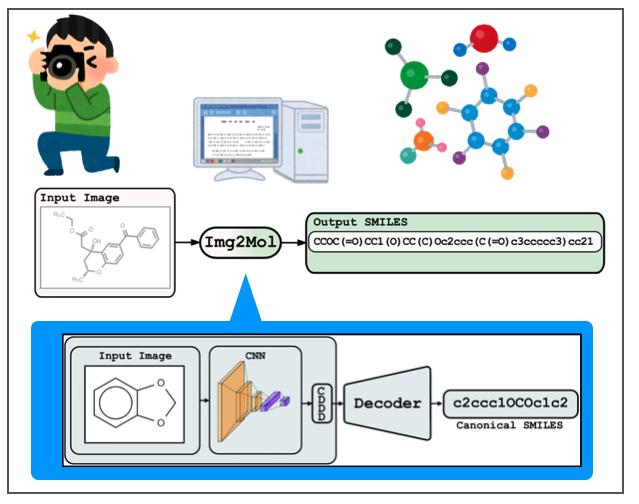
Img2Molでは画像を畳み込みニューラルネットワーク(CNN)で認識し、化学構造式の特徴を表現する空間(CDDD)に一度埋め込んだあと、線形表記(Canonical SMILES)として取り出す、という2段階の構成をとっているようです。

CDDDはcontinuous and data-driven molecular descriptorsの略で、同じくバイエルのチームが開発した記述子です。こちらの論文もOpen Accessになっています。
- Learning continuous and data-driven molecular descriptors by translating equivalent chemical representations Chem. Sci., 2019,10, 1692-1701
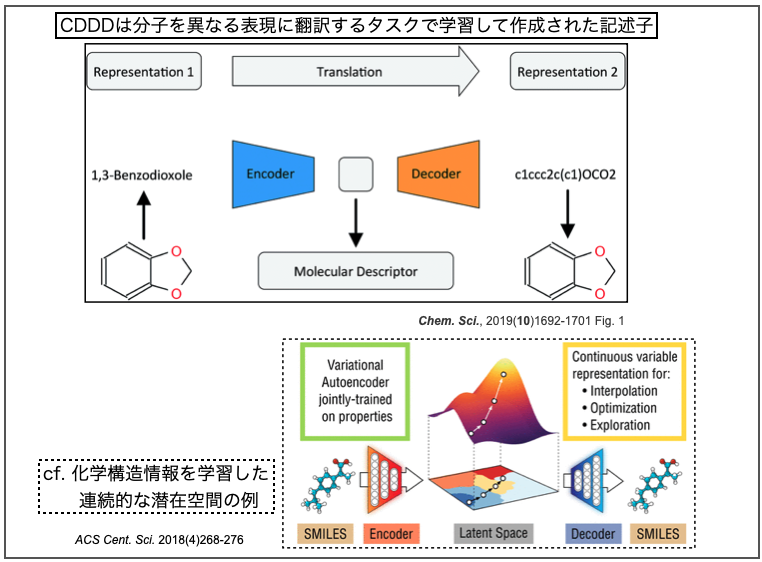
オートエンコーダ(Autoencoder、自己符号化器)はニューラルネットワークの一つで、「入力されたデータを一度圧縮し、重要な特徴量だけを残した後、再度もとの次元に復元処理をするアルゴリズム」だそうです*3。
ある化学構造を表すSMILESには複数通りの書き方があり、それらを入力として(一意の)canonical SMILESを出力する翻訳のタスクをこなすオートエンコーダを学習します。このネットワークは化学構造の特徴を学習した潜在空間(latent space)となっています。なので、化学構造を表す記述子として使うことができ、異なるタスクにも色々と応用できるよ!(とかそんな話です。たぶん。)
上図で引用した下半分はAspuru-Guzik先生のチームによる以下の論文です。*4
- Automatic Chemical Design Using a Data-Driven Continuous Representation of Molecules ACS Cent. Sci. 2018, 4, 2, 268-276
ところで、バイエルのチームはImg2Molと同様のアプローチを以前にも報告しています。以下の論文では「ECFP記述子を入力として、もともとの分子構造を逆に推定するリバースエンジニアリング」を行っています。
- Neuraldecipher – reverse-engineering extended-connectivity fingerprints (ECFPs) to their molecular structures Chem. Sci., 2020,11, 10378-10389
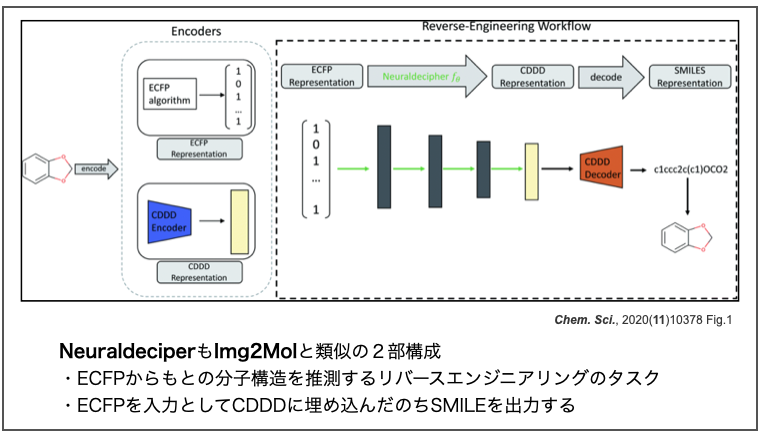
このタスクでも一度CDDDに埋め込んだ後SMILESとして出力しています。応用範囲が広い記述子を開発できると便利だなー。
Img2Molのパフォーマンス
Img2Molのネットワーク構造やメソッドの詳細はよくわからないので割愛します。ごめんなさい。
結果っぽいのだけ貼っておきます。
他の手法よりパフォーマンスが良かったよの図(ベンチマーク)。
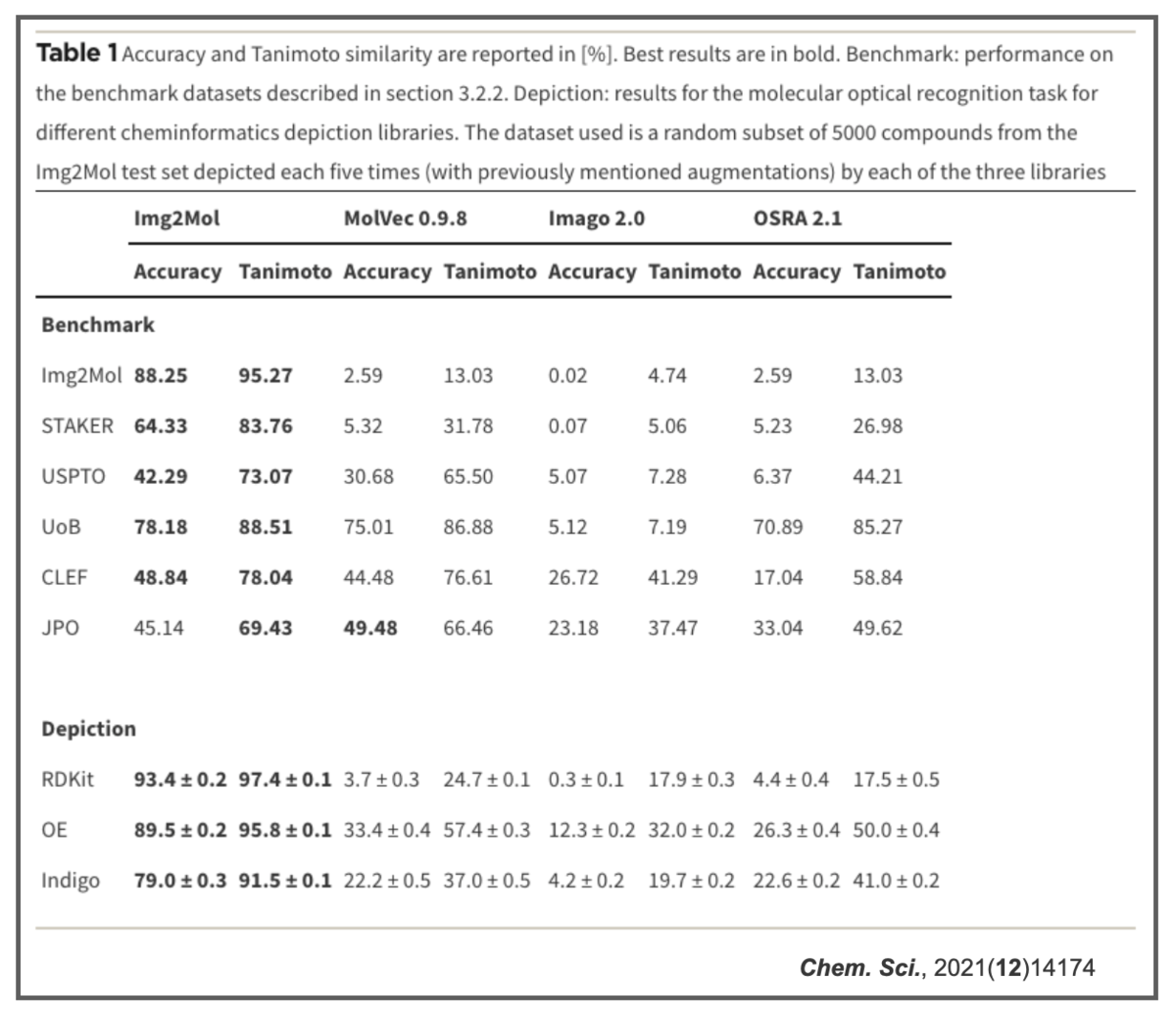
AccuracyだけでなくTanimoto類似度でも評価しているのが面白いです。
学習に使う化学構造を描くツール(depiction)によっても結果が変わっているのも興味深いです。同じ化学構造でもソフトによって表現・相性が変わるところに、コンピュータで化学を扱う難しさを感じます。
さらに面白い結果はこちら。画像の解像度(Resolution)とパフォーマンスの関係です。なんとImg2Mol (no aug)は入力の解像度が上がると精度が落ちています。
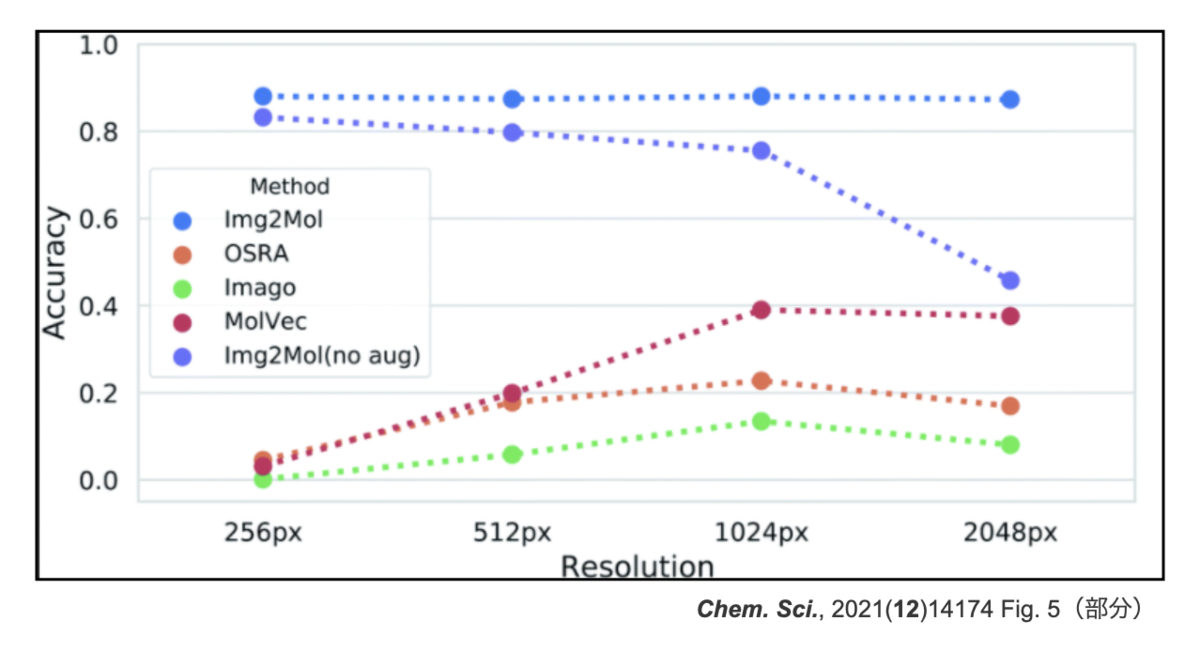
「入力の質が上がって精度が悪くなるの??逆じゃないの??」となりますが、どうやらネットワークへの入力の仕方によるもののようです。
ネットワークの構造は決まっているので、入力画像は全て同じ形(224 x 224 px)にリサイズされます。高解像度の画像は、このリサイズの処理で失われる情報が逆に多くなるため、ぼんやりとした入力になってしまうようです。
この解像度の問題や分子サイズの大きな構造の認識が難しいという問題を解決するため、data augmentationをおこなって学習し直しています。これで得られたよりロバストなネットワークが上図の「Img2Mol(無印)」に相当するものです。高解像度でもパフォーマンスが落ちていません。
分子サイズとパフォーマンスとの関係も貼っておきます。こちらもaugmentationで改善はしていますが、原子数が30を超えたあたりから精度が落ちてきています。
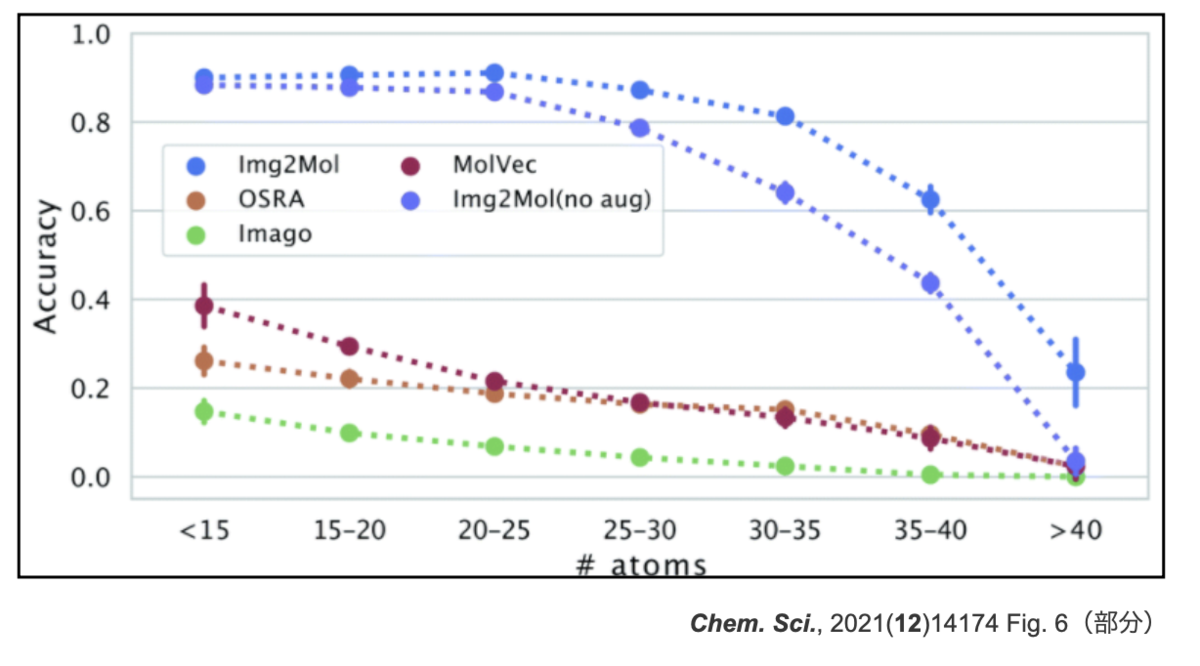
以上、Img2Molのざっとした文献紹介でした。正確な情報は論文やRDKit UGMの発表動画などをご参照ください。
Img2Molで遊ぼう
それでは早速、Img2Molで遊んでみましょう!素晴らしいことにGitHubで公開してくださっています!
但し、学習済みパラメータのライセンスはCC BY-NC 4.0でnon-commercial use onlyですので商用利用できないことには十分ご注意ください(念のため)。*5
インストール
インストール方法はcondaでOKで簡単です。GitHubのREADMEの以下をコピペするだけ。
git clone git@github.com:bayer-science-for-a-better-life/Img2Mol.git cd Img2Mol conda env create -f environment.yml conda activate img2mol pip install .
・・・が、Macユーザーの方はご注意ください。Img2Molに必要なソフトとして「cudatoolkit=11.0」の指定がありますが、NVIDAがmacOSのサポートを終了したらしく、「CUDA ToolKit v11」はmac版がありません。*6
Img2Molのインストールに使う「environment.yml」ファイルは以下のようになっています。

このままだと「condaチャンネルにそんなの無いよ!」って言われます。なので「cudatoolkit=11.0」のバージョン指定「=11.0」を消して「cudatoolkit」だけにして上書き保存してから再実行するとインストールできました。
旧バージョンのCUDA ToolKitが入るので動作の保証はできませんが、とりあえず動いたのでヨシ!*7
学習済みパラメータファイルはGoogleDriveからダウンロードできるようにしてくださっています(→ https://drive.google.com/file/d/1pk21r4Zzb9ZJkszJwP9SObTlfTaRMMtF/view )。大きい(~2.4GB)ので空き容量とインターネット回線にご留意を!
「model.ckpt」というファイルがダウンロードされるので、Img2Molディレクトリの中の「model」ディレクトリの中に放り込めば準備完了です。
Exampleで遊ぼう
準備ができたらJupyter notebookを開いて遊びましょう!
使い方がexample_inference.ipynbに、画像例がexamplesディレクトリにあるのでそのまま使わせていただきます。
# 必要なライブラリのインポート import torch from img2mol.inference import * from IPython.display import display # img2molを使う準備 device = "cuda:0" if torch.cuda.is_available() else "cpu" img2mol = Img2MolInference(model_ckpt="model/model.ckpt", # パラメーターファイルの指定 device=device) cddd_server = CDDDRequest() # > Initializing Img2Mol Model with random weights. # > Loading checkpoint: model/model.ckpt # > Setting to `self.eval()`-mode. # > Sending model to `cpu` device. # > Succesfully created Img2Mol Inference class.
これでImg2Molを使って画像からSMILESを予測するためのクラスが作成できました!
あとは変換したい画像のファイルパスを指定すれば予測を実行できます。
# exaplesディレクトリのdigital_example1.pngファイルを入力 res = img2mol(filepath="examples/digital_example1.png", cddd_server=cddd_server)
結果は辞書型で以下のようになっています。
print(type(res)) # > <class 'dict'> res # > {'filepath': 'examples/digital_example1.png', # > 'cddd': None, # > 'smiles': 'Cn1c(=O)c2c(nc(Sc3ccccc3)n2C)n(C)c1=O', # > 'mol': <rdkit.Chem.rdchem.Mol at 0x7fb09587eda0>}
入力ファイルとCDDD、結果のSMILESとRDKit Molオブジェクトも入っています。
今回入力に使ったのは以下の画像。
input_img = Image.open(res["filepath"], "r") display(input_img)
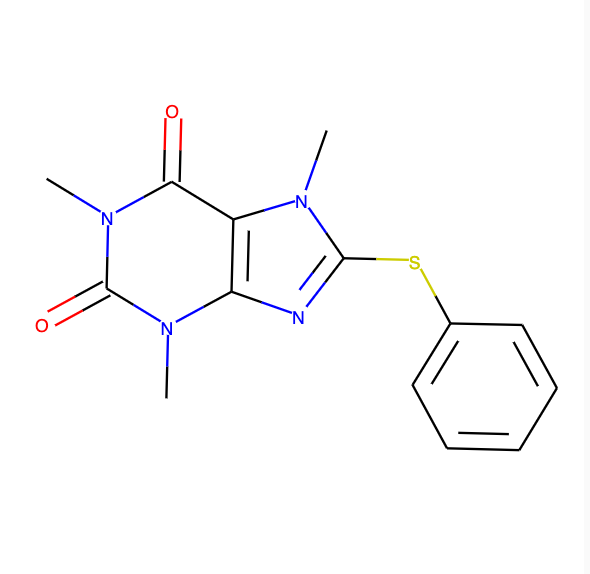
予測結果はこんな感じ。RDKit Molオブジェクトなのでjupyter notebookで構造式をそのまま描画できます。
# 変換後のSMILESを表示 print(res["smiles"]) # > Cn1c(=O)c2c(nc(Sc3ccccc3)n2C)n(C)c1=O # RDKit Molオブジェクトの構造式を描画 res["mol"]
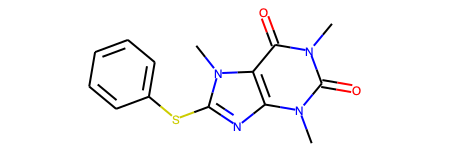
おお!正しく認識できてます!
私はSMILESを読めないのでわかりませんが構造式で確認できるので便利!!
手書きの構造式の例も用意されています。こういうのです。
# handwritten_example2.pngファイルを入力 res = img2mol(filepath="examples/handwritten_example2.jpg", cddd_server=cddd_server) input_img = Image.open(res["filepath"], "r") # 入力画像の確認 display(input_img)
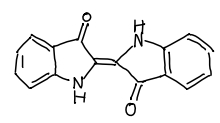
結果はこんな感じ
# 変換後のSMILESを表示 print(res["smiles"]) # > O=C1C(=C2Nc3ccccc3C2=O)Nc2ccccc21 # RDKit Molオブジェクトの構造式を描画 res["mol"]
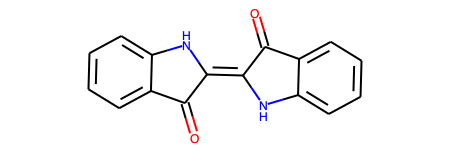
こっちもうまく認識できています!手書きの認識ができるなら授業のメモの電子化にも使えそうです!すごい!!!
ちなみに失敗例も提供されてたりします。コードは同じなので省略します。
こんなのです。
真ん中の環構造がフェニルではなくインドールとして認識されています。入力画像の右側ヘテロ環の結合関係がうまく認識できていないようです。
手書きの構造式は結合の長さや角度がまちまちだったり、元素記号のアルファベットが崩れていたりと、色々難しいポイントが多そうです。
いろいろな構造で遊ぼう
使い方が分かったので色々な画像を認識させてみましょう!
今年も色々な分子が話題となりましたが、なんといってもノーベル化学賞が有機触媒の開発に贈られたことは一大ニュースでしたよね!
というわけでプロリン画像をWikipediaから持ってきました(Wikipedia-プロリン)。こんな結果です。
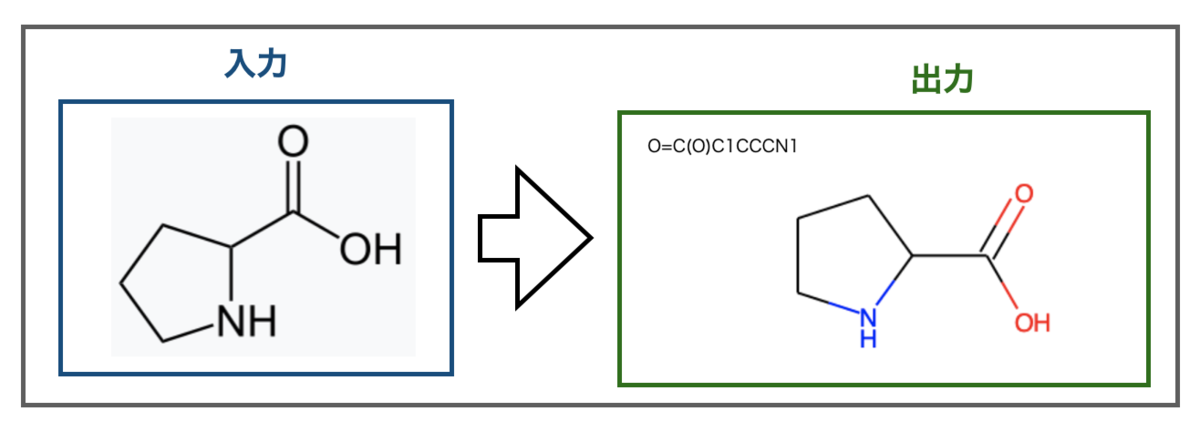
うまくできました!
マクミラン触媒はどうでしょうか?PubChemの構造を使ってみます((5S)-5-Benzyl-2,2,3-trimethylimidazolidin-4-one)。こんな感じです。
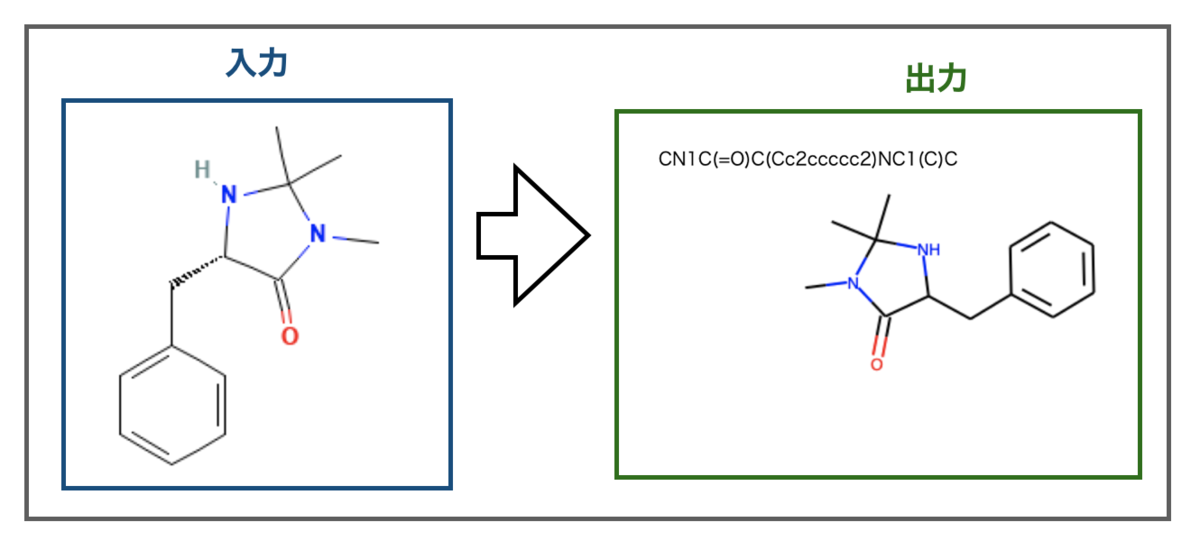
上手くいきました・・・が、不斉の情報までは認識できていないようです。出力のSMILESにキラル情報(@文字)がなく、構造式も不斉がありません。
ぱっとみた感じCDDDの学習で不斉の取り扱いまでは行っていないようなので、不斉情報は記述子に埋め込まれていないのかもしれません。
ところで化学構造の描画方法には今まで見てきたもの以外にも色々あります。Ball and StickやSpace-Fillingといったモデルで化学構造を表した画像はどのように認識されるのでしょうか?
同じくPubChemから画像を作成してImg2Molに投げてみました。
Ball and Stick描画したマクミラン触媒はこんな結果になりました。
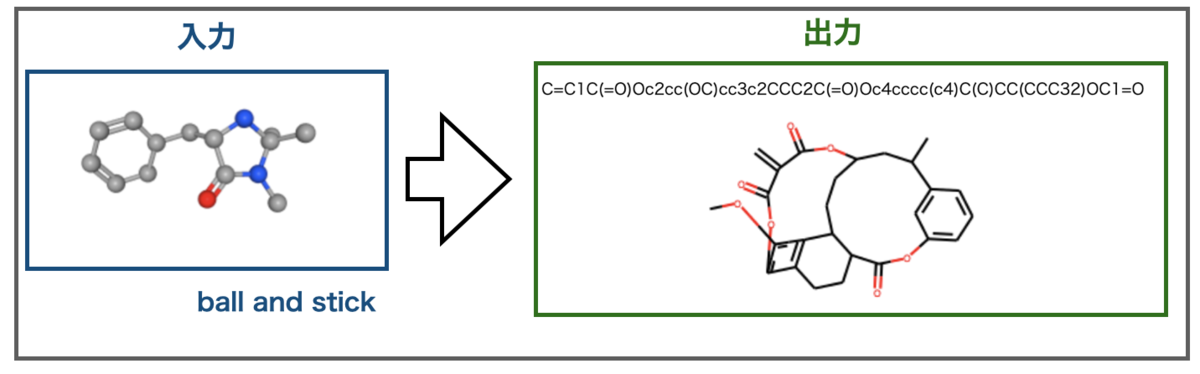
すごい構造。。。出力には窒素原子が含まれていませんが、色で原子を区別した描画では区別できないのでしょうか?
次にSpace-Fillingで描画したマクミラン触媒はこんな結果でした。
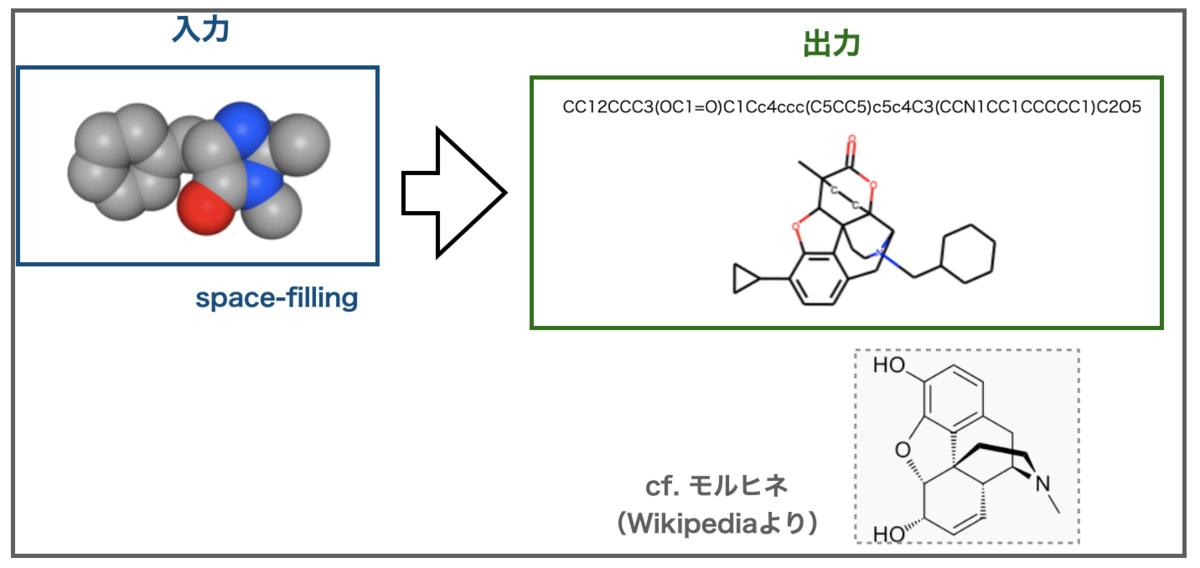
面白いことにSpace-Fillingの出力はモルヒネ様の骨格に認識されました。さらに今度は窒素原子も入っています???
出力に含まれる酸素原子の数を考えると、色で窒素を認識できていると思えないのでモルヒネ様に誤認識して引きづられた結果かもしれません。Img2Mol内部でどの様に認識されているのでしょうか???
不適切な入力構造はここまでにして。。。。
さて論文中では分子のサイズ(原子数)が大きくなると精度が落ちるとの記載がありました。最後に試してみましょう!
今年話題になった分子といえばKRAS G12C阻害剤として承認されたAmgenのSotorasibがありますね(Wikipedia - Sotorasib)。組成式C30H30F2N6O3なので、水素原子を除いても原子数41となります。
さてどうなるでしょうか?
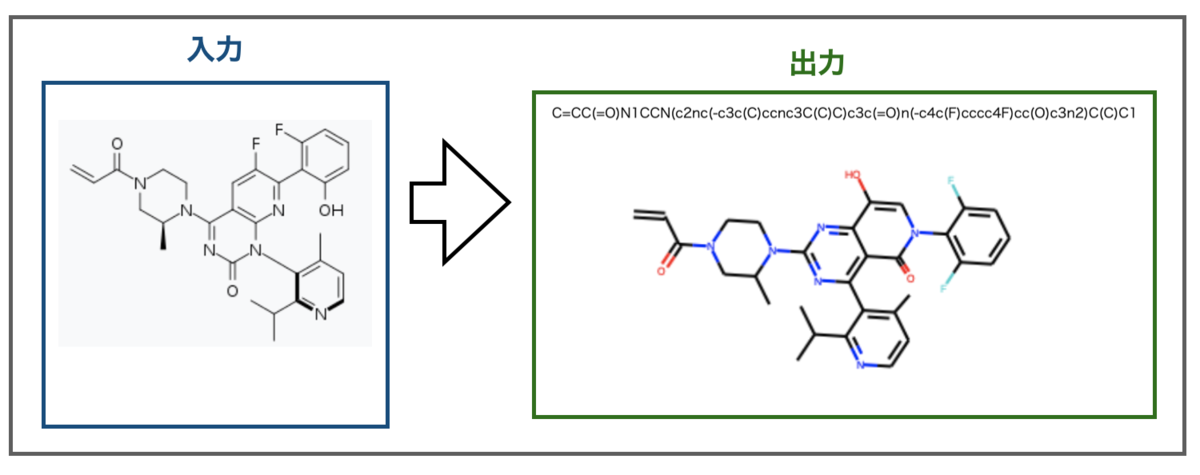
ぱっと見認識できている様にも見えますが、ちょっとずつ違います。
骨格の真ん中の環構造はカルボニルの位置や他の環との接続の仕方が異なります。また右端のフェニル基はオルト位に置換があるのは良いもののどちらもフッ素原子になってしまっています。入力画像の真ん中の環のフッ素原子と位置が近いので誤認識したのかもしれません。
画像からの構造の読み取りが失敗したとしても大枠が正しいなら細かい修正だけで済むので良いかな?とも思いましたが、微妙にちょっとずつ違うと間違い探しみたいで逆に難しくなってしまうかもしれません。
課題
いくつか遊んでみた結果、かなり精度が高く便利なツールという印象ですがいくつかできないこともわかってきました。
- 不斉の認識
- 分子サイズの大きなもの
また、利用を想定されていないであろう描画方法(ex. Ball and Stick, Space-Filling) 、色で表された原子の識別はできませんでした。
化学構造の特徴を学習した潜在空間にうつすならひょっとしていけるかも…と思いましたがダメでした。無茶を言ってすみません。
この他にRDKit UGMの動画で指摘されていたものにはキャプションつきの画像がありました。化学構造式以外の情報(化合物名など)が同じ画像の中にあると区別して認識できないとのこと。
「画像のセグメンテーションをどうするか?」が次の課題のようです。
おわりに
以上、今回は化学構造式のOCRソフト、Img2Molで遊んでみました。課題はあるもののかなり便利なツールだなぁと思いました。
ところで国語の成績も悪かった私ですがいまだに覚えている短歌があります。
「のみ込めぬままに図表は消されゆき 遠き席にて聴き終えて立つ」
詠み手*8が学生の時、講義の内容が理解できないうちに板書が消されてしまい、そのまま講義が終了してしまって「あーあ、やんなっちゃったよ」みたいな内容だった気がします。
なぜ覚えているか???
同じ経験をたくさんしたからです!
そう!化学構造式が超高速で現れては消えていく有機化学の授業でね!!
Img2Molがあればパシャーと写真撮って、後で画像からいい感じで構造式を抽出できたかもしれません。。。
今回も色々と間違いが多そうです。特に論文の中身。ご指摘いただけると嬉しいです。
おしまい。
*1:DeNAのデータサイエンティストの方々が参加されたチームが3位入賞・金メダルを獲得されたそうです(DeNA News)。格好良い!
*2:CC BY-NC 3.0
*3:オートエンコーダ(Autoencoder)とは|意味、仕組み、種類、活用事例を解説
*4:CDDD文献中にも引用されています
*5:ちなみのコードのライセンスはApache-2.0です。但しcddd_server.pyもCC BY-NC 4.0なので注意が必要です。
*6:NVIDIA、macOSのサポートを正式に終了した「CUDA Toolkit v11」をリリース。MacではCUDAアプリの開発と実行は出来ない状態に
*7:ネットワークの学習も自分でやろうとしたら不具合が出るのかもしれません。いつも通り無理矢理押し切っているのでおすすめしません。