化学構造認識(OCSR) DECIMERプロジェクトとDECIMER 1.0で遊んだ話
前回の記事で化学構造式のOCRソフトImg2Molで遊んでみました。「画像に含まれる化学構造式を認識してSMILESに変換」してくれるというものでした。
Img2Molと同時期に同様のタスクを扱うソフト DECIMER 1.0が公開されています。
- 文献①(オープンアクセス) : DECIMER 1.0: deep learning for chemical image recognition using transformers J. Cheminform. 2021(13)61
- GitHub : DECIMAR-Image_Transformer
こちらもコードや学習済みパラメータを公開してくださっています‼︎ Img2Molとの比較も兼ねて遊んでみましょう!
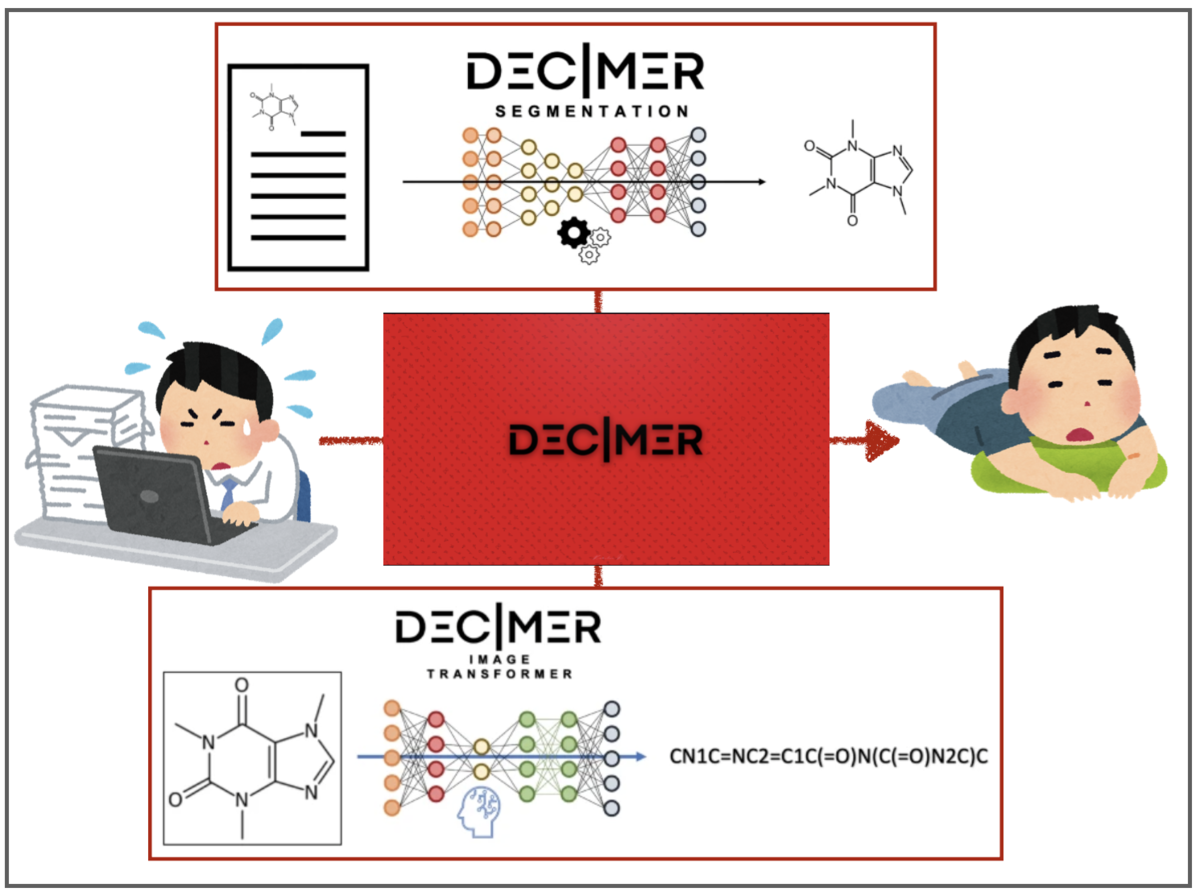
1. DECIMER文献の流し読み
1-1. DECIMERプロジェクトについて
さて、Img2Molは製薬会社バイエルの機械学習チームによるものでしたが、DECIMERはドイツのFriedrich-Schiller大学Chirstoph Steinbeck教授の研究室(HPはこちら)で開発されたアカデミックな研究に由来するものです。
こちらもディープラーニングによる手法で、DECIMERはDeep lEarnig for Chemical ImagE Recognitionの略です。
このソフトは「化学構造式の光学認識(Optical Chemical Structure Recognition, OCSR)」の課題について、「最新の人工知能技術を使ったオープンソースの自動化されたソフトウェアを開発しよう!」というDECIMERプロジェクトの中の一つです(プロジェクトWebサイト)。
関連する文献として、今回取り上げる文献の前のバージョン(DECIMER(無印))や、、、
- 文献②:DECIMER: towards deep learning for chemical image recognition J. Cheminform. 2020(12)65
テキストと画像の入り混じった文献を区分けして、画像部分をとりだすセグメンテーションのためのツール、、、
- 文献③:DECIMER-Segmentation: Automated extraction of chemical structure depictions from scientific literature J. Cheminform. 2021(13)20
があります(どちらもオープンアクセス)。後者のツールはウェブアプリとして利用可能になっています。
全て筆頭著者はKohulan Rajanさんとなっていますので、こちらの方が中心になって進められているようですね。すごい。
1-2. DECIMER 1.0のモデル
それではDECIMER 1.0の具体的な中身を見てみましょう!Img2Molと同じくディープラーニングに基づく手法ですが、具体的な中身は結構違います。
おさらいですが、Img2Molは画像を畳み込みニューラルネットワーク(CNN)で認識し、化学構造式の特徴を表現する空間(CDDD)に一度埋め込んだあと、線形表記(Canonical SMILES)として取り出す、という2段階の構成でした。またCDDDはオートエンコーダーによるものでした。
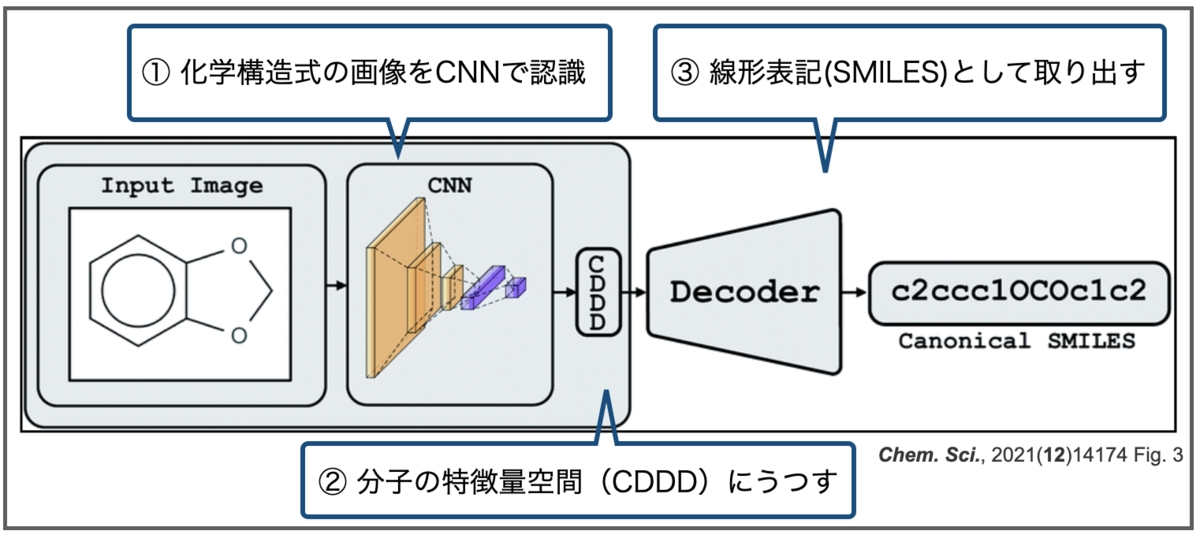
対してDECIMER 1.0も、画像を認識するための入力ネットワークと、認識した情報から化学構造式を取り出すためのネットワークの2段階の構成となっています。
ですが、化学構造を取り扱うネットワークを構築するために線形表記 SELFIESも利用していること、またTransformerベースのモデルになっていること、といった異なる点もあるようです。
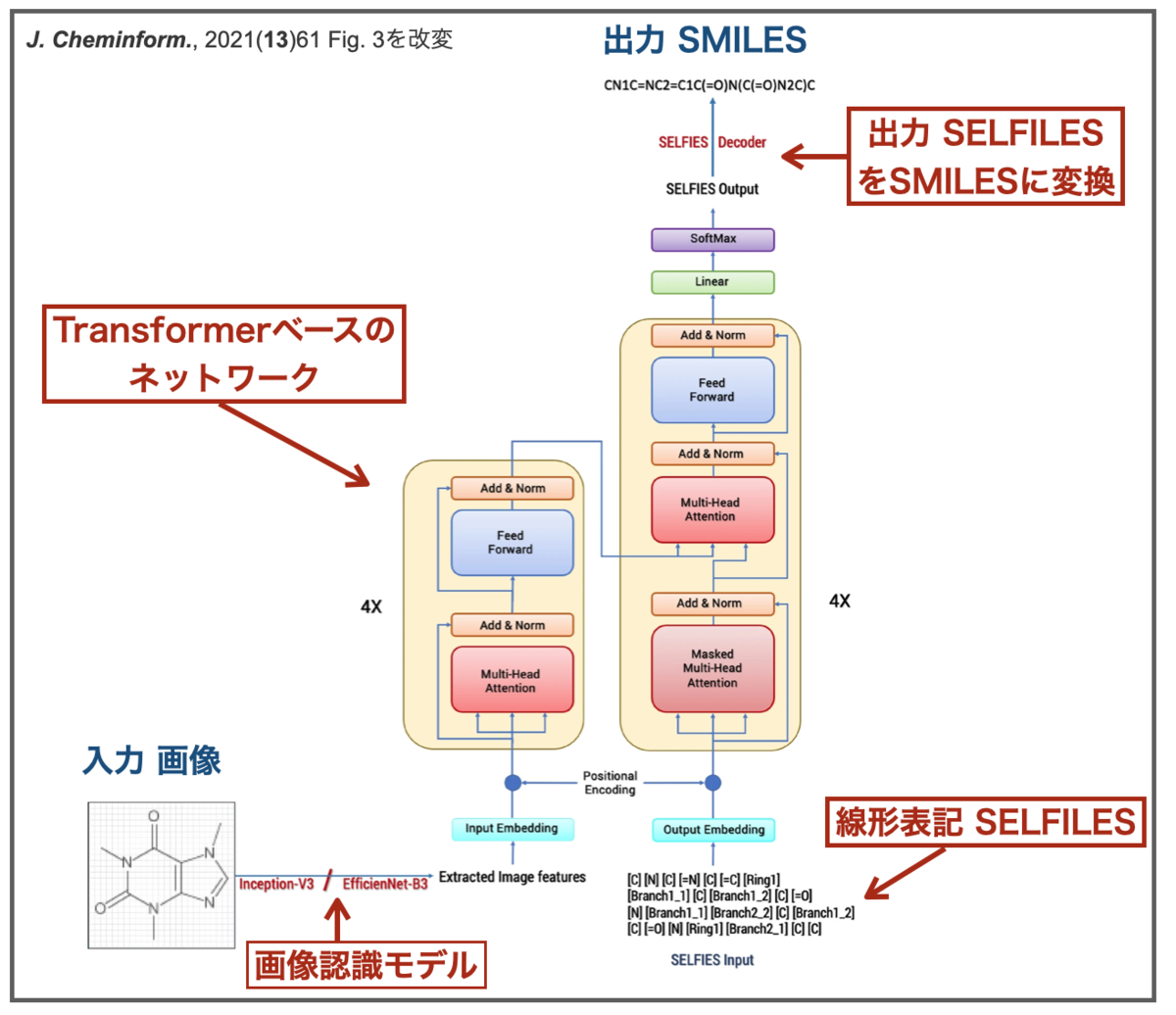
SELFIES (Self-referencing embedded strings)はAspuru-Guzik教授らのチームが開発した新しい化合物の線形表記方法で、この文法に従えばどんなふうに書いても100%分子として意味をなすという特徴があるそうです。
以下の記事が日本語でわかりやすく解説してくださっているのでおすすめです。
また、モデルのベースとなっているTransformerですが、こちらはAlphaFold2のベースにもなっていたやつですね。自然言語処理の分野で高い性能を発揮したらしく、よく使われているらしいです。
なお、前報DECIER(無印)ではオートエンコーダーをベースとしたモデルが使われています。DECIMER 1.0でもencoder-decoder型のモデルとTransformer型モデルが比較されており、結果、後者の方が精度が良かったそうです。
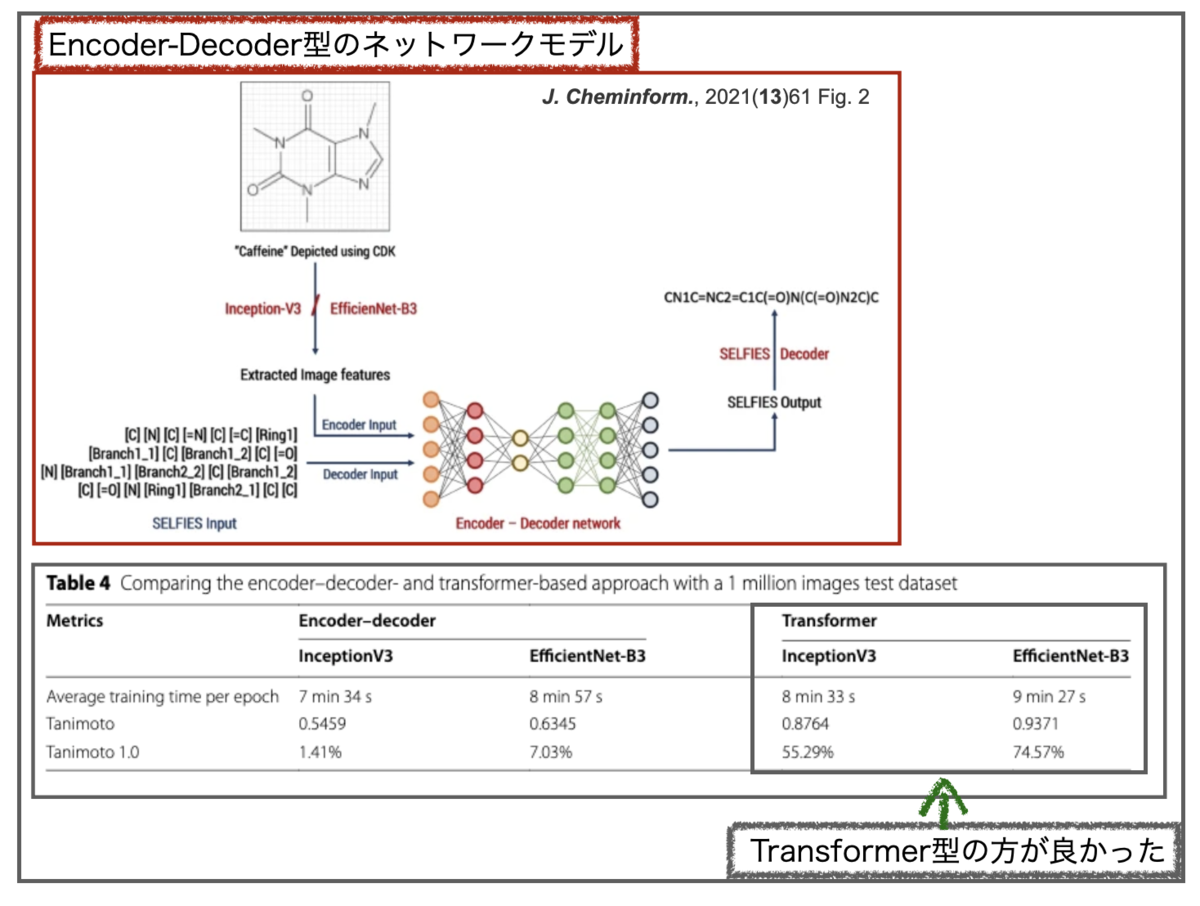
入力の画像認識 CNNのネットワーク構造としては、InceptionV3とEfficientNet-B3の2つを主に比較して、後者の方がパフォーマンスが良かった様です。
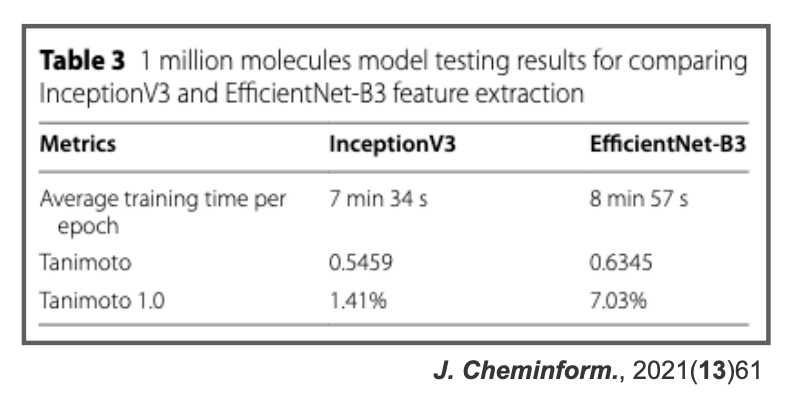
詳しくないのでよくわからないですが、各分野で最新の技術を色々と盛り込んだ感じでしょうか?最先端を攻める感じがアカデミックでいいですね!
1-3. DECIMER 1.0のデータセットと守備範囲
モデルの枠組みが大体わかったので、DECIMER 1.0の守備範囲についても参照しておきましょう!
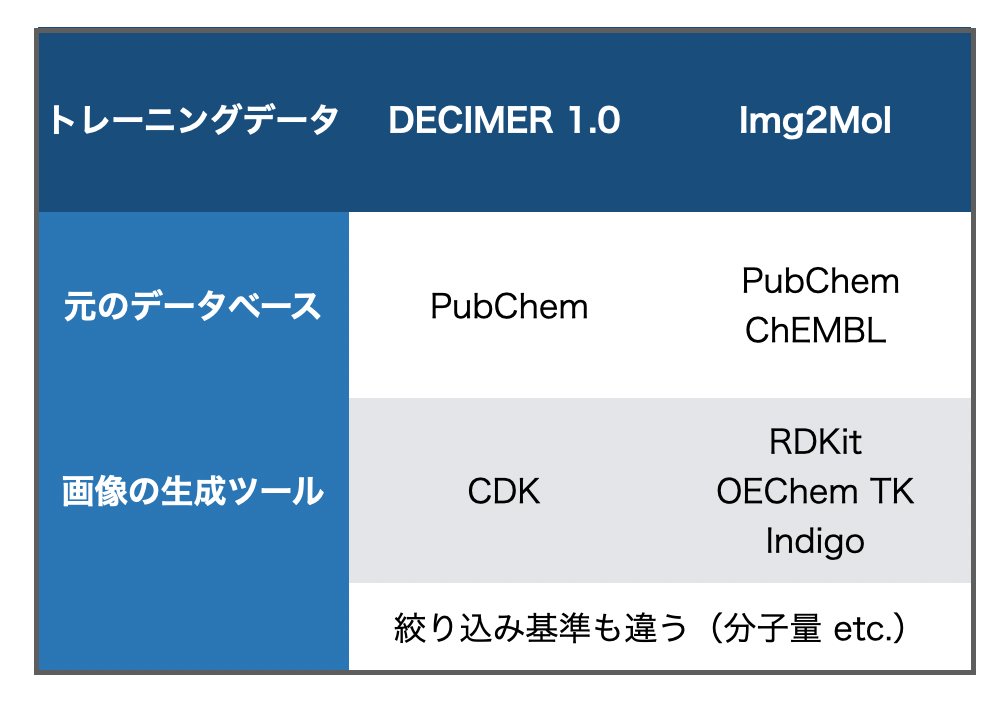
前回の記事では触れませんでしたが、Img2MolはPubChemとChEMBLから抽出し、一定の基準で絞り込んだ化合物をトレーニングに利用しています。また、トレーニング用の画像は複数のツール(RDKit、OpenEyeのOEChem TK、Indigo)を使って生成しています。
一方DECIMER 1.0は、対象データベースはPubChemのみ、トレーニング画像の生成はCDKのみを使用するといった、トレーニングデータセット準備段階での違いがあります。*2
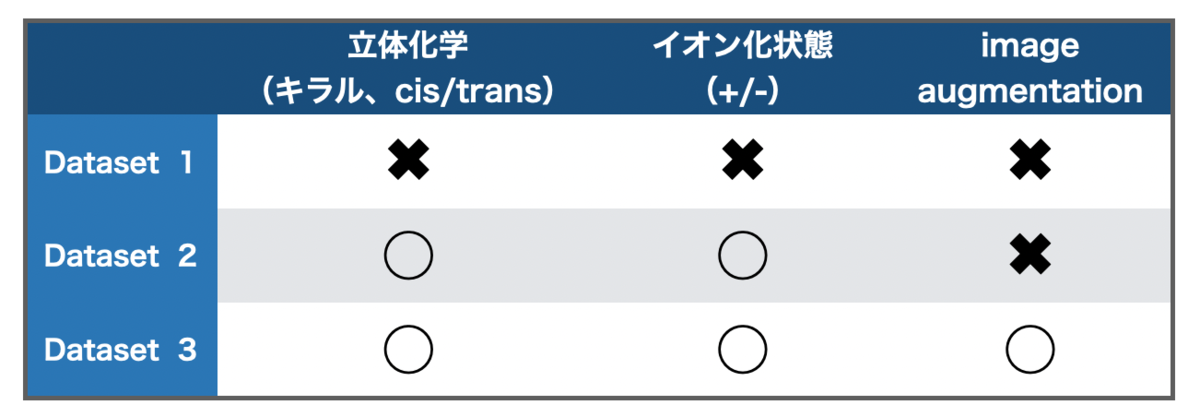
個人的に面白かった点は、DECIMER 1.0は「立体化学やイオン化状態を含めるとどうなるのかか?」という検証にも挑戦しているところです。
論文中で使われているデータセットは以下の様に「立体化学/イオン化の情報があるか?」、「Image augmentationをしたか?」で3つに分かれています。
以下のTable 8がDataset 1に対する結果、Table 12がDataset 2に対する結果です。データセットのサイズが近いsubset同士を比較すると、Table 12の方がパフォーマンス(Tanimoto、Tanimoto 1.0)が低下する傾向にあるようです。
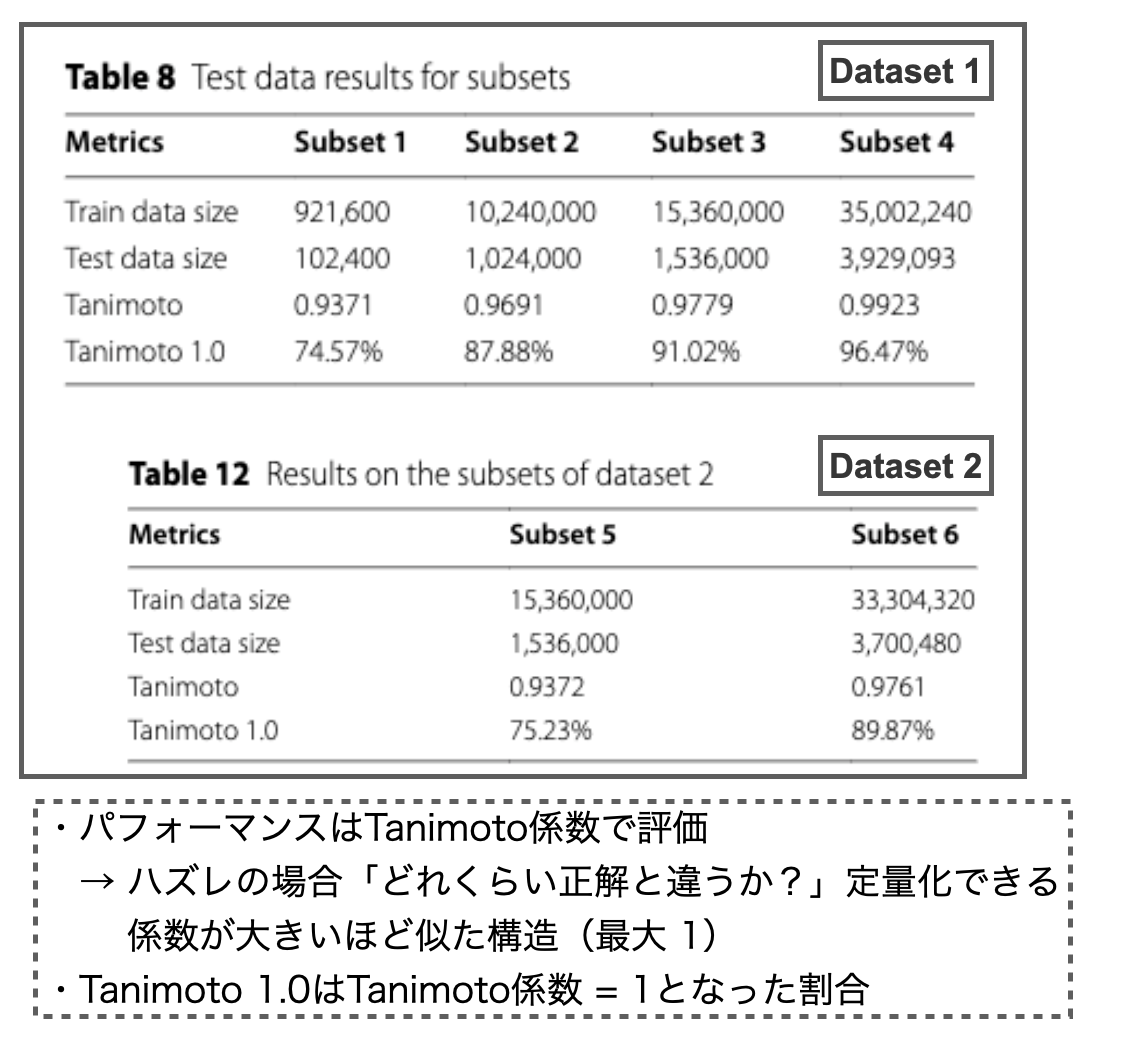
立体化学やイオン化状態の識別も考え始めると、問題が一気に複雑化するのでパフォーマンスが落ちるのは納得です。それでも結構良い結果が得られているように思いますが、皆さんはどう思われますか?
なおImage augmentationは、データ拡張を行ってDataset 2のデータを追加する(Dataset 3)とパフォーマンスが良くなるかを検証したものです。興味のある方は論文のFig. 10やTable 15あたりをご参照ください。
2. DECIMER 1.0で遊ぼう!
DECIMER 1.0の概要とImg2Molの違いがだいたい分かってきたので早速遊んでみましょう!
DECIMER 1.0はMITライセンスで公開されているので企業の方にも手を伸ばしやすいかもしれません。
後ほど記載しますが、学習済みパラメータはGoogle cloudのStorageにあり、プログラムを初めて利用するときにダウンロードする仕組みとなっているみたいです。
2-1. インストール
インストール方法はGitHubのREADMEの通りです。conda環境での利用が簡単なのでオススメとのことです。
# pipでgithubからインストール $ pip install git+https://github.com/Kohulan/DECIMER-Image_Transformer.git # もしくは以下でPyPiからインストールしてもOK $ pip install decimer
ところが、Img2Molの時と同様、Macユーザーはちょっとだけ面倒です。
MacOSでNvidia GPUがない場合は「tensorflow==2.3.0」を指定して入れなければいけないとの記載があります。上のコマンドでは私のMacにDECIMERをインストールできませんでした。
そこでgit cloneでDECIMERを落としてきた後、setup.cfgを少し書き換えるとインストールできました。
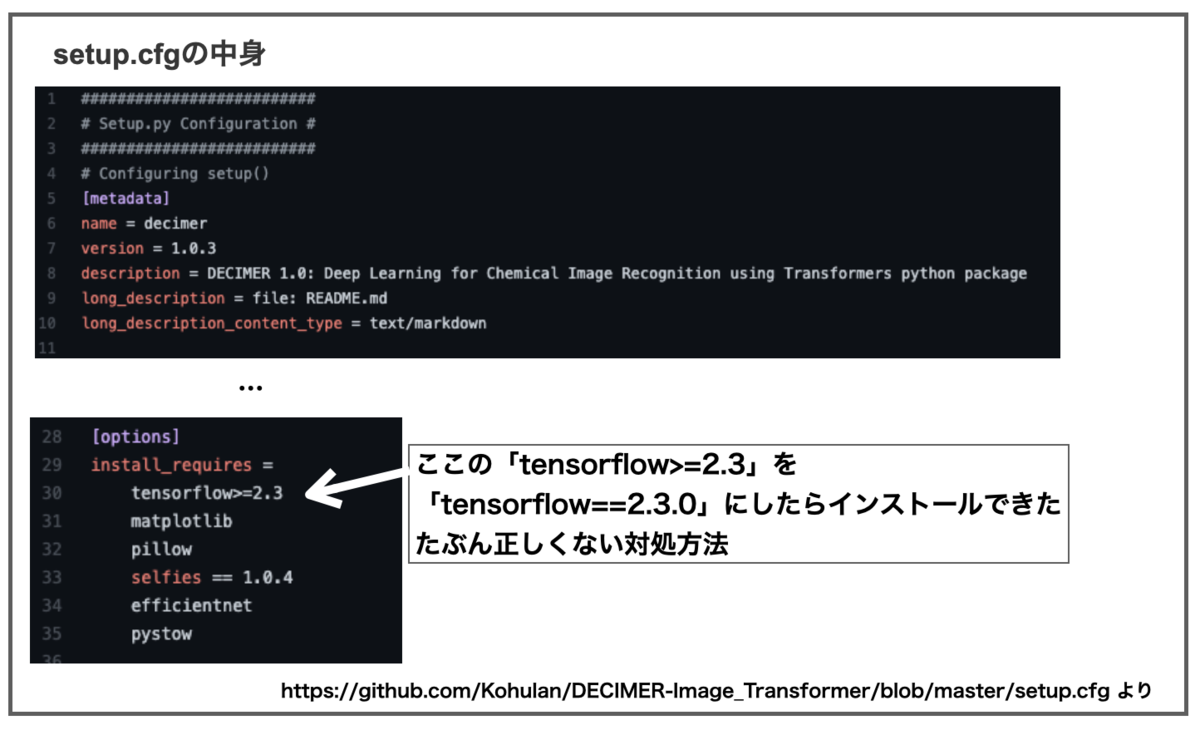
こういう感じの作業でした。
# githubからディレクトリを落としてくる $ git clone https://github.com/Kohulan/DECIMER-Image_Transformer.git decimer # setup.cfgを上図の様に書き換えて保存 # ディレクトリを移動 $ cd decimer/ # インストール開始 $ pip install -e.
どなたか正しい方法を教えてください。
2-2. 訓練済みパラメータの入手
先に書いた様に、DECIMER 1.0は学習済みパラメータも公開されていて、初回利用時にダウンロードされます。
私はパラメータの保存場所などが後でわからなくなるのを防ぐために、手動でダウンロードしてdecimerディレクトリの中に入れることにしました。
decimerのsrc/decimer/ディレクトリにあるDECIMER.pyをみると以下の記載があります。

デフォルトのディレクトリにパラメータファイルがなければmodel_urlからダウンロードしてくるみたいなことが書いてあります(たぶん)。
というわけでmolde_url = "https://~~~~" となっているURLにアクセスしてパラメータのzipファイルをダウンロードしました(1GBくらい)。
パラメータ保存場所として、DECIMERディレクトリにTrained_Modelsという新しいディレクトリをつくり、そこにzipを展開したファイルを格納しました。あとはDECIMER.pyやDECIMER_V1.0.pyの中にあるモデルのパスの箇所を書き換えて保存したら終わりです。
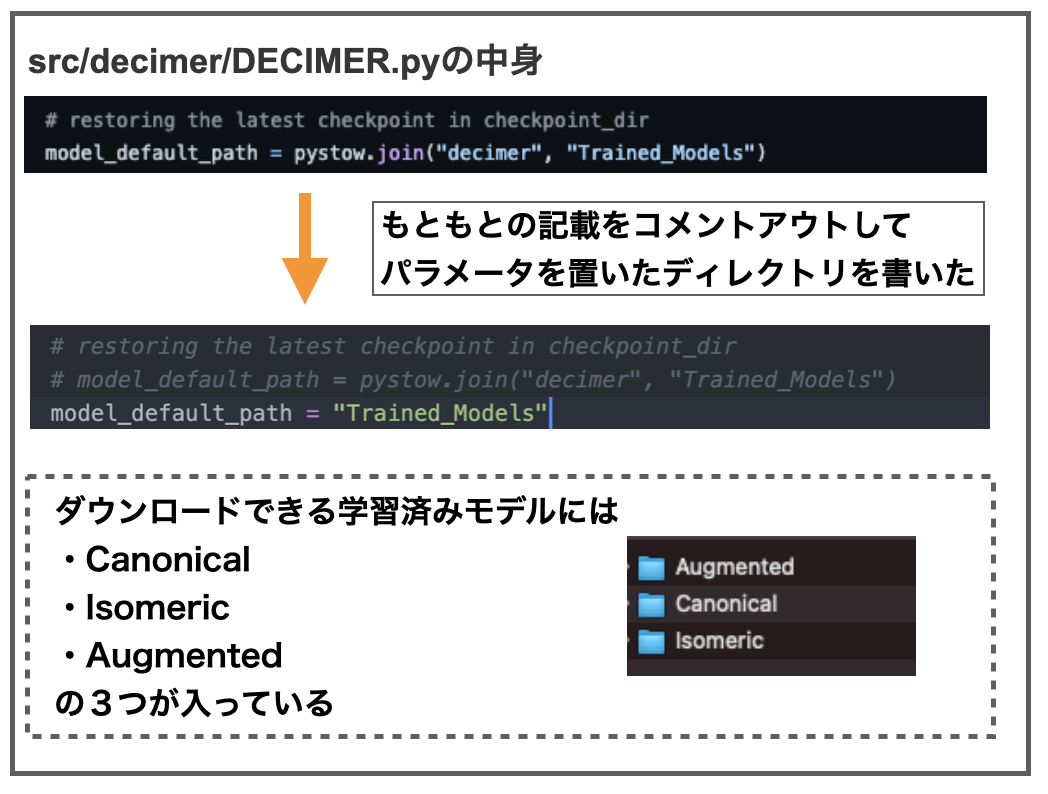
訓練済みのモデルにはCanonical、Isomeric、Augmentedの3つがあります。おそらく文献で触れられていた3つのデータセットに対応するものだと思います。*3
ダウンロードファイルの保存先を覚えておけばこんな余計なことをする必要はないと思います。私はすぐ忘れるんです。。。
何はともあれインストールとパラメータの準備ができました!
2-3. サンプルで遊んでみよう!
とりあえずJupyter notebookを起動して遊んでみましょう。Sample_Imagesディレクトリに画像例も準備してくださっているので利用します。
ライブラリをインポートしたあと、画像ファイルのパスと使いたいモデルを指定するだけでSMILESに変換して返してくれます。
from decimer import DECIMER # モデルの指定 model_name = "Isomeric" # 画像のパスを指定 img_path = "Sample_Images/caffeine.png" # 変換前に元々の画像を確認する from IPython.display import display from PIL import Image display(Image.open(img_path))
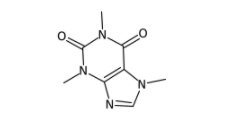
上のような画像を入力とします。さてDECIMERは無事SMILESに変換できるでしょうか??
# DECIMERでSMILESを予測 res = DECIMER.predict_SMILES(img_path, model_name) print(res) # > CN1C=NC2=C1C(=O)N(C)C(=O)N2C
SMILESができました!でもあってるのかわからない!読めない!
RDKitで変換した図をみてみます。(DECIMERと共存させられなかったので別のnotebookでやりました。)
from rdkit import Chem from rdkit.Chem import Draw m = Chem.MolFromSmiles("CN1C=NC2=C1C(=O)N(C)C(=O)N2C") Draw.MolToImage(m)
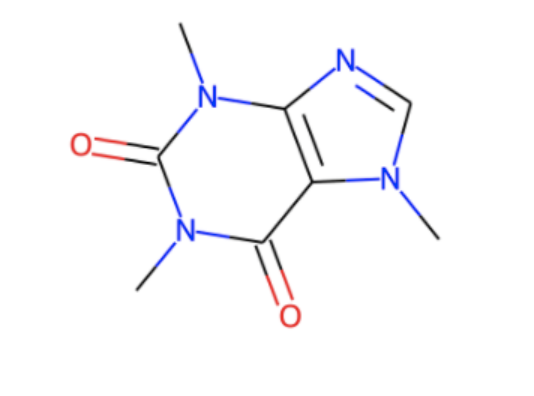
どうやらDECIMERはカフェインを認識してうまくSMILESに変換できたようです!
他のmodelではどうでしょうか?CanonicalとAugmentedで行った結果はこんな感じです。
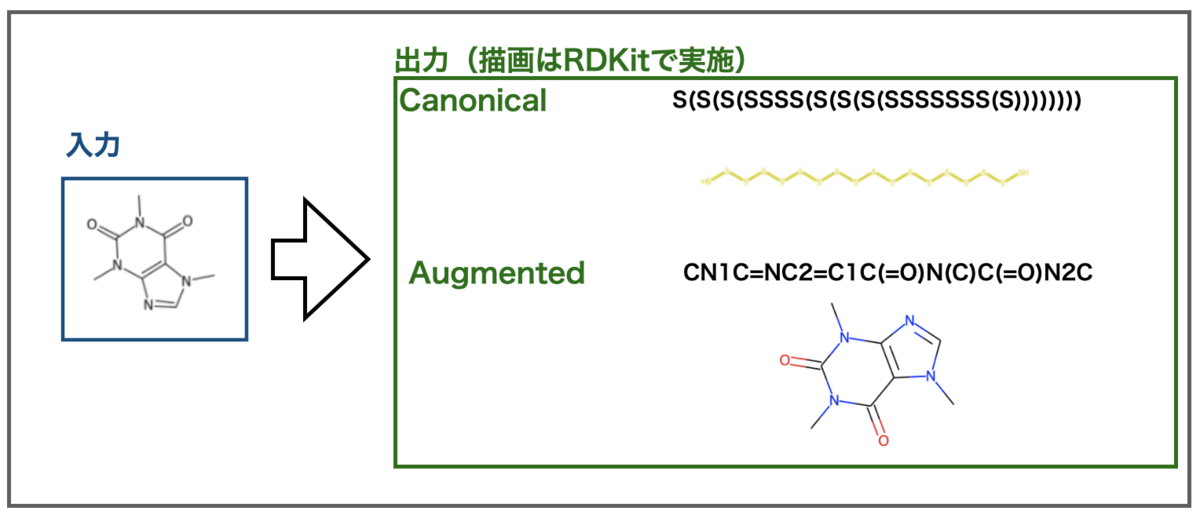
AugmentedはIsomericと同様正しく認識できました!残念ながらCanonicalは全ての原子が硫黄の鎖状構造と誤認識してしまったようです。
論文を見る限りAugmentedが一番良さそうなので、以降の検証はこちらで行ってみます。
気になるのは立体化学の認識です。サンプルファイルに立体化学を含む例がありました。
どうだ!
おお!不斉点が認識されました!!SMILESに@が入っているだけでテンションが上がります!!
RDKitでの描画の都合上向きが変わってしまっていますが、立体化学も正しいようです。また、不斉の明記されていない炭素原子もありますが、ここも不斉情報がないものとして正しく認識できています。すごい!!
次に電荷を帯びたイオンはどうでしょうか??こんな例がサンプルにありました。
プラス電荷が認識されました!サンプルなのでうまくいくだろうとは思っていても実際に結果が出ると嬉しいですね!
話がそれますが、描画を並べてみるとRDKitの原子を色分けするスタイルはわかりやすくて良いですね。
2-4. 別のサンプルでテストしよう!
さてDECIMERの使い方がわかってきました。
次に気になるのはソフトの汎用性です。ソフトウェアのサンプル画像以外でもうまく機能するでしょうか???
比較対象としてちょうど良いのでImg2Molに提供されていたexamplesでテストしてみましょう!
カフェインに類似した構造の画像例です。
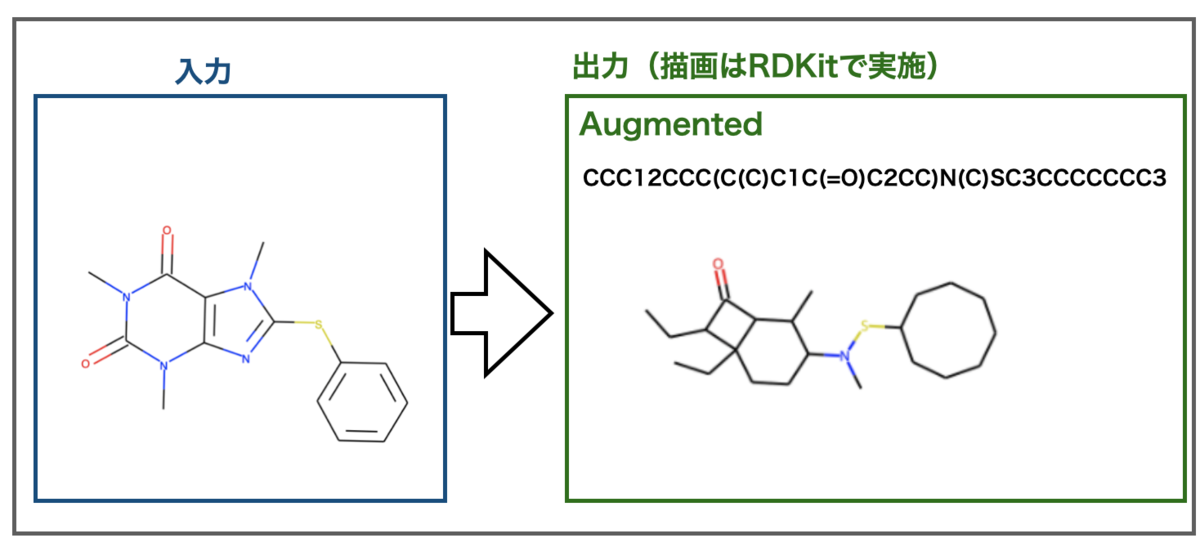
残念!だめでした。。。出力は芳香族がうまく認識できておらず、窒素-硫黄結合といった構造もできてしまっています。
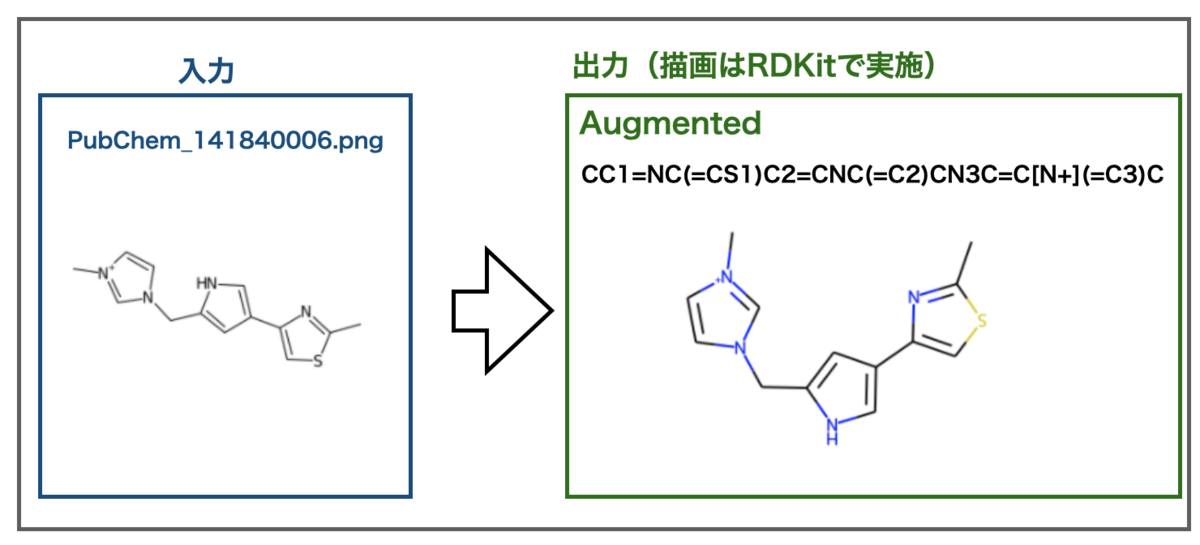
別の例ではどうでしょうか?
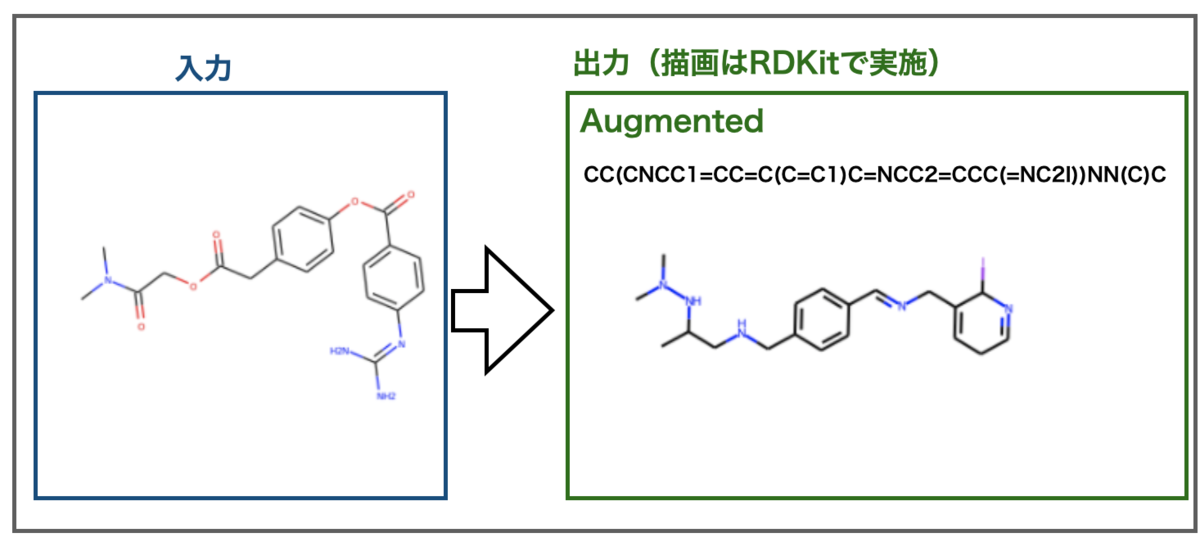
こちらもだめでした。今回は芳香環を一つ認識できたましたが、なんとなく似ていそうで全然違う結果になってしまいました。
ひょっとして画像の化学構造式に色が含まれているのが悪かったりするのでしょうか?
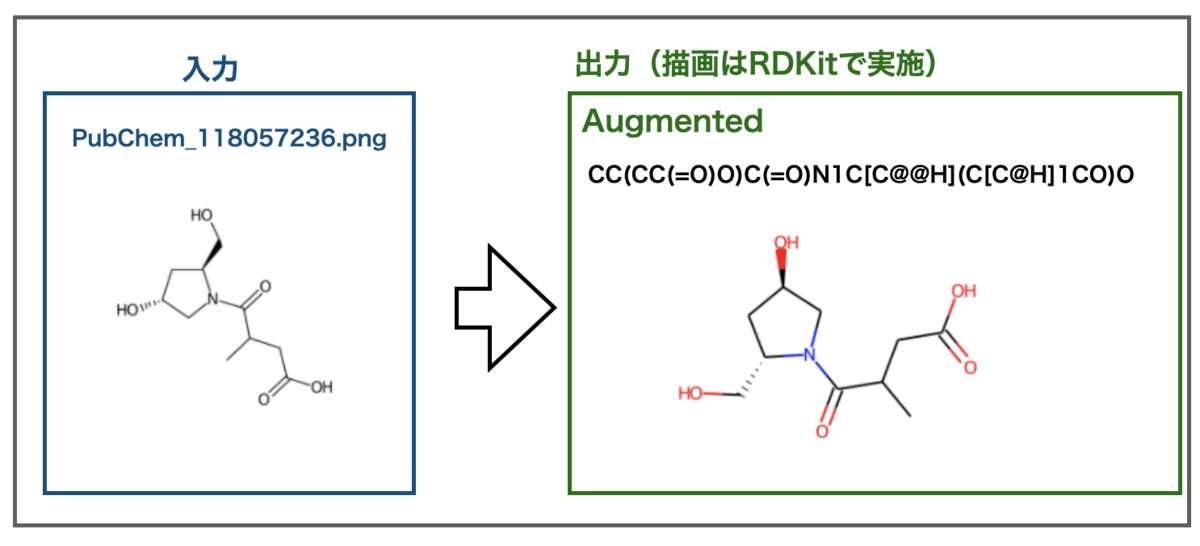
別の画像例としてWikipediaのプロリンを使ってみましょう。元素記号が大きく、原子の色分けがない画像です。こちらもImg2Molではうまくいったものです。
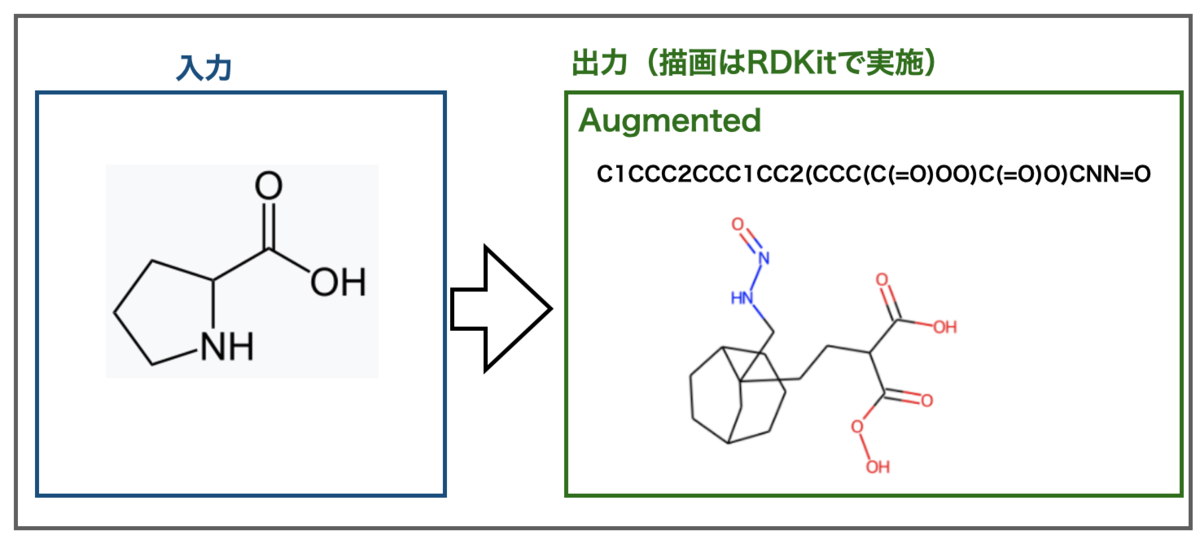
なんだかすごい構造になりました。。。こんなアミノ酸は嫌だ。
最後に手書き画像の例を見てみましょう。Img2Molのサンプルです。
こんな結果になりました。
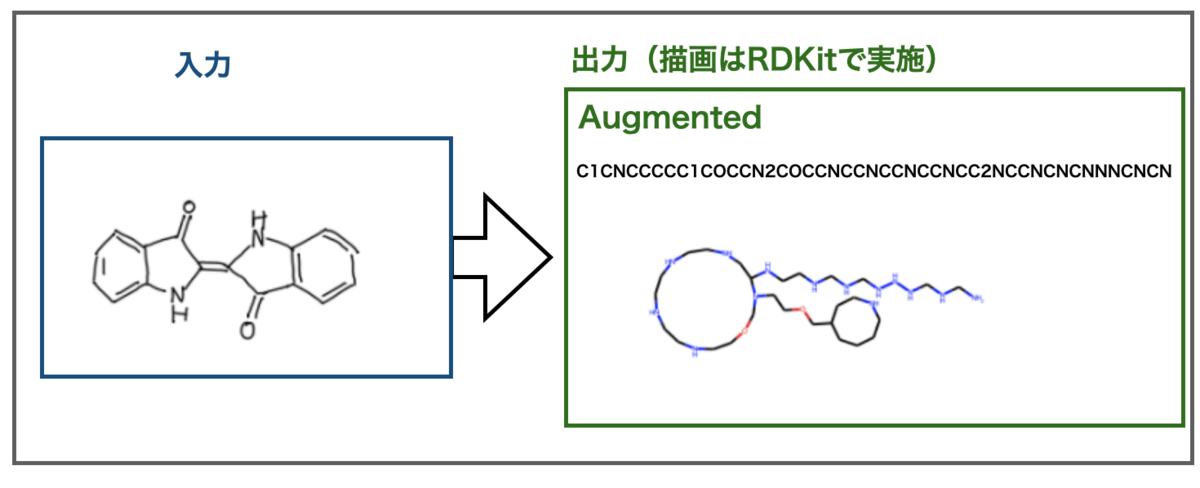
DECIMER 1.0の文献中にも記載がありましたが、環構造の認識に失敗すると、ヒトの目にはとても大きな間違いにみえますね。
上手くはいきませんでしたが手書きの構造式も化学構造として認識できいるという点はなかなかすごいと思います。
3. おわりに
以上、今回は化学構造式のOCRソフト DECIMER 1.0で遊んでみました。Img2Molと比較していかがだったでしょうか?
個人的には立体化学の認識、電荷の認識にも挑戦していて、サンプル画像ではうまく認識できているという点が驚きでした。Img2Molは立体化学を全て無視するようでしたのでDECIMERの長所だと思いました。
一方で、サンプル画像以外の画像では認識があまり上手くいっていないという印象でした。Img2Molは小さな分子であればWikipediaやPubChemのキャプチャ画像でも認識できたので、汎化性能(?)という点では課題があるかもしれません。
このあたりトレーニングデータセットの作り方による差もありそうです。Img2Molでは複数のツールで入力画像を作っていたのに対し、DECIMERはCDKのみでした。それぞれ異なる描画のクセがあるソフトを組みあせたデータセットの方がバラエティがあり、汎化性能が高くなりそうです。
DECIMER 1.0をImg2Molのデータセットでトレーニングしたらどうなるか?気になるところです。
あくまで素人の感想です。すみません。。。
また、DECIMERプロジェクトはImg2Molで今後の課題となっていたセグメンテーションにも既にWebアプリを公開しています。OCSRに関わる複数のソフト群全体の開発として取り組まれているので、シナジー効果によりさらに精度が改善していくかもしれません。次報が楽しみですね!
今回も色々と間違いが多そうです。ご指摘いただけると嬉しいです。
ではでは!