アラート ≠ 禁止 〜ZINCデータベースの医薬品にPAINSフィルターをかけてみた話〜
創薬(dry)アドベントカレンダーを埋めてみます。アドベントカレンダーの記事を見ると「いよいよ年末かー」となりますね。
さて、今年も色々な格好いい分子が出てきてケミカルスペースは広大だなぁと思いました。
というわけで発表します!びっくりモルキュールオブザイヤー!
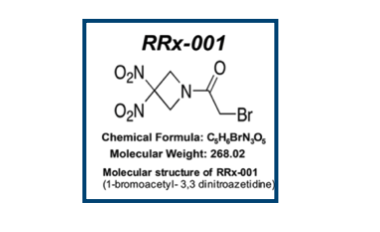
抗がん剤として臨床開発中だそうです。論文は以下。
- Discovery of RRx-001, a Myc and CD47 Downregulating Small Molecule with Tumor Targeted Cytotoxicity and Healthy Tissue Cytoprotective Properties in Clinical Development
J. Med. Chem. 2021(64)7261
ニトロ基2つにアミドのα位にハロゲン原子。。。見ただけで涙がちょちょ切れそうです。
これを臨床開発するなんて本当にびっくりですが、逆にいうと常識(?)みたいなものに囚われて有用性が見過ごされている分子がまだまだあるかもしれません。
そこで今回は「承認済みの医薬品の中にどれくらい忌避構造(アラート構造)が含まれているか?」調べてみることにしました。
1. この記事でやること
やることは単純です。ZINC15データベースにあるFDA approved drugsの一覧を使って、RDKitでPAINSフィルターにかかる分子がどれくらいあるか探してみるだけです。
2. データの準備
「ZINCデータベースは、バーチャルスクリーニングのために特別に準備された、市販の化学物質に関する精選されたコレクション」だそうです(Wikipedia - ZINCデータベースより)。
カリフォルニア大学サンフランシスコ校(UCSF)のJohn J. Irwin博士の研究室とBrian K. Schoichet博士の研究室により開発・維持されているそうで、現在はZINC20です。
- ZINC20—A Free Ultralarge-Scale Chemical Database for Ligand Discovery
J. Chem. Inf. Model 2020(60)6065
サーバーが混んでいてアクセスできなかったので今回はZINC 15を使います。
- ZINC 15 – Ligand Discovery for Everyone
J. Chem. Inf. Model 2015(55)2324)
よく知らないけどよく見るからこれを使っとけば良いんでしょう!知らんけど。
こんなページです。
ここにSubstancesがあるじゃろ。これをこうして。こうじゃ。
これでFDA承認済みの医薬品構造のSDFが手に入りました!
2015年に公開されたデータベースのようなので、少し古いかもしれませんが今回はこれで。。。
3. PAINS
3-1. PAINSフィルターって?
PAINSフィルターについては「化学の新しいカタチ」さんのブログ(RDKitのPAINSフィルターで化合物をスクリーニング)でわかりやすく解説してくださっています(RDKitのコードも!) 。
PAINSはPan-Assay INterference compoundsの略だそうです。
- New Substructure Filters for Removal of Pan Assay Interference Compounds (PAINS) from Screening Libraries and for Their Exclusion in Bioassays
J. Med. Chem. 2010(53)2719
ある種の部分構造を持つ化合物は、ハイスループットスクリーニング(HTS)を実施した際に、色々なアッセイ(pan-assay)で活性化合物としてヒットしてくるそうです。このような化合物は偽陽性である可能性が高くなるため要注意です。
そんなアッセイの妨害になる化合物(interference compounds)をフィルタリングして取り除けるように、「予め要注意な部分構造をリスト化しておこう!」というわけで作られたのがPAINSフィルターみたいです。
3-2. PAINSフィルターをもう少し
PAINSフィルターの具体的な中身についてもう少し見てみましょう!
2018年 ACS Chemical BiologyにPAINS公開後7年を経て再度その内容を振り返るといった趣旨の文献が出されています。こちらはオープンアクセスとなっています*2
- Seven Year Itch: Pan-Assay Interference Compounds (PAINS) in 2017—Utility and Limitations
ACS Chem. Biol. 2018(13)36
「PAINSは数秒で簡単に100個も1000個も大量に化合物選別できて便利だけどブラックボックスのまま使うのはやめてね!」ってな感じです。
そもそもPAINSに上げられている構造はどうしてアッセイの邪魔になってしまうでしょうか? 文献では以下の様な特徴が指摘されています。

タンパク質や試薬と反応してしまう構造(求電子剤、光反応剤)は非特異的な不可逆反応を起こしそうなのでわかりやすいですが、キレート剤やフォトクロミズムといったものは考えてもみませんでした。確かにアッセイの原理と相性が悪そうです。なるほど!
さてPAINSフィルターをブラックボックスとしないために把握しておくべき注意点ですが、PAINSが「商用化合物を使ってPPI(protein-protein interaction)阻害を対象に行われた6つのHTS(AlphaScreen)における観察」に基づくものであることに由来する様です。*3
注意点はこんな感じ。

PAINSフィルターを使うときは用法・用量を守って適切に!
参考までに②のそもそもライブラリに含まれていなかった構造の例としてFig. 1を引用しておきます。

ついでにFig. 2も面白いので引用しておきます。メカニズムも用語も色々あってわかりにくいから整理して分類したよ!って図です。
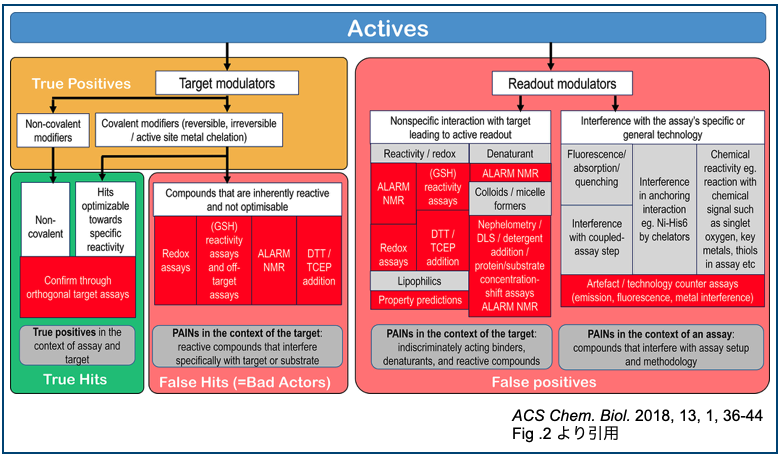
promiscuous binderとかfrequent hitter、bad actorってさらっと言えたら業界人っぽくないですか?・・・そうでもないですか。すみません。
4. RDKitのPAINSフィルターをつかってみよう
4-1. 簡単な使い方 ~ RRx-001を例に ~
だいたいPAINSのやりたいことがわかったので早速使ってみましょう!
まずは記事冒頭にあげたRRx-001を例にRDKitのPAINSフィルターを使ってみます。コードは化学の新しいカタチさんの記事とRDKitのブログ記事を参考にさせていただいています。
とりあえず化合物構造を準備します。
from rdkit import Chem from rdkit.Chem import AllChem, Draw # PubChemよりRRx-001のSMILESを取得(PubChem ID : 15950826) RRx_001_smiles = "C1C(CN1C(=O)CBr)([N+](=O)[O-])[N+](=O)[O-]" # Molオブジェクトを作って描画 RRx_001_mol = Chem.MolFromSmiles(RRx_001_smiles) Draw.MolToImage(RRx_001_mol)
RDKitのPAINSフィルターには4つ(PAINS、PAINS_A、PAINS_B、PAINS_C)あります。ABCはそれぞれPAINSを作る際のHTSで150以上の化合物に含まれた構造(Family_Filter_A)、15 - 149化合物(Family_Filter_B)、14以下(Family_Filter_B)に対応しているようです。
よくわからないので「PAINS(無印)」を使ってみす。
FilterCatalogに実装されており、FilterCatalogParamsオブジェクトに使いたいフィルタリング要素を追加してフィルターを作った後、目的の化合物に適用すれば良いそうです。
# インポート from rdkit.Chem import FilterCatalog # パラメータのオブジェクトを作る param = FilterCatalog.FilterCatalogParams() # PAINSフィルターをパラメータに追加する param.AddCatalog(FilterCatalog.FilterCatalogParams.FilterCatalogs.PAINS) # フィルターを作成 filt = FilterCatalog.FilterCatalog(param)
フィルターができたので適用します。
HasMatchで「フィルター中の部分構造が含まれているかどうか?」を確認することができます。
print(filt.HasMatch(RRx_001_mol)) # False
False !!! RRx-001はPAINSフィルターにはかからない化合物でした!見た感じヤバいのに。。。
他のフィルターではどうでしょうか?RDKitには他に3つのフィルター(BRENK、NIH、ZINC)が用意されています。それぞれ適用してみましょう。
# フィルター名のリスト filter_names = ["BRENK", "NIH", "ZINC"] # 順番に適用 for name in filter_names: p = FilterCatalog.FilterCatalogParams() fc = "FilterCatalog.FilterCatalogParams.FilterCatalogs." + name p.AddCatalog(eval(fc)) f = FilterCatalog.FilterCatalog(p) res = f.HasMatch(RRx_001_mol) print("Does filter " + name + " detect RRx-001? : " + str(res) ) # Does filter BRENK detect RRx-001? : True # Does filter NIH detect RRx-001? : True # Does filter ZINC detect RRx-001? : False
BRENKとNIHではRRx-001はフィルターにかかりました!どの構造が認識されたのでしょうか?
GetMatchesで「具体的にどのようなフィルターにひっかかったか?」取り出せるそうです。NIHを例に行ってみます。
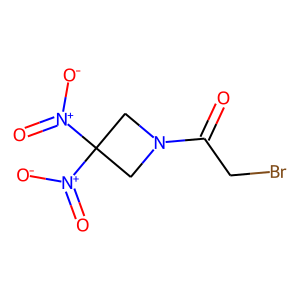
# NIHフィルターを作成 p2 = FilterCatalog.FilterCatalogParams() p2.AddCatalog(FilterCatalog.FilterCatalogParams.FilterCatalogs.NIH) f2 = FilterCatalog.FilterCatalog(p2) # フィルターに認識された構造情報を取り出す matches = f2.GetMatches(RRx_001_mol) for match in matches: print(match.GetProp("description")) print(match.GetProp("Reference")) print(match.GetProp("Scope")) print("------------") """ alpha_halo_carbonyl Jadhav A, et al. Quantitative Analyses of Aggregation, Autofluorescence, and Reactivity Artifacts in a Screen for Inhibitors of a Thiol Protease. J Med Chem 53 (2009) 37D51. doi:10.1021/jm901070c. annotate compounds with problematic functional groups ------------ non_ring_ketal Jadhav A, et al. Quantitative Analyses of Aggregation, Autofluorescence, and Reactivity Artifacts in a Screen for Inhibitors of a Thiol Protease. J Med Chem 53 (2009) 37D51. doi:10.1021/jm901070c. annotate compounds with problematic functional groups ------------ primary_halide_sulfate Jadhav A, et al. Quantitative Analyses of Aggregation, Autofluorescence, and Reactivity Artifacts in a Screen for Inhibitors of a Thiol Protease. J Med Chem 53 (2009) 37D51. doi:10.1021/jm901070c. annotate compounds with problematic functional groups ------------ """
「alpha_halo_carbonyl、non_ring_ketal、primary_halide_sulfate」の3つの部分構造が認識されること、NIHフィルターが以下の文献をもとにしていることがわかりました。
- Quantitative Analyses of Aggregation, Autofluorescence, and Reactivity Artifacts in a Screen for Inhibitors of a Thiol Protease
J. Med. Chem. 2010 (53)37
このままではわかりにくいので構造式上で確認してみましょう。フィルターにかかった構造のアトム番号はGetFilterMatchesで取り出せるそうです。
# descriptionのリスト descriptions = [match.GetProp("description") for match in matches] # アトム番号のリストのリスト atom_nums_list = [] for match in matches: atom_nums = [x[1] for x in match.GetFilterMatches(RRx_001_mol)[0].atomPairs] atom_nums_list.append(atom_nums) # 描画 Draw.MolsToGridImage([RRx_001_mol for _ in range(len(matches))], highlightAtomLists=atom_nums_list, legends=descriptions)
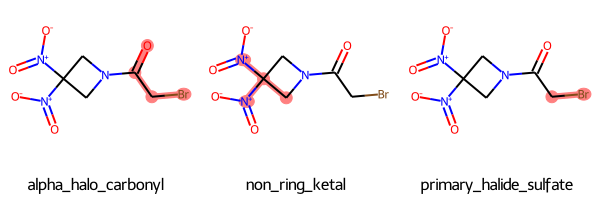
構造を描画するとわかりやすいですね!臭素原子はカルボニルα位のハロゲンや、primary-halideとして認識されています。意外にも(?)、ニトロ基はニトロ基としてではなく、同一炭素原子に2つの窒素原子が結合した構造(non_ring_ketal)として認識されている様です。
同じことの繰り返しなので省略しますがBRENKフィルターでは以下の結果が得られました。
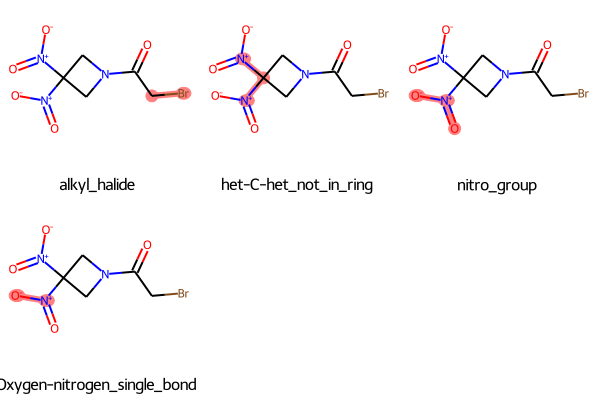
今度はニトロ基はnitro_group、Oxygen-nitrogen_single_bondとしても認識されている様です。見比べると面白いですね!
4-2. いざ承認化合物に適用!
RDKitのPAINSフィルターの使い方が大体わかりました。
それではようやく本題!ZINCデータベースから取り出したFDA承認化合物に適用してみましょう!
フィルターとしてはPAINSを使い、それぞれの化合物に「フィルターにマッチする部分構造がいくつ含まれているか?」検証してみます。
化合物数が多いのでPandasToolsでDataFrameとして扱います。
from rdkit.Chem import PandasTools import pandas as pd # SDFをデータフレームに読み込む df = PandasTools.LoadSDF("fda.sdf") print(df.shape) # (1615, 4) df.head(2)
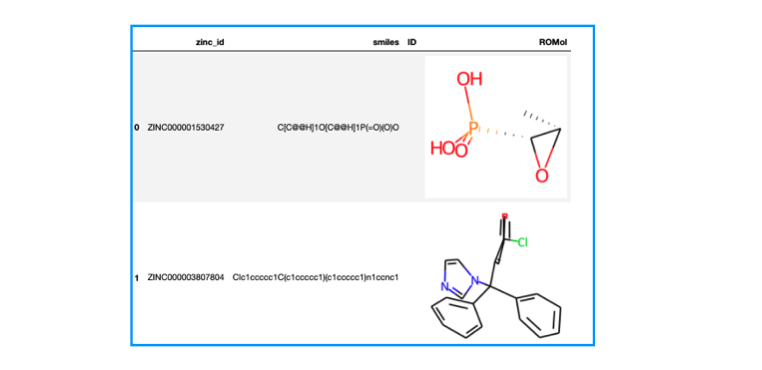
1615個の化合物がありました。zinc_id、smilesといったプロパティが含まれており、化合物は3次元に起こされた座標が入っている様です。
PAINSフィルターとマッチする構造を数える関数を作ってmap()でDataFrameに適用します。
# PAINSフィルター作り直し param = FilterCatalog.FilterCatalogParams() param.AddCatalog(FilterCatalog.FilterCatalogParams.FilterCatalogs.PAINS) filt = FilterCatalog.FilterCatalog(param) # フィルターにかかった数を数える関数 def match_num(mol): if filt.HasMatch(mol): matches = filt.GetMatches(mol) num = len(matches) else: num = 0 return num # PAINSフィルターにかかる化合物か否か?(True or False) df['PAINS'] = df.ROMol.map(filt.HasMatch) # フィルターにかかった回数は? df['filter_match_num'] = df.ROMol.map(match_num)
そもそもFDA承認化合物にPAINSフィルターにかかったものはあったのでしょうか?
# PAINSフィルターでTrueになった化合物の数 PAINS_drugs_num = df['PAINS'].sum() print("PAINS detected {} compounds.".format(PAINS_drugs_num)) # PAINS detected 79 compounds.
# 割合 PAINS_ratio = round((PAINS_drugs_num / len(df))*100) print("ratio : {} %".format(PAINS_ratio)) #ratio : 5 %
79化合物がPAINSフィルターにひっかかることがわかりました!
承認済み1615化合物の約5% にあたります。結構多いですね。
フィルターにかかった回数はどうでしょうか?
print(df['filter_match_num'].value_counts()) """ 0 1536 1 72 2 5 3 2 Name: filter_match_num, dtype: int64 """
72化合物で1回、5化合物で2回、2化合物で3回ひっかかっているようです。
3回かかった2化合物を取り出してみましょう。
df[df['filter_match_num'] == 3]
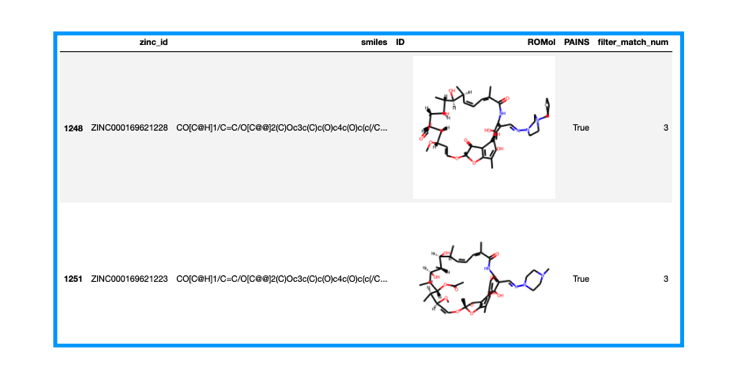
3次元だとよくわからないので2次元に描画しなおしてみます。マクロサイクルの化合物みたいなのでSetPreferCoordGenで描画の調整を行います。
# 該当化合物のSMILESを取り出してMolオブジェクトを作りなおす df3 = df[df['filter_match_num'] == 3] mols = [Chem.MolFromSmiles(smi) for smi in df3["smiles"]] # ZINC idを取り出す zinc_ids = [zinc_id for zinc_id in df3["zinc_id"] ] # マクロサイクルの描画を綺麗にする設定 Draw.rdDepictor.SetPreferCoordGen(True) # 描画 Draw.MolsToGridImage(mols, legends = zinc_ids)
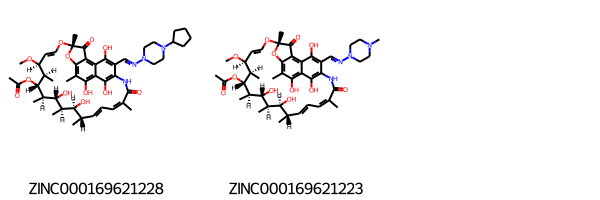
PubChemで調べたところ、それぞれCyclopentylrifampicinとRifampicineのようです。
大体同じ構造なのでRifampicineでPAINSフィルターに合致した構造をみてみます。コードは繰り返しなので省略します。
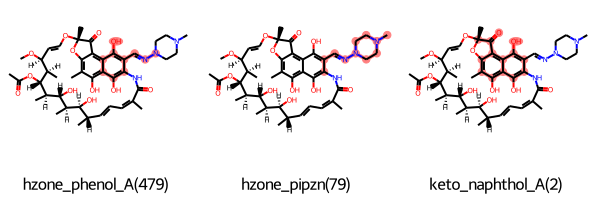
最初の二つはヒドラゾン構造とその周辺が認識されている様です。もう一つはケトンと共役したナフトール構造でした。
二重結合と共役したアミド構造やビニルエーテルなど他にも気になる構造はありますがこれらは認識されていない様ですね。
5. おわりに
以上、今回は見た目のインパクトが強いRRx-001を題材にRDKitのPAINSフィルターの使い方をお勉強し、ついでにZINC15から取得したFDA承認済み化合物にも適用してみました。
途中で引用したACS Chemical Biology「PAINS振り返り論文」中に、「PAINSは網羅的なものではない」という注意点が指摘されていました。実際に、RRx-001はPAINSには認識されないものの、他のフィルター(BRENK、NIH)ではアラートとして認識されていました。
また、FDA承認済み化合物とPAINSの関係についても上記文献に以下の記載がありました。
A small proportion (ca. 5%) of FDA-approved drugs contain PAINS-recognized substructures, these comprising both natural products and synthetic drugs.
ZINC15の例でも確かに5% でフィルターに引っかかりました。中でも3回フィルターにひっかかった化合物はRifampicineとCyclopentylrifampicin(リファペンチン)でした。
この2つは抗結核薬で、放線菌から取り出されたリファマイシンをリード化合物としたリファマイシン系化合物に分類されるもののようです。確かにいかにも天然物由来な構造です。
天然物由来の化合物には部分構造だけを取り出してみたら「そんな構造大丈夫なの?」と不安になるものが他にも多そうです。人間がデザインしたライブラリには絶対に入らなさそうな構造に有用性が見出されるのが天然物の面白さですよね!
先入観にとらわれなければライブラリにも自然界にもまだまだお宝が眠っているかもしれません。フィルターは適切に使いたいですね!
ところで、昨年に引き続き今年も対面で人に会うのが難しい一年でした。
来年は会えん 痛みがなくなるといいですね!
おしまい!