タンパク質構造のまとめサイト⁉︎ 3D-Beacons Networkで遊んだ話
AlphaFold2、AlphaFold DBの公開から4ヶ月ほどですが、まさに日進月歩という感じで次々と新しいニュースを目にしますね。
複合体予測のAlphaFold-Multimer*1や、Baker研からRosettaFoldと組み合わせた大規模な複合体予測結果*2、さらにAlQuraishi研究室からPyTorch版 OpenFoldが公開されました*3。ますます広く多様な応用が出てきそうですね。
実験によるタンパク質構造解析も「え、そんな構造とけるんだ!複合体すぎるやろ!」みたいな研究がたくさん報告されてびっくりです。*4
発展著しいタンパク質構造解析&予測の分野ですが、専門外の素人からすると「色々ありすぎてよくわかんない。まとめて見られるようにして!」ってなります。
そんな我々に朗報!素敵なまとめサイト(?)が公開されました!
その名も3D-Beacons Network!!

実験データも予測モデルもタンパク質3D構造ならまとめて探せてしまうぜ!しかも安心のEMBL-EBI運営。。。AlphaFold DB公開元なら信頼して遊べるね!
ってなわけで遊んでみましょう!
3D-Beacons Networkって?
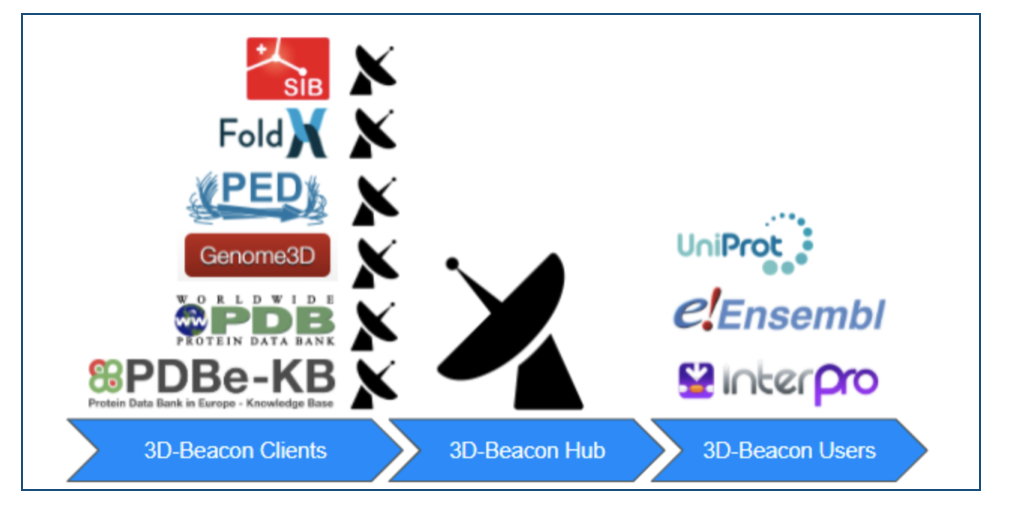
3D-Beacons Networkは複数のリソースから公開されている実験で決定されたタンパク質構造と予測モデルをまとめて一箇所で無料で利用できるようにしたものです。WebページだけでなくAPIも用意されているので、簡単なコードだけでプログラミングによる情報収集・解析も可能です。

データの提供元はこんな感じ。*6
- Protein Data Bank in Europe (PDBe)
- AlphaFoldDB
- SWISS-MODEL
- Protein Ensemble Database (PED)
- Small Angle Scattering Biological Data Bank (SASBDB)
- Genome3D
- PDBe Knowledge Base (PDBe-KB)
これらのデータが統一フォーマットで参照できます。PDBとAlphaFoldしかわからない。。。
Webページをみてみよう
ではさっそく3D-Beacons NetworkのWebページにアクセスしてみましょう。

使い方は簡単!検索したいタンパク質のUniProt IDを検索窓に入れて「Search」するだけです。試しにヒトのEGFR(Epidermal growth factor receptor)「UniProt ID: P00533 (EGFR_HUMAN)」をいれてみました*7。
こんな結果になりました。

実験データに基づく構造が235個、テンプレートに基づくモデルが3個、ディープラニングのモデルが1個の計239構造が登録されているようです。
どんな構造があるでしょうか?
検索結果のページにインタラクティブな3Dビューワーも用意してくれています。さすがEMBL-EBI。

上の図は「PDB ID : 5ug9」が表示されている様子です。「Click to download」をクリックすれば構造データファイルをすぐに取得できます。ここではcifファイルでした。
個人的に便利だと思ったのは3Dビューワーの下にある各構造エントリーとそれぞれがカバーする配列範囲のグラフです。興味のある部分がそのエントリに含まれているか一目瞭然です。

AlphaFold DBがとにかく配列全長をつっこんでいるのが潔くて良いですね!
気になった構造があればエントリー横の目のアイコンをクリックすれば画面上部の3Dビューワーに表示させられます。また、先と同様に下矢印アイコンでダウンロードすることもできます。

またマウスオーバーすると以下のように配列範囲や分解能といった情報も見られます。もちろん元のデータベースへのリンクもあるので、移動して詳細を確認することもできます。

EGFRの例で言えば抗EGFR抗体との相互作用に興味があれば細胞外ドメインを含む構造をしたり、キナーゼ活性部位・低分子阻害剤との相互作用に興味があれば細胞内ドメインを含みそうな構造をチェックしたり、といった遊び方がありそうですね。
APIをつかってみよう
Webページの使い勝手が大体わかりました。Web版はエントリをそれぞれチェックするには便利ですが、「まとめてデータを取得したい!」とか「まとまったデータを別の解析につかいたい!」というときには手間です。
「退屈なことはPythonにやらせよう」ということでAPIをさわってみたいとおもいます。*8
3D Beacons HUB APIのページはこんなでした。

URLを指定してGETすればJSON形式でデータを得られるみたいです。
Pythonならrequestsとかいうのを使えば良いそうです。標準ライブラリではないようですがcondaとかpipで簡単にインストールできます。*9
とりあえずjupyter notebookで試してみます。
# ライブラリのインポート import requests # サーバーURLを指定 Server_url = "https://www.ebi.ac.uk/pdbe/pdbe-kb/3dbeacons/api" # 検索したいUniprot ID(EGFR_HUMAN : P00533) Uniprot_number = "P00533" # APIに従ってURL全体を指定 url = Server_url + "/uniprot/summary/" + Uniprot_number + ".json" # requestsでURLにアクセスしてデータを取得 response = requests.get(url) # HTTPステータスコードの確認 print(response.status_code) # > 200と出力された
HTTPステータスコード 200となりました。無事URLヘのアクセスが成功してデータを取得できたようです。
responseオブジェクトに返ってきているJSONデータはrequestsのjson()メソッドで簡単に確認できるそうです。
json_data = response.json()

情報が多すぎてそのまま表示してもよくわかりませんね。
print(type(json_data)) # <class 'dict'> print(json_data.keys()) # dict_keys(['uniprot_entry', 'structures'])
json()メソッドで変換したデータは辞書型で、keyとしてuniprot_entry、structuresの2つがあるようです。ここで興味があるのは3D-Beacons Networkに含まれている構造情報なので、structuresのvalueを確認すれば良さそうです。
PandasのDataFrameにすれば見やすそうです。
# 辞書型からstructuresのkeyに対応するvalueを取り出す structures_data = json_data['structures'] # PandasのDataFarameに変換 import pandas as pd df = pd.DataFrame(structures_data) df.head()

上図のようなDataFrameができました。「model_identifier」がPDB ID等に相当しそうなので、各行がそれぞれの構造エントリになっているようです。
データ全体を確認してみます。
print(df.shape) # (239, 16)
全239行のDataFrameでした!3D-Beacons NetworkのWebページでEGFRを検索した結果の構造も239エントリーだったのでうまくデータ取得できているようです。
このデータ全体をcsvで出力しておくとプログラミング苦手な私でもExcelなどで気楽に眺められそうです。
df.to_csv("P00533_3D_Beacons.csv", sep=",")
エントリーごとに含まれてる情報はこんな感じ。DataFrameのcolumn名です。
print(df.columns) # Index(['model_identifier', 'model_category', 'provider', 'created', 'sequence_identity', 'uniprot_start', 'uniprot_end', 'resolution', 'coverage', 'model_url', 'model_format', 'experimental_method', 'model_page_url', 'confidence_version', 'confidence_avg_local_score', 'confidence_type'], dtype='object')
model_categoryに「実験によるデータか?予測モデルか?」、model_URLに「データ元のURL」、model_formatに「ファイルフォーマット」といった情報が記載されているようです。
構造データの元ファイルをまとめて取得できるかやってみましょう。
URLから情報を取得するにはライブラリurllibのrequestに含まれるurlopenを使えば良いようです。*10
DataFrameの上から5つを対象にしてみます。
import urllib, time for i in range(5): # IDとフォーマットを使ってダウンロード後のファイル名にする model_ID = df.at[i, "model_identifier"] model_format = df.at[i, "model_format"] if model_format == "MMCIF": extension = ".cif" elif model_format == "PDB": extension = ".pdb" file_name = model_ID + extension # ダウンロードするファイルのURL model_url = df.at[i, "model_url"] # urlretrieveでURLからデータをダウンロードしファイル名をつける model_data = urllib.request.urlopen(model_url).read() with open(file_name, mode="wb") as f: f.write(model_data) # サーバーに負担をかけすぎないようにちょっと待つ time.sleep(20)
無事ファイルが5つダウンロードできました!
上ではDataFrameの各エントリーのmodel_urlにアクセスしてデータを取得しているだけですが、ファイル名をわかりやすくするためにmodel_identifierと、拡張子(extension)の設定にmodel_formatの情報をつかっています。
ついでに連続でアクセスして公共データソースに迷惑をかけないようにちょっと待ち時間(20秒)をはさんでいます。これであっているかはわかりません。すみません。
PyMolでファイル5つを表示してみました。alignするとこんな感じ。

うまく重なっているので目的のタンパク質の構造が取得できていそうですね。
おわりに
以上、実験データと計算による予測モデルの両方を横断したタンパク質立体構造のデータベース3D-Becons Networkで遊んでみたお話でした。
直感的に利用できるWebページが良いですね。また、APIが用意されているのでプログラミングが得意な方であれば、統一フォーマットの情報を色々な用途に利用できるのではないかな?と思います。
私は見様見真似でrequestsを使ってみましたが全く自信がないです。サーバーURLの指定とかアクセスに時間を置くやり方とか間違っていたらごめんなさい。
相変わらず他にもいろいろと間違いが多そうなので、正しい使い方を教えていただけると嬉しいです。
余談①
ところでUniProt IDを使ってPDBとAlphaFold DBのデータをまとめて見るだけなら、UniProtのStructure項でも確認可能です。
ですが、AlphaFold2公開以来のタンパク質構造予測分野の発展のスピードをみていると、これからもどんどんと新しいモデル、データベースが増えていくこと予測されます。
3D-Beacons Networkの利点は統一のフォーマットが用意されており、ソースコードも公開されているため、誰でも3D-Becons Registoryに沿った形にデータを整えて共有できる拡張可能性にあることではないかな?と思います。
また、運営元がEMBL-EBIなので、作っておしまいではなく継続的な運営・発展が期待できるのも魅力かな?とおもいます。
このあたり実際にタンパク質科学に携わられている専門の先生方のご意見を伺いたいところです。
余談②
余談ついでに、先日のサイエンスアゴラ2021でERATO胡桃坂クロマチンアトラスプロジェクトセッションを拝見しました。
クライオ電子顕微鏡を用いたRNAポリメラーゼによる転写メカニズムの解析が非常に印象的でした。
Science文献へのリンク www.science.org
ライフサイエンス統合レビュー 著者による日本語解説
「クロマチンにおいてヒストンに硬く巻きついたDNAをどのようにほどきながら読み取っているか?」という基本的な生命現象がそもそも最近になるまで分かっていなかったということ、また、そのメカニズムがcryoEMでスナップショットをとるように明らかにされていく様子にタンパク質構造解析の威力を感じました。
CryoEMの威力もあってか、どんどん複雑な複合体構造のデータが出てきて、またAlphaFoldやRosettaFoldによる複合体予測も公開されてきています。複合体に割り当てるIDはどうするんだろう?など素人的には気になるところです。
ますます発展する構造解析・予測の分野の共通のインフラとして3D-Beacons Networkがこの先どのように使われていくかも楽しみですね。
*1:https://www.biorxiv.org/content/10.1101/2021.10.04.463034v1
*2:https://www.science.org/doi/10.1126/science.abm4805
*3: OpenFoldには公開版AlphaFold2にはなかったトレーニング部分も含まれているそうです GitHub、colab
*4:10年前はGPCRが一つ公開されると異分野の私でもニュースを見るくらい大変な大騒ぎだった気がするのに。。。科学の発展はすごいです。
*5:図中のいらすとは「いらすとや」さんから、ロゴは各サイトのロゴを利用させていただいています
*6:EMBL Newsより 3D-Beacons Network: protein structure data, all in one place
*7:EGFR_HUMANのUniProtページはこちら UniProtKB - P00533
*8:そんな名前の書籍があった気がしますが未読です。すみません。
*9:note.nkmk.me 「Python, Requestsの使い方」