UniProt の使い方から構造情報の一覧へ
前回よくわからないままUniProtを参照してしまいましたので少し調べて見たいと思います。
UniProt (別称: The Universal Protein Resource )
アミノ酸配列とその機能情報を掲載している代表的なデータベースです。「UniProtKB」、「UniRef」、「UniParc」の3種類のデータベースから構成されています。
UniProtKB: 文献情報などを元に手作業で高品質のアノテーションを付けたSwissProtと、機械的にアノテーションを加えたTrEMBLを公開しています。
UniRef: あらかじめ行われた配列相同性検索の結果を提供しています。
UniParc: 配列IDごとに他のデータベースのID等の情報をまとめています。
タンパク質のアミノ酸配列の情報を得る上で最も信頼性の高いデータソースの一つ、とのことです。
PD-L1 の場合、UniProtKB におけるID は 「Q9NZQ7」。アミノ酸配列は以下の通りです。
CD274 - Programmed cell death 1 ligand 1 precursor - Homo sapiens (Human) - CD274 gene & protein
| >sp|Q9NZQ7|PD1L1_HUMAN Programmed cell death 1 ligand 1 OS=Homo sapiens OX=9606GN=CD274 PE=1 SV=1 MRIFAVFIFMTYWHLLNAFTVTVPKDLYVVEYGSNMTIECKFPVEKQLDLAALIVYWEME DKNIIQFVHGEEDLKVQHSSYRQRARLLKDQLSLGNAALQITDVKLQDAGVYRCMISYGG ADYKRITVKVNAPYNKINQRILVVDPVTSEHELTCQAEGYPKAEVIWTSSDHQVLSGKTT TTNSKREEKLFNVTSTLRINTTTNEIFYCTFRRLDPEENHTAELVIPELPLAHPPNERTH LVILGAILLCLGVALTFIFRLRKGRMMDVKKCGIQDTNSKKQSDTHLEET |
|---|
Fig. 1 PD-L1 isoform 1 アミノ酸配列 ("Canonical" sequence, [ID: Q9ZQ7-1] )
上記はFASTA形式とよばれるフォーマットで、1行目( ">"で始まる行)にその配列に関する情報、2行目以降にアミノ酸配列が1文字表記で記載されています。
まずはヘルプに従って、Fig. 1のヘッダ行の情報をみてみます。( FASTA headers )
">" 以降左から順番に下記テーブルに記載しました。
| 項目 | 説明 | Fig. 1 該当部位 | |
|---|---|---|---|
| ① | データベース(db) | 配列が Swiss-Prot ("sp") と TrEMBL ("tr") のいずれのデータベース由来かを示す | sp |
| ② | ID (Unique Identifier) | UniProtKBの各エントリーに割り当てられた ID | Q9NZQ7 |
| ③ | エントリー名 (EntryName) | PD1L1_HUMAN | |
| ④ | タンパク質名 (ProteinName) | Programmed cell death 1 ligand 1 | |
| ⑤ | 生物種名 (OrganismName) |
どの生物種に由来する配列か(学名) | OS=Homo sapiens |
| ⑥ | 生物種ID (OrganismIdentifier) |
NCBIにより各生物種に割り当てられたID ヒトは9606 |
OX=9606 |
| ⑦ | 遺伝子名(GeneName) | GN=CD274 | |
| ⑧ | タンパク質の確かさ(ProteinExistence) | タンパク質の存在の証拠を数値で表現 | PE=1 |
| ⑨ | 配列のバージョン(SequenceVersion) | SV=1 |
人類、9606と呼ばれていたのか・・・。
また、⑧タンパク質の確かさ(PE)の数値は下記を表します。
| 数値 | レベル | 説明 |
|---|---|---|
| 1 | protein | タンパク質レベルで実験的証拠がある ex. MS、NMR、X-ray |
| 2 | transcript | 転写産物レベルでの実験的証拠 ex. cDNA、RT-PCR、Northern blots |
| 3 | homology | 近接種オルソログの存在から推定される |
| 4 | protein predicted | どのレベルでも実験的証拠がない |
| 5 | protein unsure | 存在が不確か |
PD-L1はタンパク質レベルで存在が証明されているので 「PE = 1」です。
ところで、前回部分構造と判明したPD-L1の結晶構造ですが、解かれた部分は配列全体のどの位置に相当するのでしょうか?
アミノ酸の文字列をながめてもさっぱりわからないので、ClustalWに探してもらいました。( ClustalW | DDBJ )
各種設定はデフォルトのまま、sequence として
・UniProtKB から取得した配列[Q9NZQ7]
・結晶構造の配列([PDB id: 4ZQK] Chain A)
を投入したところ、Fig. 2 のような結果が出ました。

Fig. 2 PD-L1 アミノ酸配列全体にしめる結晶構造の配列 (ClustalW 2.1を使用)
Fig 2 右半分がアライメントで、各段の上がUniProtKB、下がPDBの配列です。
「*」が一致している配列なので、結晶構造はN末端側(~A132)に相当するみたいです。
上記に含まれないC末端側 (P133~)の構造はどうなっているのでしょうか?
UniProtKB にちょうど良い項目が(Fig. 3)。
こちらによると結晶構造に含まれないP133以降の大部分は「Ig-like C2-type」とよばれるドメインとなっているようです。

Fig. 3 UniProtKB ID Q9NZQ7:Family & Domains項目
ドメイン名がわかってもどんな構造なのか想像がつかない・・・
さらにUniProtページを下ると素敵なテーブルがありました。
Cross-References 項目の 3D Structure databases です。こちらにPD-L1の3D構造の一覧が含まれるアミノ酸配列([Positions])とともにまとまっていました。
折角なので情報を付け足して下記にまとめました。
| PDB entry | Method | Resolution (Å) | Chain | Positions | 複合体 | 文献 | |
|---|---|---|---|---|---|---|---|
| 1 | 3BIK | X-ray | 2.65 | A | 18-239 | PD-1 (Mus musuculus) | PNAS 2018(105)3011 |
| 2 | 3BIS | X-ray | 2.64 | A/B | 18-239 | なし | PNAS 2018(105)3011 |
| 3 | 3FN3 | X-ray | 2.70 | A/B | 19-238 | PD-L1 二量体 | Protein Cell 2010(1)153 |
| 4 | 3SBW | X-ray | 2.28 | C | 19-239 | PD-1 (Mus musuculus) | To be published |
| 5 | 4Z18 | X-ray | 1.95 | A/B | 19-239 | なし | To be published |
| 6 | 4ZQK | X-ray | 2.45 | A | 18-132 | PD-1 (Homo sapiens) | Structure 2015(23)2341 |
| 7 | 5C3T | X-ray | 1.80 | A | 18-134 | なし | Structure 2015(23)2341 |
| 8 | 5GGT | X-ray | 2.80 | A | 18-134 | BMS-936559 Fab (抗体) | Nat. Commun. 2016(7)13354 |
| 9 | 5GRJ | X-ray | 3.21 | A | 18-238 | avelumab (抗体) | Cell Res. 2017(27)151 |
| 10 | 5IUS | X-ray | 2.89 | C/D | 18-239 | PD-1 mutant (Homo sapiens) | Structure 2016(24)1719 |
| 11 | 5J89 | X-ray | 2.20 | A/B/C/D | 2-134 | BMS-202 (低分子) | Oncotarget 2016(7)30323 |
| 12 | 5J8O | X-ray | 2.30 | A/B | 18-134 | BMS-8 (低分子) | Oncotarget 2016(7)30323 |
| 13 | 5JDR | X-ray | 2.70 | A/B | 18-239 | なし | Cell Discov. 2017(3)17004 |
| 14 | 5JDS | X-ray | 1.70 | A | 18-132 | KN035 (抗体) | Cell Discov. 2017(3)17004 |
| 15 | 5N2D | X-ray | 2.35 | A/B/C/D | 2-134 | BMS-37 (低分子) | J. Med. Chem. 2017(60)5857 |
| 16 | 5N2F | X-ray | 1.70 | A/B | 18-134 | BMS-200 (低分子) | J. Med. Chem. 2017(60)5857 |
| 17 | 5NIU | X-ray | 2.01 | A/B/C/D | 18-134 | BMS-1001 (低分子) | Oncotarget 2017(8)72167 |
| 18 | 5NIX | X-ray | 2.20 | A/B/C/D | 18-134 | BMS-1166 (低分子) | Oncotarget 2017(8)72167 |
| 19 | 5O45 | X-ray | 0.99 | A | 17-134 | Macrocyclic inhibitor | Angew. Chen. Int. Ed. Engl. 2017(56)13732 |
| 20 | 5O4Y | X-ray | 2.30 | B/C/E | 18-132 | Macrocyclic inhibitor | Angew. Chen. Int. Ed. Engl. 2017(56)13732 |
| 21 | 5X8L | X-ray | 3.10 | A/B/C/D/E | 18-134 | atezolizumab (抗体) | Sci. Rep. 2017(7)5532 |
| 22 | 5X8M | X-ray | 2.66 | A | 18-134 | durvalumab (抗体) | Sci. Rep. 2017(7)5532 |
| 23 | 5XJ4 | X-ray | 2.30 | A | 19-238 | durvalumab (抗体) | Protein Cell 2018(9)135 |
| 24 | 5XXY | X-ray | 2.90 | A | 18-133 | atezolizumab (抗体) | Oncotarget 2017(8)90215 |
・・・これがあるならClustalWとか持ち出さなくて良かったやん。っていうかC末端側他のエントリーでは解かれているやん。
今まで一つのグループから出ている構造のみ見ていたため、見落としていましたがPD-L1については全体構造が明らかになっていそうです。
では PD-L1 / PD-1(ハツカネズミ )複合体 [PDB id:3BIK] とPD-L1 / PD-1(ヒト)複合体 [PDB id:4ZQK] をならべてみます。

Fig. 4 PD-L1 Ig-like C2-typeドメインの有無による全体像の違い
「Ig-like C2-type」 ドメインは 「Ig-like V-type」同様、主にβシートからなる構造のようです。Fig. 4 ではこのC2-typeドメインの有無にかかわらず、V-typeドメインにPD-1が結合している様子がわかります。
ちなみに、Protein Feature View から判断する限り、より大きなこちらの構造においても細胞内ドメインまでは含まれていないようです。

Fig. 5 [PDB id: 3BIK] Protein Feature View
これまでの記事では、低分子リガンドとの共結晶構造ばかり眺めてきましたが、抗PD-L1抗体との共結晶構造も複数とかれているようなので見てみます。
こんな感じ・・・

Fig. 6 PD-L1/PD-1 複合体(左)とPD-L1/抗PD-L1抗体複合体 (右)
Fig. 6 右は、アストラゼネカ durvalumab と PD-L1との共結晶ですが、PD-1の結合側面を抗体が大きくブロックしている様子がわかります。
こちらの構造が報告された論文(Open Access)では複数の抗体の結合様式の比較がされています。
文献中に利用可と記載されていたので、お言葉に甘えて図を拝借。。。

Fig. 7 Protein Cell 2018(9)135 Figure 2より一部抜粋
Fig. 7A は PD-L1 (surface diagram) と PD-1(marine)、抗体3種類 avelumab (magenta)、BMS-936559 (cyan)、durvalumab (limon) の複合体を重ね合わせた図です。
また、Fig. 7BはPD-L1の表面にそれぞれの複合体の接触面が投影されています。
抗体の結合様式全体としては3種の抗体でかなり異なっていますが、結合面においては重なっている部分が大きいことがわかります。
詳細に関しては下記をご参照ください。
* Tan, S., Liu, K., Chai, Y., ZHang, C.W., Gao. S., Gaso, G.F., Qi, J.
(2018) Protein Cell 9: 135-139
Distinct PD-L1 binding characteristics of therapeutic monoclonal antibody durvalumab
Above article is distributed under the terms of the Creative Commons Attribution 4.0 International License ( https://creativecommons.org/licenses/by/4.0/ )
ところで、Fig. 4 ではハツカネズミ由来のPD-1とヒト由来のPD-1を並べましたが、この二つのアミノ酸配列は似ているのでしょうか?
種差が大きい場合、最悪「ヒトの薬をつくるはずが、ネズミの薬をつくっていた!」とか、「ヒトでは効くはずだけどネズミの試験で薬効が見えないせいで高次評価に進めない!」といった悲劇になりかねません。
今度こそClustalWの本領を発揮させるべく比較して見ます。
使用したsequence
① Human PD-1 [UniProtKB id : Q15116]
② Mouse PD-1 [UniProtKB id : Q02242]

Fig. 8 PD-1 アミノ酸配列の種差 (ClustalW 2.1を使用)
格段の上がHuman 、下が Mouseです。
記号は
・「*」完全に一致
・「:」強い類似性のあるグループに属している
・「.」弱い類似性のあるグループに属している
・「(空欄)」上記以外
ということのようです。( ClustalW で3種類の記号 "*", ".", ":" の意味は何ですか )
ぱっと見、かなり類似性が高いように見えますが、Fig 8緑色で示したアミノ酸残基に注目です(Human Y68、Mouse N68)。
この残基はPD-L1との結合部位に面しており、Mouse PD-1とHuman PD-1におけるPD-L1との相互作用の違いに寄与していると下記論文中で指摘されています。
結晶構造を比較する際にこの辺りの残基が重要になってきそうです。
( Structre 2015 (23) 2341 特にSupplemental Information Figure S3 )
https://www.cell.com/structure/fulltext/S0969-2126(15)00402-5 [PDB id: 4ZQK 元文献]
それでは最後に、PD-1についてももっと大きな3D構造が報告されていないか確認したいと思います。
[UniProt KB id: Q15116] Cross-References / 3D Structure databases を元に作成
| PDB entry | Method | Resolution (Å) | Chain | Positions | 複合体 | 文献 | |
|---|---|---|---|---|---|---|---|
| 1 | 2M2D | NMR | - | A | 34-150 | なし | J. Biol. Chem. 2013(288)11771 |
| 2 | 3RRQ | X-ray | 2.10 | A | 32-160 | なし | To be published |
| 3 | 4ZQK | X-ray | 2.45 | B | 33-150 | PD-L1 (Homo Sapiens) | Structure 2015(23)2341 |
| 4 | 5B8C | X-ray | 2.15 | C/F/I/L | 32-160 | pembrolizumab Fv(抗体) | Sci. Rep. 2016(6)35297 |
| 5 | 5GGR | X-ray | 3.30 | Y/Z | 26-150 | nivolumab Fab (抗体) | Nat. Commun. 2016(7)13354 |
| 6 | 5GGS | X-ray | 2.00 | Y/Z | 26-148 | pembrolizumab Fab (抗体) | Nat. Commun. 2016(7)13354 |
| 7 | 5IUS (mutant) | X-ray | 2.89 | A/B | 26-146 | PD-L1 (Homo Sapiens) | Structure 2016(24)1719 |
| 8 | 5JXE | X-ray | 2.90 | A/B | 33-146 | pembrolizumab Fab (抗体) | Cell Res. 2017(27)147 |
| 9 | 5WT9 | X-ray | 2.40 | G | 1-167 | nivolumab Fab (抗体) | Nat. Commun. 2017(8)14369 |
Table 4. はHumanのPD-1なので[PDB id: 3BIK] は含まれていません。
一番アミノ酸配列の長い[PDB id: 5WT9] でドメインを確認して見たいと思います。

Fig. 9 [PDB id: 5WT9] Protein Feature View
PD-1の細胞外ドメインはほぼ「Ig-like V-type」となっており、こちらは結晶構造中に含まれていますが、PD-L1と同じく細胞内ドメインについては含まれておりません。
細胞膜を挟む領域にまたがって結晶構造を取得するのは依然として難しいのでしょうか?
以上、UniProtに情報がよくまとまっていたので、抗体との複合体まで足をのばして眺めて見ました。既知構造の一覧は初めに作っておけばいろいろ落とし穴にはまらずに済んだかもしれない・・・
ClustalWの設定や結果(スコア)の見方などわからないところをすっ飛ばしているので、詳しい方教えていただければ幸いです。
************************************************************************************************
おまけ
[Surface]表示がどう見てもバッハ


[PDB id : 5J8O] / Structure view [ Style : Surface] / [ Color : By Molecule Type]
結晶構造で全部ではなかった!
これまでPD-L1 / PD-1 の複合体結晶 [ PDB id : 4ZQK ]などで遊んできました。
ところが、気になるつぶやきが、、、
どうやら私が見ている構造は部分構造にすぎないとのこと。
(すみません元ツイートを貼ろうと思いましたがTwitterの貼りつけ方わかりません)。
そこでまずは、アミノ酸残基を眺めてみることにしました(Table 1)。
Table 1. アミノ酸残基数の比較 (PDB vs. UniProt)

PDB のアミノ酸残基数はUniProtの半分近くしかありません。
私が見ていた構造はなんだったんでしょうか?
・・・PDBのページをスクロールしたらどうやらヒントらしきものが。
[PDB id : 4ZQK]
項目: Macromolecules 内に Protein Feature View という機能がありました。

図1. Protein Feature View 全体 (RCSB PDBページより https://www.rcsb.org/structure/4ZQK )

図2. 4ZQK Chain A (PD-L1) 一部拡大
こちらに答えがありそうなので、RCSB PDB のヘルプ( RCSB PDB - Help ) に従い図2をみます。
図2は左端のバー(緑色 / 青色)で上下2つにざっくり分かれ、それぞれ
・上段(緑色)・・・ UniProtKBのデータに基づき作成
・下段(青色)・・・ PDBのデータに基づき作成
されており、下段(PDB)は上段(UniProt)に対応するよう配置されているようです。
この対応をみると、
・[PDB id: 4ZQK] 中のPD-L1 ( Chain A / 図2下段 4ZQK_A 行の横バー) は、
・UniProtKB アミノ酸配列中の細胞外( Extracellular ) [Ig-like V-type] と呼ばれるドメイン(図2上段 Motif行の横バー)
のみ対応しており、タンパク質の一部のみしか結晶構造に含まれていないようです。
図2は拡大しているため途切れていますが、UniProt Motif行を右にたどっていくと、細胞質内(Cytoplasmic)ドメイン の文字が見えます。
PD-1 (4ZQK Chain B)については省略しますが、Chain Aと同様Extracellular ドメインのみに対応していました。
以上が、「細胞外ドメインのみで細胞内についての構造は明らかになっていない」という議論の前提にあたりますでしょうか?
間違い等ご指摘いただければ幸いです。
PDBにリンクがはってあったからUniProtを参照しましたがよかったのだろうか??
結合サイトをさがしてみる(実践編)
それでは前記事でご紹介したCASTpをPD-1/PD-L1に使ってみたいと思います。
まずはBMSリガンドとPD-L1の共結晶構造から。[PDB id: 5J8O]
下図左がPDBのviewerのキャプチャ、下図右がCASTpで認識されたポケットです。


認識されたポケットの上位3つを、赤色(Red)、青色(Blue)、緑色(Green)としました。
BMSのリガンドは3つめのポケット(PocID 3:緑色)に結合しています。
それぞれポケットの表面積、体積はこんな感じ。

次にもう少し大きなリガンドの共結晶構造を見てみます。[PDB id: 5NIU]


色の指定は [PDBid: 5J8O] と同じです。
PDBのviewerで見た時にはわかりませんでしたが、こちらの構造はリガンド 2つ とPD-L1 4つの 2 : 4 の複合体を形成しているようです。
PocID 2(青色)とPocID 3(緑色)がリガンド結合部位と重なっています。

論文中で大きなリガンドではPD-L1の構造変化によりbinding pocket が binding tunnelになったと記載されていましたが、実際に [PDBid : 5NIU] のポケットは [PDBid: 5J8O]の二つのポケット (PocID 2 と PocID 3) がつながったような貫通した形となっています。
この表面積、体積が良いのか悪いのかわからないので PPI 阻害剤 の代表例、p53-MDM2の場合を見てみたいと思います。
MDM2 と阻害剤 Nutlin-3a の共結晶構造です。 [PDBid : 4HG7]


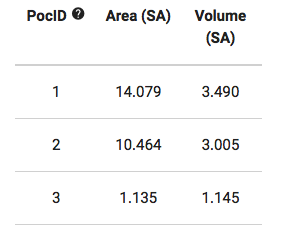
・・・ちっさい!! これではポケットというよりHot Spot です。
あらためてPPI 阻害の標的としての難しさがわかります。よくこんなターゲット相手に開発化合物を作り上げられますね。
もう少し普通(?)のターゲットを見てみたいと思います。
ALK (Anaplastic Lymphoma Kinase)とクリゾチニブ (PF-02341066) の共結晶構造です。[PDB id: 2XP2]

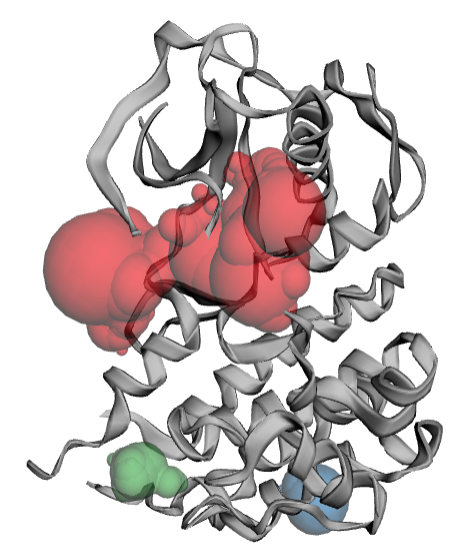

クリゾチニブの結合部位はPocID 1、表面積、体積ともにPD-L1の場合の3倍近いです。
それでは、話を元に戻しPD-1/PD-L1の複合体を見てみたいと思います。[PDB id: 4ZQK]
いままでの例はリガンドとの共結晶構造だったので、結合サイトがあるという前提でした。リガンドのない構造ではどうなるでしょうか?

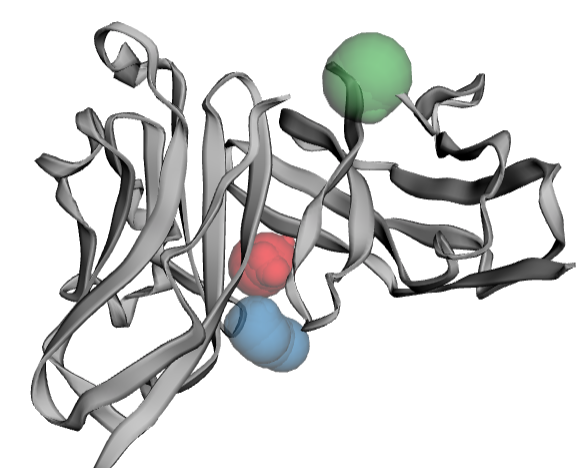
各図の左側が PD-L1 ( chain A ) 右側がPD-1 ( chain B )です。


PocID 1は小さいですが、アミノ酸残基としてTyr 56 を含む空間です。このアミノ酸残基はBMSリガンド結合部位のポケットの大きさを左右する重要な残基として論文中で注目されていたものです。
BMSリガンドの結合サイトはPD-1/PD-L1複合体においてもポケットとしての可能性があるかもしれません。
最後にPD-L1 単体の場合をみてみます。[PDB id: 5C3T]



向かって右側のβシート面がBMSリガンドの結合している部分ですので、PD-L1単量体の場合、リガンド結合サイトは上位3つのポケットに含まれませんでした。
PocID 1、PocID 2、PocID 3 いずれも小さく、リガンド結合部位としては適していないようにみえますが、PocID 1のような場所をアロステリック部位として狙えたら格好いいですね。
以上、結合サイトの探索についてでした。
CASTp のコンセプト「浅いくぼみはポケットの候補から外す」からすると、そもそもPPI阻害剤に適用するのが間違いだったかもしれません・・・。
より良い手法、誤りがあればご指摘いただければ幸いです。
*******************************************************************************************************
*引用
CASTp
Tian et al. Nucleic AcidRes. 2018. PMID: 29860391 DOI: 10.1093/nar/gky473.
PDB viewer
AS Rose et al. (2018) NGL viewer: web-based molecular graphics for large complexes. Bioinformaticsdio:10.1093/bioinformatics/bty419
*******************************************************************************************************
*余談
大きすぎる結合サイト
巨大タンパク質 イカのヘモシアニン [PDB id: 4YD9]

大きすぎてタンパク質すらとりこめます。こちらはCASTpにはデータがありませんでした。
この構造を解いた田中良和先生(東北大学生命科学研究科)は、巨大な穴と配列する特性を利用し、藤田誠先生(東京大学大学院工学系研究科)のすばらしい研究、結晶スポンジ法のタンパク質版をつくれるのではないかと講演されていました。
構造解析の世界もすごい勢いで進化していますね。
結合サイトをさがしてみる(背景編)
これまで共結晶構造をながめてきました。「結合サイトはここでOK!」という感じですが、一歩立ち止まってもっと良い結合サイトがないか検証してみたいと思います。
以下のサイトを使用したいと思います。
CASTp 3.0: Computed Atlas of Surface Topography of proteins
(思ったより長くなってしまったので今回は背景?編です。)
PDBのIDを投げ込むと、タンパク質のポケットを探してくれるサイトです。
Backgroundにポケットを探索する手順が記載されているので頑張って翻訳してみます。
( http://sts.bioe.uic.edu/castp/background.html )
以下の図も上記ページから拝借しました。
基本的な目的はタンパク質の表面から、溶媒がアクセス可能な「空の凹面(empty concavity)」を探すことです。
この際に"より使える深いポケット"を探すため、
・溶媒がアクセスできないようなポケット (cavity or void)
ex. 図1c の中央下のような完全に周囲を青丸で囲まれた白い空間
・浅いくぼみ(shallow depression)
溶媒接触面(口、mouth)がポケットの中で一番ひらいているようなもの (ex. 図2b)
は除くようにするとのことです。

図1:ポケットをみつける手順 (a :ボロヌイ図、b: ドロネー図、c: アルファ図)

図2: 深いポケット(a) と浅いくぼみ(b)の選別 (Discrete Flow Method)
ポケット探索の手順は下記の通りです。(2次元での説明です)
1. 単純な分子モデルの作成(alpha shape:アルファシェイプ - Wikipedia)
まずタンパク質の各原子に一定の大きさの円盤をおきます (図1a : 青丸)
この円盤の集合が単純な分子のモデルとなっており、集合の領域内にタンパク質の原子を全て含む凸包(convex hull)が含まれています。( 凸包 - Wikipedia )
円盤の大きさが各原子の大きさを反映していたら、青い部分の外周がタンパク質の表面に一致するのだと思いますが、ここでは全て同じ大きさの単純化したモデルとしているようです。
2. ボロノイ図 (Voronoi diagram)の作成 ( ボロノイ図 - Wikipedia )
次に1.で用いたタンパク質の各原子を母点とするボロノイ図を描きます。(図1a: 点線)
ボロノイ図は平面を平面内のどの点に最も近いかで分割した図です。
図1a の場合、各点線に囲まれた領域は原子を一つ含み、その原子に対してもっとも近い領域となります。
3. ドロネー図(Delaunay diagram)の作成 ( ドロネー図 - Wikipedia )
2.で作成したボロノイ図をもちいることでドロネー図(ドロネー三角形分割)が作成できます。(図1b)
具体的には、隣接するボロノイ領域の境界をまたぎ、二つの原子を繋ぐ線を描いていきます。
図1bの三角形の各頂点が原子(ドロネー図の母点)に対応しており、複数の三角形からなる多角形(緑の領域)のなかに、タンパク質の凸包が含まれています。
逆にいうと、タンパク質を大きく取り囲むような多角形領域を作成し、隙間も、重なりもないように三角形をしきつめて分割した形とになります。
4. アルファコンプレックス(alpha complex)の作成
1. で作成した単純な分子モデル(図1a: alpha shape)と、3.で作成したドロネー図(図1b)を重ね合わせ、アルファコンプレックス(図1c)を作成します。
これにより円弧で囲まれた分子モデルの多角形版が作成されたことになります。
図1bと図1aの差分、ドロネー図の多角形領域のなかで青丸を含まない領域が、少なくとも空いている空間、ポケットの候補となります。
空いている空間をみつけるため、ドロネー図のなかから「空の三角形("empty" triangle)」を見つけ出します。
再度、図1aに戻り、ドロネー図を作成する時に用いたボロノイ図のうち、ボロノイ領域の辺(edge)と頂点(vertex)が、分子モデル(青丸の領域)の完全に外部にある物を見つけ出します。ドロネー図を作成する際に、ボロノイ図の辺をまたぐようにドロネー三角形の辺を描いていいきましたが、このうち分子モデル外部となるボロノイ辺に対応するドロネー三角形の辺を外部として、点線とします(図1c 点線)。この点線を、1つ以上ふくむドロネー三角形が「空の三角形("empty" triangle )」と定義されます。
ここまででようやく下準備が完了です。空の三角形の領域がポケット候補となる空いた空間となるのですが、その中から確からしいポケットの選別とその大きさを求めるのが次なる課題です。
5. cavity (void)の選別
まず、空いてはいるけど、リガンドが入り込めないような空間を除きます。
図1c の中央下あたりを見ていただくと、青い領域に囲まれた小さな白い領域があります。確かにタンパク質とはぶつかりませんが、リガンドがアクセスできないのでは結合サイトとしては使えません。このようなcavityをどう判別すれば良いかが問題となりますが、これはドロネー図の辺のうち実線と点線をくみあわせることで可能となります。
まず上記の空間は、点線を含むempty triangle 3つにより空いた空間と認識されます。しかしながら、この3つのempty triagle 全体は全て実線の辺で囲まれています。これによりアクセス不能な空間としてポケット候補から外れます。
6. 浅いくぼみ(shallow depression)の選別
次に浅いくぼみの選別ですが、こちらはempty triangleに、"Discrete Flow" method と呼ぶ方法を適用することで行なっています(図2)。
empty triangle が複数隣接している領域において、鈍角三角形(obtuse triangle)からは隣のempty triangeへに向かう流れ(flow)を、鋭角三角形(acute triangle)は隣のempty triangle からflow が流れ込んでくると考えます。
ここで再度「浅いくぼみ」の定義に戻りますが、「溶媒接触面(口、mouth)がポケットの中で一番ひらいているようなもの」でした。こちらはポケット候補の空間のうち溶媒接触面となるドロネー三角形の辺が、同一空間の他のempty triageの辺よりも長いと解釈できます。図2b のように、この時鈍角三角形1から始まったflow は流れ続けて無限へと発散していきます。
一方で、深いポケット(図2a)では、鈍角三角形からのflow は鋭角三角形2にいずれも流れ込み収束します。このempty triangle の集合として我々の探していた有望なポケットが見つけ出されたことになります。
7. ポケットの大きさ
さて、ようやく見つかったアクセス可能な深いポケットですが、次なる問題は「リガンドが入れるくらいに十分大きいか?」ということです。
つまり空間の体積ですが、こちらは「空間を成すempty triagleの合計」から、その中に含まれる「分子モデル(青い領域)」の分を引くことで求まります。
以上がCASTpのポケット探索の手順となります。
ここまでお付き合いいただいた皆さんはそろそろお気づきと思いますが、書いている人はよく理解できていません。
「ボロノイ図? あー、サッカーの試合の解説で何か見た気がする。」レベルです。
雑にサッカーに例えると、
「リガンドが入れられる結合サイトを探す」=「相手のフィールド(タンパク質)の奥にボールを投げ込むパスコースを探す」
ということでしょうか?
1. ディフェンダー(原子の点)の守備範囲(ボロノイ図、ドロネー三角形)見極めて、誰からも遠い空間(パスコースの候補)を見つける。
2. その中から守備陣に囲まれていて味方が走りこめないスペース(ドロネー三角形の実線で囲まれたcavity)は、候補として外す。
3. 次に、相手ゴールにできるだけ近づきたいのでより深くまで到達する空間を探す(ポケットの体積)。
4.できるだけ守備の壁の裏の空間に通すような(浅いくぼみでない)パスは、多数のディフェンダーを一気に抜けるので良いパスコース
みたいな?? すみません。サッカーわかりません。
英語、数学能力ももれなく低いので、CASTpの解析手順について間違いがあったらご指摘いただければ幸いです。
結論、CASTpはペップ・コードだった。
引用:Nuclei Acids Research 2018 (46) W363-W367
https://academic.oup.com/nar/article/46/W1/W363/5026264
PDBにおけるPD-1/PD-L1結晶構造をもう少しながめてみる
前記事でピックアップしたPDBファイルについてもう少しながめてみようと思います。
久しぶりにPDBにアクセスしたらいろいろな機能が追加されていました。
先ずはPDB id: [5J8O]
こんな化合物がPD-L1に結合しています。化合物のID は [6GZ] です。

(2R)-1-({3-bromo-4-[(2-methyl[1,1'-biphenyl]-3-yl)methoxy]phenyl}methyl)piperidine-2-carboxylic acid
SMILES: Cc1c(COc2ccc(CN3CCCC[C@@H]3C(O)=O)cc2Br)cccc1-c1ccccc1
全体の外観としてはこんな感じ。


真ん中の化合物(Ball & Stick)を挟み込む形でPD-L1が2量体化しています。
PDBの3D view で動かしてみると、βシート構造の面でペタっと化合物が挟まれています。
描画方法をかえてみるとこんな感じ。[PD-L1 : Licorice、Ligand : Sapcefill]


化合物の疎水性部位(灰色)がPD-L1に挟み込まれており、外側に向かって親水性部位が伸びている様子です。
より具体的なアミノ酸残基との相互作用をみてみます。
論文中には疎水性相互作用について色々と記載がありましたが、PDBの[2D Diagram & Interactions] では下図左のようにざっくりと疎水性ポケットと認識されているようです。


明示的に描かれている2つのTyr ですが、3Dでみてみると化合物との相互作用形式は異なっており、一方はπ-πスタッキング ( Tyr56A - BrPh側 )、もう一方は直交するようなT-stacking interactionとなっています(Tyr56B - ビフェニル構造末端側)。
論文中では後者のTyr56B の動きが注目されており、PD-L1のapoと、共結晶で大きくシフトしている様が描かれています。さらに、ビフェニル末端に置換基の入ったより大きなリガンドとの共結晶構造(PDB id: 5NIU 、5NIX)では、Tyr56Bが押しのけられリガンド結合部位が広がっています。論文著者らはbinding pocket が binding tunnel になったと呼んでいます。
さて、リガンド逆末端のカルボキシ基ですがタンパク質との明確な相互作用はみられず溶媒面に露出しています。特許中の化合物もアミドやヒドロキシ基等、様々な置換基をふっており、いくつか強い活性をもつものがありそうでした。
もう一つの親水性部位、アミノ基はどうも重要そうです。なぜならWO2015160641 のクレーム、マーカッシュ構造が下図のようなものだから!(雑)

これだけ見たら保護基みたいですね。酸化とか強酸性条件で外れてしまいそうです。
疎水性のビフェニル部位と比較して親水性部位は不斉点が多く凝った構造が多いという印象ですが、特許の合成スキームをみてみるとアミノ酸のようなビルディングブロックをひたらすら還元的アミノ化でバシバシくっつけています。
手堅い&クリーンな反応に最終工程をもってくるあたり、効率的かつ合成速度重視の探索合成という感じで格好良いですね。
共結晶構造に話を戻し、今度はより大きなリガンドとの共結晶構造を眺めます。
PDB id: [5NIU]
リガンド id: [8YZ]

(2R)-2-[[2-[(3-cyanophenyl)methoxy]-4-[[3-(2,3-dihydro-1,4-benzodioxin-6-yl)-2-methyl-phenyl]methoxy]-5-methyl-phenyl]methylamino]-3-oxidanyl-propanoic acid
SMILES: Cc1cc(CN[C@H](CO)C(O)=O)c(OCc2cccc(c2)C#N)cc1OCc1cccc(c1C)-c1ccc2OCCOc2c1
さきのリガンドとの大きな違いは、ビフェニル構造末端の拡大(dioxaneみたいな構造)と中央のフェニル基に新たにシアノベンジル基が導入されていることです。
共結晶構造では前者が上述のPD-L1 Tyr56 を押しのける役割を、後者があらたな相互作用の獲得に機能しています。
全体的な結合モードは先の共結晶と同じです。


2D の Ligand interactionの図がなかったので3Dを貼ります。


上左図ではリガンドのビフェニル末端側がPD-L1のTyr 56を押しのけている様子を、
上右図ではリガンド左下、シアノベンジル基があらたなπ -π相互作用 ( Tyr123 )を獲得している様子がみえます。
(透けて見えるタンパク質はpocketの opacity という値を変えると表示されました。格好良いかと思いましたが見づらくなった感もあります。)
以上、共結晶構造をみてきましたが、重要な問題が、、、それはPD-L1の2量体と低分子の相互作用形式で、PD-1 と PD-L1 のProtein-protein interaction 阻害とはまた話が違うのではということです。
そこでついでにPD-L1 と PD-1の複合体の構造を眺めたいと思います。
PDB id: [4ZQK] ( chain A : PD-L1、chain B: PD-1 )
先ずは外観です。


色の変え方が分からなかったのでわかりにくくなっていますが、それぞれ複合体の左側にPD-L1 がくるようにしています。
PD-L1二量体とリガンドの共結晶構造ではPD-L1は「両手を指先であわせて三角形を作った」ような見た目でしたが、PD-L1 : PD-1複合体は「掌を合わせて握手した」ようなみためです(上左図)。
ですが、上右図のようにPD-L1 : PD-1 複合体の境界面にはPD-L1 2量体の共結晶でみえたような隙間があります。これはリガンド結合部位として期待できるかもしれません。
これ以上の細かな設定はPDBのviewer では難しそうだったので論文中の構造を引用させていただきます。(文献:Structure 2015(23)2341 よりFigure 2)

図の緑色がPD-L1、青色がPD-1です。PD-L1の黄色でハイライトされたアミノ酸残基をみると、Tyr56やTyr 123があります。また、Fig. 2B 左端にはGln66(緑色)がありますが、こちらは[PDB id : 5J8O] においてBMSリガンドのアミノ基との相互作用が認識されていた残基です。
PD-L1 : PD-1相互作用とPD-L1 2量体 : リガンドの相互作用に機能するアミノ酸残基は共通するものが多く、同じ結合位置をしめているようです。
と、いうわけでBMSリガンドとの共結晶構造はStructure Based Drug Design を行う上で役に立ちそうです。
以上、いつの間にか機能が増えていたPDBで遊んだみたという記事です。
図は主にRCSB PDB の NGL viewer を用いて作成しました*。
私はタンパク質結晶の専門家でもなんでもないので、「これは変だ」というのがあればご指摘いただければ幸いです。
*引用
AS Rose et al. (2018) NGL viewer: web-based molecular graphics for large complexes. Bioinformatics dio:10.1093/bioinformatics/bty419
PDBにおけるPD-1/PD-L1結晶構造について
この記事はPD-1/PD-L1に対する低分子創薬に関する記事です。
ノーベル賞関連をぼんやりながめていたら以下のイベントをみつけました。
薬学部にいた遠い記憶を掘り起こし、ちょっと調べてみることに。。。
とはいえ、ボーナスで買ってしまったmac 1台以外になにもなくin silico創薬なるものになんの知識もない凡人ではどうしていいかわからず、とりあえず何か投げ込めば頭のいい人たちが教えてくれると期待して書きます。
(ちなみに薬剤師の資格もありません...orz)
とりあえずタンパク質の構造といえばPDBということで、PDBでタンパク質-化合物の共結晶構造をしらべてみました。
「創薬ちゃん」さんがtwitterであげていた構造、「PDB id :4zqk」(ありがたいことに元文献はopen accessです) の研究室から、複数の共結晶構造が報告されていました。
元文献→
https://www.ncbi.nlm.nih.gov/pubmed/?term=26602187
Prof. Tad Holak→
http://www.mcb.uj.edu.pl/prof.-tad-holak
(こんな素敵なタイトルのreivew もでてきましたが、課金が必要みたいです...
https://www.tandfonline.com/doi/full/10.1080/13543776.2018.1512706 )
さて、共結晶構造ですが、いずれもBMS社の特許文献中の化合物との共結晶構造ということです。
1. PDB id: [5J89] [5J8O] 元文献(open access) [Oncotarget. 2016; 7:30323-30335.]
2. PDB id: [5NIU] [5NIX] 元文献(open access) [Oncotarget. 2017; 8:72167-72181. ]
https://doi.org/10.18632/oncotarget.20050]
3. PDB id: [5N2D] [5N2F] 元文献(課金が必要) [J. Med. Chem. 2017, 60, 13, 5857-5867]
[ https://pubs.acs.org/doi/abs/10.1021/acs.jmedchem.7b00293 ]
(JMCは埋め込みができなかった)
1 , 2はopen access (?課金なしで見られた)のですが、軽くまとめるとBMSの特許記載中の化合物との共結晶構造をとってみたよ、ということみたいです。
1) BMSの特許: WO201503482、WO2015160631
(特許についてはrkakamilanさんのすばらしい記事をお読みください)
2) 文献の内容
BMSの化合物とPD-L1の共結晶をとったところ、下記の内容がわかった。
・BMS低分子化合物との共結晶では、「化合物 : PD-L1 = 1 : 2」、すなわちタンパク質が低分子と結合し、2量体化した構造となっていた。
・BMS低分子の構造が大きくなる(BMS-8, BMS-202 vs. BMS-1001, BMS1166)とPD-L1のアミノ酸残基(Tyr-56)の向きが大きく変化し、低分子化合物を許容するような結合サイトの変化がみられた(「文献中ではトンネル構造がでできた」と言った記載がされています)・・・文献のFigの貼り方がわからないので雑な記載ですがご容赦ください。
・共結晶構造ではPD-L1の2量体となっているが、著者らが既に報告しているPD/PD-L1複合体の構造と比較すると、だいたい重なっており、PD/PD-L1の相互作用阻害としても同様の結合モードで機能しているのでは??
ということみたいです。
3) PPI
上記文献ではPD/PD-L1の相互作用、低分子とPD-L1化合物の相互作用形式について「2~3のHot Spot」という形式を提唱していました。
3点のHot Spotを提唱するあたり、MDM2にたいする阻害剤(PPI: Protein-protein interaction inhibiter)を意識しているのかなぁという印象です。
余談ですが、ペプチドリームの枡屋先生はNovartisで絶対無理だといわれながらもMDM2を標的としたPPI創薬にとりくみ、見事臨床試験に到達した化合物をしあげたとのお話をされていました。「ちょっと格好良すぎるのでは、、、」と思う次第です、、、。
以上、わずかながら情報提供できれば、、、とおもい記載してみました。
インターネットの賢者たちが行く末を示してくれると信じ、この記事を終えます。
お付き合いありがとうございました。
ps. レイドバトルと聞いて、下が思い浮かんだ私は負け組...
シラット...hoo!!!