結合サイトをさがしてみる(実践編)
それでは前記事でご紹介したCASTpをPD-1/PD-L1に使ってみたいと思います。
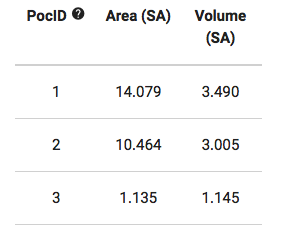
まずはBMSリガンドとPD-L1の共結晶構造から。[PDB id: 5J8O]
下図左がPDBのviewerのキャプチャ、下図右がCASTpで認識されたポケットです。


認識されたポケットの上位3つを、赤色(Red)、青色(Blue)、緑色(Green)としました。
BMSのリガンドは3つめのポケット(PocID 3:緑色)に結合しています。
それぞれポケットの表面積、体積はこんな感じ。

次にもう少し大きなリガンドの共結晶構造を見てみます。[PDB id: 5NIU]


色の指定は [PDBid: 5J8O] と同じです。
PDBのviewerで見た時にはわかりませんでしたが、こちらの構造はリガンド 2つ とPD-L1 4つの 2 : 4 の複合体を形成しているようです。
PocID 2(青色)とPocID 3(緑色)がリガンド結合部位と重なっています。

論文中で大きなリガンドではPD-L1の構造変化によりbinding pocket が binding tunnelになったと記載されていましたが、実際に [PDBid : 5NIU] のポケットは [PDBid: 5J8O]の二つのポケット (PocID 2 と PocID 3) がつながったような貫通した形となっています。
この表面積、体積が良いのか悪いのかわからないので PPI 阻害剤 の代表例、p53-MDM2の場合を見てみたいと思います。
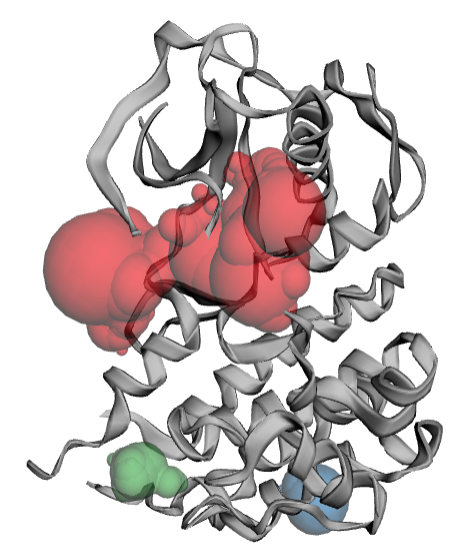
MDM2 と阻害剤 Nutlin-3a の共結晶構造です。 [PDBid : 4HG7]


・・・ちっさい!! これではポケットというよりHot Spot です。
あらためてPPI 阻害の標的としての難しさがわかります。よくこんなターゲット相手に開発化合物を作り上げられますね。
もう少し普通(?)のターゲットを見てみたいと思います。
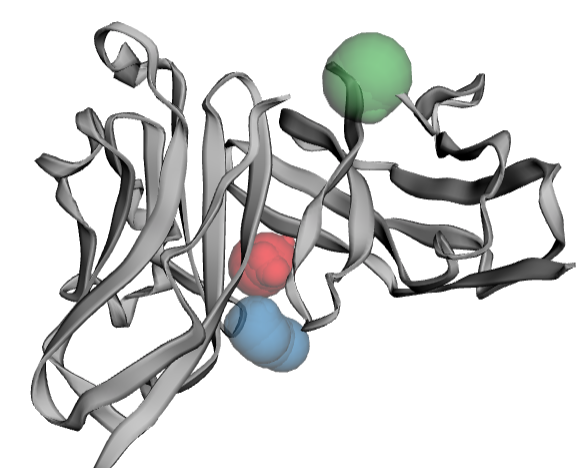
ALK (Anaplastic Lymphoma Kinase)とクリゾチニブ (PF-02341066) の共結晶構造です。[PDB id: 2XP2]


クリゾチニブの結合部位はPocID 1、表面積、体積ともにPD-L1の場合の3倍近いです。
それでは、話を元に戻しPD-1/PD-L1の複合体を見てみたいと思います。[PDB id: 4ZQK]
いままでの例はリガンドとの共結晶構造だったので、結合サイトがあるという前提でした。リガンドのない構造ではどうなるでしょうか?

各図の左側が PD-L1 ( chain A ) 右側がPD-1 ( chain B )です。


PocID 1は小さいですが、アミノ酸残基としてTyr 56 を含む空間です。このアミノ酸残基はBMSリガンド結合部位のポケットの大きさを左右する重要な残基として論文中で注目されていたものです。
BMSリガンドの結合サイトはPD-1/PD-L1複合体においてもポケットとしての可能性があるかもしれません。
最後にPD-L1 単体の場合をみてみます。[PDB id: 5C3T]



向かって右側のβシート面がBMSリガンドの結合している部分ですので、PD-L1単量体の場合、リガンド結合サイトは上位3つのポケットに含まれませんでした。
PocID 1、PocID 2、PocID 3 いずれも小さく、リガンド結合部位としては適していないようにみえますが、PocID 1のような場所をアロステリック部位として狙えたら格好いいですね。
以上、結合サイトの探索についてでした。
CASTp のコンセプト「浅いくぼみはポケットの候補から外す」からすると、そもそもPPI阻害剤に適用するのが間違いだったかもしれません・・・。
より良い手法、誤りがあればご指摘いただければ幸いです。
*******************************************************************************************************
*引用
CASTp
Tian et al. Nucleic AcidRes. 2018. PMID: 29860391 DOI: 10.1093/nar/gky473.
PDB viewer
AS Rose et al. (2018) NGL viewer: web-based molecular graphics for large complexes. Bioinformaticsdio:10.1093/bioinformatics/bty419
*******************************************************************************************************
*余談
大きすぎる結合サイト
巨大タンパク質 イカのヘモシアニン [PDB id: 4YD9]

大きすぎてタンパク質すらとりこめます。こちらはCASTpにはデータがありませんでした。
この構造を解いた田中良和先生(東北大学生命科学研究科)は、巨大な穴と配列する特性を利用し、藤田誠先生(東京大学大学院工学系研究科)のすばらしい研究、結晶スポンジ法のタンパク質版をつくれるのではないかと講演されていました。
構造解析の世界もすごい勢いで進化していますね。