2Dも3Dも!Panel-Chemistryでまとめて描画しよう!
前回の記事でJupyter notebookで構造式エディタを使う方法を調べました。
もっといい方法がありました!その名もPanel-Chemistry!!
2Dの構造式(JSME Editor)だけでなく、3Dの描画(NGL Viewer, Py3DMol)もカバーしてくれています。こりゃ便利!
1. Panel-Chemistry?
とりあえずデモが格好いいですよ!*1
Panel-Chemistryは、化学分野の探索的データ解析・ビジュアリゼーションをサポートするためのプロジェクトです。Pythonでデータ可視化するためのオープンソースライブラリであるHoloViz - Panelに化学ドメインを組み込んで構築されているのでPanel-Chemistryと名付けられているみたいです。
Panel-Chemistryがサポートしている可視化ツールは3つです。
- JSME Editor (化学構造式描画&編集、2D)
- NGL Viewer (タンパク質、DNA/RNA etc.の描画、3D)
- py3DMol (タンパク質、低分子化合物の描画、3D)
それぞれ対応可能なファイルフォーマットが異なります。2D、3Dの生物学・化学描画をカバーしてくれているのはありがたいですね!
# pipの場合 pip install panel-chemistry # condaの場合 conda install -c conda-forge panel-chemistry # もちろんmambaでもOK mamba install -c conda-forge panel-chemistry
とりあえず使用感を知りたいと言う方は、GitHubレポジトリのREADMEにBinderへのリンクが用意されているのでそちらをご利用ください。
2. 使ってみよう!
GitHubの例(examples)ほぼそのままで恐縮ですが、どんな感じに使えるかご紹介します*2。
サポートする可視化ツールそれぞれについて、①Jupyter notebook上で描画、あるいは②サーバーモードでアプリケーションとして利用する方法の2つの使い道があります。
2-1. JSMEの場合
2-1-1. Jupyter notebook上で使う
まずはJSMEをJupyter notebookで使う場合です。JSMEについては以下をご参照ください。
- Bienfait and P. Ertl, JSME: a free molecule editor in JavaScript, J. Cheminformatics 5:24 (2013)
panelとpanel_chemistryから使いたいツール(今回はJSMEEditor)をインポートしておきます。
import panel as pn from panel_chemistry.widgets import JSMEEditor
次にpanelのextension()でjsmeを読み込みます*3。以下のpn.extension()実行前に、上記の通りpabel_chemistryを読み込んでおく必要があることに注意です。
pn.extension("jsme", sizing_mode="stretch_width")
panel可視化スペースのレイアウトはcolumn()を使って設定できます。JSMEEditorを以下のようにレイアウトに追加するだけでもう使えます。
editor = JSMEEditor(height=500)
pn.Column(editor)

JSMEが起動しました!簡単!
さらに便利な機能は、JSME起動時に同時に指定した構造式が描画された状態で起動できることです。例えばSMILESで構造を指定するなら以下のようにすればOKです。
smiles="N[C@@H](CCC(=O)N[C@@H](CS)C(=O)NCC(=O)O)C(=O)O" editor = JSMEEditor(value=smiles, height=500, format="smiles") pn.Column(editor, editor.param.value)

上図の通りグルタチオンが描かれた状態でJSMEが起動しました。またpn.Column()でeditorに加えてeditor.param.valueもレイアウトに追加しているので、上図下部のようにvalueに格納されたSMILESも表示されています。
valueは他にもmolやsdf形式も使えます。例えば以下のようにRDKitで作成したMol形式の座標を読み込むこともできます。
from rdkit import Chem toluene = Chem.MolFromSmiles("Cc1ccccc1") toluene_mb = Chem.MolToMolBlock(toluene) print(toluene_mb) """ RDKit 2D 7 7 0 0 0 0 0 0 0 0999 V2000 3.0000 0.0000 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 1.5000 0.0000 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 0.7500 -1.2990 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 -0.7500 -1.2990 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 -1.5000 0.0000 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 -0.7500 1.2990 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 0.7500 1.2990 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 1 2 1 0 2 3 2 0 3 4 1 0 4 5 2 0 5 6 1 0 6 7 2 0 7 2 1 0 M END """
# mol形式の座標を読み込んで起動する editor = JSMEEditor(value=toluene_mb, height=500, format="mol") pn.Column(editor)

トルエンのように単純な構造だと有り難みは少ないですが、より複雑な分子を描画する時、SMILESから構築するとツールに依存した描画スタイルになってしまいます。複雑な分子を好みの向き・スタイルで描きやすいと言う点で、座標情報のあるMol形式が使えるのは良いですね!
2-1-2. アプリケーションとして使う
つづいてアプリケーションとして使う場合です。ビューのレイアウトといった諸々の設定をJupyter notebook上に書き込んだあと、コマンドラインでpanel serveコマンドを使うことでアプリケーションとして起動することができます。
この方法ではPanelのFastLstTemplateテンプレートを利用してレイアウトを作成します。最後に.servabl()でアノテーションをつけておくことでコマンドラインから起動できるようになります。
Panelのアプリケーションサーバーの仕組みはドキュメントのServer Deploymentをご参照ください。デフォルトのポートは5006なので、起動後http://localhost:5006にブラウザでアクセスすればアプリを利用できます。
設定例はこんな感じ。
# アプリケーションの見た目の色調設定 accent="#00A170" # メイン描画部位の設定 values = pn.Param(editor, parameters=["value", "jme", "smiles", "mol", "mol3000", "sdf"], widgets={ "value": {"type": pn.widgets.TextAreaInput, "height": 200}, "jme": {"type": pn.widgets.TextAreaInput, "height": 200}, "smile": {"type": pn.widgets.TextAreaInput, "height": 200}, "mol": {"type": pn.widgets.TextAreaInput, "height": 200}, "mol3000": {"type": pn.widgets.TextAreaInput, "height": 200}, "sdf": {"type": pn.widgets.TextAreaInput, "height": 200}, }) # サイドバーの設定 settings = pn.Param(editor, parameters=["height", "width", "sizing_mode", "subscriptions", "format", "options", "guicolor"], widgets={ "options": {"height": 300}, }) # テンプレートを利用したレイアウトの作成 + servable() pn.template.FastListTemplate( site="Panel Chemistry", title="JSME Editor", sidebar=[settings], main=[editor, values], accent_base_color=accent, header_background=accent, ).servable();
上記設定が完了したら、コンソール(私はmacなのでターミナル)でpanel serve ノートブック名を打ち込めばサーバーアプリケーションとして起動します*4。「PanelChemistry_JSME.ipynb」というノートブックならこんな感じ。
panel serve PanelChemistry_JSME.ipynb
もちろんJupyter notebook上で最初に「!」をつけてコマンドを実行してもOKです*5。
実行後「サーバーを起動したぜ!」「アプリケーションはここだぜ!( Bokeh app running at: http://localhost:5006/PanelChemistry_JSME )」みたいなことを言ってくるのでこのURLをブラウザにコピペすればアプリが使えます。
こんな感じ!

緑を基調にしてるのが素敵ですね!
JSMEを使う場合は以上です。
2-2. NGL viewerの場合
2-2-1. Jupyter notebook上で使う
つぎにNGL viewerの場合です。NGL Viewerについては以下をご参照ください。
AS Rose, AR Bradley, Y Valasatava, JM Duarte, A Prlić and PW Rose. Web-based molecular graphics for large complexes. ACM Proceedings of the 21st International Conference on Web3D Technology (Web3D '16): 185-186, 2016. doi:10.1145/2945292.2945324.
AS Rose and PW Hildebrand. NGL Viewer: a web application for molecular visualization. Nucl Acids Res (1 July 2015) 43 (W1): W576-W579 first published online April 29, 2015. doi:10.1093/nar/gkv402.
やることは大体一緒です。まずはライブラリのインポートとngl_viewerのpanelへの読み込みです。
import panel as pn from panel_chemistry.pane import NGLViewer from panel_chemistry.pane.ngl_viewer import EXTENSIONS
pn.extension("ngl_viewer", sizing_mode="stretch_width")
次いで、ビューワーの設定です。
描画する目的の構造はobjectパラメーターで設定し、①url like、②non-url like、③blobの3種類から選べます。①と②はPDBに対応するもので、①なら「'rcsb://1NKT'」、②なら「'1NKT'」のようにします。②はPDB IDだけで、自動的に「rcsb://」を保管してURLにして構造を持ってきてくれます。
③blobの時は合わせて拡張子(extension)パラメーターを設定する必要があります。pdbやcif等色々と使えるのでGitHubなどをご参照ください。
今回はGitHubのexample通りやってもRCSBからPDBファイルを持ってきてくれなかったので、別途ダウンロードした「1nkt.pdb」を使っています*6。
# ビューワーと読み込む構造の設定 viewer = NGLViewer(object="1nkt.pdb",background="#F7F7F7", min_height=700, sizing_mode="stretch_both") # パラメーター設定 settings = pn.Param( viewer, parameters=["object","extension","representation","color_scheme","custom_color_scheme","effect",], name="⚙️ Settings" ) # 入力ファイルの設定 file_input = pn.widgets.FileInput(accept=','.join('.' + s for s in EXTENSIONS[1:])) def filename_callback(target, event): target.extension = event.new.split('.')[1] def value_callback(target, event): target.object = event.new.decode('utf-8') file_input.link(viewer, callbacks={'value': value_callback, 'filename': filename_callback}) # レイアウトの設定 header = pn.widgets.StaticText(value='<b>{0}</b>'.format("💾 File Input")) file_column = pn.layout.Column(header, file_input) layout = pn.Param( viewer, parameters=["sizing_mode", "width", "height", "background"], name="📐 Layout" )
pn.Row(
viewer,
pn.WidgetBox(settings, layout, width=300, sizing_mode="fixed",),
)

できました!
2-2-2. アプリケーションとして使う
つづいてアプリケーションとして使う場合です。コマンドラインで起動する際にノートブック単位で指定するので、JSMEを起動したノートブックとは別のもの(PanelChemistry_NGL.ipynb)を作っています。
アプリケーションにおける設定は上のJupyter notebook上で使う場合と同様です。viewerやsettings、flie_column、layoutといった変数の部分なのでそのまま引き継ぎます。
こんな感じで起動します。
# アプリケーションの見た目の色調設定 accent="#D2386C" # テンプレートを利用したレイアウトの作成 + servable() pn.template.FastListTemplate( site="Panel Chemistry", title="NGLViewer", sidebar=[file_column, settings, layout], main=[viewer], accent_base_color=accent, header_background=accent, ).servable();
あとはコンソールでコマンドを打ち込むだけです。
panel serve PanelChemistry_NGL.ipynb
ポートもデフォルト「5006」で変わらずなので「http://localhost:5006/PanelChemistry_NGL 」にブラウザでアクセスすればアプリを利用できます。

できました!起動時にファイルを指定しているので構造を読み込んでいますが、左上のファイルを選択から新しく構造を読み込むこともできます。
2-3. Py3DMolの場合
2-2-1. Jupyter notebook上で使う
最後にpy3Dmolの場合です。py3Dmolについては以下をご参照ください。
- Nicholas Rego and David Koes, 3Dmol.js: molecular visualization with WebGL, Bioinformatics (2015) 31 (8): 1322-1324 doi:10.1093/bioinformatics/btu829.
また、化学の新しいカタチさんの記事「py3Dmolを使って化学構造をJupyter上で美しく表示する」で詳細に解説してくださっています。
今までと同様にまずはライブラリを読み込みます。
import py3Dmol import panel as pn from panel_chemistry.pane import Py3DMol
pn.extension()
基本的な使い方は以下です。1行目py3DMol.view()のqueryを設定するとデータベースから構造をダウンロードして描画してくれます。下記ではmmtf(macromolecular transmission format)形式でPDB ID 1nktの構造をとってきています。
p = py3Dmol.view(query="mmtf:1nkt") p.setStyle({"cartoon": {"color": "spectrum"}}) pviewer=Py3DMol(p, height=400, sizing_mode="stretch_width", name="Basic") pviewer

できました!これ以外にもグリッド表示やインタラクティブなプロット(低分子の動くやつ)の例がGitHub上にありますが割愛します。
2-3-2. アプリケーションとして使う
つづいてアプリケーションとして使う場合です。ここでも新しいノートブックを(PanelChemistry_py3Dmol.ipynb)を作っています。
今までと同様、設定を書き込んだあと起動するだけです。
# 設定 background = pn.widgets.ColorPicker(value="#e6f6ff", name="Background") style=pn.widgets.RadioButtonGroup(value="sphere", options=["line", "cross", "stick", "sphere"], name="Style", button_type="success") # アプリケーションの見た目の色調設定 accent = "#0072B5" # テンプレートを利用したレイアウトの作成 + servable() pn.template.FastListTemplate( site="Panel Chemistry", title="Py3DMol Pane", sidebar=[background, style], main=[pviewer], header_background=accent, accent_base_color=accent ).servable();
あとはコンソールでコマンドを打ち込むだけです。
panel serve PanelChemistry_py3Dmol.ipynb
ポートもデフォルト「5006」で変わらずなので「http://localhost:5006/PanelChemistry_py3Dmol 」にブラウザでアクセスすればアプリを利用できます。

できました!
2-4. 3つのアプリを同時に
最後に、3つのアプリを同時に使う方法です。
それぞれのノートブックでservable()にした状態で、コマンドラインで以下の通り3つ並べて起動すればOKです。
panel serve PanelChemistry_JSME.ipynb PanelChemistry_NGL.ipynb PanelChemistry_py3Dmol.ipynb
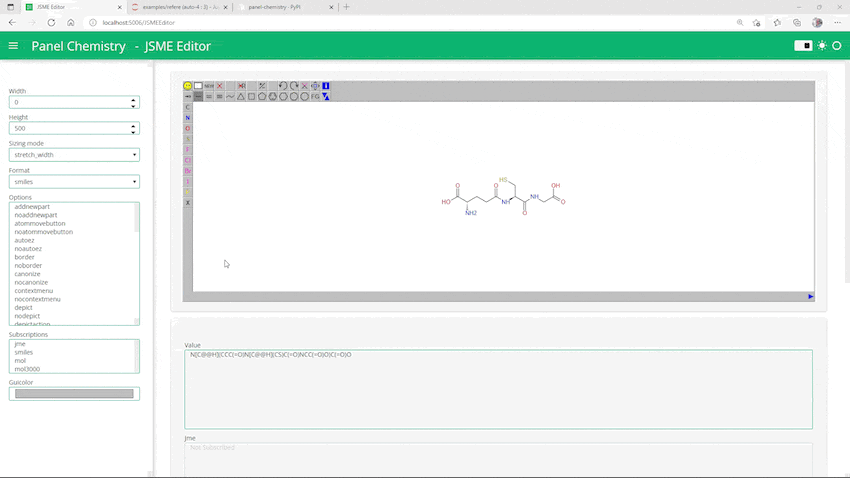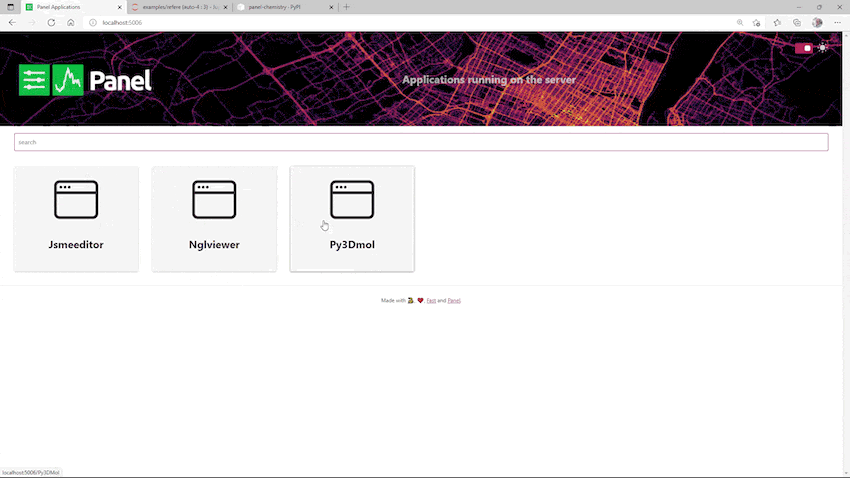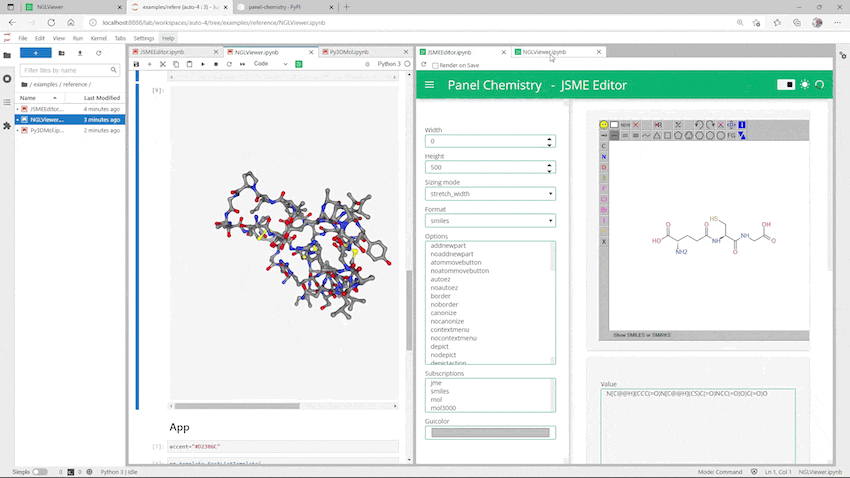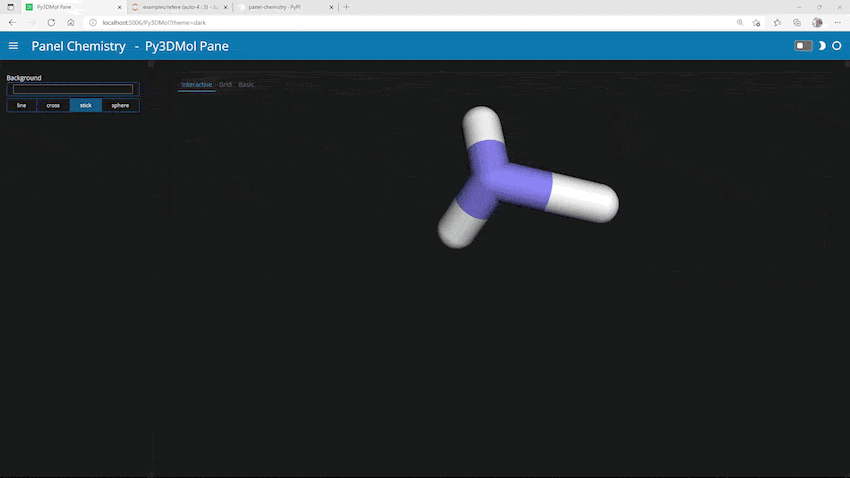
この状態で「http://localhost:5006/ 」にアクセスすると以下のようになります。
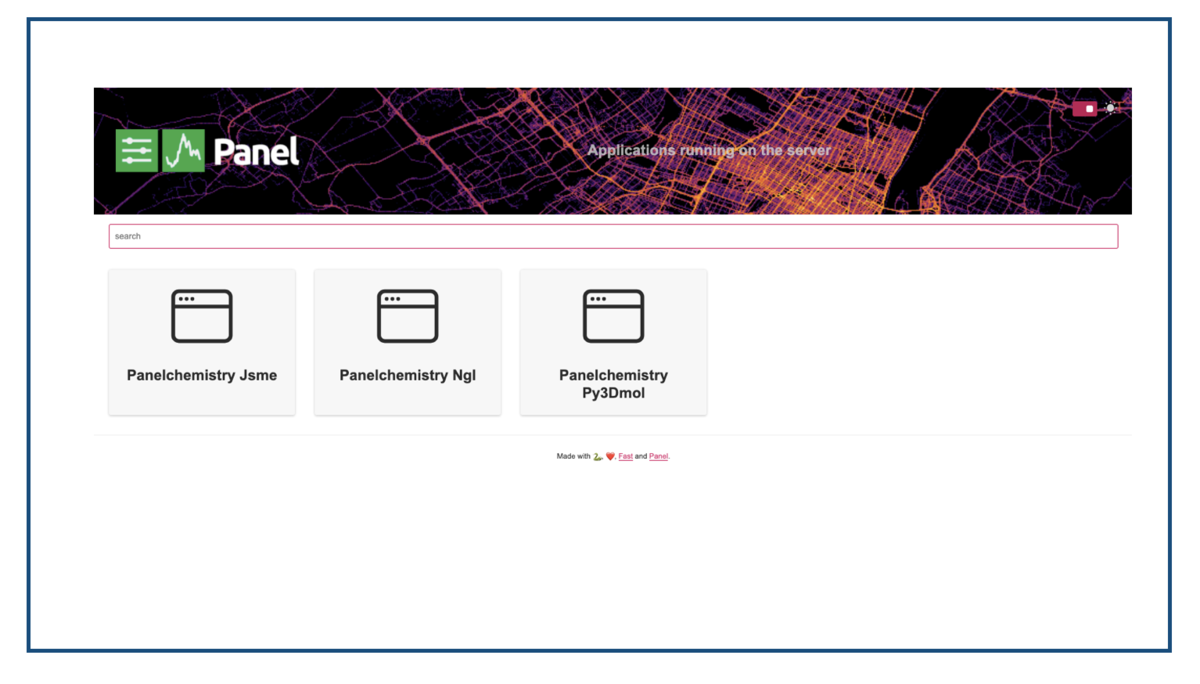
真ん中に並んでいるブロックがそれぞれJSME、NGL viewer、py3Dmolそれぞれのアプリに対応するので、クリックすれば開けます。もちろん別々のタブで同時に開くことも可能です。
3. おわりに
以上、今回は描画ツールPanel-Chemistryのご紹介でした。
化学構造式を描画・編集できるJSMEと、3Dの描画が得意なNGL Viewer、py3Dmolを並べて扱えるので便利ですね!一度コピー&ペーストしてノートブックを作っておけば、気軽にアプリとして起動できそうです。
まあ私はサーバーとかアプリとか全くわかってないんですけどね!
備忘録がわりのメモをはさんでいるのでGitHubのexampleよりわかりづらくなっているかもしれません。間違いがあったらご指摘いただけると嬉しいです。
ではでは!