ファーマコフォアスクリーニングをする話
前回の記事で共結晶構造を元にしたRDKitのファーマコフォアの作成を実施しました。いよいよこちらを用いてスクリーニングを実施したいと思います。
ファーマコフォアの準備
前回の再掲ですが、SAR News No.19の吉森氏による記事「Chemoinformatics Toolkits を用いた創薬システム開発における ラピッドプロトタイピング」での流れを確認します。
- 座標を使って特性基を定義
- 距離情報を使ってファーマコフォアを設定
- ファーマコフォアサーチ
- 対象のフィーチャーをそもそも含んでいるか?
- フィーチャー間の距離が条件を満たすか?
- 距離情報を拘束条件にして3D構造を発生させる。
このうちファーマコフォアの設定までは、前回のものをそのまま利用します。

import os from rdkit import RDConfig from rdkit.Chem import AllChem from rdkit import Chem from rdkit.Chem import ChemicalFeatures from rdkit.Chem.Pharm3D import Pharmacophore from rdkit import Geometry from rdkit.Chem.Pharm3D import EmbedLib from rdkit.Chem import rdDistGeom Ffactory = AllChem.BuildFeatureFactory(os.path.join(RDConfig.RDDataDir,'BaseFeatures.fdef')) feat_1=ChemicalFeatures.FreeChemicalFeature('Aromatic',Geometry.Point3D(-7.625, 10.407, -21.653)) feat_2=ChemicalFeatures.FreeChemicalFeature('Aromatic',Geometry.Point3D(-1.851, 11.051, -24.019)) feat_3=ChemicalFeatures.FreeChemicalFeature('Acceptor',Geometry.Point3D(-0.100, 13.586, -28.137)) feats = [feat_1,feat_2,feat_3] pcophore = Pharmacophore.Pharmacophore(feats) # ファーマコフォアの設定 d_upper = 1.5 # 特性基間の距離の許容範囲(上限値) d_lower = 0.5 # 特性基間の距離の許容範囲(下限値) # feat_1とfeat_2の距離 6.3Åを基準に、下限と上限を設定 pcophore.setLowerBound(0,1, 6.3-d_lower) pcophore.setUpperBound(0,1, 6.3+d_upper) # Acceptor(feat_3)の代表点の選び方によってfeat_2との距離は[5.0~5.6]の値をとる pcophore.setLowerBound(1,2, 5.0-d_lower) pcophore.setUpperBound(1,2, 5.6+d_upper) # 同様にfeat1とfeat_3は[9.8~11.2]の値をとる pcophore.setLowerBound(0,2, 9.8-d_lower) pcophore.setUpperBound(0,2, 11.2+d_upper)
ファーマコフォアをpcophore、フィーチャーファクトリーをFfactoryとして用意しました。molオブジェクトが与えられた場合に、ファーマコフォアサーチを行う関数を作成します。
ファーマコフォアサーチの関数作成(関数1)
距離情報を拘束条件にした3D構造がうまく発生できた場合、その構造をファーマコフォア合致構造とすることとします。また、合致構造間での順位づけとして、構造がうまく生成できた数(条件を満たすコンフォマーの数)をスコアとしてもちいることとしたいと思います。生成させる構造の最大数(count)は10としました。
具体的な関数を以下のように作成しました。
def PSearch(mol): #水素原子を付加(3次元構造利用のため) molH = AllChem.AddHs(mol) #水素原子を綺麗に整える座標計算 AllChem.Compute2DCoords(molH) #フィーチャーをそもそも持っているか? match, mList = EmbedLib.MatchPharmacophoreToMol(molH, Ffactory, pcophore) print(match) if match == True: #距離の検証 #距離行列の取得 bounds = rdDistGeom.GetMoleculeBoundsMatrix(molH) #ファーマコフォアのマッチング pList = EmbedLib.GetAllPharmacophoreMatches(mList,bounds,pcophore) #pListのそれぞれについてFeatureとマッチする原子のidを取得する num_match = len(pList) phMatches = [] for i in range(num_match): num_feature = len(pList[i]) phMatch = [] for j in range(num_feature): phMatch.append(pList[i][j].GetAtomIds()) phMatches.append(phMatch) #原子のidとファーマコフォアをもとに3次元構造を埋め込む Generated_embeds = [] for phMatch in phMatches: bm,embeds,nFail =EmbedLib.EmbedPharmacophore(molH, phMatch, pcophore,count=5, silent=1) Generated_embeds.append(len(embeds)) return sum(Generated_embeds) else: return 0
関数1の検証とエラー
この関数をもちいて動作確認のため、共闘用シェアデータ ) から取り出した分子のうち、前回用いなかったものを含めて検証します*1。
chain_suppl = Chem.SDMolSupplier('./chain_compounds.sdf', removeHs=False) chain_mols = [x for x in chain_suppl if x is not None]
この化合物群に適用したところ早速以下のエラーで止まりました。
--------------------------------------------------------------------------- KeyError Traceback (most recent call last) <ipython-input-40-74609285009c> in <module>() ----> 1 PSearch(test_m3) <ipython-input-38-904fad3ab3c2> in PSearch(mol) 34 35 for phMatch in phMatches: ---> 36 bm,embeds,nFail =EmbedLib.EmbedPharmacophore(molH, phMatch, pcophore,count=20, silent=1) 37 38 Generated_embeds.append(embeds) ~/.pyenv/versions/anaconda3-4.4.0/lib/python3.6/site-packages/rdkit/Chem/Pharm3D/EmbedLib.py in EmbedPharmacophore(mol, atomMatch, pcophore, randomSeed, count, smoothFirst, silent, bounds, excludedVolumes, targetNumber, useDirs) 427 for idx, stereo in mol._chiralCenters: 428 if stereo in ('R', 'S'): --> 429 vol = ComputeChiralVolume(m2, idx) 430 if (stereo == 'R' and vol >= 0) or (stereo == 'S' and vol <= 0): 431 keepIt = False ~/.pyenv/versions/anaconda3-4.4.0/lib/python3.6/site-packages/rdkit/Chem/Pharm3D/EmbedLib.py in ComputeChiralVolume(mol, centerIdx, confId) 1241 nbrRanks = [] 1242 for nbr in nbrs: -> 1243 rank = int(nbr.GetProp('_CIPRank')) 1244 pos = conf.GetAtomPosition(nbr.GetIdx()) 1245 nbrRanks.append((rank, pos)) KeyError: '_CIPRank'
EmbedPharmacophoreの部分で止まっているようです。ComputeChiralVolumeという箇所のようです。
エラーの出た構造はこちら、
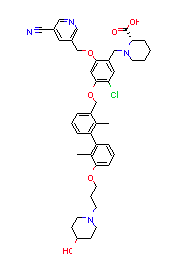
不斉点をもつ構造でエラーが出たようです。
エラーの原因は不斉点?
pythonファイルの該当部分の周辺をみると、nbrは不斉中心の近隣(neigbhbor)の原子を指定しているように見えました。
エラー箇所、
1243 rank = int(nbr.GetProp('_CIPRank'))
は、近隣原子の特性(atom property)を取得した際に、マジックプロパティの一つ_CIPRankの情報が見当たらないと言う内容のようです。
不斉に関する設定なので、CIPはカーン・インゴルド・プレローグ順位則(wikipedia: Cahn–Ingold–Prelog priority rulesの略と推測できます。不斉中心の近隣の原子のCIPRank、ということですから、CIP順位則に従って各原子に割り当てられた番号(rank)、を意味していそうです。
描画を見る限り光学活性体として認識されているのに、この情報が見当たらない、設定されていないというのはなぜ???
おそらくですが、一番最初に水素原子を付加(AllChem.AddHs())した際に、不斉中心(3級炭素)の周りの原子のプロパティ情報(_CIPRank)失われているように見えます。
検証してみたいと思います。
水素原子の取り扱いが原因?
まずは水素原子を付与する前のMolオブジェクトでプロパティを確認します。
不斉中心の情報を取得します。
Chem.FindMolChiralCenters(test_m3)
#[(31, 'S')]
AtomID: 31の原子に不斉 S が割り当てられています。GetNeighbors()を使って近隣の原子のidを取得します。
chiral_center = test_m3.GetAtomWithIdx(31)
nbrs = chiral_center.GetNeighbors()
取得した近隣の原子について、AtomIDと元素記号、問題の_CIPrankを出力します。
for x in nbrs: print('AtomID: ', x.GetIdx(), 'Symbol: ', x.GetSymbol(), 'CIPRank: ', x.GetProp('_CIPRank')) #AtomID: 30 Symbol: C CIPRank: 4 #AtomID: 32 Symbol: C CIPRank: 39 #AtomID: 26 Symbol: N CIPRank: 42
水素原子を付与する前のMolオブジェクトでは問題なくCIPRankが割り当てられています。値の意味はわかりません・・・・(1,2,3,4の順位かと思っていましたが、なんらかの基準でもっと広い範囲の値が割り当てられ大小を比較していそう・・・)
続いて水素を付与した場合の確認をします。
test_m3H = AllChem.AddHs(test_m3)
AllChem.Compute2DCoords(test_m3H)
Chem.FindMolChiralCenters(test_m3H)
#[(31, 'S')]
不斉中心を持つと言うことは認識されていそうです。
chiral_centerH = test_m3H.GetAtomWithIdx(31)
nbrsH = chiral_centerH.GetNeighbors()
for i in nbrsH:
print('AtomID: ', i.GetIdx(), 'Symbol: ', i.GetSymbol())
#AtomID: 30 Symbol: C
#AtomID: 32 Symbol: C
#AtomID: 26 Symbol: N
#AtomID: 79 Symbol: H
CIPRankを確認します。
nbrsH[0].GetProp('_CIPRank') """ --------------------------------------------------------------------------- KeyError Traceback (most recent call last) <ipython-input-99-89378a1786f7> in <module>() ----> 1 nbrsH[0].GetProp('_CIPRank') KeyError: '_CIPRank' """
付与した水素原子だけでなく、もともとあった炭素原子からも_CIPRankの情報が失われていました。
立体化学を再度割り当てれば良いのかと思い、Chem.AssignStereochemistry()を使ってみましたが、_CIPRankの情報は付与されていませんでした。
そもそも水素原子を付加していたのは、3次元構造を扱うためでした。今回、embedできたか否かを判定するだけ(3次元構造の最適化といった処理をしない)ですので、水素原子付加をしなくても良さそうです。
したがって、最初の2つの処理
molH = AllChem.AddHs(mol) AllChem.Compute2DCoords(molH)
を除いてしまえば良さそうです。
エラーを回避した関数作成(関数2)
上記2つを除いた関数をPSearch2として作成しました。(コード省略)
この関数を用いたところエラーで止まることなく最後まで計算できました。
PScore2_list = [] for x in chain_mols: num_embeds = PSearch2(x) PScore2_list.append(num_embeds) print(num_embeds) x.SetIntProp('PScore', num_embeds) print(PScore2_list) #[4, 62, 0, 7, 7, 2, 11, 78, 0, 0, 0, 0, 0, 60, 0, 2, 0, 0, 51, 0, 75, 0, 0, 29, 157, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 14, 0, 0, 20]
0の数が多いようなので確認してみます。
#リストから要素が0となっている数を求める print( len([i for i, x in enumerate(PScore2_list) if x == 0]) ) #24
活性化合物群のなかでもembedできた数が0となる構造が多数ありました(24個)。
実行結果で気になる点として、
RDKit DEBUG: [22:55:56] DEBUG: Removed embedding due to chiral constraints.
という表示が多く出力されました。不斉の制限のせいで弾かれている構造が多そうです。かなり荒い3次元構造の取り扱いをしているので、不斉点にそこまで強い制約を加えない方が良い気がします。
不斉点を考慮しない関数の作成(関数3)
今回の計算にあたって、不斉点を無視して処理したいと思います。具体的にはSMILESに変換(オプションでisomericSmiles=Falseとする)して不斉の情報を除いてからMolオブジェクトに戻すという処理を加えます。
#そのままSMILESに変換した場合 test_iso_smi = Chem.MolToSmiles(test_m3) print('chiral:', test_iso_smi) #出力: chiral: Cc1c(COc2cc(OCc3cncc(C#N)c3)c(CN3CCCC[C@H]3C(=O)O)cc2Cl)cccc1-c1cccc(OCCCN2CCC(O)CC2)c1C #立体の情報を除いてSMILESに変換した場合 test_smi = Chem.MolToSmiles(test_m3, isomericSmiles=False) print('achiral: ', test_smi) #出力: achiral: Cc1c(COc2cc(OCc3cncc(C#N)c3)c(CN3CCCCC3C(=O)O)cc2Cl)cccc1-c1cccc(OCCCN2CCC(O)CC2)c1C #もう一度Molオブジェクトに戻す test_mol_re = Chem.MolFromSmiles(test_smi) AllChem.Compute2DCoords(test_mol_re) #立体の確認 print(Chem.FindMolChiralCenters(test_mol_re)) #空のリストが出力された
isomericSmiles=Falseとしたものでは@Hがなくなっており、またChem.FindMolChiralCenters()が空のリストを返すことから、不斉の情報がなくなっていることが確認できました。
上記の処理を加えた関数を再定義します。
def PSearch3(mol): #水素原子を付加しない smi = Chem.MolToSmiles(mol, isomericSmiles=False) mol_re = Chem.MolFromSmiles(smi) AllChem.Compute2DCoords(mol_re) #フィーチャーをそもそも持っているか? match, mList = EmbedLib.MatchPharmacophoreToMol(mol_re, Ffactory, pcophore) if match == True: #距離の検証 #距離行列の取得 bounds = rdDistGeom.GetMoleculeBoundsMatrix(mol_re) #ファーマコフォアのマッチング pList = EmbedLib.GetAllPharmacophoreMatches(mList,bounds,pcophore) #pListのそれぞれについてFeatureとマッチする原子のidを取得する num_match = len(pList) phMatches = [] for i in range(num_match): num_feature = len(pList[i]) phMatch = [] for j in range(num_feature): phMatch.append(pList[i][j].GetAtomIds()) phMatches.append(phMatch) #原子のidとファーマコフォアをもとに3次元構造を埋め込む Generated_embeds = [] for phMatch in phMatches: bm,embeds,nFail =EmbedLib.EmbedPharmacophore(mol_re, phMatch, pcophore,count=5, silent=1) Generated_embeds.append(len(embeds)) return sum(Generated_embeds) else: return 0
関数3の検証
活性化合物群に適用します。
PScore3_list = [] for x in chain_mols: num_embeds = PSearch3(x) PScore3_list.append(num_embeds) #Molオブジェクトのプロパティに結果を保持させる x.SetIntProp('PScore', num_embeds)
今回はRDKit DEBUGは出ませんでした。期待した挙動を示していそうです。 embedできなかった数を数えます。
False_list = [i for i, x in enumerate(PScore3_list) if x == 0] print(False_list) #出力: [8, 14, 21, 25, 27, 28, 29, 30, 31, 32, 33, 34] print(len(False_list)) #出力: 12
今回はembedできた数が0となった構造は12個でした。キラルの制約を無くしたことで、活性化合物群のファーマコフォアベースのヒット率が上がったようです。
embedできなかった化合物について具体的な構造を一つ見てみます。
test_m8 = chain_mols[8] print(test_m8.GetProp('PScore')) # 0 Draw.MolToImage(test_m8)

Property「'PScore'」に値が保持されていることが確認できました。
ipywidgetsをつかって順番に眺めてみます。
from ipywidgets import interact,fixed,IntSlider import ipywidgets def False_mols(idx): mol_idx = False_list[idx] mol = chain_mols[mol_idx] return(display(Draw.MolToImage(mol))) interact(False_mols, idx=ipywidgets.IntSlider(min=0,max=len(False_list)-1, step=1));
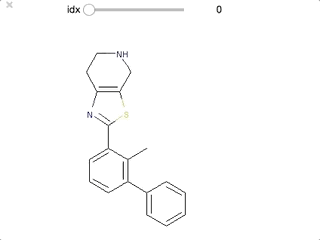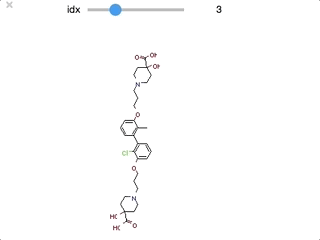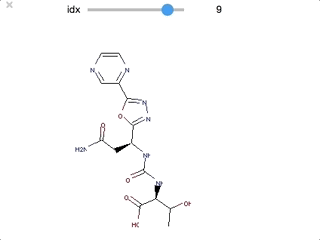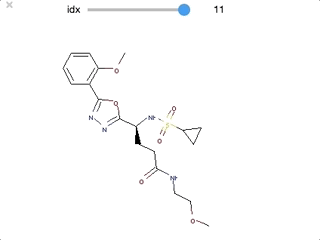
最初のいくつかはファーマコフォアを満たすとして認識されても良さそうですが、後半は芳香環を一つしかもたないなど、確かに要件を満たしていない、ということがわかります。
スクリーニングの実施
スクリーニング対象の再確認
関数の動作確認ができたので、いよいよ本題のスクリーニングに進みたいと思います。
スクリーニング対象の化合物群に関しては、以下の処理をこれまでに行いました。(だいぶ日があいたので再確認)
| 処理 | 内容 | |
|---|---|---|
| ① | 指標による絞り込み | Rule of Five & フラグメントライクな化合物の除去 |
| ② | 部分構造で絞り込み-1 | ビフェニル構造をもつもの |
| ③ | 部分構造で絞り込み-2 | オルト位に置換基を持つもの |
指標としては以下を用いています。
| 指標 | 分子量 | LogP | 水素結合供与体数 | 水素結合受容体数 | 回転可能結合数 | 極性表面積 |
|---|---|---|---|---|---|---|
| Rule of 5 | ≦500 | ≦5 | ≦5 | ≦10 | ||
| 基準 | >300 | >3 | >3 | >3 | >60 |
上記の結果として4220個の化合物まで絞り込み、biphenyl_library.sdfというSDFで保存しています。
スクリーニング本番
この化合物群にファーマコフォアサーチを実施します。
biphenyl_suppl = Chem.SDMolSupplier('./biphenyl_library.sdf') biphenyl_mols = [x for x in biphenyl_suppl if x is not None] biphenyl_PScores = [] for x in biphenyl_mols: num_embeds = PSearch3(x) biphenyl_PScores.append(num_embeds) x.SetIntProp('PScore', num_embeds)
ヒット(一つ以上ファーマコフォアを満たす構造がembedできた化合物)の数を確認します。
Hit_list = [i for i, x in enumerate(biphenyl_PScores) if x != 0] print(len(Hit_list)) # 2463
合計2463個の化合物が残りました。約半数に絞り込めたようです。
上記の計算過程で、
RDKit ERROR: [23:37:56] DEBUG: Removed embedding due to c[00:09:28] UFFTYPER: Unrecognized charge state for atom: 1
といったエラーが出ましたが、4つだったので今回は深追いするのをやめようと思います。(困難から逃げるダメな大人)
embedの数が最も多かった化合物を確認してみます。
print([i for i, x in enumerate(biphenyl_PScores) if x == max(biphenyl_PScores)]) # [1351] max_mol = biphenyl_mols[1351] max_mol.GetProp('PScore') #'99' Draw.MolToImage(max_mol)
数は99で、構造は以下でした。
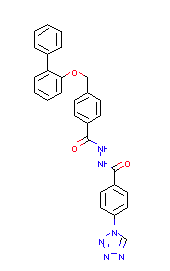
・・・ほとんどsp2です。溶解度悪そう、、、
とりあえず結果をSDFで出力しておきます。
#保持するためのpropertyを確認 check_mol = biphenyl_mols[0] prop_names = [p for p in check_mol.GetPropNames()] print(prop_names) # ['original_id', 'MW', 'MolLogP', 'NumHDonors', 'NumHAcceptors', 'NumRotatableBonds', 'TPSA', 'PScore'] writer = Chem.SDWriter('PSearched.sdf') writer.SetProps(prop_names) for mol in biphenyl_mols: writer.write(mol) writer.close()
おしまい。
まとめ
今回は、前回作成したファーマコフォアをもちいて、スクリーニングを実施してみました。不斉点を持つ化合物に対して期待した挙動をしめさず、いくつか修正が必要となりましたが、最終的に約4000個の化合物から、2500個程度に絞ることができました。思っていたほど減りませんでしたが、ファーマコフォアの設定ポイント(フィーチャー)として、ビフェニル骨格の芳香環2つを選んでいることに由来しているかもしれません。すでに「ビフェニル構造を部分構造として持つ」という絞り込みを行なったリストに対してスクリーニングを実施しているので、「より数を絞り込む」と言う観点からはビフェニル以外のフィーチャーを選択すべきであったかもしれません。