オープンソースで逆合成解析 ~AiZynthFinder~ part 2. 遊んだ
オープンソースの自動逆合成解析ツール、AiZynthFInder。前回は背景を知るために文献を読んでみました。今回は実際に遊んでみたいと思います。
ツールは以下のGitHubに、
ドキュメントはこちらにあります。
インストール
GitHubのREADMEそのままにインストールしていきます。
Prerequisites
サポートされているOSはLinux、Windows、Macです。ツールの開発はLinuxで、Windows 10、macOS Catalinaではテスト済みとのこと。
ちなみに私のPCはmacOS High Serraです。後ほどいくつか躓いた点を書きますが、他のOSでは当てはまらないかもしれません。
あと、Python 3.6か3.7のanacondaかminicondaのインストールが必要です。
Installation
まずcondaのパッケージを3つインストール。
RDkitユーザーおなじみの仮想環境 my-rdkit-env に入れてみます。アクティベート! !
conda activate my-rdkit-env # お好みの環境でどうぞ conda install -c rdkit "rdkit=>2019.09.1" -y conda install -c anaconda tensorflow>=2.1.0 -y conda install graphviz -y
ついでAiZynthFinderパッケージをインストール
python -m pip install https://github.com/MolecularAI/aizynthfinder/archive/v1.0.0.tar.gz
簡単! でもこれでも失敗するんですよ、私は。
インストールの注意点
起動するとこんな感じになる予定でしたが、私はSMILESを入力しても構造式が描画されませんでした。
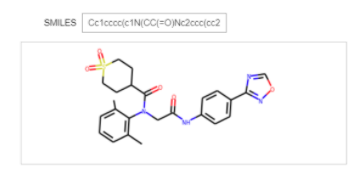
RDKitのモルオブジェクトがある、みたいな表示はされましたが、、、
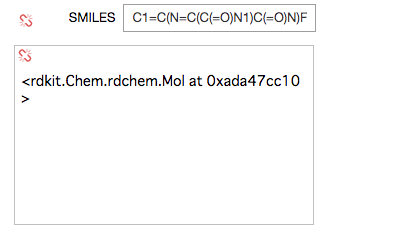
READMEを見直すと、Graphvizに問題がありそうでした。インストールの際に代わりに-c anacondaを使ってくださいとのことだったので、こんな感じでやってみたら解決しました。
conda install -c anaconda graphviz -y
違いがわからない…
他に必要なもの
ツールを使うには以下の3つ用意する必要があります(ファイル的には4つ)。
- stock file : 逆合成の終点となる前駆体(購入可能な試薬)のリスト
- 訓練済みの rollout policy network : Kerasモデルと、ユニークなテンプレートのリスト(計2ファイル)
- configuration file
ありがたいことに1と2はfigshare からダウンロードできるようにしてくださっています。
以下の3つのファイルがダウンロードできます。結構大きいのでちょっと時間がかかります...
- zinc_stock_17_04_20.hdf5 (約650MB)
- full_uspto_03_05_19_rollout_policy.hdf5 (約300MB)
- full_uspto_03_05_19_unique_templates.hdf5 (約45MB)
ファイル名からstockはZINC、rollout policy model と template はUSPTOをベースとしてそうな感じがわかりますね。
で、figshareからはダウンロードした場合はconfiguration fileは自分で用意する必要があります。
ドキュメンテーションのConfiguration file を参考に、こんな感じのテキストファイルを書いて「config.yml」として保存しました。
policy:
files:
full_uspto:
- full_uspto_03_05_19_rollout_policy.hdf5
- full_uspto_03_05_19_unique_templates.hdf5
stock:
files:
zinc: zinc_stock_17_04_20.hdf5
面倒!! という方のためにもっと簡単な方法も用意されています。
次のコマンドで指定したフォルダにデータのダウンロードとconfigファイルの作成まで行ってくれます。「my_folder」のところはお好きなフォルダ名に変えてください。
download_public_data my_folder
著者親切すぎる・・・。
さて、ファイルが4つとも用意できたら、これらを全部作業ディレクトリに入れます。ディレクトリを移動して起動だ!!
・・・の前に、configの注意点を少し。
configの注意点
configuration fileはインターフェースにpolicy modelとstockへのファイルパスと、探索の詳細なパラメータを追加で指定するためのものです。
ですので、ファイルを作業ディレクトリ直下やパスの通っていない場所に置いておくとエラーが出てしまいます。
私は最初、作業ディレクトリ内に「data」というフォルダを作ってそこに「~.hdf5」ファイルを入れていました。この状態で先の「config.yml」を使うと以下のエラーが出ました。
OSError: SavedModel file does not exist at: full_uspto_03_05_19_rollout_policy.hdf5/{saved_model.pbtxt|saved_model.pb}
だよね! 見つかんないよね! 指定してないもんね!
相対パスに直したら動きました(「./data/」を追加)。
# config.yml
policy:
files:
full_uspto:
- ./data/full_uspto_03_05_19_rollout_policy.hdf5
- ./data/full_uspto_03_05_19_unique_templates.hdf5
stock:
files:
zinc: ./data/zinc_stock_17_04_20.hdf5
わかってみれば簡単でした。パス苦手・・・。
GUIアプリ
AiZynthFinderはJupyter Notebook上で使えるGUIと、コマンドベースのCUI両方が提供されています。
GUIの利点は何と言っても構造式を見ながら使えることでしょうか?
一方、CUIはBatchモードが使えるので複数の分子を一度に処理できるという利点があります。
ここは、コマンド苦手だからGUI一択で!
まずは作業ディレクトリに移動してJupyter notebookを起動します。
ちなみに、aizynthapp --config config_local.ymlとするとノートブックを自動で作ることもできるらしい。至れり尽くせり。。。
以下、2行をコピペしてCtrl+Enterでインターフェースが起動します。
from aizynthfinder.interfaces import AiZynthApp app = AiZynthApp("./config.yml")
出た!! さあ、逆合成開始だ!・・・と思いましたが、ここでもまた問題が! Kernelが死ぬ。。。
OMPとKernelの死(Mac限定?)
さて、インターフェースが起動したので早速検索を実行したところ、直ぐに画面が灰色になり「kernel die」と表示されnotebookが止まってしまいました。
・・・ YOU LOSE 的な?
ターミナルをみると以下のエラーが出てました。
OMP: Error #15: Initializing libiomp5.dylib, but found libiomp5.dylib already initialized. OMP: Hint This means that multiple copies of the OpenMP runtime have been linked into the program. That is dangerous, since it can degrade performance or cause incorrect results. The best thing to do is to ensure that only a single OpenMP runtime is linked into the process, e.g. by avoiding static linking of the OpenMP runtime in any library. As an unsafe, unsupported, undocumented workaround you can set the environment variable KMP_DUPLICATE_LIB_OK=TRUE to allow the program to continue to execute, but that may cause crashes or silently produce incorrect results. For more information, please see http://www.intel.com/software/products/support/.
どうもOpenMPというのがよろしくないらしい。原因がわからない。。。
ヒントに安全じゃないけど、と断りつつ回避方法も書いてあるのでそのままやってみます。Notebook上で環境変数を書き換えます、、、 *1
import os os.environ['KMP_DUPLICATE_LIB_OK']='TRUE'
パス苦手・・・。
この後、再度GUIを起動するコマンドをうつと今度は「kernel die」することなく検索できました!
I win !!!
もうちょっと調べるとmacOSでcondaを使っていることが原因な疑惑が出てきました。( stack overflowの記載より )
雰囲気こんな感じ・・・
- Condaは IntelのMKL optimizationsを使っていてそれにOpenMPが使われている。
- macOSはMKLではなく、独自のアルゴリズムを使っていてそこに既にOpenMPが使われいる。
- なのでOpenMPが重複することになり先のエラーが出る
解決方法として、MKLサポートなしでパッケージを再イントールすること(conda install nomkl)が書かれていました。試してませんが備忘録として・・・。
詳しい方ご存知でしたら教えていただけると嬉しいです。
逆合成してみた
やっと準備が整ったので、気を取り直して再度実行。
まとめると私の場合これを打ち込めばOK!
import os os.environ['KMP_DUPLICATE_LIB_OK']='TRUE' from aizynthfinder.interfaces import AiZynthApp app = AiZynthApp("./config.yml")
Favipiravir
まずは小さなものから、ということで話題のアビガン(Favipiravir)!
PubChemによるとSMILESは「C1=C(N=C(C(=O)N1)C(=O)N)F」ということみたいです。
SMILESをコピペ + Enterで構造出ました!
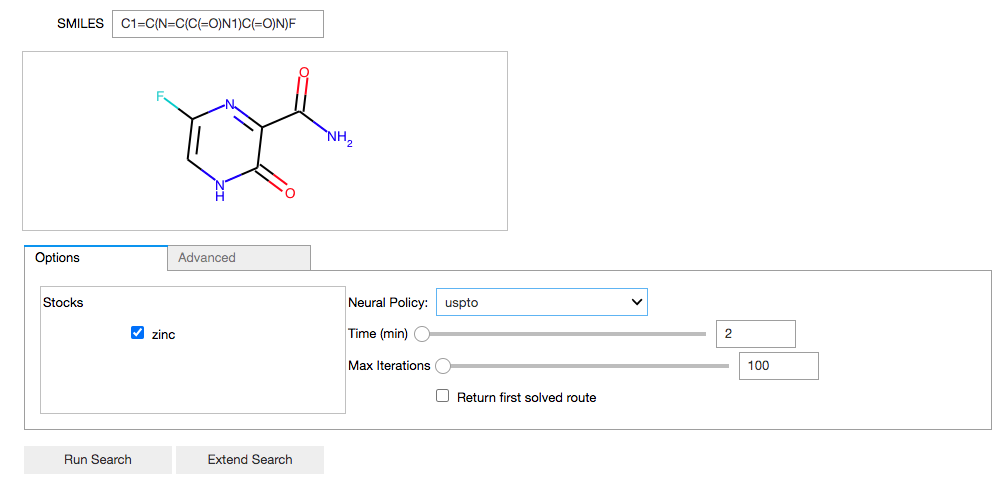
さあ、逆合成解析の実行だ! Run Search!!
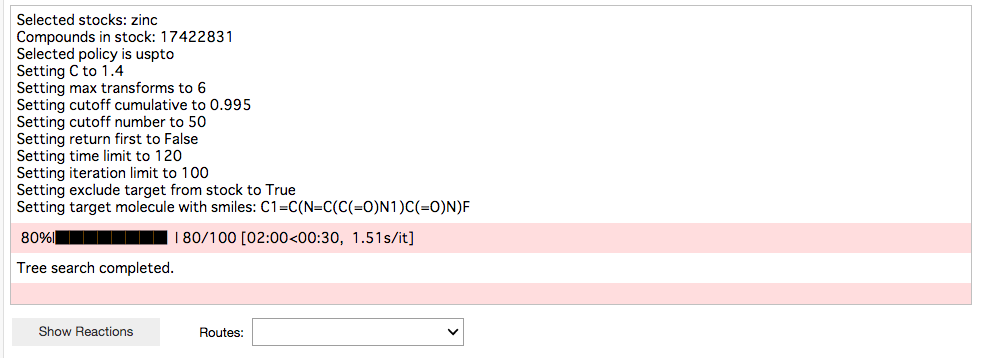
終わった。探索の最長時間 2分、最大イテレーション 100となっていたので、今回は2分経った時点で探索終了。イテレーションは80でした(80/100)。
どんな結果かみてみましょう。Show Reactionsをクリック!
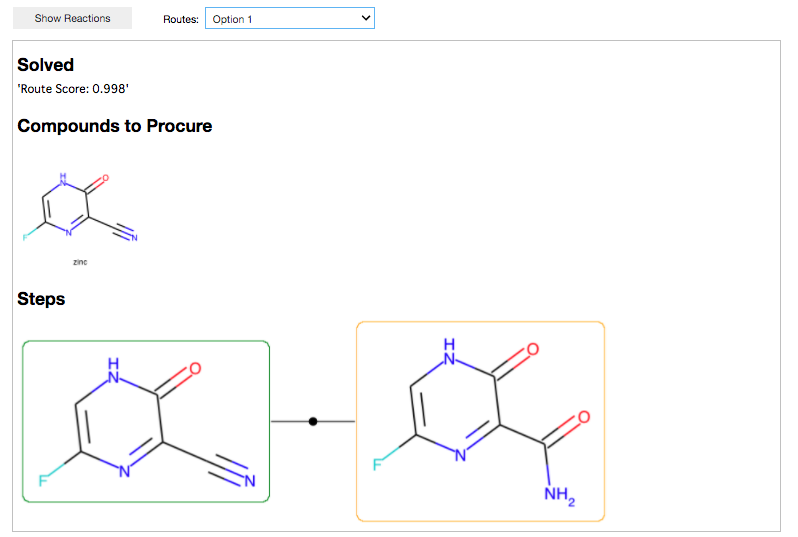
上位5つの結果から選択でき、上は最も良いスコアのルートです。
Compounds to ProcureがStock fileで指定したZincの中で見つかった手に入りそうな原料、Stepsが合成ルートです。
シアノ基の水和で1 stepのルート。ふむ、なるほど。。。
別のルートはどうでしょうか?
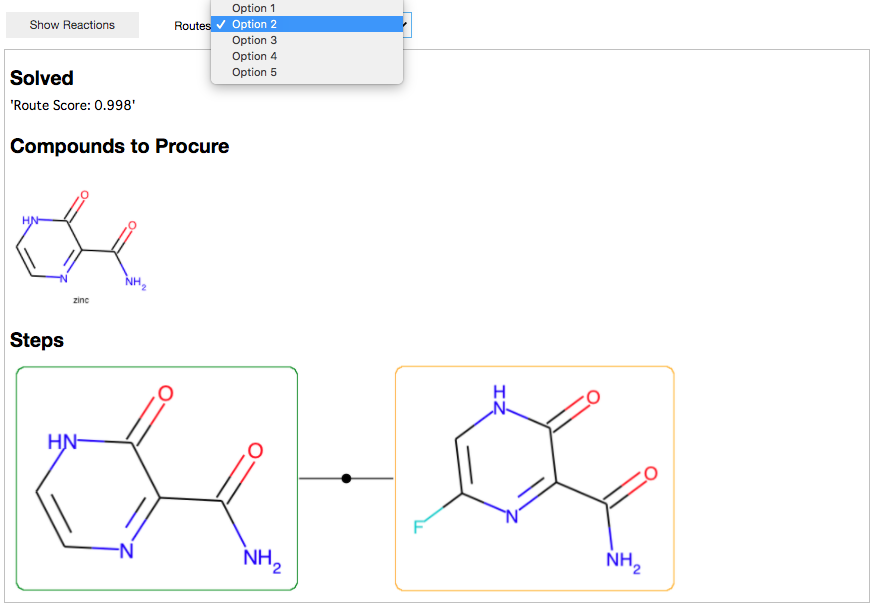
フッ素化・・・ミニスキ反応っぽいあれを使えばできる、、、のでしょうか?
3つめのルート。
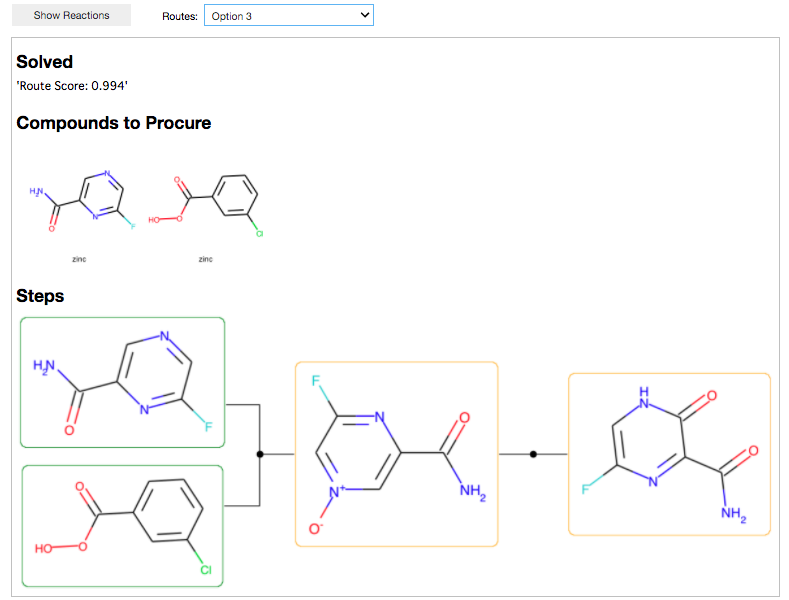
N-oxideの転位で酸素を導入! ほーそういうのもあるのか!!
選択性の考慮は今後の課題と論文にありましたが、確かにこのルートでは酸化も転位も選択性が課題になりそうです。
Remdesivir
もう少し大きな分子、ということで今度はレムデシビルを試してみます。
立体は考慮しないみたいですがとりあえずIsomeric SMILESは「CCC(CC)COC(=O)C@HNP@(OC[C@@H]1C@H(C#N)C2=CC=C3N2N=CN=C3N)O)O)OC4=CC=CC=C4」だそうです。
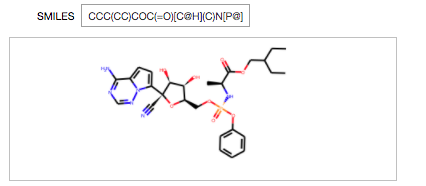
今回は探索時間2分でイテレーション78回でした。で、残念ながら解けなかったらしい(Not Solved, Route score: 0.715)。
最もスコアが良いものはこんな感じ、、、。
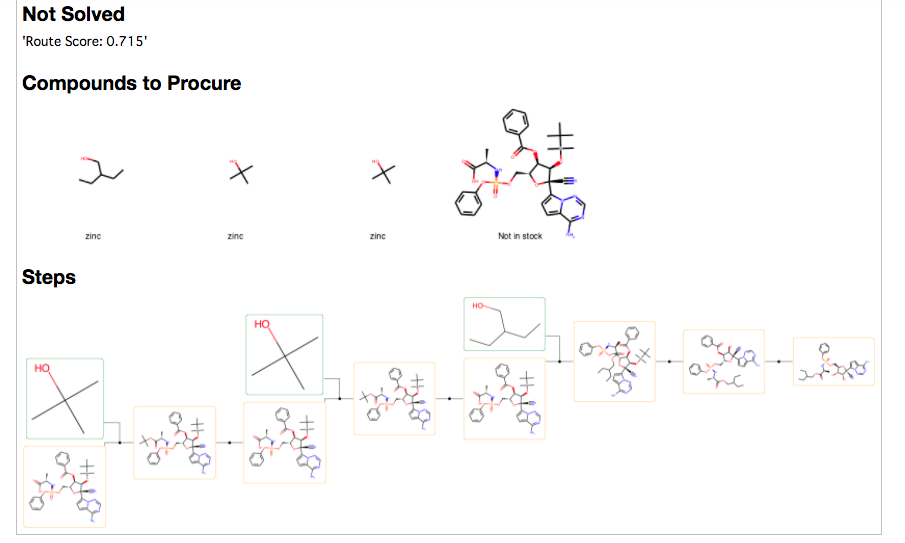
画像が小さいですが、Compounds to Procureの一番右の化合物はNot in stockとなっています。
Stock file内の構造まで分解できなかったみたいのでNot Solvedとなるようです。
ルートを見ると最初はtert-Butylエステルを作っては分解、作っては分解してます。
文献中に保護-脱保護の合成戦略はまだ組み入れられていないと記載されていましたが、確かに苦戦している様子が伝わってきます。
核酸アナログや糖って合成特殊な感じしますよね。ジョーンズ有機化学にも糖の合成しんどいみたいなこと書いてあった気がします。記憶の彼方ですが。。。
Ledipasvir
目標はもっと高く、大きな分子!ノーベル賞関連でC型肝炎治療薬、レジパスビルを逆合成!
SMILESも長い!! 「CC(C)C@@HN1CC2(CC2)C[C@H]1C3=NC=C(N3)C4=CC5=C(C=C4)C6=C(C5(F)F)C=C(C=C6)C7=CC8=C(C=C7)N=C(N8)[C@@H]9[C@H]1CCC@HN9C(=O)C@HC)NC(=O)OC)NC(=O)OC」
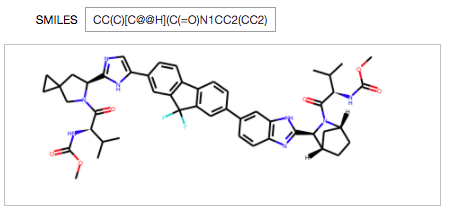
イテレーション3までしか進まずに探索が終了してしまいました。全くもってすごい分子ですよ。どうやって見つけたんだろう??
これもNot Solved(Route score: 0.534) で、分解の結果にZincのStock fileにないものが含まれていました。提案ルートはこんな感じ、、、

真ん中あたりで切って、左右ユニットに分かれたルート提案となっています。
構造の右半分ユニットのルートは図の上部、左半分ユニットは下部に分かれています。
どちらもイミダゾール環(右はベンゾイミダゾール)を構築しています。
なかなかヘテロ環、芳香環を切断-構築する逆合成って難しいのかな、と思っていたのですが、合成のプロからしたら普通なのでしょうか?
イミダゾールの特殊性みたいなものがあるのでしょうあ?ちょっとわからないので専門家の方コメントいただけると嬉しいです。
ここで気づいてしまいました。正解(実際の合成ルート)を知らず、妥当な合成を判断できない私が眺めていてもツールの性能は評価できない・・・・
答え合わせ?
というわけで合成ルートをググりました。巨人の肩の上に立つ!
実際の製造方法はわかりませんが一つのルートとして比較してみると面白いかもしれません。
Favipiravir
富士フィルム株式会社の特許から(JP2012180336)。以前Twitterで見かけて印象に残ったルートです。
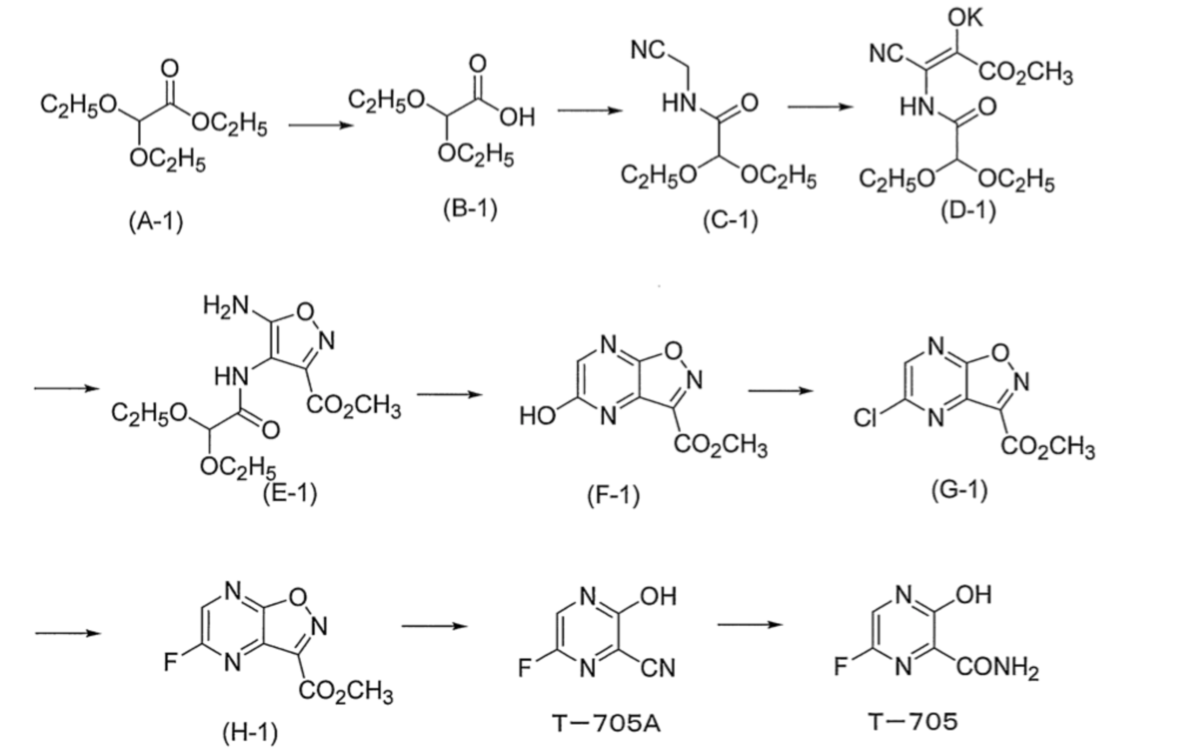
H1からT-705Aへは塩基性条件で加熱撹拌してます。加水分解、脱炭酸、ヘテロ環の開裂の3段階が起こってるんですかねー?お洒落!
・・・というか最終工程(T-705A → T-705)、AiZynthFinderが最初に予想したルートそのものです。えっ、凄い!!!
Remdesivir
New Drug Approvalsというサイトに合成ルート含めて色々と記載がありました。が、ちょっと長いのと、今回はAiZynthFinderの提案がうまくいってなかったので割愛させていただきます。
気になる方はサイト(New Drug Approcals: Remdesivir)をご覧ください。こちらによるとGilead Sciences社の特許に記載があるらしいです(WO2016069826)。
Ledipasvir
こちらも先のサイトに記載がありました。 リンクはこちら New Drug Approvals:Ledipasvir。
Gilead Sciences社の特許WO2013184702が引用されています。ルートの全体は上記サイトをみていただくとして、特許の一部を抜粋します。
右側ユニット・・・ベンゾイミダゾール巻いてます!っていうかそのまんま!
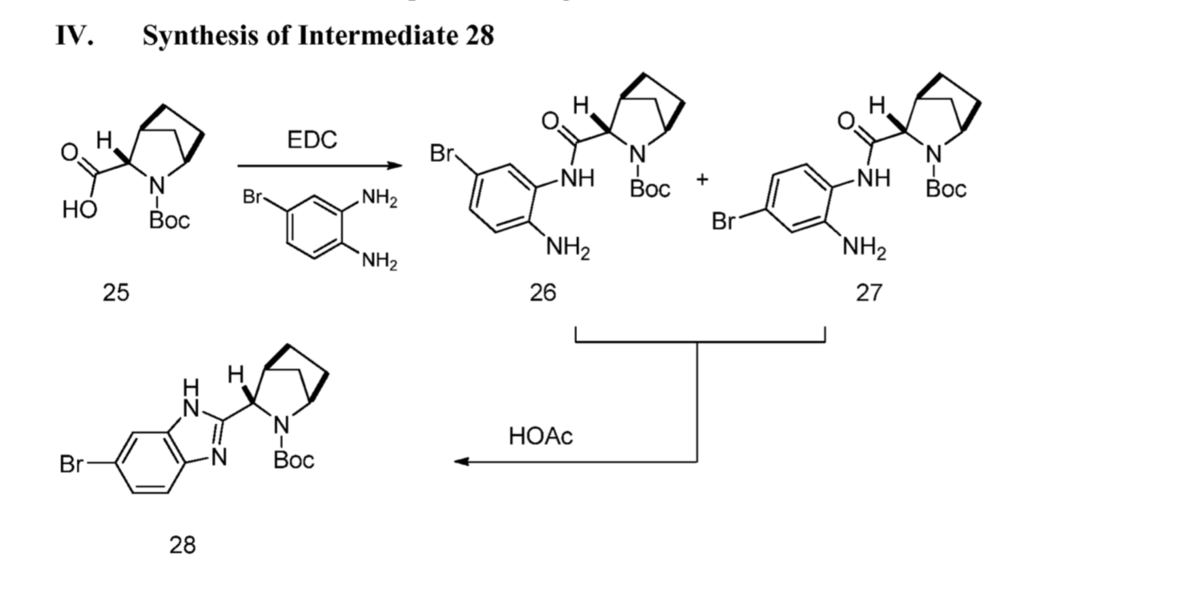
左側ユニット・・・こっちもイミダゾール巻いてます!保護基が特許はN-Boc基、AiZynthFInderはN-Cbz基という違いはありますがだいたい一緒!
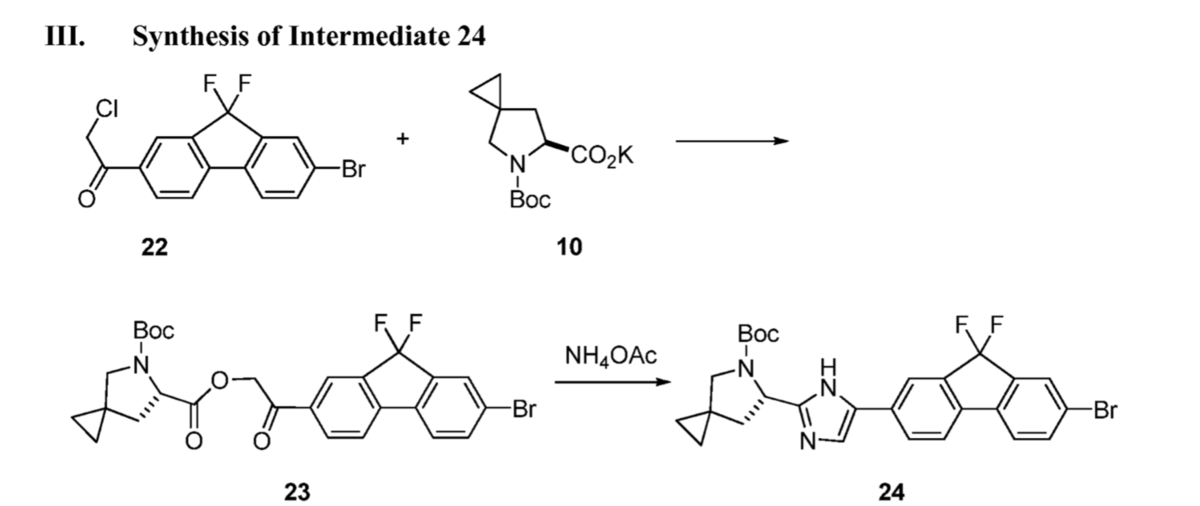
そんでもって右側と左側の連結は・・・ベンゾイミダゾールユニットのボリル化を経た鈴木-宮浦クロスカップリング!反応の選択一緒!
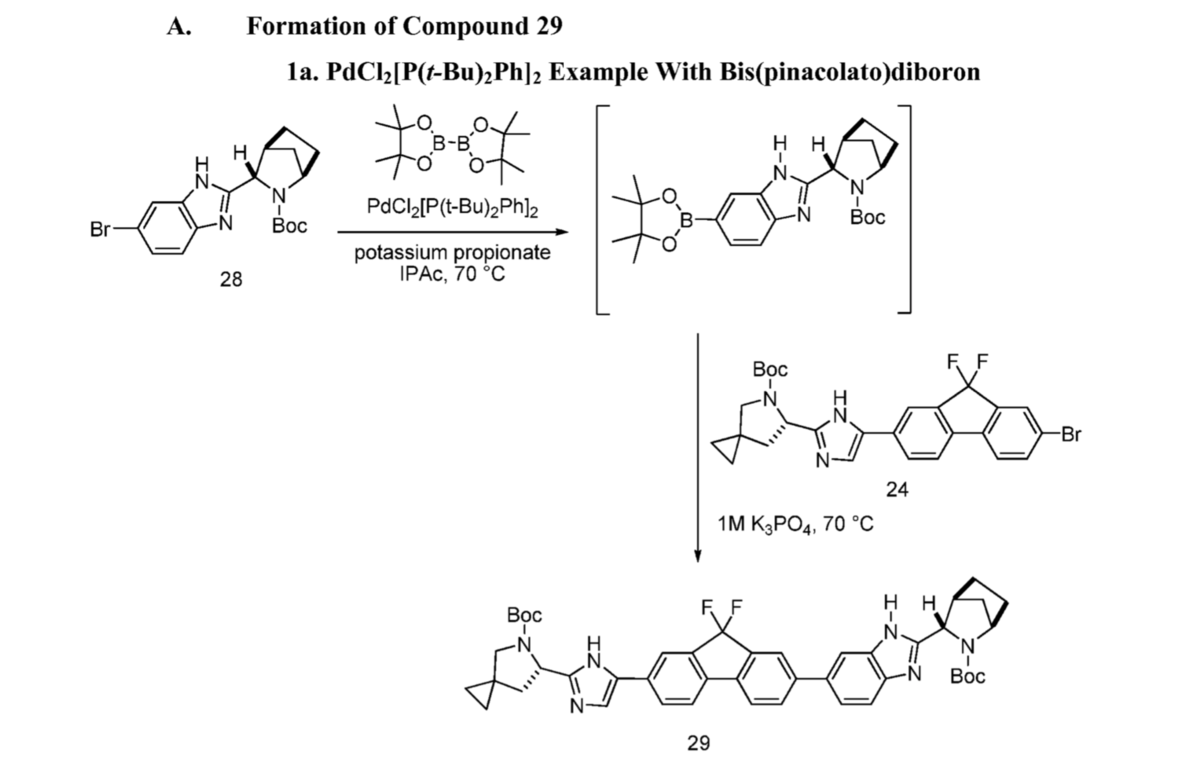
ここから特許では両端のBoc基を外し、2つともバリンメチルエステルとアミド化することでLedipasvirに導いています。
AiZynthFInderは、バリンユニットの導入のタイミングをのぞいて、ほぼ反応のコンセプトとしては同じルートを提案しているようです!
すごい!!!
まとめ
他にも機能はあるようですが、とりあえず今回はGUIで遊んでみました。これだけ親切設計なのに使えるまでに色々躓いてしまいました・・・。
実際に遊んで見るまでは、データから学習しているといっても限界があるのではないかな・・・と思ってましたが、AiZynthFInder予想以上に良い提案を出している、というのが私の感想です。
というか少なくとも私よりはるかに賢いです。。。ワタシ、チョットカナシイ。。。オベンキョウタイヘンダッタノニ。。。
しかし、これがオープンソースで公開されるというのは本当に凄いですね。私は遊ぶことしかできないので、みなさんぜひ使ってみてGitHubに改善を提案してください。合成の専門家からみたルートへのつっこみとか聞いてみたいです。
ではでは