cootで電子密度を眺める

前回、結晶の質を眺めるにあたってPDB viewer を使用しましたが、cootというソフトウェアをご紹介いただきました。
cootで電子密度見ましょう!リガンドの化学構造が誤っている部分は、電子密度もけっこうプアでした。
— torusengoku (@torusengoku) 2018年11月13日
Sengoku先生、ありがとうございました!
早速導入(無料!)
以下のページからダウンロードしてきました。
Stand-Alone Coot - OS X Scientific Computing
オフィシャルHPはこちら
→ https://www2.mrc-lmb.cam.ac.uk/personal/pemsley/coot/
ターミナルで 「coot」と打ち込むと無事立ち上がりました。
ソフトを問題なくインストールできたのは初めてかも・・・
インターネット上でみつけたtutorialを参考に、課題の構造 [PDB id: 5NIX ]を眺めてみました。
tutorial 1 → Coot Tutorial X-ray Methods, CSHL 2016 Dec. 18, 2017 (pdf)
tutorial 2 → X線結晶構造解析における構造バイオインフォマティクス (pdf)
- 1. 設定の変更
- 2. ファイルの準備
- 3.ファイルの読み込み
- 4. 構造のおかしな場所を簡単に探す
- 5. アミノ酸側鎖の補正
- 6. リガンド部位の確認
- 7. validationのグラフ
- 8. 別の構造でも確認(PDB id: 5NIU)
1. 設定の変更
tutorial 1 のオススメに従い設定を変更。
① 分子の動かし方 "virtual trackball" (2.2章)
・"Sperical Surface" → "Flat" modeへ変更
② 動きを加速するための設定 (2.4章)
・ Preferences から "Active Map on Draggeing" を "No" へ変更
・"Smooth Recentering" の "Number of Steps" を 40 から 20 へ変更
③ 注目している原子の周辺の原子との距離を表示 (2.4章)
"Measures" の "Environment Distances" で
・"Show Residue Environment?"
・ "Label Atom?"
を表示させるように変更
2. ファイルの準備
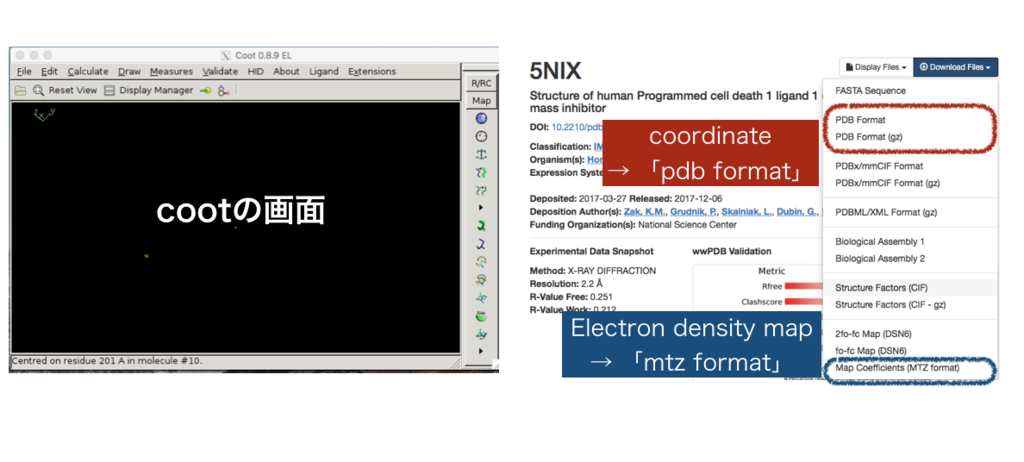
・原子座標ファイル(coordinate)・・・「PDB format」
・X線回折データ (電子密度)・・・「Map Coeficients (MTZ format)」
を取得しました。
3.ファイルの読み込み
File の [Open Coordinates…] から PDB format を、[Auto Open MTZ…]から mtz format を読み込みます。
電子密度マップの表示領域を半径 20Åとすると以下のような図が得られました。(Fig. 2)
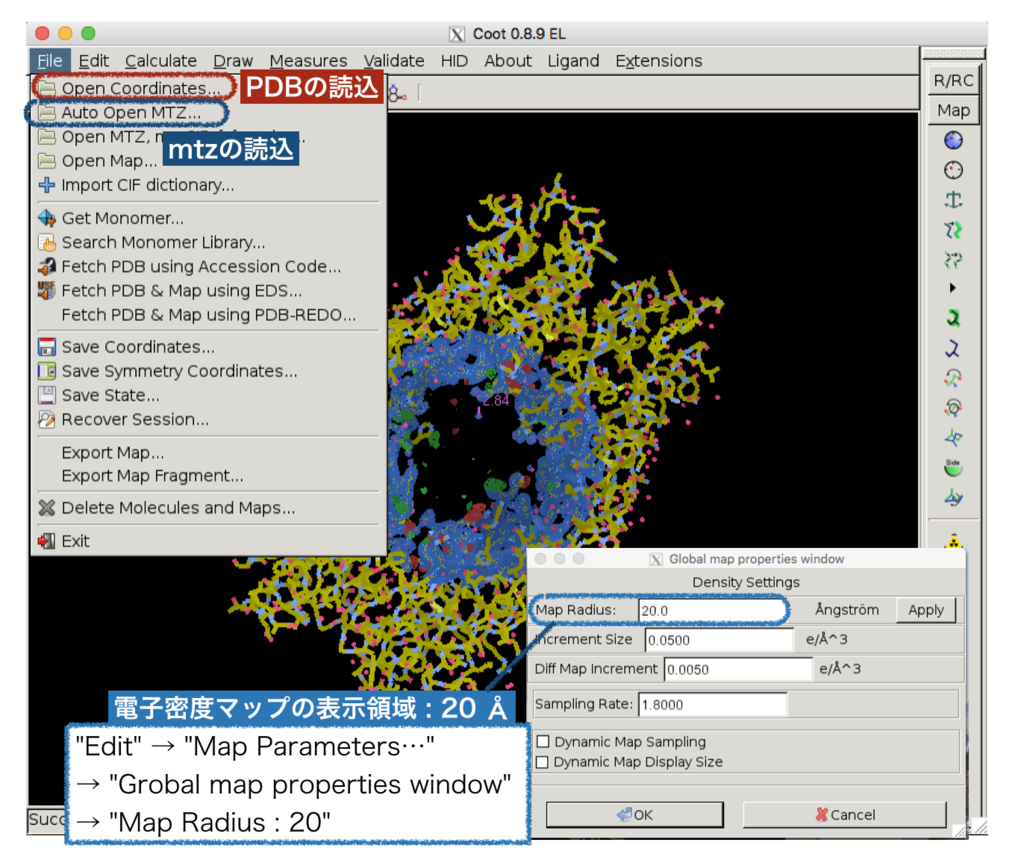
電子密度の色はそれぞれ、
・青 : 2Fo - Fc マップ → 電子の存在位置
・赤 : Fo - Fc マップ(正)→ 本来電子密度がないはずなのに構造が置かれている
・緑 : Fo - Fc マップ(負)→ 本来電子密度があるはずなのに構造が置かれていない
を表すそうで、つまり赤と緑がモデルのまずい場所だそうです。
4. 構造のおかしな場所を簡単に探す
電子密度の色の違いはわかりましたが、アミノ酸残基一つ一つを確認するのは面倒です。
Coot の 「Density Fit Analysis」をもちいると、残基ごとのモデルの良し悪しを簡便に確認可能でした。
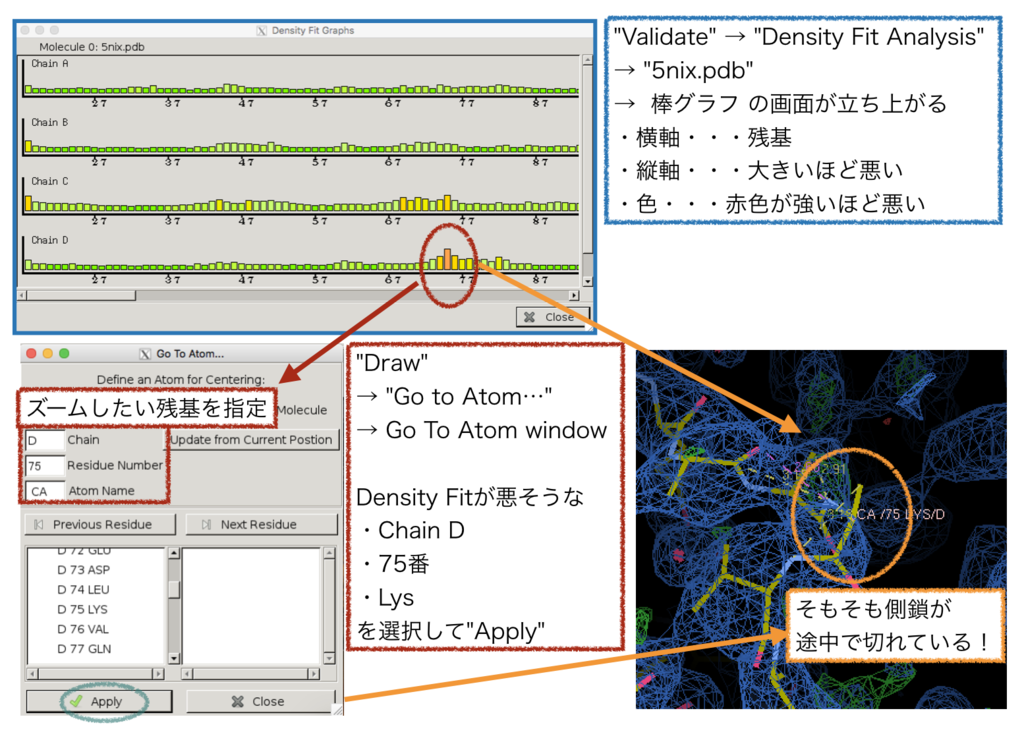
chain D の 残基番号 75 Lys が「Density Fit Analysis」でもっともFit が悪そうです。
確認したい残基の棒グラフをクリックすると、自動的に目的のアミノ酸残基がズームされます。また、「Go To Atom…」機能をもちいても、残基(原子)を指定したズームが可能です。
どうやらLys 側鎖に電子密度が見えておらず、そもそも側鎖もモデル化されていないようです。
途中で切れてる・・・
他にもChain A で Fit の悪い 82 ARG などでも側鎖が途中で切れており、モデル化されていませんでした。
長く、自由度の高いアミノ酸残基が特に溶媒露出面に位置している場合、電子密度がうまく観測されずモデル化も難しい、という感じでしょうか??
5. アミノ酸側鎖の補正
Chain A : 82 : ARG は側鎖の延長線上に緑色の電子密度があります。緑色は「構造が置かれていない電子密度」なので、試しに側鎖を伸ばしてみたいと思います。
「Mutate & Auto Fit…」機能を利用することで簡単に側鎖を伸ばすことができました。

「Rotamers」機能を使うと側鎖の取りうるRotamerの候補が出てきますが、今回は「Mutate & Auto Fit …」で導入したconformerが電子密度と最も重なりが良さそうに見えます。
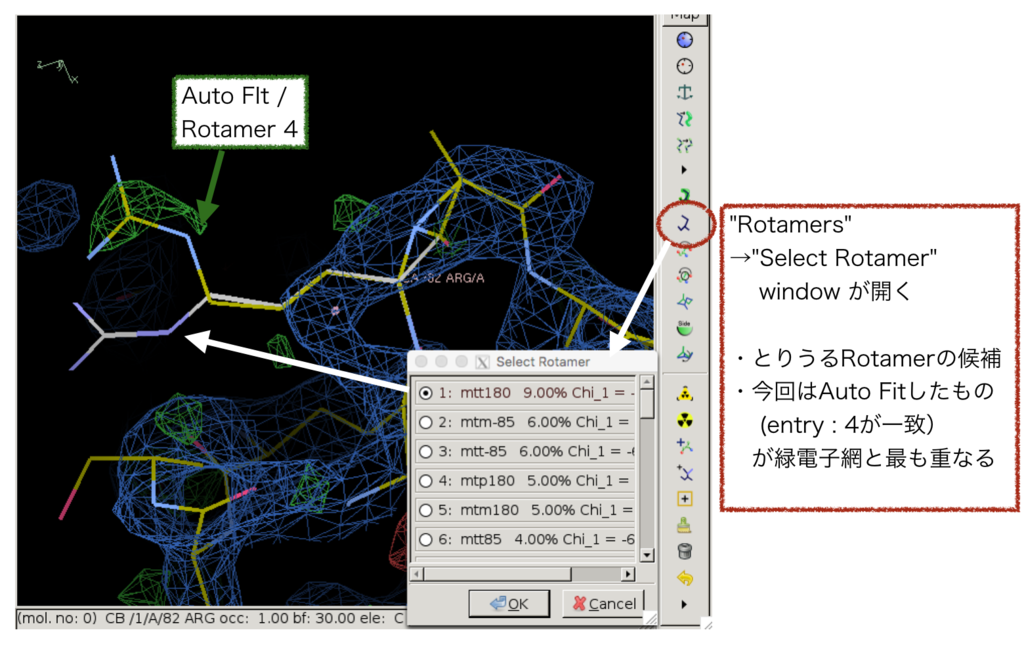
ARG 側鎖については問題ないように見えますが「Auto Fit」した主鎖はおかしな様子です。「Real Space Refine Zone」という機能をもちいると、該当の残基をクッリックしてドラッグすることで簡単に構造の修正を行うことができました。以下のような感じです。
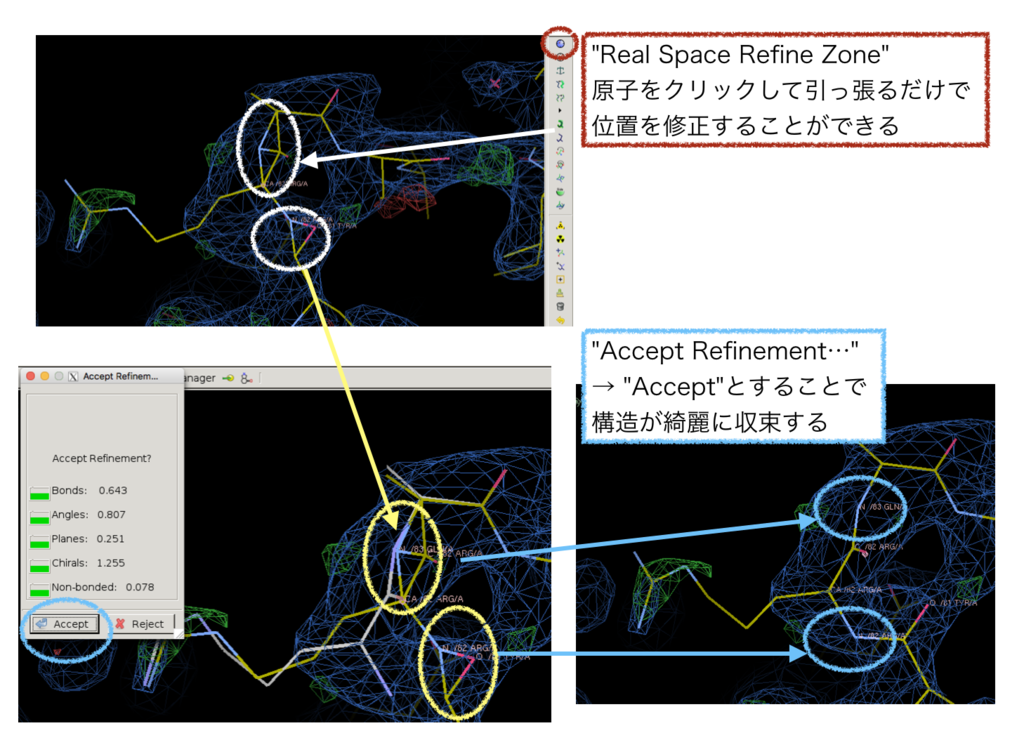
6. リガンド部位の確認
問題のリガンド部位ですが、残基番号 201 として登録されています。
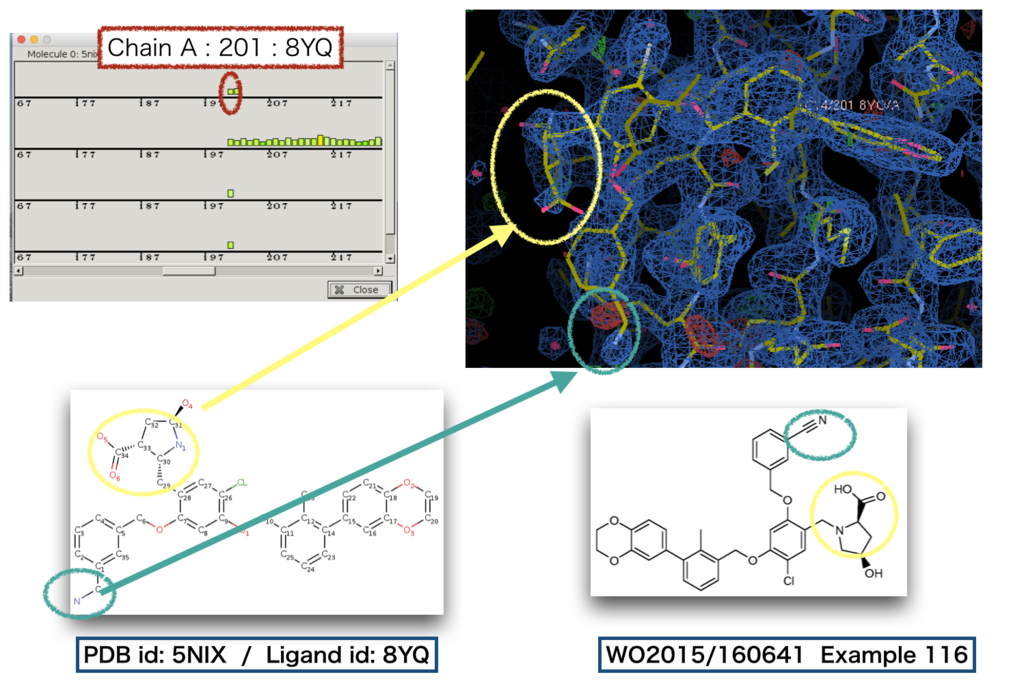
PDBに登録されている構造で元の文献と異なっている部位のうち
・ピロリジン部位(黄丸)は電子密度が薄い(poor)
・シアノ基(緑丸)は 赤い電子密度 とぶつかっています。(電子密度がないのに構造がモデル化されている)
と、なっています。どうやら、Fitさせた構造に無理がありそうです。
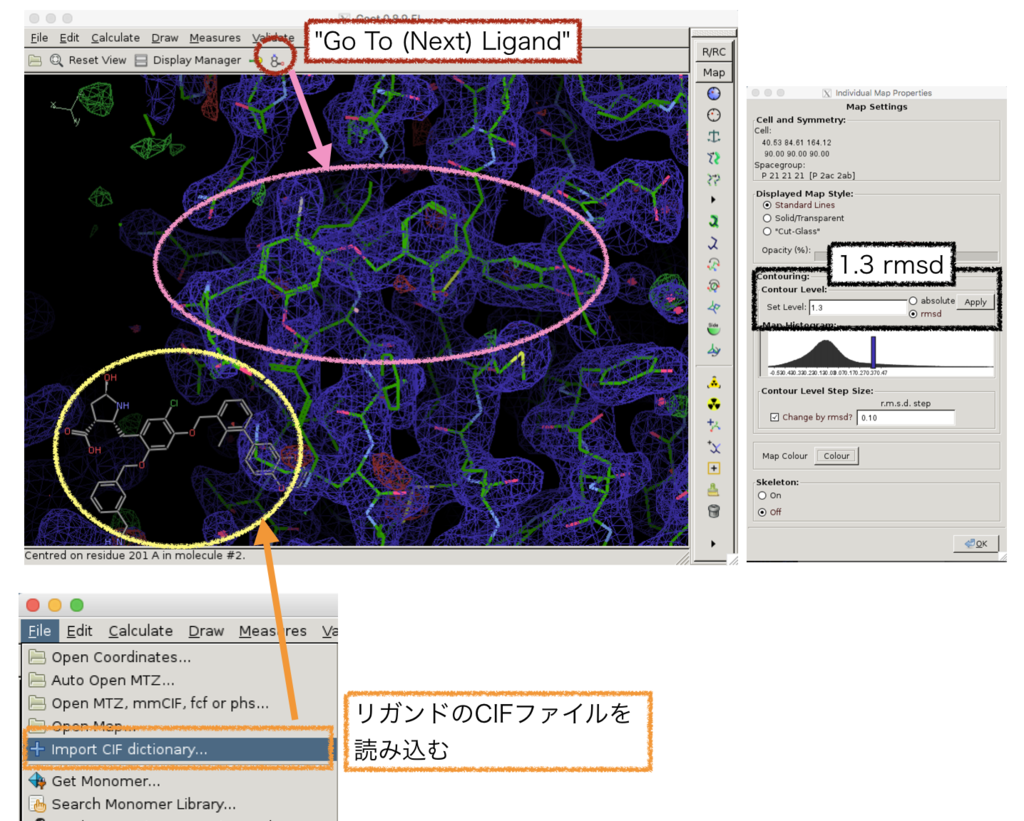
上記では残基番号からリガンドを辿りましたが、「Go To (Next) Ligand」機能を使用した方が、リガンドのズームが容易でした。
また、「import CIF dictionary…」からリガンドのCIFファイルを読み込むことで画面左下に2次元構造が表示されるようになりました。
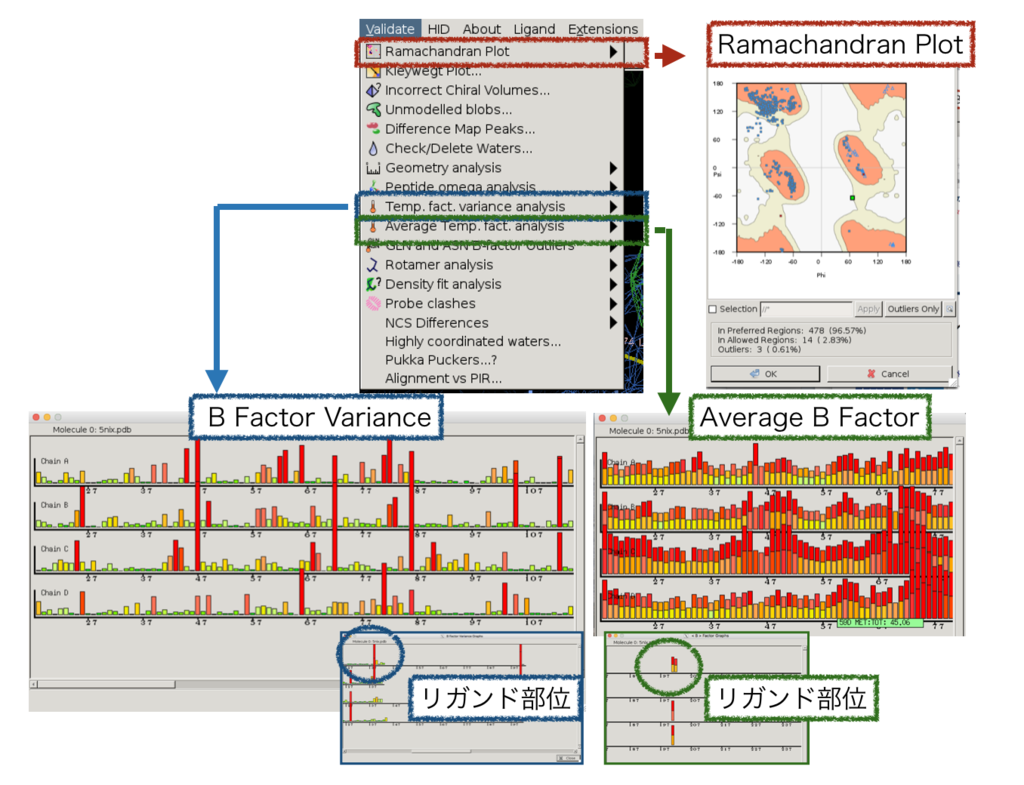
7. validationのグラフ
ちなみにその他のvalidation に関係しそうな指標は以下のように確認できます。
8. 別の構造でも確認(PDB id: 5NIU)
以上、構造式の誤っているPDB id : 5NIX の確認を行ってきましたが、同じ文献で報告されており別の構造 PDB id : 5NIUについても確認しておきたいと思います。
まずは2次元構造から・・・
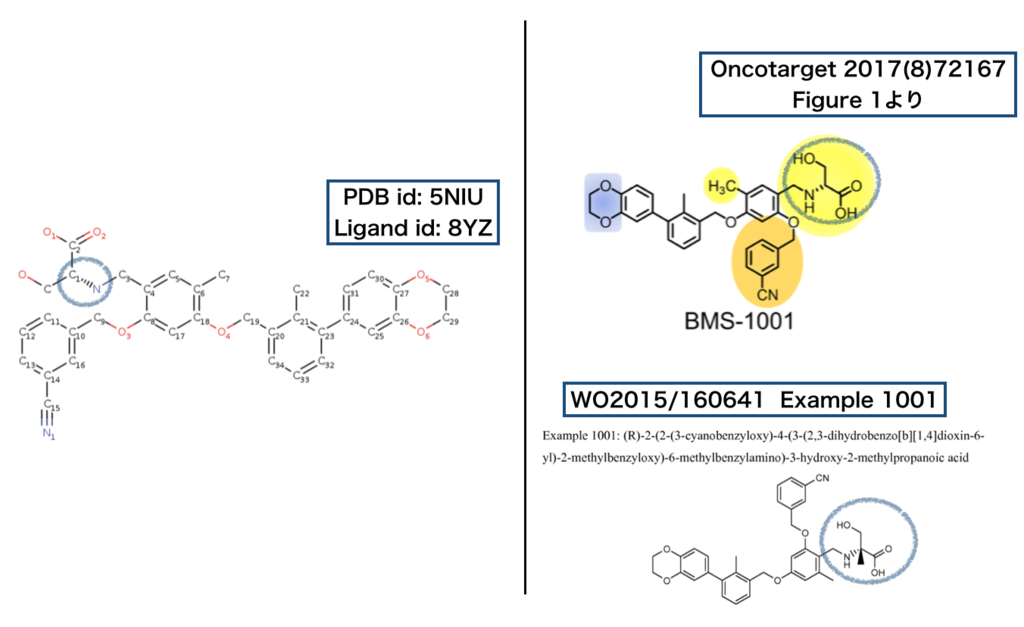
・・・今度は文献とPDBに登録された構造はあっていますが、さらに元となるBMSの特許とは構造が異なっており、メチル基が欠けています。
なんだか色々怪しくなってきました・・・
coot でリガンド部位を見てみましょう。
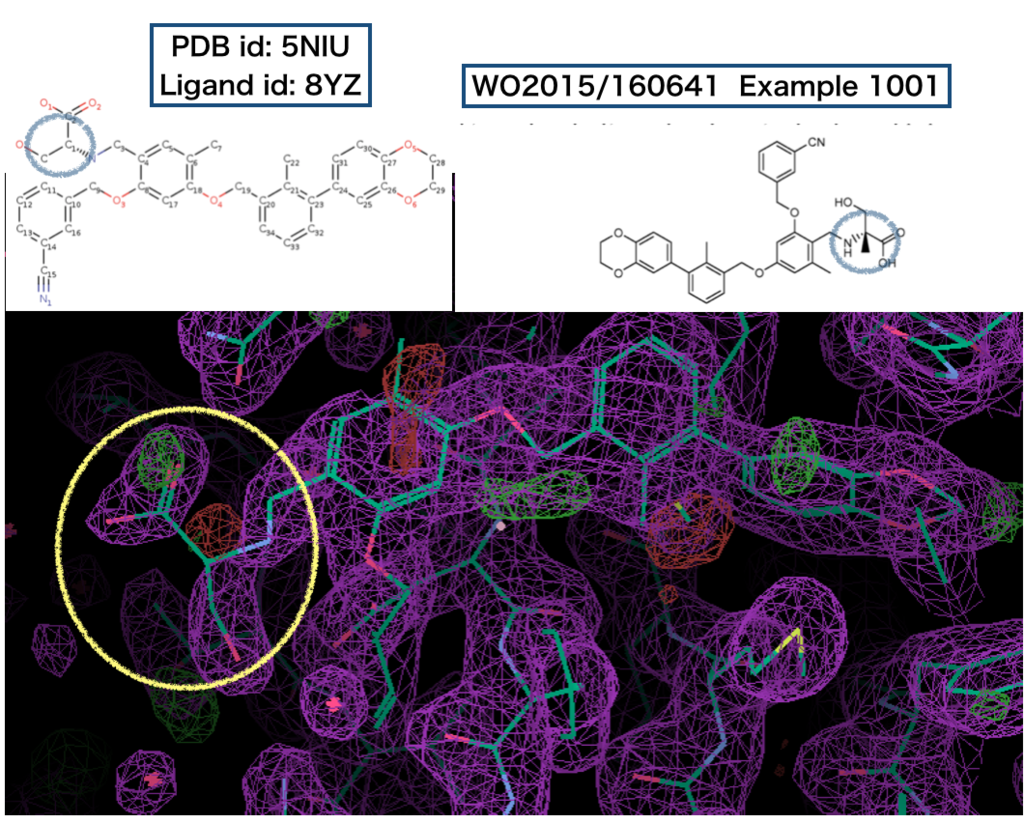
こちらもタンパク質の奥深くまで結合している部位は電子密度がはっきりしているように見えますが、溶媒に露出している部分は不明瞭になっているように見えます。
また、特許と構造の異なる部分(メチル基有無が異なる炭素)については特に電子密度がはっきりしていません。
先の構造で誤って登録されていた、シアノ基についてはどのようになっているでしょうか?
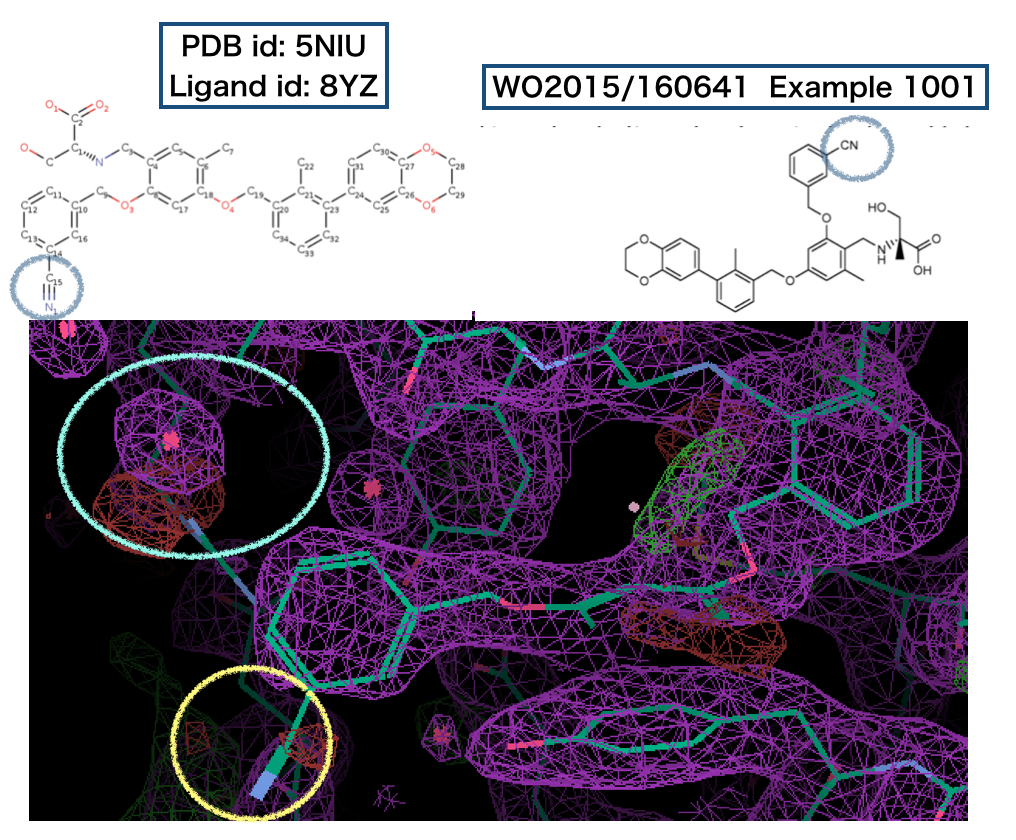
こちらは、シアノ基として登録されていますが少し曲がっており、赤い電子密度とも重なっているのが気になります。シアノ基の置かれた電子密度と対称の位置に、水(302 / HOH)が置かれた部位がありますが、こちら側にシアノ基が伸びていても良いようにみえます。
まとめ
以上、cootを利用して共結晶構造を眺めて見ました。今回取り上げた構造はどちらもそもそもの構造式に疑惑があり、適切な例ではなかったかもしれませんが、電子密度と比較してみることで構造の問題がよりクリアになったように思います。
結晶構造の解析等を実際に行ったことがないため、そもそも眺めている電子密度がどれほど質のものなのかもわかっておりません。誤り等あればご指摘いただければ幸いです。
あと、このページすごい・・・
*********************************************************************************************************
*1)
coot の tutorial
https://www2.mrc-lmb.cam.ac.uk/personal/pemsley/coot/web/tutorial/tutorial.pdf
"Features and Development of Coot" P Emsley, B Lohkamp, W Scott, and K Cowtan Acta Cryst. (2010). D66, 486-501 Acta Crystallographica Section D-Biological Crystallography 66: 486-501
*2)
使用にあたって参考にさせていただいたページ、ファイル
Refmac/cootでrefinement (finish編)
Introduction to ligand chemistry in protein X-ray structures
http://www.tsurumi.yokohama-cu.ac.jp/xtal-mls/coot/COOT.pdf
*3) MTZファイルについて
RCSB PDB: X-ray Electron Density Maps
*4) 共結晶構造の報告されている文献
Oncotarget. 2017; 8:72167-72181.