リガンドを重ね合わせてファーマコフォアを作る話
前回の記事でファーマコフォアスクリーニングを実施し、約2500個の化合物までリストを絞り込みました。思った以上に減らなかったので、さらなる絞り込みのため、ファーマコフォアを作成した流れを振り返りたいと思います。
- リガンド-タンパク共結晶構造を取得(PDB, 6構造)
- PLIPを使って相互作用を解析
- RDKitでPLIF作成
- 複数の構造で相互作用に関与する残基に着目
- 4.の残基と近接するリガンドの部分構造を選定(フィーチャー、3つ)
- 5.のフィーチャーの位置関係からファーマコフォアを作成
このうち、Step 5、Step 6の部分では、共結晶構造1つ(PDB id: 5NIX)をもとに作成をおこなっています。課題として「フィーチャー3つの選択基準がかなり恣意的だった」、ということが挙げられると思います。そこで今回はもう少し、根拠(?)をもってフィーチャーを選択できるか、残りの共結晶構造5つを眺めたいと思います。
1. 今回の流れ
といっても、ただ単に眺めるだけでは仕方ないので、今回は「複数の構造を重ね合わせて眺める」という点に主眼を置きたいと思います。
モチベーションは2つ・・・
- AI創薬のためのケモインフォマティクス入門 *1 6章、SBDDの項目で異なる化合物の複合体結晶構造の重ね合わせが紹介されていたこと
- ファーマコフォアについてのよもやま話 *2 において、結晶構造以外のアプローチとしてリガンド重ね合わせのアプローチが紹介されていたこと
です。折角、異なるリガンドを含む共結晶構造が複数あるのだから、タンパクごと重ね合わせて、そこに含まれているリガンドの重なりを比較してみれば、リガンドのみの重ね合わせよりもより実際の結合のポーズを意識したヒントが得られるかもしれません。
ということで、まずは
- Pymolで複合体構造をアラインメント
- アラインメント後のリガンドの座標を出力
をやってみます。
2. Pymolでアラインメント
用いる複合体構造を再掲します。①、②、③のグループは以前の記事でPLIFをベースに分けてみたものです。
| Group | PDB entry | Chain | リガンド | リガンドID | リガンド分子量 | 文献 |
|---|---|---|---|---|---|---|
| ① | 5J89 | A/B/C/D | BMS-202 | 6GX | 419.52 | Oncotarget 2016(7)30323 |
| 5N2D | A/B/C/D | BMS-37 | 8J8 | 448.55 | J. Med. Chem. 2017(60)5857 | |
| ② | 5J8O | A/B | BMS-8 | 6GZ | 494.42 | Oncotarget 2016(7)30323 |
| 5N2F | A/B | BMS-200 | 8HW | 497.49 | J. Med. Chem. 2017(60)5857 | |
| ③ | 5NIU | A/B/C/D | BMS-1001 | 8YZ | 594.65 | Oncotarget 2017(8)72167 |
| 5NIX | A/B/C/D | BMS-1166 | 8YQ | 643.13 | Oncotarget 2017(8)72167 |
pymolの設定などに関しては或る化みす途さんのブログ*3を参考にさせてきただきました。
具体的には、以下3ステップです。
- PDBファイル6つをpymolで読み込む
- chain C/Dを除いてA/Bのみにする
「remove (chain C,D)」と打ち込む - アラインメントをとる
例)「align 5j89, 5nix」
意) 5j89を5nixに重ね合わせる
そのままの重ね合わせではうまくいかなかったため、ステップ2(chain A/Bのみを残す)を行なっています。全て5nixに重ね合わせました。
次いで、下記の設定で描画を行いました。
- set cartoon_transparency, 0.8
- リガンドを中心に持ってくる(Command+左クリック)
- 水を消す(object panelのallでH(hide)からwatersを選択)
以下のような見た目となりました。比較は後回しにして、座標の出力へと進みます。

3. リガンドの座標を出力
ついで、重ね合わせからリガンドの座標のみを取り出します。以下のように一つずつpdbファイルで出力しました。
「aligned_5nix.pdb」という名前で、5nixのリガンドのみを含むpdbファイルが出力されました。
出力の方法に関してはPymolWikiの Save *4を参照しました。
- 「save file [,(selection) [,state [,format]] ]」 という形でつかう
- formatとしてpdb以外にもmol、sdfなど多数選択可能
- stateは「Default = -1」(現在の状態のみを保存)
stateに関しては、formatを指定する場合に省略すると、引数がおかしいと怒られたので、Defaultで良くても書き加える必要がありそうです。
出力するリガンドを選択する際に、pymolのwindow上でクリックすると、意図しない残基を選択してしまうことがあったので、「Sequence」でリガンドIDを選択した方がやりやすいと思います。

出力したpdbファイルを再度読み込んで確認します。
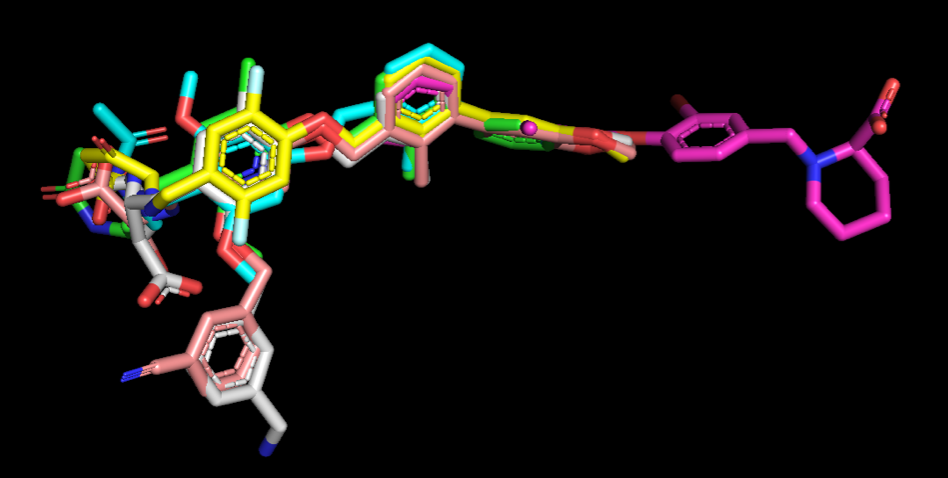
アラインメントの情報(変換された座標)を保ったまま、構造が出力されていそうです。
今度はまとめてsdfで出力して見ます。拡張子をファイル名に記載していれば自動でformatを認識して処理してくれる、ということだったので
「save aligned_ligands.sdf」
とだけ、打ち込みました。
ちなみに、pymol上で座標を確認するには目的の構造を選択した後、
- xyz = cmd.get_coords('sele', 1)
- print xyz
とすることで、各原子のxyz座標の3要素のリストを要素とする、リストのリストとして座標が出力されました。PyMolWikiの Get Coordinates I *5によると、他にも色々と方法があるそうです。
4. 重なりの比較
準備ができたのでリガンドの重なりの比較を行なっていきたいと思います。
4-1. 5J89と5N2D (グループ1)
5J89(リガンドID: 6GX)と 5N2D(リガンドID: 8J8)を表示させました。前回もちいたファーマコフォアの3点をメインとする構造のように見えます。

芳香族の重なりはかなり良いようですが、末端の親水性領域についてはズレが大きくなっています。この辺りは溶媒露出面にもなっていそうなので、許容される動きの幅が大きいのかもしれません。
・・・ということは、この親水性部位の座標を狭い範囲で厳格に決めすぎてファーマコフォアを作ることはよくない・・・、ということでしょうか?フィーチャー3点の一つとして3角形をつくる前にもう少し慎重になった方が良かったかもしれません。
4-2. 5N2D (グループ1) と5N2F (グループ2)
同一の論文で報告されている5N2D と 5N2F(リガンドID: 8HW)を表示させました。

5N2Dと5N2Fの大きな違いは、後者において図右端のフェニル環にさらに環構造が付加していることです。これにともない、タンパク側の chain A 残基56 Tyrの位置が大きく動いています。押しのけられた、、、という感じでしょうか?(Induced Fit?)
残念ながら構造が報告されている論文(J. Med. Chem. 2017(60)5857)*6は読めなかったのですが、オープンアクセスの Oncotarget 2017(8)72167で、構造の伸長に伴うTyrの動きが議論されていました。
4-3. 5J89(グループ1) と5J8O (グループ2)
もう一つのペア、5j89 と 5j8O(リガンドID: 6GZ)を表示させました。
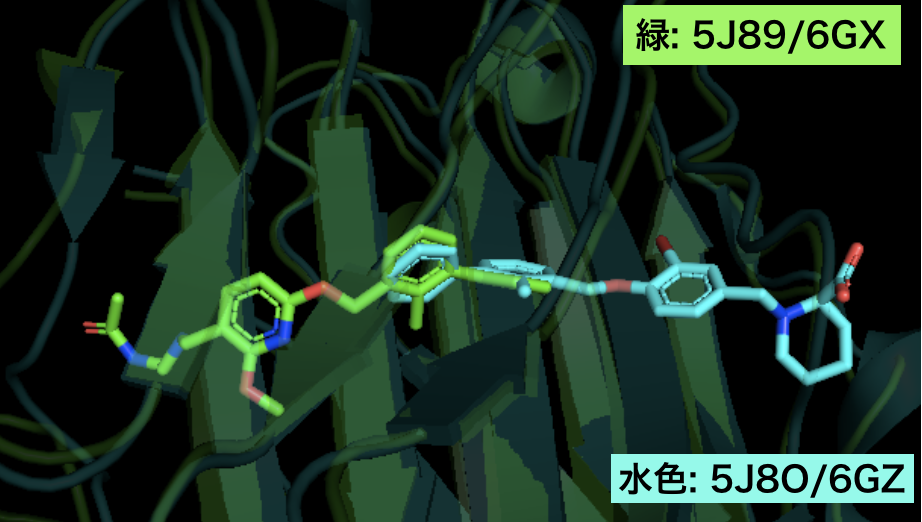
興味深いことに、このペアではビフェニル骨格で重なっているものの対称的な位置に広がっており、リガンド分子全体としての重なりはとても悪くなっています。リガンドベースではなく、タンパク質を基準としたアラインメントになっているため、このような結果になっていると思われます。共結晶構造がPD-L1の対称的な二量体となっていることを踏まえると、確かに対照的な位置に結合してもおかしくないという気もします。(結晶の群とか対称とかわかりません。すみません)
予期せぬ重ね合わせ構造でびっくりしましたが、これはこれで示唆に富む結果だと思います。なぜかというと、共闘用シェアデータに抜き出されている特許構造をながめても、最近のものに移るにつれて、中心に疎水性、両末端に親水性基をもつ対称性のある構造へと変化しているように見えるからです。(構造式は MarvinSketch 18.24.0で作成)

Twitterで拝見したDeNA 望月氏の講演資料「コンペティションから見るAI創薬」において、創薬レイドバトルに関して化合物の線対称に近い構造に着目しているとのコメントがありました(スライド p27)。*7 ご自身で記述子を開発されているとのこと、一体どんな工夫をされているのか?とても結果が楽しみです。
"コンペティションから見るAI創薬" というタイトルで DeNA の AIエンジニアが日本オミックス医学会シンポジウムに登壇しました。
— DeNA Tech (@DeNAxTech) 2019年3月20日
コンペという軸で、AI創薬の近年の動向について紹介しています!https://t.co/EQx48elkL6
対称性のある構造の医薬品
ちょっと脱線しますが、上述の対称性のある構造を見た際にC型肝炎治療薬を思い出しました。C型肝炎治療薬といえば、ハーボニー配合錠が薬価や偽造品流通などで話題となりました。こちらはNS5A阻害剤 レジパスビル と NS5B阻害剤 ソホスブビルを含む合剤となっています。 NS5A阻害剤は複数ありますが、例えば以下のように、分子量が大きく対称的な構造をとっている、という印象です。

経口薬というのが驚きの巨大分子ばかりです。
Wikipedia(英語)の記事「Discovery and development of NS5A inhibitors」に、BMSの研究者によるNS5A 結晶構造の論文(PDB id: 4CL1) (Protein Sci. 2014(23)723)が引用されていました。共結晶構造ではなかったのですが、
- 2量体の構造解析
- ドッキングシミュレーション
等の結果を示すFigureがありました(・・・詳しく読んでません。すみません)。
2量体の形成する疎水性の溝に、対称性の高いリガンド(Daclatasvir : DCV)が結合している図は、これまで見てきたPD-L1の共結晶構造を彷彿とさせるもので、ひょっとしてNS5B阻害剤、PD-L1にもヒットするのでは???という気もしてきます。・・・まあ、無理か。。。
当てずっぽうはさておき、LipinskiのRule of 5からは真っ先に弾かれてしまいそうなこれらの分子が経口薬として上市に至っている、という成功例がPD-1/PD-L1 結合阻害剤においても巨大化、対称化の流れを進める背景にもあったりするのかな、と想像してしまいます。創薬の現場からみたらどうなんでしょう???
4-4. 5J89(グループ1) と5NIU、5NIX (グループ3)
閑話休題。
残りの5NIU(リガンドID: 8YZ)、5NIX(リガンドID: 8YQ)を5J89と重ね合わせます。
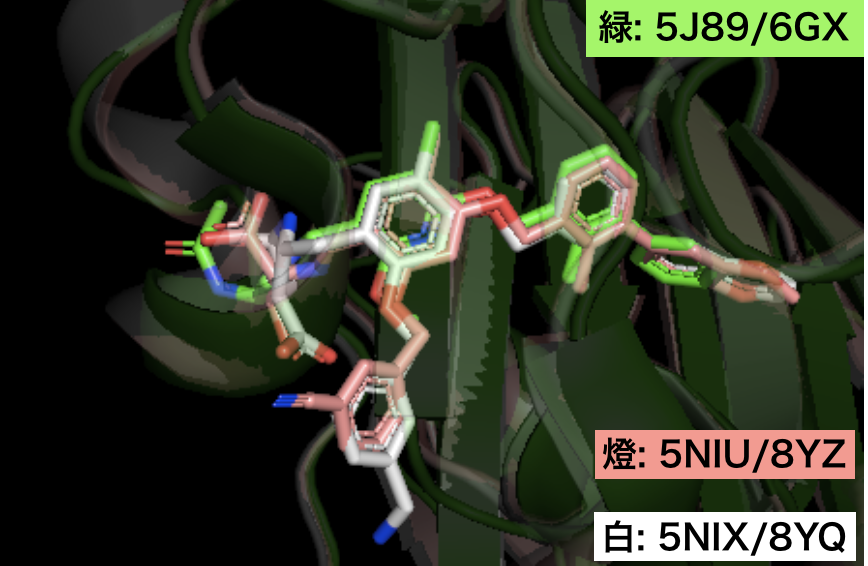
全体として重なりがよく、グループ3の大きな違いはグループ1、2と異なりベンジル基が左下方向にむかって付け足されていることです。文献(Oncotarget 2017(8)72167)*8中では、この置換基が新しい相互作用の獲得に寄与しているとの指摘もなされていました。
重ね合わせ結果の考察
以上、重ねあわせ構造を順番にながめてみました。6つの構造で、前回ファーマコフォアとして選択した3つのフィーチャーを満たしているようにみえ、とりあえずの選択としては十分要件を満たしていたのではないかな?という印象です。
・・・つまり、選択に根拠はあった!(・・・後付け)
共結晶構造中のリガンドをベースに考える場合、更なるポイントとして前回のファーマコフォアに加えて、
- 右末端フェニルからの置換基伸長(疎水性ポケット)
- 左下方向への芳香環伸長
- 左端、親水性基は座標の自由度がある
といった点を考慮していくと良いかもしれません。
5. ファーマコフォアを再作成
5-1. 今回の目標
観察をもとに、更なる絞り込みのためのファーマコフォアを作りたいと思います。
2.左下への芳香環伸長は新しいポケットを埋めるという意味で魅力的ですが、「共闘用シェアデータ」の活性化合物群の流れをみる限り、こちらのポケットは活用しない方向に流れがシフトしているように見えます。そこで、今回は1.右端疎水性ポケットと3. 親水性基の自由度を考慮したファーマコフォアを作成したいと思います。
5-2. Pymolで出力したSDFをRDKitで読み込む
まずは、Pymolで出力したファイルをRDkitで読み込めるか確認して見ます。
from rdkit import Chem aligned_lig_sup = Chem.SDMolSupplier('./aligned_ligands.sdf') aligned_ligs = [x for x in aligned_lig_sup if x is not None]
以下のようなエラーがたくさん出てきました。
RDKit ERROR: [20:29:00] ERROR: non-ring atom 14 marked aromatic RDKit ERROR: [21:10:06] non-ring atom 4 marked aromatic RDKit ERROR: [21:10:06] ERROR: Could not sanitize molecule ending on line 72
MarvinViewでは開き、構造を確認することができたので、どうもRDKitとpymolの相性が悪そうです。
RDKitのエラーメッセージによると、環に含まれていない原子(non-ring atom)に芳香族性が紐づけられていることが問題のようです。SDFの中身を色々とみた結果、エラーがでた構造ではすべてカルボキシ基を有しており、結合表(bond block)においてこの官能基を構成する原子間の結合タイプが「4 = aromatic」となっていることが原因(?)、という結論に達しました。*9
aromatic bondと紐づけられているためaromatic atomと認識された原子が、ring atomではないためエラーとなり、sanitizeもできなかったと思われます。(推定)
5-3. Pymolで出力したpdbをRDKitで読み込む
SDFの中身を修正するのは私には難しいので、PDBファイルとして出力した個々の構造の検証に移りたいと思います。
from rdkit.Chem import Draw pdb_5j8o = Chem.MolFromPDBFile('./aligned_ligands/aligned_5j8o.pdb') Draw.MolToImage(pdb_5j8o)

こちらは読み込み自体はうまくいきましたが、PDBフォーマットに結合次数が記載されていないため、全て単結合、カルボキシル基に至ってはジェミナルジオール構造のように認識されています。
RDKitメーリングリストのディスカッション *10を参考に結合次数を割り当てたいと思います。
手順としては
- smilesからMolオブジェクトを作成(テンプレート)
- AssignBondOrdersFromTemplate(refmol, mol)でテンプレートから次数を割り当て
となります。
from rdkit.Chem import AllChem smi_5j8o = 'Cc1c(COc2ccc(CN3CCCC[C@@H]3C(O)=O)cc2Br)cccc1-c1ccccc1' template_5j8o = Chem.MolFromSmiles(smi_5j8o) mol_5j8o = AllChem.AssignBondOrdersFromTemplate(template_5j8o, pdb_5j8o) Draw.MolToImage(mol_5j8o)

うまくいったようです。残りの5つも同様に処理します。
#pdb idをkey、smilesをvalueとする辞書を作成 smi_dict = {} smi_dict['5j89'] = 'COc1nc(OCc2cccc(c2C)-c2ccccc2)ccc1CNCCNC(C)=O' smi_dict['5n2d'] = 'COc1cc(OCc2cccc(c2C)-c2ccccc2)cc(OC)c1CNCCNC(C)=O' smi_dict['5n2f'] = 'Cc1c(COc2cc(F)c(CNCC(=O)CC(O)=O)cc2F)cccc1-c1ccc2OCCOc2c1' smi_dict['5niu'] = 'Cc1cc(CN[C@H](CO)C(O)=O)c(OCc2cccc(c2)C#N)cc1OCc1cccc(c1C)-c1ccc2OCCOc2c1' smi_dict['5nix'] = 'Cc1c(COc2cc(OCc3cccc(CN)c3)c(C[C@H]3N[C@H](O)C[C@H]3C(O)=O)cc2Cl)cccc1-c1ccc2OC=COc2c1' mol_5j8o.SetProp('PDB_id', '5j8o') #結合次数を割り当てたMolオブジェクトを格納するリストを作成 mols = [mol_5j8o] for k, v in smi_dict.items(): # aligned_xxxx.pdb という名前(xxxxはPDB id)のファイルから作成 path = './aligned_ligands/aligned_' + k + '.pdb' tmp = Chem.MolFromPDBFile(path) template = Chem.MolFromSmiles(v) mol = AllChem.AssignBondOrdersFromTemplate(template, tmp) # PDB idをプロパティとして持たせる mol.SetProp('PDB_id', k) mols.append(mol) #確認 Draw.MolsToGridImage(mols)

#SDFとして書き出しておきます writer=Chem.SDWriter('aligned_pdb_molecules.sdf') writer.SetProps(['PDB_id']) for mol in mols: writer.write(mol) writer.close()
5-4. 重ね合わせでフィーチャーを眺める
出力したSDFをつかって、pymol + RDKitでフィーチャーを描画して見ます。
import os from rdkit import RDConfig fdef = AllChem.BuildFeatureFactory(os.path.join(RDConfig.RDDataDir,'BaseFeatures.fdef')) from rdkit.Chem.Features import ShowFeats from rdkit.Chem import PyMol import subprocess from IPython import display showfeatpath = os.path.join(RDConfig.RDCodeDir, 'Chem/Features/ShowFeats.py') v = PyMol.MolViewer() v.DeleteAll() process = subprocess.Popen(['python', showfeatpath, '--writeFeats', 'aligned_pdb_molecules.sdf'], stdout=subprocess.PIPE) stdout = process.communicate()[0] png=v.GetPNG() display.display(png)

この構造をもとに、5-1. 今回のファーマコフォアの狙いと合わせると、下図のようなフィーチャーの選択が考えられそうです。

"Aromatic" 2つと "Acceptor" 1つの3点という点は以前と同じですが、選択する場所を変えています。
- 右側疎水性ポケットを意識した"Aromatic"の選択
- 複数のリガンドの親水性基が集まる場所を意識した"Acceptor"の選択
としています。
Acceptorの位置を決めるにあたっては、複数のリガンドの親水性基の中心にPseudoatomを作成することとしました。具体的には各リガンドで、該当部位の近辺の親水性基をなす原子を選択し、すべて選んだあとで「pseudoatom hyd, sele」とコマンドを打ち込みました。
Aromatic 2点についてもそれぞれ中心にPseudoatomを作成し、meshで表示すると以下の図のようになりました。

「Wizard → Measurement」で測定した距離と、別に取得した座標情報「例) xyz = cmd.get_coords('hyd', 1)」とを合わせてまとめると以下のようになりました。

なんだか格好良くなってきました!それっぽいぞ!(意味不明)
この情報を使って、再度ファーマコフォアサーチを行います。
5-5. ファーマコフォアを作成
前回と設定値以外変わってませんが念のため・・・
from rdkit.Chem import ChemicalFeatures from rdkit.Chem.Pharm3D import Pharmacophore from rdkit import Geometry feat_1=ChemicalFeatures.FreeChemicalFeature('Aromatic',Geometry.Point3D(-11.796, 10.999, -22.536)) feat_2=ChemicalFeatures.FreeChemicalFeature('Aromatic',Geometry.Point3D(-7.625, 10.407, -21.653)) feat_3=ChemicalFeatures.FreeChemicalFeature('Acceptor',Geometry.Point3D(1.222, 9.145, -28.891)) feats = [feat_1,feat_2,feat_3] pcophore = Pharmacophore.Pharmacophore(feats) # ファーマコフォアの設定 d_upper = 1.5 # 特性基間の距離の許容範囲(上限値) d_lower = 0.5 # 特性基間の距離の許容範囲(下限値) # feat_1とfeat_2の距離 4.3Åを基準に、下限と上限を設定 pcophore.setLowerBound(0,1, 4.3-d_lower) pcophore.setUpperBound(0,1, 4.3+d_upper) # feat_2とfeat_3の距離 11.5Åを基準に、下限と上限を設定 pcophore.setLowerBound(1,2, 11.5-d_lower) pcophore.setUpperBound(1,2, 11.5+d_upper) # feat_1とfeat_3の距離 14.6Åを基準に、下限と上限を設定 pcophore.setLowerBound(0,2, 14.6-d_lower) pcophore.setUpperBound(0,2, 14.6+d_upper) print(pcophore) """ Aromatic Aromatic Acceptor Aromatic 0.000 5.800 16.100 Aromatic 3.800 0.000 13.000 Acceptor 14.100 11.000 0.000 """
新しいファーマコフォアの準備ができました。 関数は前回作成したPSearch3というものを用います。
6. ファーマコフォアサーチ
6-1. サーチ
前回、ファーマコフォアサーチを実施したSDFファイルを読み込みます。
PSerched_suppl = Chem.SDMolSupplier('./PSearched.sdf') PSerched_mols = [x for x in PSerched_suppl if x is not None] print(len(PSerched_mols )) #4220
Embedできた数をPScoreというプロパティに格納しているので、こちらの値が1以上のものを、ファーマコフォアにマッチしたもの(P_Matched)、0のものをマッチしなかったもの(P_MissMatched)として別々のリストに分けます。
P_Matched = [] P_MissMatched = [] for x in PSerched_mols : PScore = int(x.GetProp('PScore')) if PScore >= 1: P_Matched.append(x) else: P_MissMatched.append(x) print('P_Matched:', len(P_Matched)) #2463 print('P_MissMatched:', len(P_MissMatched)) #1757
前回のファーマコフォアにマッチした約2500個について、今回新しく作成したファーマコフォアスクリーニングを適用します。
for x in P_Matched: num_embeds = PSearch3(x) x.SetIntProp('PScore2', num_embeds)
マッチしたものを取り出します。(上と同じなので省略)
#取り出したリストをP_Matched_2、P_MissMatched_2としている。 print('P_Matched:', len(P_Matched_2)) #997 print('P_MissMatched:', len(P_MissMatched_2)) #1466
前回と今回のファーマコフォア両者を満たすものは約1000個でした。 「P_Matched2.sdf」という名前でSDFを出力しました。
6-2. 確認
PandasのDataFrameで読み込んで見ます。
from rdkit.Chem import PandasTools import pandas as pd PSearched2_df = PandasTools.LoadSDF('./P_Matched2.sdf')
今回のスクリーニング(PScore2)の値の分布を取得して見ます。
print(PSearched2_df['PScore2'].describe()) """ count 997 unique 16 top 1 freq 254 Name: PScore2, dtype: object """
最大値や最小値が返ってくるのを期待していたのですが、どうも様子がおかしいです。データ型を確認して見ます。
PSearched2_df.dtypes """ ID object MW object MolLogP object NumHAcceptors object NumHDonors object NumRotatableBonds object PScore object PScore2 object ROMol object TPSA object original_id object dtype: object """
PandasToolsで読み込んだものは全てobject型になっています。こちらの記事「pandasのデータ型dtype一覧とastypeによる変換*11」によると、要素に文字列を含むDataFrameの列はobject型となるらしく、また、化学の新しいカタチさんの記事RDKitのPandasToolsでデータ分析を加速する*12にもPandasToolsで読み込んだ場合は「プロパティが全て「文字列」として認識」されると記載されていました。
astypeで整数型(int)に変換します。
PSearched2_df = PSearched2_df.astype({'PScore': int, 'PScore2': int})
PSearched2_df.describe()
"""
PScore PScore2
count 997.000000 997.000000
mean 14.343029 3.289870
std 12.517565 2.394612
min 1.000000 1.000000
25% 5.000000 1.000000
50% 10.000000 3.000000
75% 20.000000 4.000000
max 99.000000 31.000000
"""
無事出力できました。それぞれの最大値の構造を確認して見ます。
#最大値のindexを取得 PScore_max = PSearched2_df['PScore'].idxmax() PScore2_max = PSearched2_df['PScore2'].idxmax() #Molオブジェクトを取り出し max_mol = PSearched2_df.loc[PScore_max, 'ROMol'] max_mol2 = PSearched2_df.loc[PScore2_max, 'ROMol'] #スコアを構造とともにlegendとして描画する準備 legends = [] for x in [PScore_max, PScore2_max]: score1 = str(PSearched2_df.loc[x, 'PScore']) score2 = str(PSearched2_df.loc[x, 'PScore2']) legend = 'PScore:' + score1 + '/ PScore2:' + score2 legends.append(legend) Draw.MolsToGridImage([max_mol, max_mol2], legends = legends)

前回と今回で同じものがEmbed数最大となっていたようです。
7. まとめ
今回は、より良いファーマコフォアの作成を目指して複数の共結晶構造を重ねあわせる、というステップを加えて見ました。タンパク質をあつかうにはPyMolが非常に使いやすく、Wikiの充実しているので少しずつ機能がわかってきました。
今回のファーマコフォアマッチングの課題としてはEmbedの数しか考慮しておらず、リガンドの3次元構造を実際に発生、最適化させることまでできていないことです。3次元構造発生 → ドッキング みたいな感じに持っていけると良いのですが、、、進捗が悪い。。。
行ったり来たりあちらこちらで躓いていたらとても冗長になってしまいました。おかしな点が多数あると思いますのでご指摘いただければ幸いです。
*1:AI創薬のためのケモインフォマティクス入門 https://github.com/Mishima-syk/py4chemoinformatics/blob/master/pdf/py4chemoinformatics.pdf
*2:ファーマコフォアについてのよもやま話 http://blog.kzfmix.com/entry/1552452149
*3: 話題のXofluzaの変異をPymolで見てみる https://keetaneblog.hatenablog.com/entry/2019/02/08/210016
*4:PymolWiki Save https://pymolwiki.org/index.php/Save
*5:PyMolWiki Get Coordinates I https://pymolwiki.org/index.php/Get_Coordinates_I
*6:https://pubs.acs.org/doi/abs/10.1021/acs.jmedchem.7b00293
*7:コンペティションから見るAI創薬 https://speakerdeck.com/m_mochizuki/ai-drug-discovery-in-the-view-of-competitions
*8:Oncotarget 2017(8)72167 http://www.oncotarget.com/index.php?journal=oncotarget&page=article&op=view&path%5B%5D=20050
*9:化学構造の表記 http://www.metabolomics.jp/wiki/Aritalab:Lecture/Compchem/MOL
*10: https://sourceforge.net/p/rdkit/mailman/message/31844451/
*11:pandasのデータ型dtype一覧とastypeによる変換 https://note.nkmk.me/python-pandas-dtype-astype/
*12: RDKitのPandasToolsでデータ分析を加速する https://future-chem.com/rdkit-pandastools/