HELMで核酸医薬をお絵描き
前2回の記事でマクロ分子の線形表現HELMについて概要と利用ツールをみてきました。
「いろいろなモダリティの表現に良さそう!!」ということですが、実際どんな感じになるのでしょうか??
今回は核酸医薬の具体例をお絵描きしていきたいと思います。
核酸医薬の概要
といっても私は核酸医薬をよくわかっていません。
国立医薬品食品衛生研究所 遺伝子医薬部 井上貴雄先生の以下2件の解説に特徴と現状がまとまっており非常にわかりやすかったです。(いずれもオープンアクセス)
- 文献1) 井上貴雄・佐々木澄美・吉田徳幸 核酸医薬開発の現状と今後の展望 Drug Delivery System 2019(34)86-98
- 文献2) 井上貴雄 核酸医薬 〜低分子、抗体に続く第3のモダリティ〜 SAR News No.38 (Apr 2020) 1-8
以下、引用させていただきつつ、基本的な内容を辿りたいと思います。
長くなってしまったのでご存知の方は飛ばしてください。
核酸医薬の特徴
まず、核酸医薬品とは一般に以下のような医薬品を指します。
上記の特徴は、核酸医薬と遺伝子治療用製品との違いを踏まえると納得しやすいかもしれません。(後者は翻訳されたタンパク質が作用(②)し、生物学的に製造される(③))
さて、従来の医薬品(低分子、抗体)と比較した核酸医薬の特徴としては、以下3点が挙げられています。
加えて、新規モダリティとして核酸医薬には従来の低分子と抗体の利点を併せ持つことが期待されています。具体的には以下2点。
- 抗体同様、高い特異性と有効性をもつ
- 低分子同様、化学合成で製造可能できる
(2.の利点はより具体的には低コスト、高均質性でしょうか??)
また、核酸医薬は一度開発スキームができれば、異なる創薬標的に対しても迅速に開発可能なことが期待されます。理由は以下3点。
核酸医薬の課題
このように魅力的な特徴のある核酸医薬品ですが、薬効本体であるオリゴ核酸に由来する、生体内での安定性・有効性という課題がありました。
これらの課題に対して修飾核酸技術や薬物送達技術(DDS)といった技術発展により改善が見られ、 現在の全身投与でも高い効果を発揮する候補品の開発に至っている、とのことです。
核酸医薬の薬物送達技術
核酸医薬の実用化に寄与した技術のうち薬物送達技術にはどのようなものがあるでしょうか?
臨床応用されているデリバリー技術は大きく①Lipid Based Nanoparticle、②Polymer Based Nanoparticle、③Conjugated Delivery Sytemに分かれるそうです。 *1
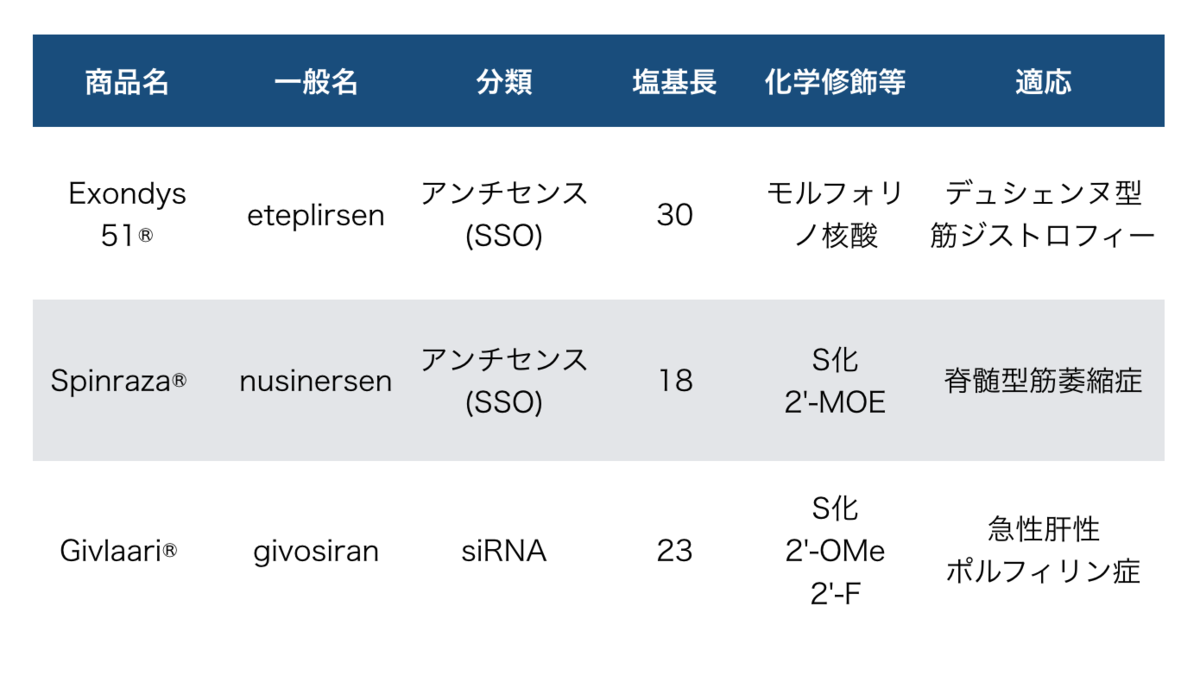
文献2 表1にはPEG、GalNac、LNPという例が記載されています。*2
| 薬物送達技術 | 商品名 | 一般名 | 分類 | 適応 | 投与 |
|---|---|---|---|---|---|
| PEG | Macugen®︎ | pegaptanib | アプタマー | 滲出型加齢黄斑変性症 | 硝子体内投与 |
| GalNAc | Givlaari®︎ | givosiran | siRNA | 急性肝性プリフィリン症 | 皮下投与 |
| LNP | Onpattro®︎ | patisiran | siRNA | 遺伝性ATTRアミロイドーシス | 静脈内投与 |
前2例(PEG、GalNac)は核酸そのものに薬物送達のためのユニットを直接(共有)結合させるもので、 後1例(LNP)は脂質ナノ粒子(Lipid Nano Particle)という粒子に核酸を内包させるDDS技術です。
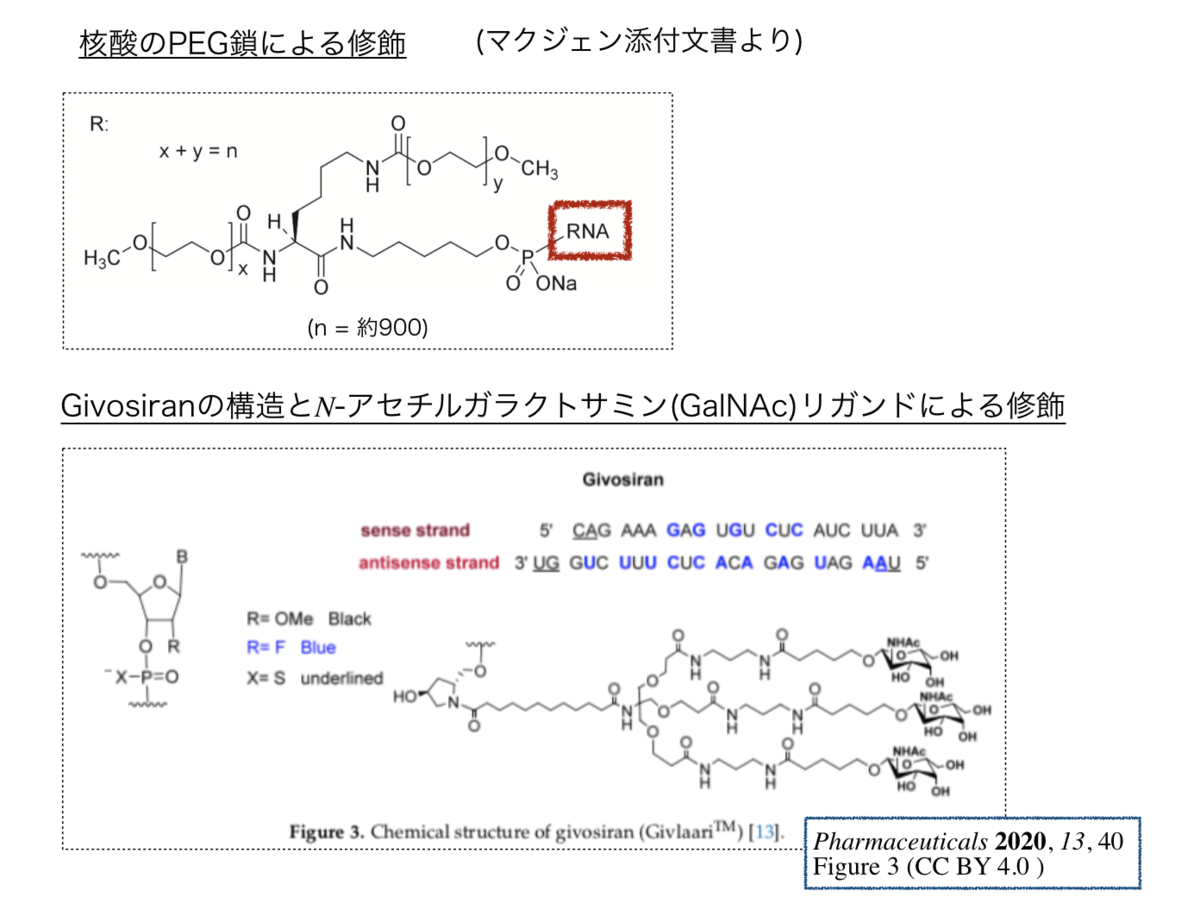
3剤の内、全身投与という観点ではgivosiran、patisiranが当てはまります。 これら2剤はどちらもAlnylam社によるもので標的臓器は肝臓となっています。
例えばgivosiranの場合、GalNAcリガンドは上図のような構造となっているそうです。*3
末端に結合するN-アセチルガラクトサミン(GalNAc)は肝実質細胞の細胞表面に発現するアシアロ糖タンパク質(asialoglycoprotein receptor, ASGPR)に認識され結合します。 そして、受容体介在型エンドサイトーシス(receptor-mediated endocytosis)によって取り込まれることで、細胞内において核酸の効果が発揮されるとのことです。 *4
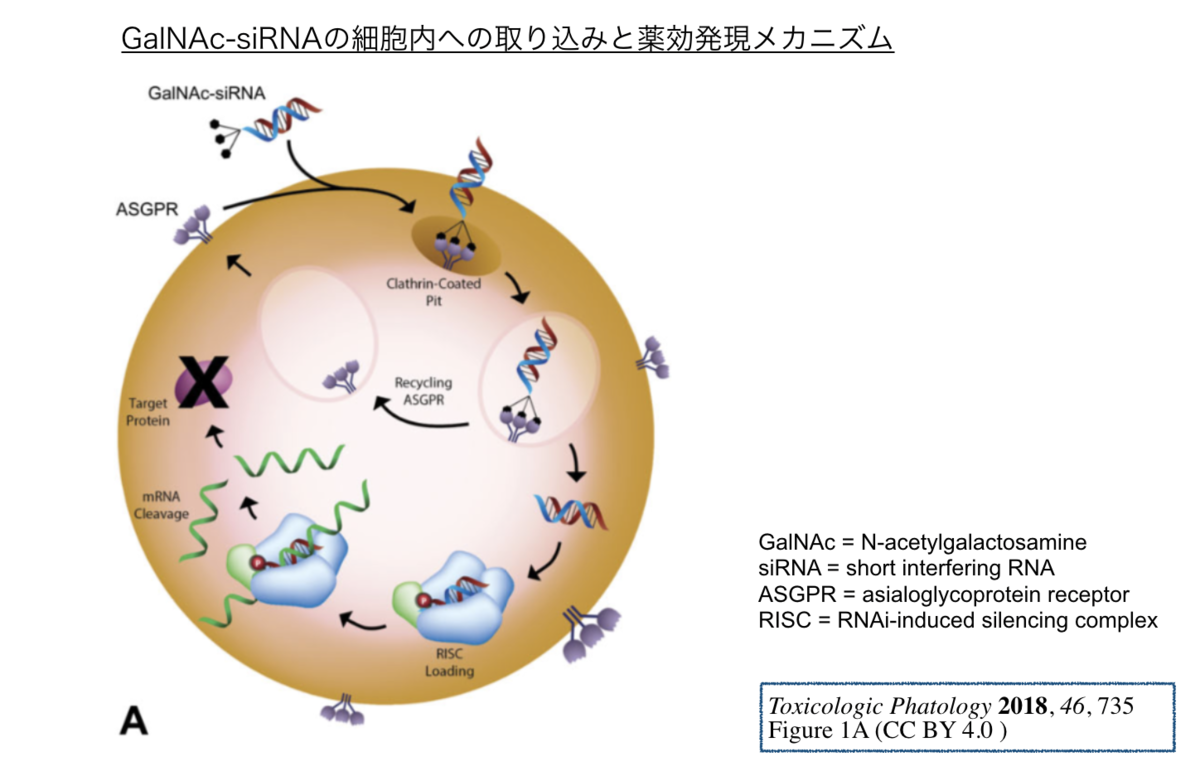
なぜhydroxyproline誘導体をかませているのか?炭素鎖の長さの影響は?GalNAcを3つぶら下げてるのは?などなど色々と興味深いところです。
脂質ナノ粒子
全身投与のもう一方patisiranは脂質ナノ粒子(LNP)によって体内動態がコントロールされています。
Alnylam社のOnpattro製品解説ページの図が非常にわかりやすいですが、 とても興味深い輸送メカニズムとなっています。
標的臓器、肝臓に到達するポイントは2点。
- 肝臓が大量の血液が潅流する臓器であること
- ApoE依存的機構
①は静脈内投与なので血液が集まりやすい肝臓に行きやすくなるのはなんとなくわかります。
面白いのは②で、肝臓分布過程でLNPに内因性アポリポタンパク質E(ApoE)が結合するそうです。 肝細胞表面の低比重リポタンパク質受容体(LDLR)などのApoE結合受容体を介して細胞内に取り込まれる、とのこと。
Alnylam社の2010年の文献ではapoE-/-マウス、LDLR-/-マウスを用いた解析が行われています。*5
中性リポソーム(neutral liposome)にアポリポタンパク質が吸着することが一般に知られていて、 その中でアポリポタンパク質Eだけが肝細胞への取り込みを促進できるから、ということに基づく研究のようです。
・・・すごい。
中性と書きましたが、ここも大事なポイントです。
実験室レベルでは核酸送達のキャリアとしてカチオン性キャリアがよく用いられるそうです。*6
カチオン性を用いることの利点は2つ
一方で核酸医薬のDDSに応用するにあたって困難な問題点も指摘されています。
- 細胞毒性:細胞内タンパク質との非特異的吸着による阻害
- キャリア正電荷により核酸が細胞内でもリリースされづらい
- 生体内組織への非特異的吸着により初回通過臓器(肝臓、肺)に捕捉されやすい
- in vivoで血中の細胞、タンパク質との非特異的結合により凝集、不活化、排出される
そこで臨床応用にあたっては非カチオン性キャリアが重要になってきます。 特にLNPでは非カチオン性脂質とpH感受性カチオン性脂質のハイブリッドといったアプローチがとられています。
このpH感受性というのがとても面白いポイントで、安全性や核酸輸送にとって以下のような利点があるそうです。*7
- 血中pH7.4ではほとんどイオン化しない(中性)ので、細胞障害性が低い
- 細胞内に取り込まれたのち、エンドソーム・リソソーム内の弱酸性pHでイオン化
- イオン化するとエンドソーム・リソソーム膜と融合し、内包核酸を細胞質に効率よく放出
patisiranで用いられているpH感受性カチオン性脂質はDLin-MC3-DMAというもので、pKa6.5前後、3級アミンを有する構造です。
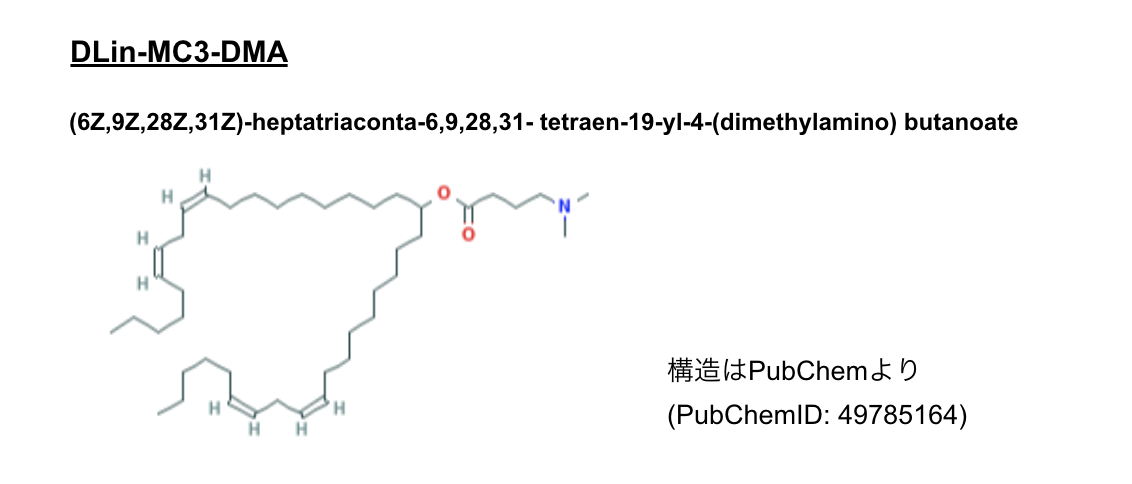
このLNP技術は元々Pieter Cullis 教授らにより2010年 Nature Biotechnolgyに報告された下記文献が鍵となっているようです。*8 LNPの要素として1,2-dilinoleyloxy-3-dimethylaminopropane (DLinDMA)構造に基づく脂質のスクリーニングを行なっています。(読めなかった)
さらに2012年、Angewante Chemie Internal Editionに報告された文献では、 種々の親水性head groupを有する脂質を用いたpKaとin vivoでの効果の相関を調べており、 pKa 6.2-6.5が最適な範囲であるとしています。(オープンアクセス。Abstractの図がとても印象的です。)*9
Jayaraman M. et. al., Maximizing the potency of siRNA lipid nanoparticles for hepatic gene silencing in vivo. Angew. Chem. Int. Ed. Engl. 2012;51:8529–8533. https://onlinelibrary.wiley.com/doi/full/10.1002/anie.201203263
こういう化学構造のデザインで好ましい性質を達成していくのとても格好いいですよね。*10
修飾核酸
こちらは核酸医薬自体の安定性に関与し、具体的には生体内に存在する核酸分解酵素(ヌクレアーゼ)等による分解への抵抗性や、 標的との結合安定性の強化といった効果が期待されます。
文献2 SAR News No.38 の図2がわかりやすので以下に引用させていただきます。
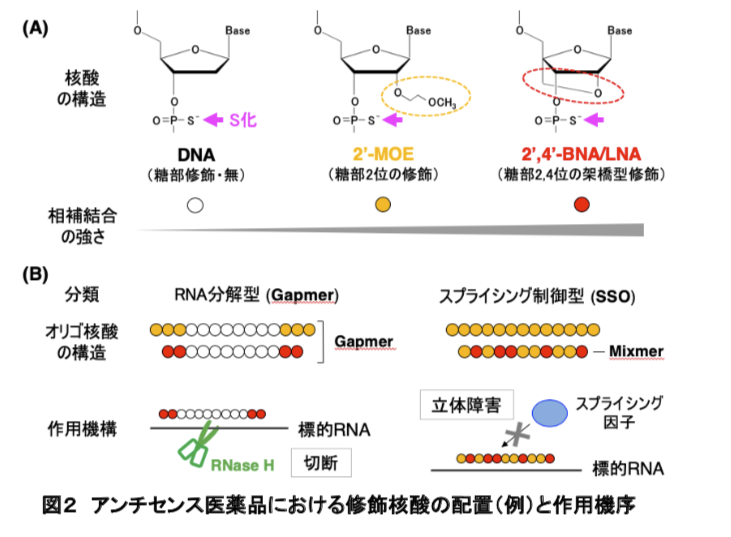
まずリン酸部位の修飾です。ここには酸素原子を硫黄原子に置換したホスホロチオアート修飾(S化)がよく用いられます(S化オリゴ核酸、Sオリゴ)。
これにより以下の利点が期待されます。
- タンパク質との結合の向上、脂溶性の増加により細胞膜上のタンパク質を介した細胞親和性、膜透過性が向上
- 血中タンパク質との結合による血中滞留性の増加
- ヌクレアーゼ耐性の付与による整体安定性の改善
化学的な注意点として、リン原子上に不斉点が発生するためSオリゴは立体異性体混合物になってしまいます。
この立体配置を厳密に制御する技術を持った企業もあるそうです(Wave Life Sciences)。
文献*11によるとリン原子に不斉補助基を結合させたモノマーを使って、伸長・キラルS化を行なっているようです。*12
・・・すごい。
続いて糖部の修飾です。これによりRNAとの結合力の強化が期待され、2'位の修飾と架橋型修飾が例としてあげられています。
2'位の修飾としては上図に引用した2'-MOE以外にもフッ素化体(2'-F)、メトキシ体(2'-OMe)などがあります。
架橋型は糖部の2'と4'を架橋することでゆらぎのある糖部の立体配座を固定化します。これにより以下が期待できます。
- 相補鎖との結合力の向上
- 立体障害によるヌクレアーゼ耐性
他にも糖部修飾そのものではありませんが糖部にモルフォリノ環を用いたモルフォリノ核酸が知られており、
- ヌクレアーゼで分解されない
- 毒性が低い
といった利点があります。
以上が、修飾核酸の概要でした。 こういった様々な非天然のユニットが活用されていることが、HELMにおけるモノマーの取り扱いの柔軟さとどのように結びつくか、気になるところです。
核酸医薬の分類
核酸医薬の構造的特徴をざっとみたので、最後に分類についても触れておきます。
文献2 SAR News No.38 の表2がわかり易いので引用させていただきます。

この分類は「RNAを標的とするか、タンパク質を標的とするか」でとらえるとわかり易い、とのこと。
表2の左3つ(アンチセンス、siRNA、miRNA)がRNAを標的、右3つ(デコイ、アプタマー、CpGオリゴ)がタンパク質標的に相当します。
RNAを標的とする中でもアンチセンスは病因となるタンパク質を減少させたり、機能的なタンパク質を増加させたりと様々な作用機序があるそうです。
ちなみに薬物送達技術のところで取り上げた3剤はそれぞれ、① Macugen®︎(pegaptanib) アプタマー、② Givlaari®︎(givosiran) siRNA、③ Onpattro®︎(patisiran) siRNA となっていました。
分類は以上です。
以上、核酸医薬の基本的な事項について辿ってみました。
面白い話題が多くてどんどん脱線してしまいましたが、構造の複雑さと背景を知るのが表現方法を考える上で大事ということで。。。
HELM表現と他の表現の比較
では漸くですがHELM表記を試してみましょう。
文献2 SAR News No.38 の表1に記載されている中から化学修飾の異なる以下の3つを試してみたいと思います。
Exsondys®︎ (eteplirsen)
まずはSarepta TherapeuticsのExsondys®︎ (eteplirsen)から。
Drugs@FDAで閲覧できるLabel(NDA 206488)記載の構造はこんな感じです。
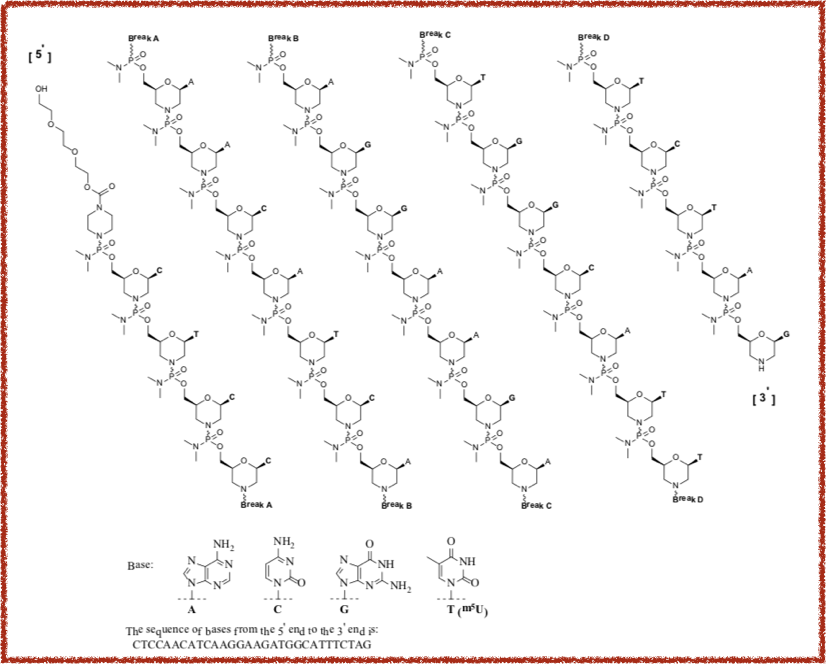
えらいごっついですわ。。。
表現の比較対象としてIUPHAR/BPS Guide to Pharamacologyの2D表現を引用してみます。 Guide to Pharmacologyは専門家によりキュレーションされたリガンド-活性-標的関係についてのデータベースで、CC BY 4.0で利用可能です。
Guide to Pharamacology; eteplirsenの核酸配列の2D表現はこんな感じ。
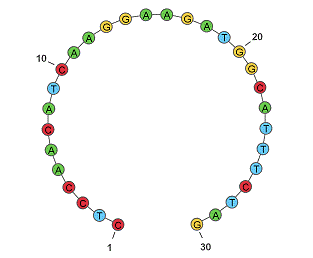
配列はわかりやすいですが、この表現ではモルフォリノ核酸であることやリン酸の修飾といった情報が表現できていないようです。
ではHELM editorをつかって構造を描いていきましょう。
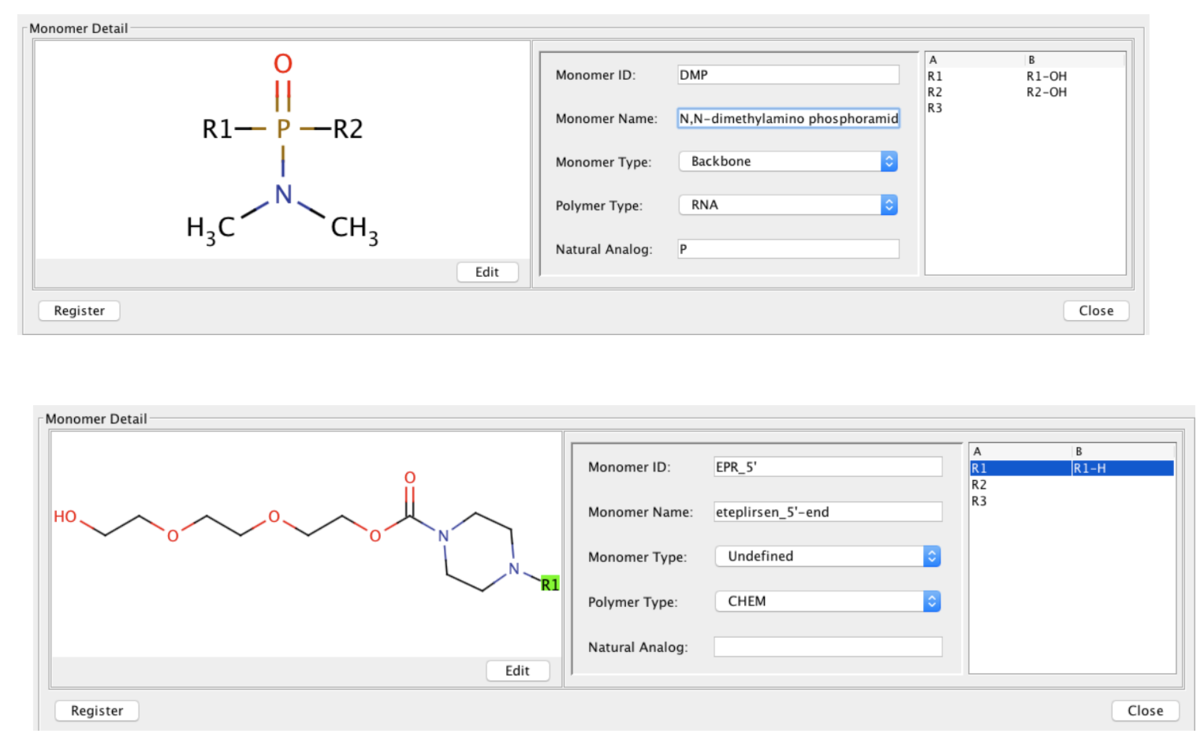
まずはmonomerの確認してみます。モルフォリノ核酸糖部はあるようです。
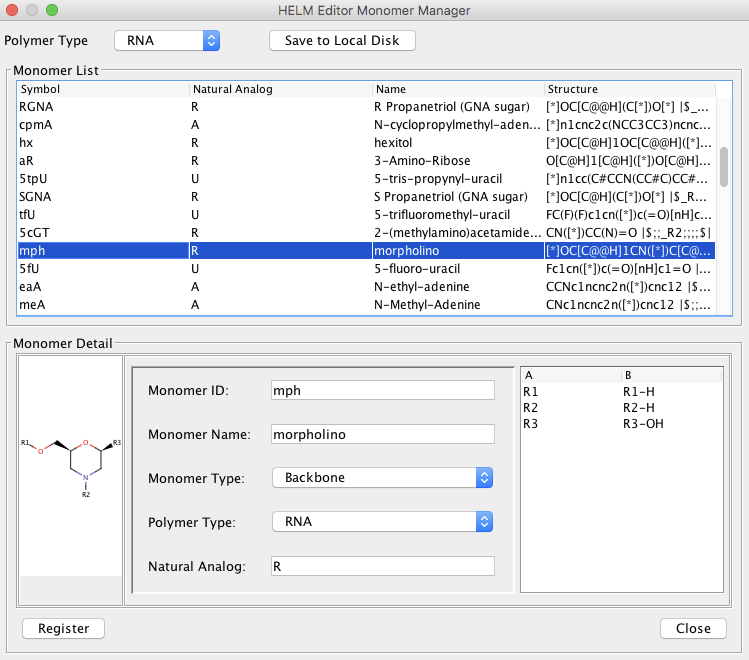
リン酸部位と5'末端の構造は無いようなのでMonomer Managerに登録しましょう。
前者はPolymer Type RNA、後者はCHEMとしました。
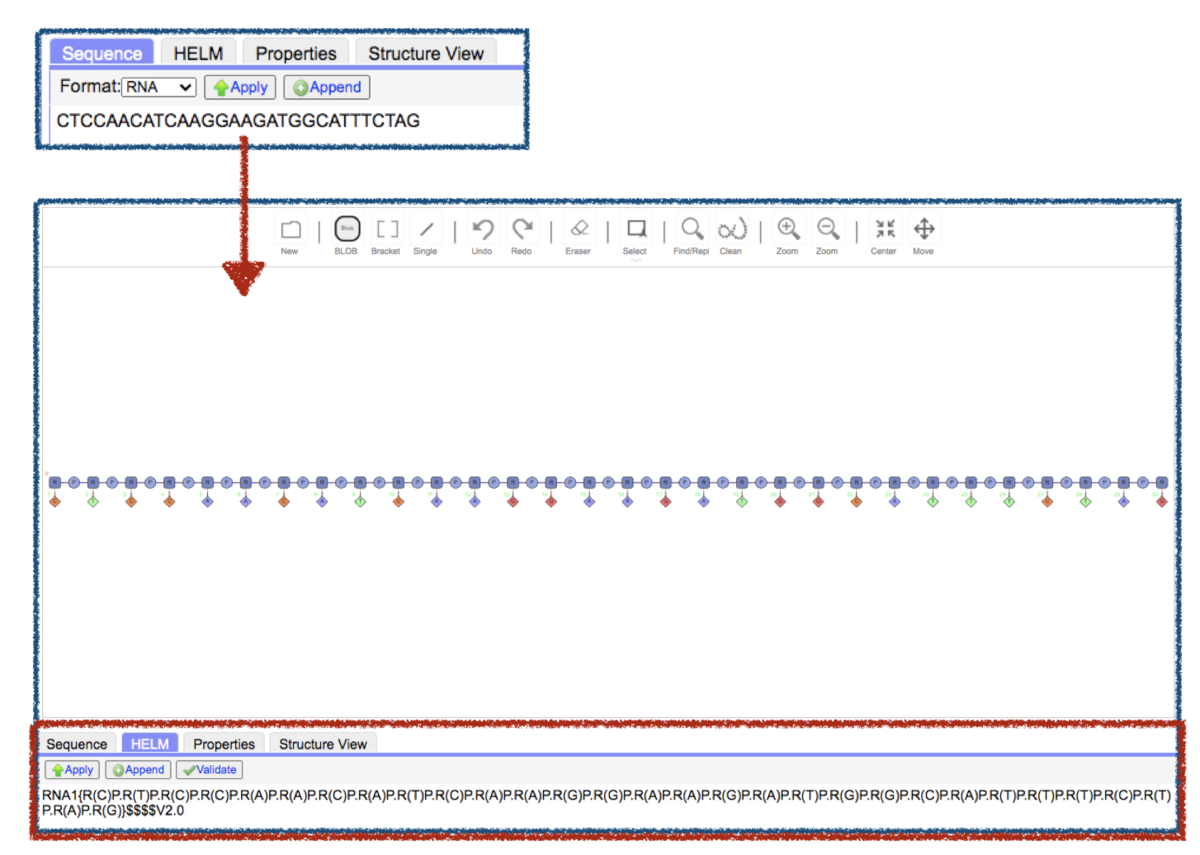
まずは天然の核酸の場合のHELMを確認します。HELM Web-editorがお手軽です。
配列を「CTCCAACATCAAGGAAGATGGCATTTCTAG」をSequenceに入れてApplyするとグラフ表現とHELMが確認できます。
糖部位はすべてモルフォリノ核酸なので[mph]で、リン酸部位は新しく作ったモノマー[DMP]で置き換えていきます。
複数文字の略記なので角括弧[]で囲むことに注意します。
Pythonのreplaceで文字列置換してみます。RではなくR(を[mph](で置換しているのはRNAのRも置換されてしまうからです。
natural_analogue = 'RNA1{R(C)P.R(T)P.R(C)P.R(C)P.R(A)P.R(A)P.R(C)P.R(A)P.R(T)P.R(C)P.R(A)P.R(A)P.R(G)P.R(G)P.R(A)P.R(A)P.R(G)P.R(A)P.R(T)P.R(G)P.R(G)P.R(C)P.R(A)P.R(T)P.R(T)P.R(T)P.R(C)P.R(T)P.R(A)P.R(G)}$$$$'
mph_analogue = natural_analogue.replace('R(', '[mph](')
DMP_analogue = mph_analogue.replace(')P', ')[DMP]')
print(DMP_analogue)
# RNA1{[mph](C)[DMP].[mph](T)[DMP].[mph](C)[DMP].[mph](C)[DMP].[mph](A)[DMP].[mph](A)[DMP].[mph](C)[DMP].[mph](A)[DMP].[mph](T)[DMP].[mph](C)[DMP].[mph](A)[DMP].[mph](A)[DMP].[mph](G)[DMP].[mph](G)[DMP].[mph](A)[DMP].[mph](A)[DMP].[mph](G)[DMP].[mph](A)[DMP].[mph](T)[DMP].[mph](G)[DMP].[mph](G)[DMP].[mph](C)[DMP].[mph](A)[DMP].[mph](T)[DMP].[mph](T)[DMP].[mph](T)[DMP].[mph](C)[DMP].[mph](T)[DMP].[mph](A)[DMP].[mph](G)}$$$$
できました!お手軽!
HELM Editorで読み込んでみます。
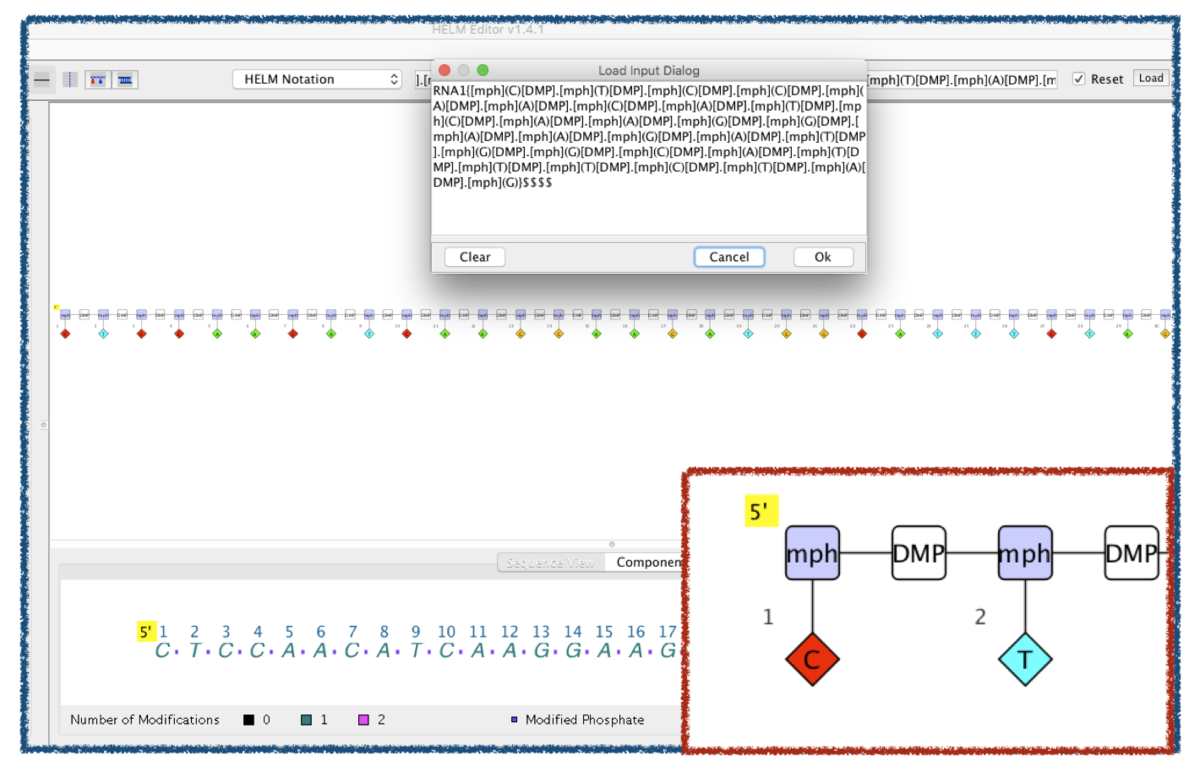
グラフ表現でました!小さいので5'末端の拡大図も載せました。
ここに5'末端の修飾を足します。リン酸の追加とChemical Modifierから先に作成したモノマーを選んで貼り付け。
こんな感じになりました。
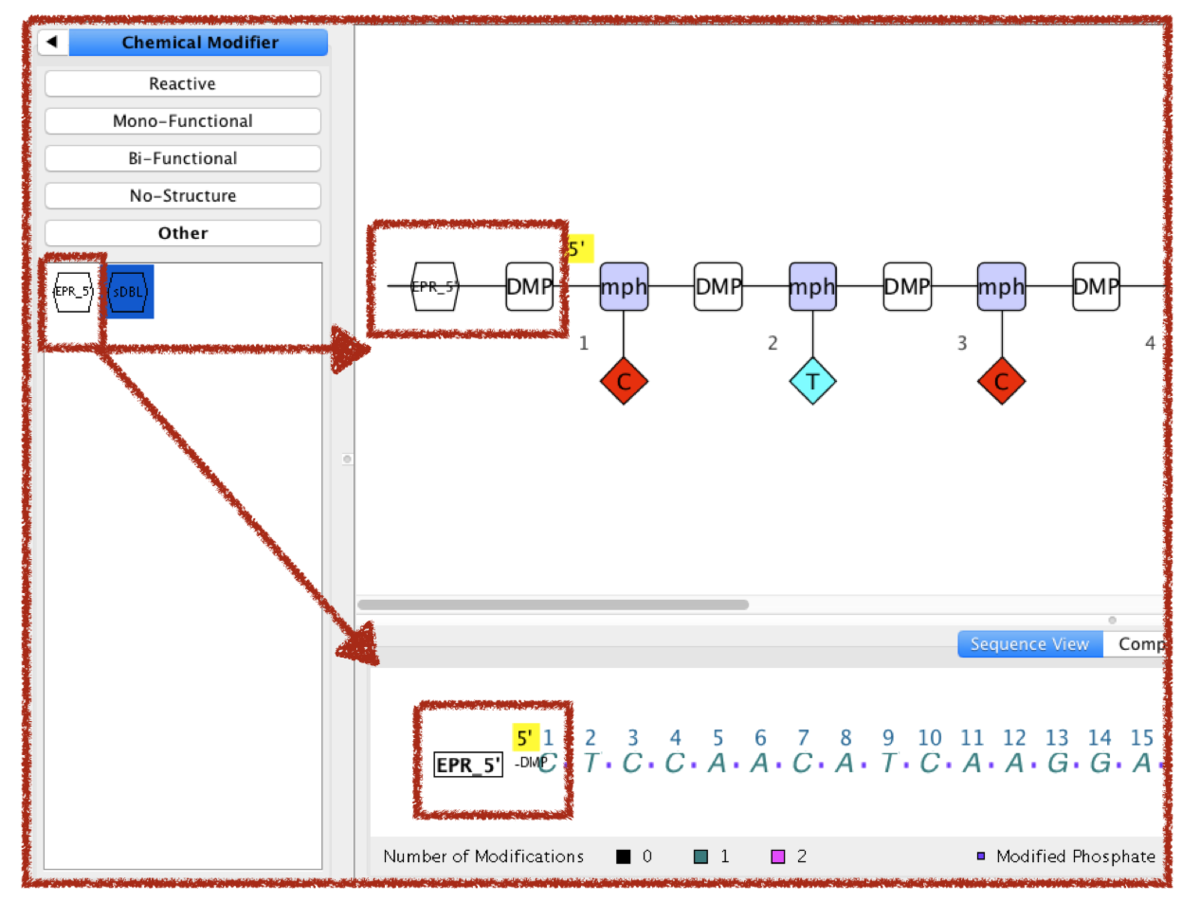
先の「Guide to Pharmacology」のような2D表現と比較してどうでしょうか?
HELMのグラフ表現ではモノマーが拡張可能かつ塩基、糖、リン酸の3つに分かれており、非天然の構造修飾をもった核酸であることが明確に示されています。
また、モノマーライブラリを介してより具体的な構造式と結びついているため、このグラフ表現は実際の構造式の情報と紐づけることが可能です。
一方で、構造式そのものよりも単純化されているため塩基の配列といった情報を把握しやすいものともなっています。
ではこのグラフ表記のHELMはどのようになっているでしょうか?
作成したグラフ表現の他の表記はEdit のViewから確認できます。
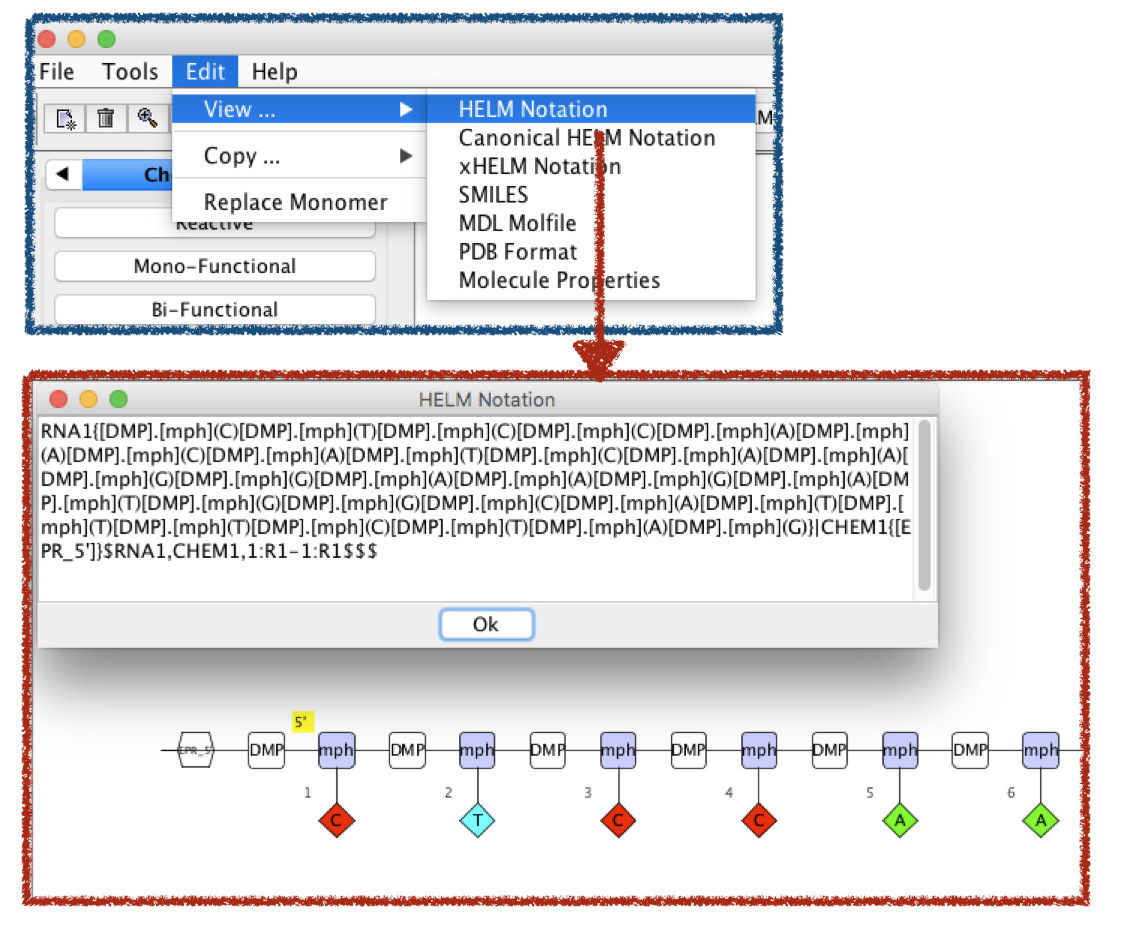
HELM表記の変更点はSimple polymer listの最初とConnection listです。 「RNA1{[DMP]. ~~~~ |CHEM1{[EPR_5']}$RNA1,CHEM1,1:R1-1:R1$$$」という表現になっています。
SMILESやMolファイルといった表記も確認できます。線形表記同士、SMILESとHELMを並べてみましょう。

HELMはモノマーの略記(ID)が使えることで簡略化されています。
SMILESで途中でてくる「%xx (xは数字)」は2桁の番号を使って閉環位置を示すためのものだそうです。*13
・・・初めて見ました。
このSMILESが他のソフトでも認識できるものなのか?念のためRDKitで構造を起こしみます。
from rdkit import rdBase, Chem from rdkit.Chem import Draw from rdkit.Chem.Draw import IPythonConsole # smi = SMILES文字列長いので省略 m = Chem.MolFromSmiles(smi) m
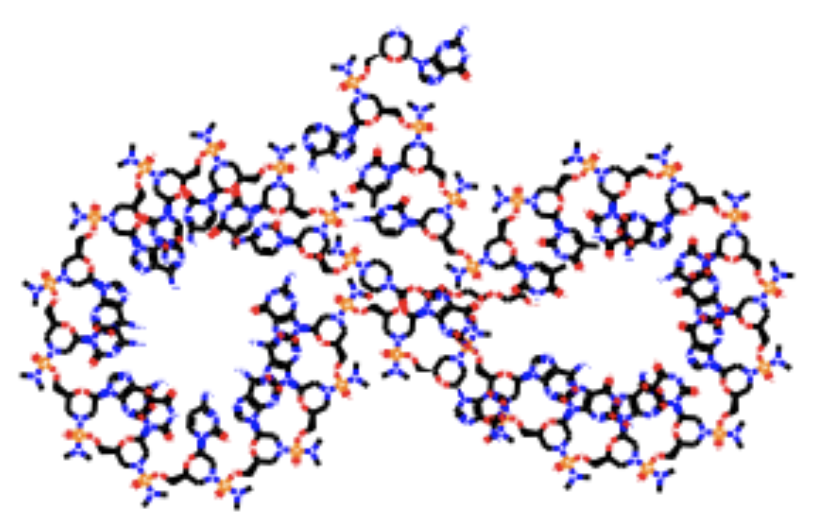
できました!わけがわからないがMolオブジェクトはできた!
Spinraza®︎ (nusinersen)
つづいてSpinraza®︎(nusinersen)です。Novaltis社のZolgensma®︎(Onasemnogene abeparvovec-xioi)との関係でも話題となりましたね。
構造は以下の通りです。
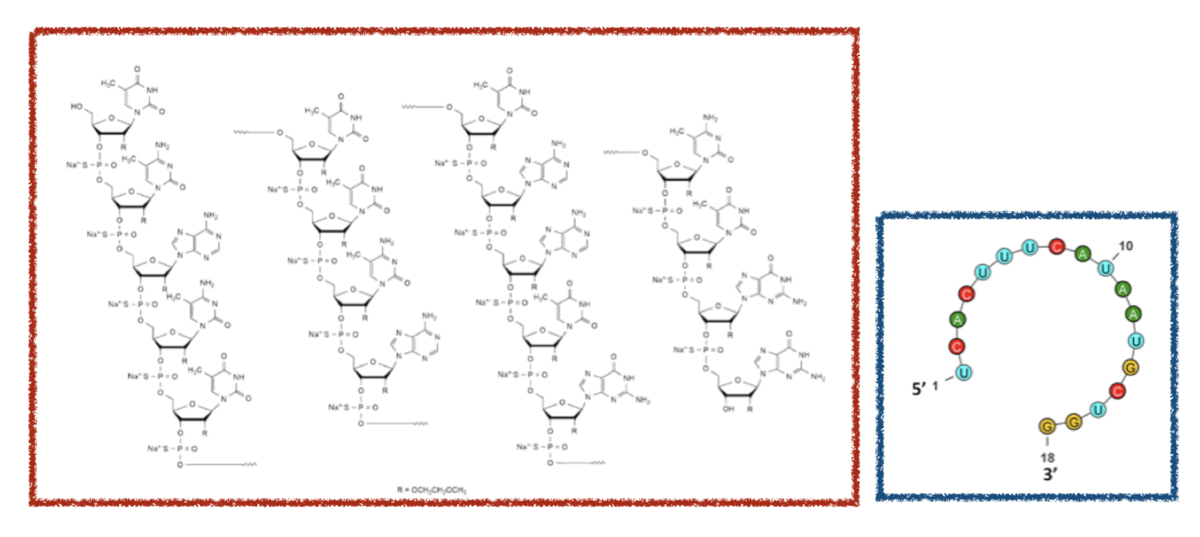
特徴としてはリン酸がS化されたSオリゴであることと、糖が2'-MOEとなっていることです。
こちらはPubChemにHELM含めて色々な表現が載っていました!こんな感じ・・・
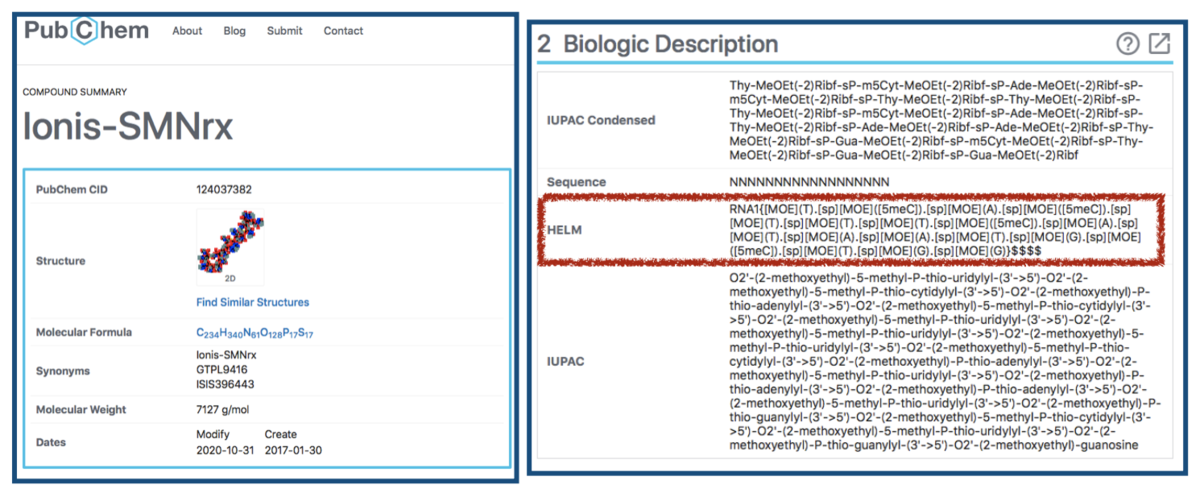
他にもInChiやCanonical SMILES、Isomeric SMILESが載っていました。情報が載っているものと見当たらないものの違いは何なのでしょう??
HELM editorに入れる前にモノマーがあるかを確認しておきます。
デフォルトで必要なモノマーが揃っていそうです。モノマーライブラリの追加が必要ないのでHELM Web-editorで描画してみます。
HELMをはりつけてApply!!
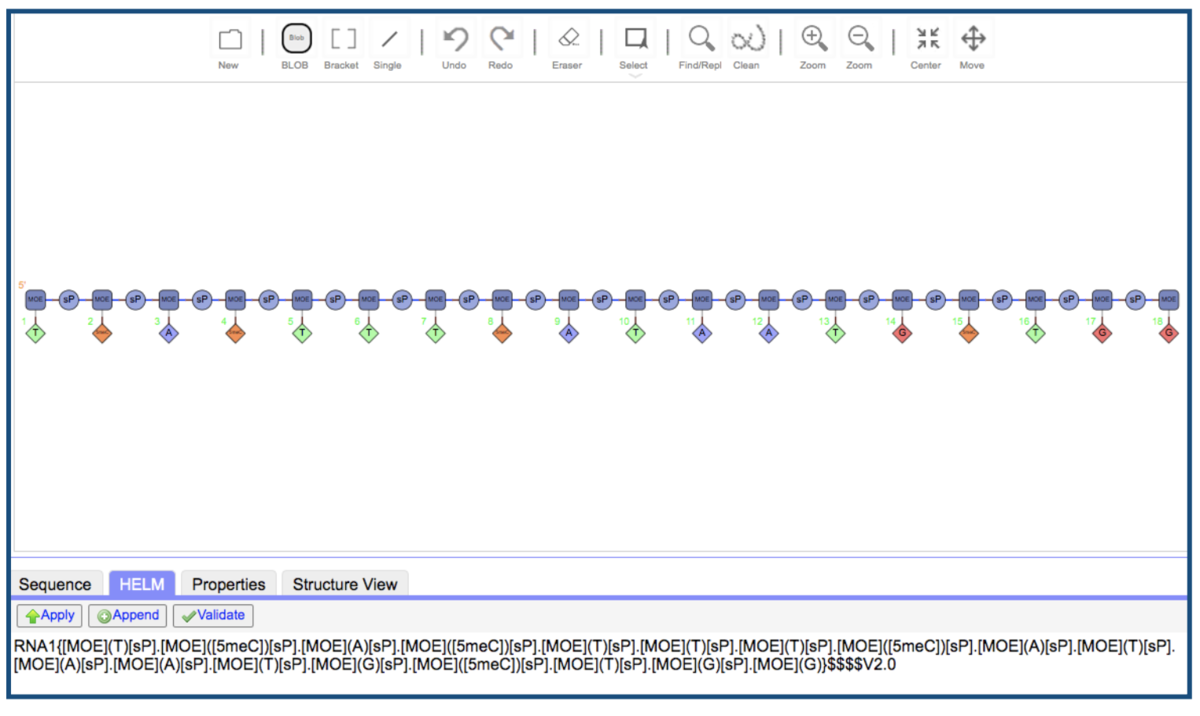
HELMがPubChem等で提供されているとお手軽ですね!
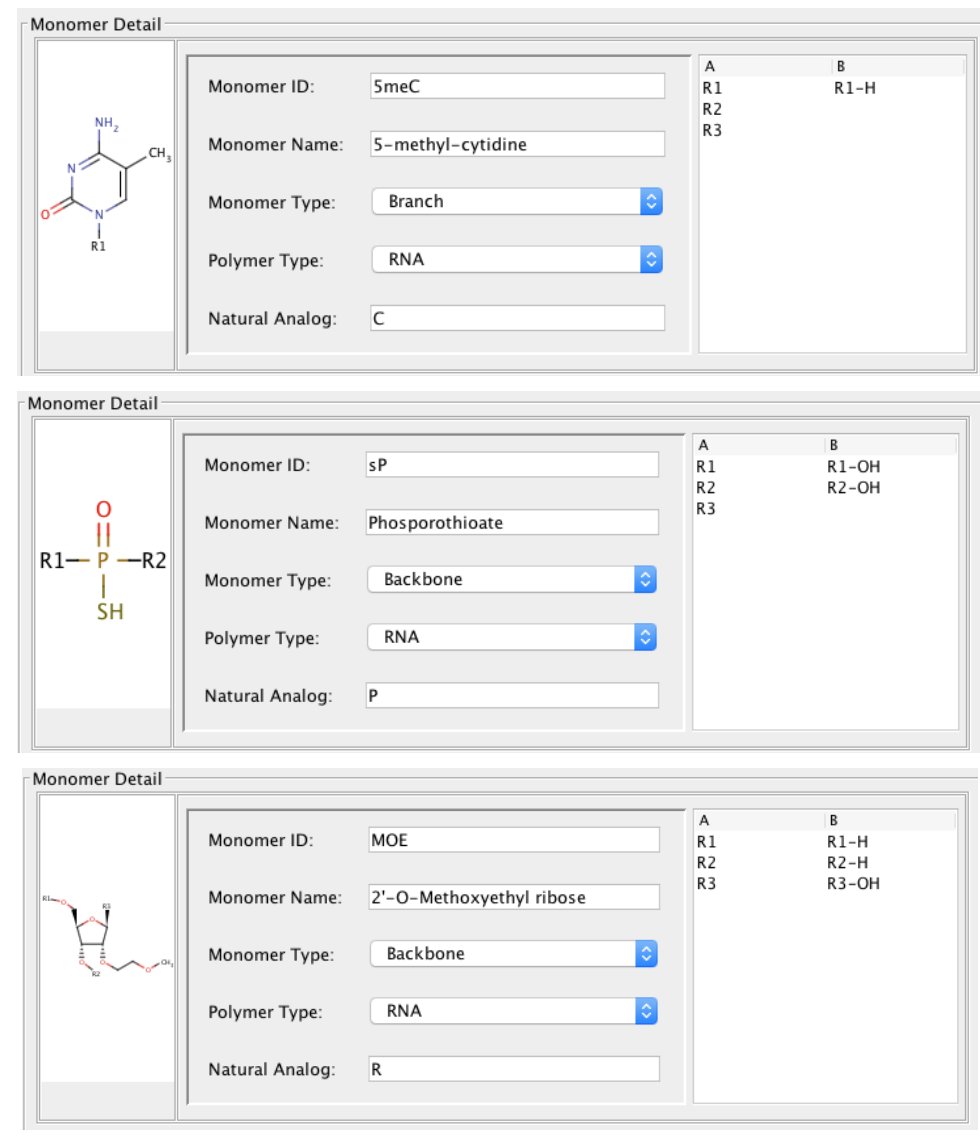
HELM表記およびグラフ表現では修飾構造(5meC、sP、MOE)といった点がわかりやすいように思います。
Givlaari®︎(givosiran)
最後、Givlaari®︎(givosiran)です。
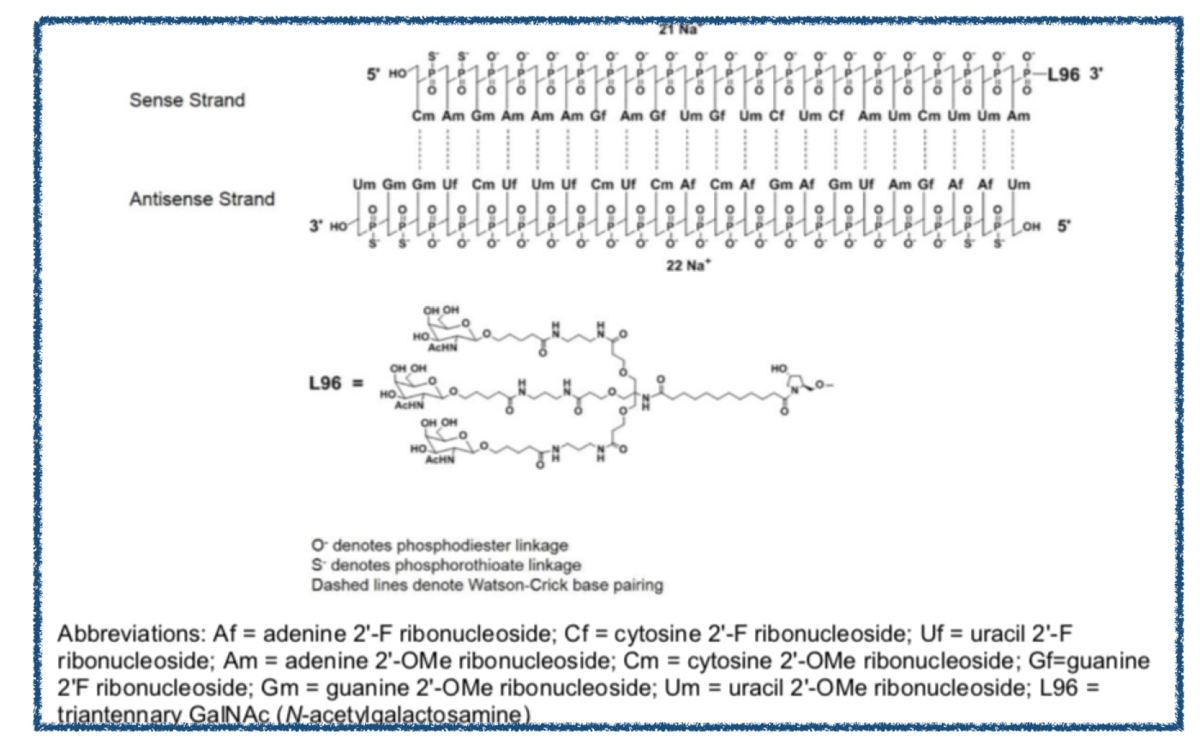
先の2つよりも構造の特徴が多いようです。
- リン酸部位にO体とS体の両方
- 糖部位は2'-F体と2'-OMe体が使われている
- 2本鎖
- GalNAcコンジュゲート結合
まずはモノマーの確認です。2'-Fと2'-OMeはあるようです。
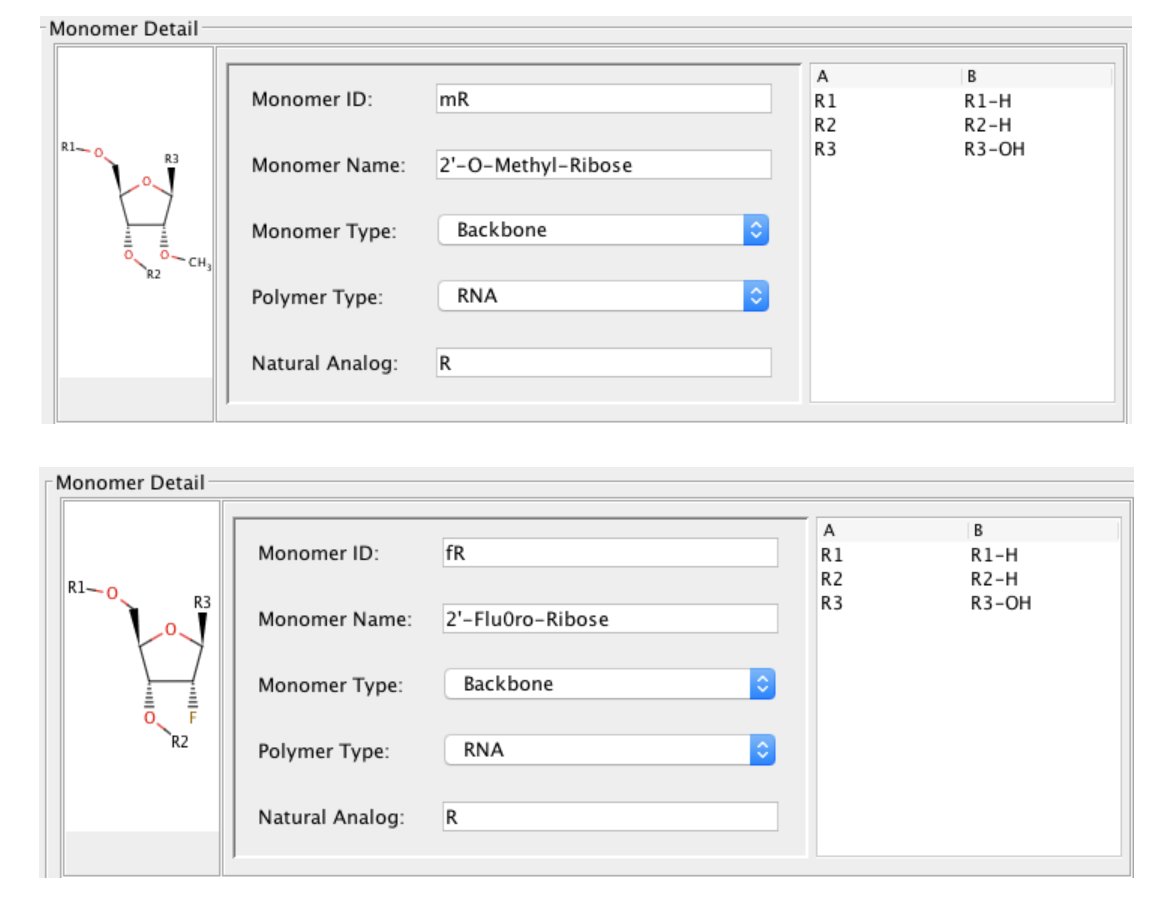
コンジュゲートを用意しましょう。こんな感じで登録してみました。
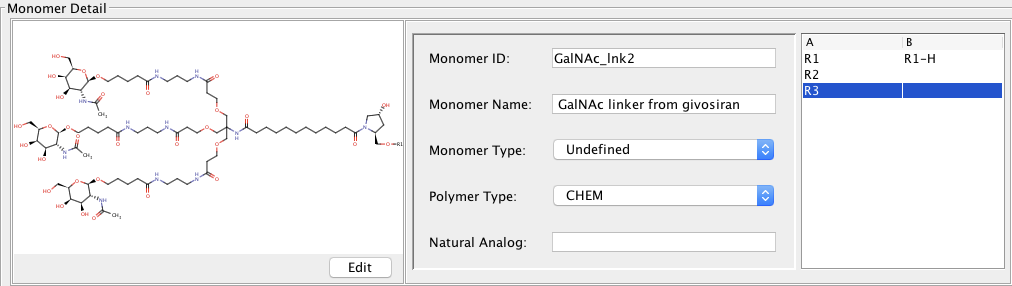
ある程度HELMの書き方がわかってきたのでStrandごとのSimple Polymerを書いてみます。

HELM editorに描いていきます。StrandごとにHELMを読み込んでいきましょう。
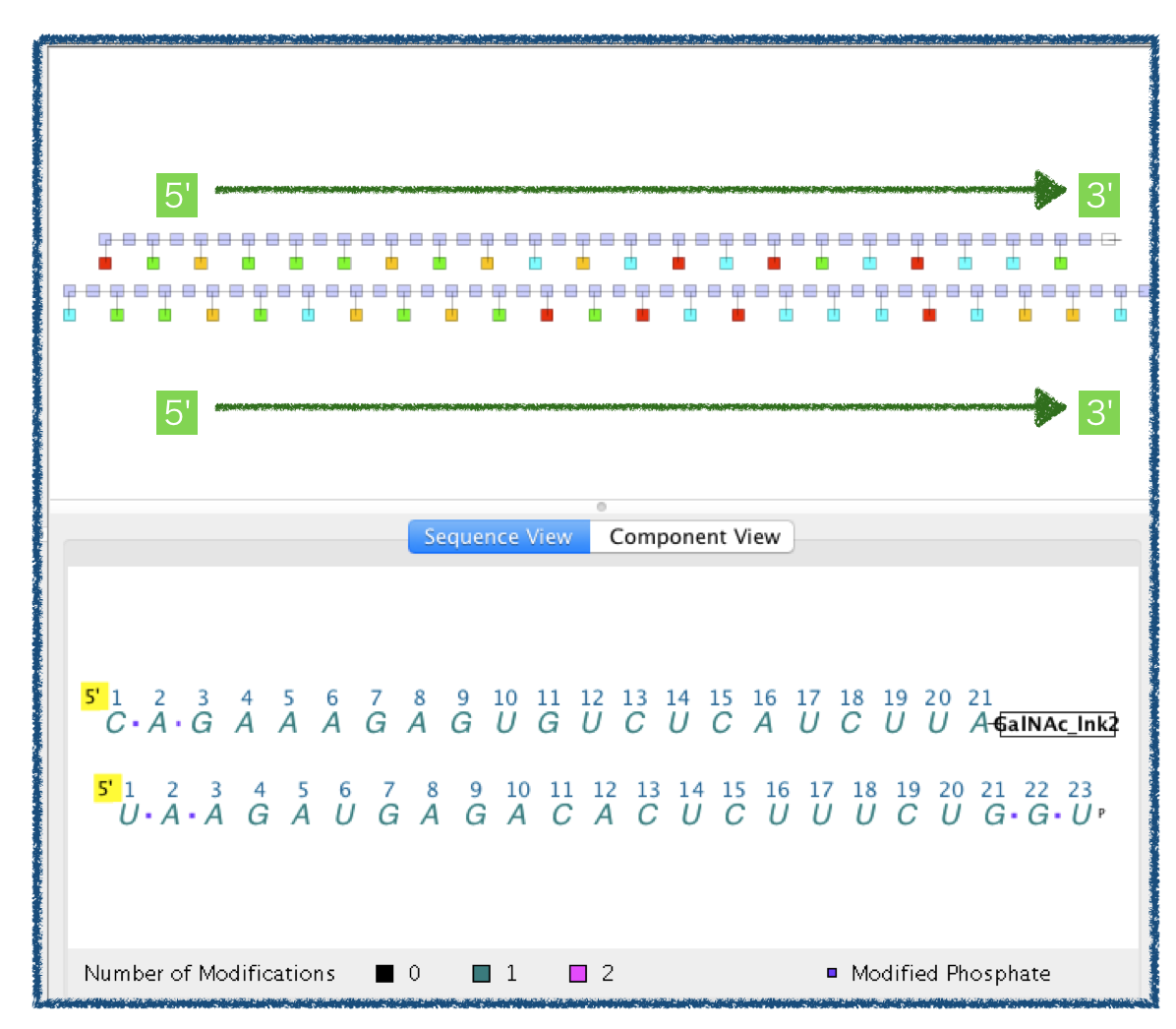
Sense StrandとAntisense Strandをどちらも読み込むと図のようになります。 どちらも左から右に向かって5'-3'の向きとなっています。
この状態で図の上にあるHybridizeボタンを押してやります。
するとAntisense Strandの左右がひっくり返り、2つのStrand間に水素結合が形成されます。
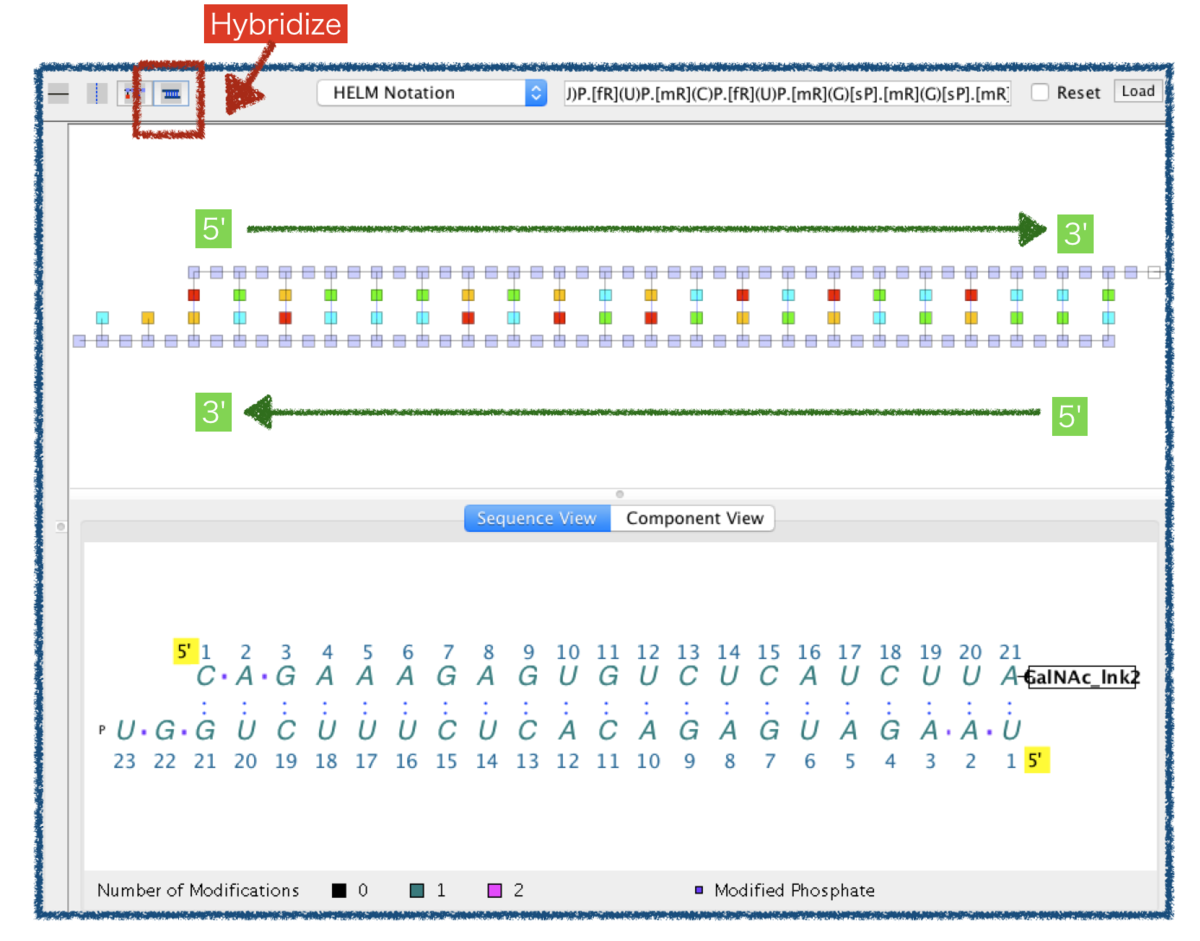
拡大してみます。塩基の間のドットが水素結合を表しています。
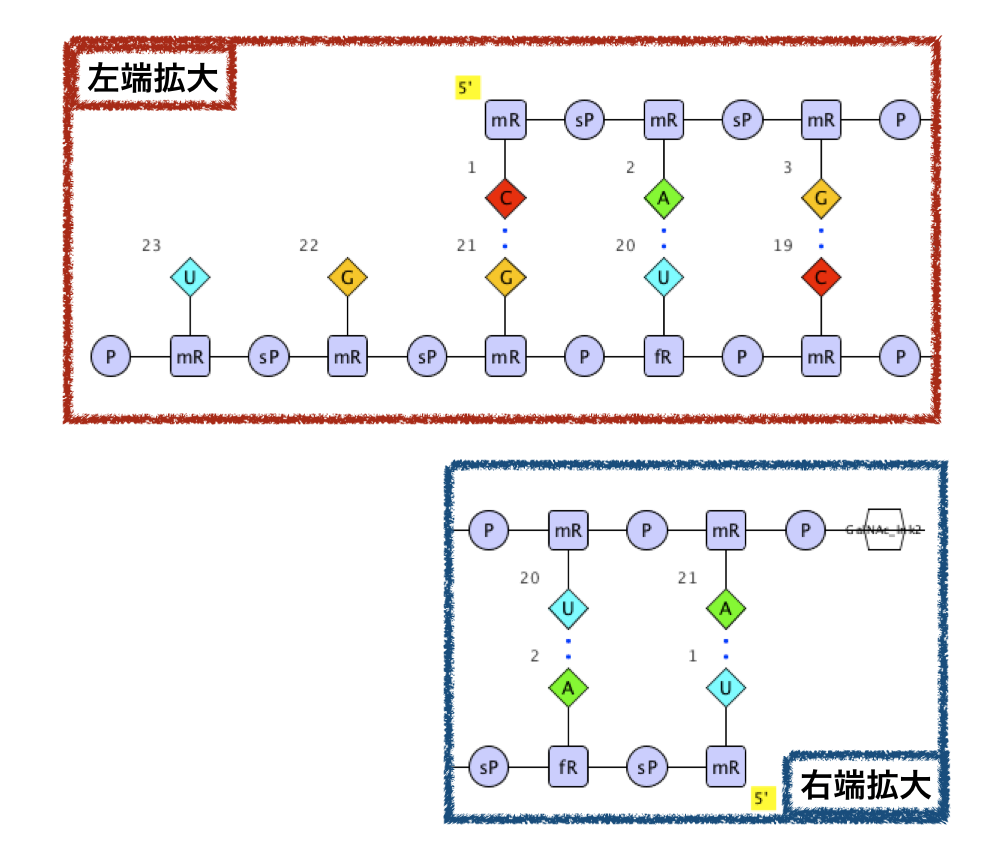
この状態でHELMは以下のような表現となっています。
水素結合がpairとして表されています。

最後にsense鎖(ss)、antisens鎖(as)を明確にするためAttribute Listに属性を付け加えることもできます。こんな感じ、、、

以上、givosiranのHELM表記とグラフ構造でした。
givosiranは添付文書の構造式の書き方がかなり分かりやすいのでHELMにしたことで可読性がよくなったか?というとちょっと微妙です。
ですが、モノマーライブラリと紐づいているので、略語の説明を個別にする必要がなく、糖、塩基の結合位置も構造式レベルで明確に指定されている、という点では HELMを使うことに利点があるのかもしれません。
またGalNAcコンジュゲートを一つのCHEMモノマーとして登録しましたが、リンカーと糖(GalNac)を別々に登録すれば、 異なる糖の組み合わせのコンジュゲートライブラリを表現し、それぞれの性質と結びつけることもできそうです。
まとめと感想
以上、今回は核酸のHELM表現を試してみました。
実際に使われている医薬品でHELM表現を試すことでいくつかメリットを感じられたように思います。
- 異なるタイプの核酸医薬に対して同じモノマーライブラリに基づいた共通の表現ができる
- 実際の構造式に紐づいており、修飾構造を明確に表現できる
- IDで表記するのでユニットの文字列置換で簡単に記述を変更できる
と、いったところでしょうか?
それぞれ単独の構造のみで記載したのであまり"旨み"がなかった感もありますが、 これらの裏側にあるであろう大量の核酸医薬候補ライブラリーを表現、管理、データベース化する、 となった場合に中々使い勝手の良いものとなるのではないでしょうか???
一方で、使いにくさを感じる点としては、コンジュゲートのような化学修飾ユニットをCHEMモノマーとしてそれぞれ登録する必要があることです。
化学修飾はSMILESで書いてハイブリッドな表現ができたらもっと便利な気もしますが、やはり難しいのでしょう。
あとPuChemやChEMBL、思ったより欲しいHELM表記あまり載ってなかったです・・・。 もう少し核酸医薬分野のデータベースも手厚くして欲しい気もします・・・が、ひょっとしたらHELMの標準ライブラリ、IDに修飾核酸をどこまで含めるか?みたいな問題があるのかもしれません。
毎度のごとく知識不足すぎて基礎事項を調べていたら本題のHELM表記に中々たどり着けませんでした。。。
まあ色々面白かったのでいいや。
以上!
今回も色々と間違いが多そうです。ご指摘いただければ幸いです。
*1:久保田恒平博士 博士論文「siRNA 担持脂質ナノ製剤の評価 に関する研究」記載より CiNii
*2:下表は文献2 表1より抜粋・改変して作成
*3:Pharmaceuticals 2020, 13(3), 40 CC BY 4.0の元に図を引用
*4:Toxicologic Phatology 2018, 46, 735 CC BY 4.0の元に図を引用
*7:菊池 寛 Drug Delivery System 2019(34)106 企業的観点からみた核酸・遺伝子医薬品開発の現状と今後の展望:DDSとの関わり
*8:Semple, S. et. al., Rational design of cationic lipids for siRNA delivery. Nat. Biotechnol. 28, 172–176 (2010).
*9:Jayaraman M. et. al., Maximizing the potency of siRNA lipid nanoparticles for hepatic gene silencing in vivo. Angew. Chem. Int. Ed. Engl. 2012;51:8529–8533.
*10:こちらのレビューの図も良いです Mol Ther. 2017(25)1467
*11:Nat. Biotecnol. 2017(35)845