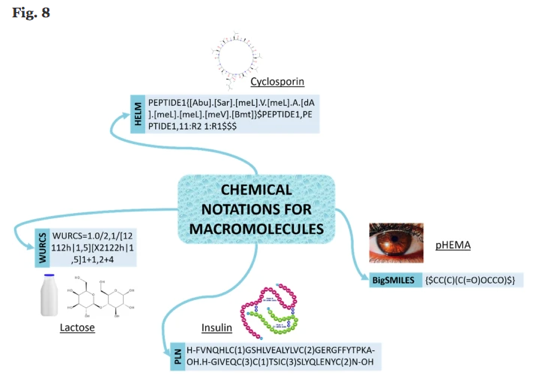
HELM editorでお絵描き
前回の記事でマクロ分子の表現方法HELMの表記ルールについて調べてみました。
今回は実際の利用にあたって便利そうなツール、特にHELMエディタの入手方法と簡単な使い方について調べてみたいと思います。
HELMのデータベース・ツール
Pistoia AllianceのHPによるとHELMはマクロ分子の表現として標準的な手法となることを目指しているそうです。 新しい表記の普及にはデータベースの整備とお手軽な読み書きのツールが大事そうです。
データベースが準備されていれば、実際の分子がその表現でどのように表せるか実例を参照したり、他の表現方法との比較が簡単にできます。 また、ツールを使って自分で表記を書いてみたり変換したりできれば、ちょっと使ってみようかなってなりますよね!
具体例としてHELM Wiki (HELM Resources)には 以下が記載されていました。ありがたいことにツールの開発・公開も行なってくださっているそうです。
- オンラインデータベース:HELM in Online Databases
- PubMed:PubChemの「biologic description」セクションにHELM表記の記載
- EBI ChEMBL:「BIOTHERAPEUTICS」テーブルや「Compound Report Card」に記載
- ツール:
HELM Toolkitはこんな感じの構成。MIT licenseライセンスの下でオープンソースとなっています。 *1

前回の記事でRDKitでもHELMの取り扱いが可能であることを確認しましたが、現在RDKitで扱える範囲は以下のような天然のモノマー+αに限定されているようです。(RDKit-discussの議論より)
HELMの表現力を活かすには新しい非天然のモノマーもどんどん取り込んでいきたいところです。 上記RDKit-disucsではより細かな内容を扱うにはPiasota Allianceが提供しているJavaツールキットを利用する方が良いとオススメされていました。
ほな公式のHELM Toolkitで遊ばしてもらいましょ!
HELM editor入手方法
ざっと見た感じeditorには3種類のアクセス方法がありそうです。一つずつ試してみましょう!
HELM editor (ローカル環境)
まずはHELM editorに記載のローカル環境にインストールする方法。
GitHubページのリンク先に「HELM Editor v1.4.1」のバイナリファイル(zip)があるので ダウンロードしてきて解凍するだけです。

「HELMEditor-1.4.1.jar」というファイルをひらけばJAVAプログラムが起動します。

HELM Web-editor (トライアルバージョン)
ついで、WebベースのHELM Web-ediorです。HELM Wikiのページはこちら 。
トライアルバージョン(v1.1.4)が用意されているので下のリンク先に飛べばすぐに試すことができます。
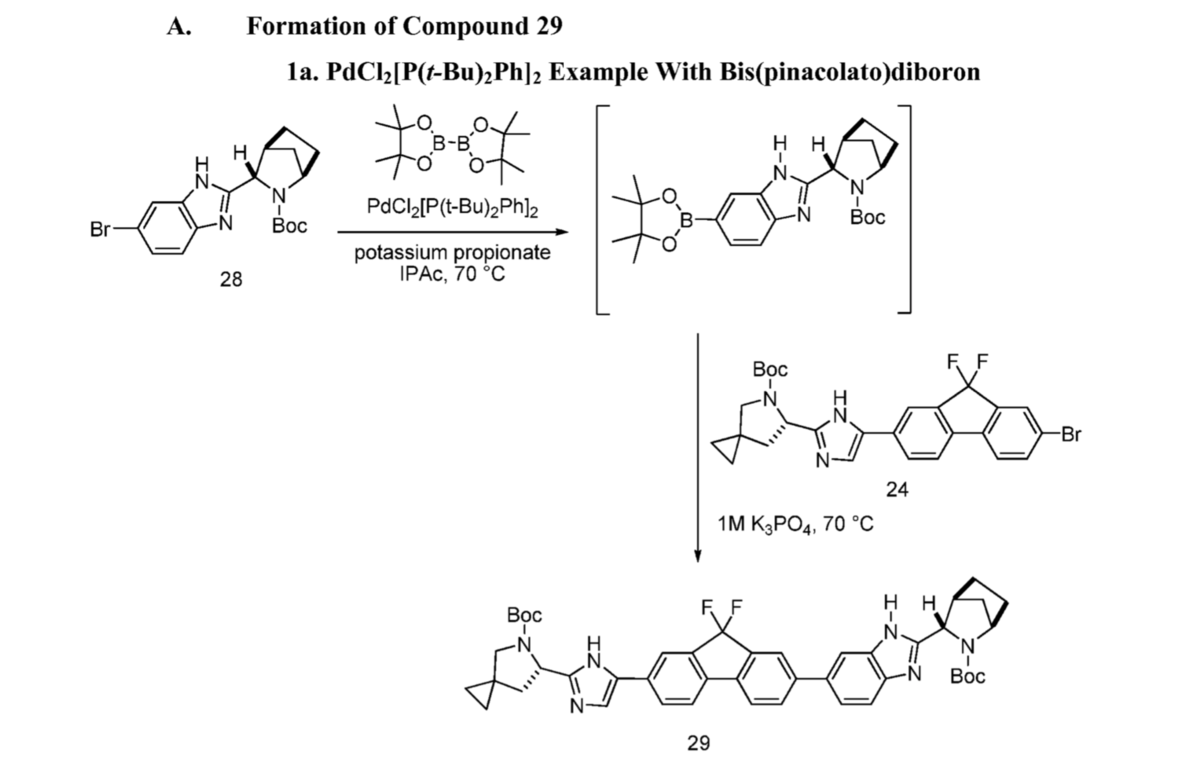
先のローカルに落としたEditorとWeb-editorはちょっと見た目が違う感じです。

HELM Web-editor (インストールバージョン)
Web-editorをインストールして利用することも可能です。GitHubにコードが公開されており、 ReadMeにインストール方法が書かれています。
HELM Web-editorはJavascriptで書かれているそうです。で、JavascriptのWebアプリを使うには、Webコンテナとかいうアプリケーションサーバーを用意してWebサーバで動かすことになるらしい。。。どういうこっちゃ?
Tomcatを使って、ということなのでダウンロードしてみます。*3
① Java SE Development Kit (JDK)のインストール
OracleのサイトからmacOS用のJDK-15.0.1というのをダウンロードしてインストールしました。 Personal useはOKですと。。。アグリーアグリー
② Apache Tomcatのインストール
ついでApache TomcatのページからTomcat 9.0.39というのをダウンロードしてきました。 Tomcat 10.0もあるようでしたがalpha版のようだったので9.0にしました。
インストール後、実行権限を確認する必要があるそうです。
ターミナルでディレクトリを移動してls -lとしたところ、binは「drxr-xr-x」となっていました。(755であってます?)
一方、binの中のファイル群は「-rw-r--r--」(これは644?)となっていたのでシェルスクリプトの権限を書き換えました。
ターミナルに「chmod 755 *.sh」とコピペします。
実行権限も設定できたので、後はbinの中にあるstartup.shというスクリプトを実行すれば良いそうです。
ターミナルで./startup.shとすると「Tomcat started」と無事出てきました。
あとはlocalhostのポート番号8080にアクセスすれば良いらしい...
「http://localhost:8080/」をChromeに貼り付けてエンター!

できた!劇団四季のライオンキングみたい!この微妙な可愛くなさがいい!
シャットダウンは「./shutdown.sh」だそうです。忘れぬうちにメモしときます。
③ HELM Web-editorのインストール
Tomcatの準備ができたのでHELM Web-editorを順番に準備して行きます。必要なファイルは3つ!
まず、「①HELM2MonomerService (HELM2MonomerService.war)」 と 「② HELM2WebService (WebService.war)」を手に入れます。
GitHubからのリンク先からはダウンロードファイルがうまく見つからなかったので、 HELM Wikiに記載の Nexus repository managerというところから それっぽいファイルを取ってきました。コンパイル済みのJAVAファイルを置いてくださっているそうです。

warファイルというのはJAVAベースのwebアプリが圧縮された形式らしいです。
ファイル名が長いのでGitHubの手順と同じ名前に変更した後、2つのwarファイルを「apache-tomcat-9.0.39」ディレクトリ配下の「webapps」ディレクトリに入れました。
デプロイ!!・・・で、あってます?
で、すぐにwarと同じ名前のディレクトリが勝手に作られます。こんな感じになりました。。。
最後に「③HELMWebEditor (hwe-1.1.0.zip)」を手に入れます。こちらはGitHubのリンクからそのままダウンロードできました。
unzipしてできた「hwe」ディレクトリを先に作った「\webapps\HELM2MonomerService\」の中に移動します。
ファイルの準備はこれでおしまい。
④ 起動
Apatche Tomcatのサーバーを起動した後、URL「http://SERVER/HELM2MonomerService/hwe/」にブラウザからアクセスすれば
Web Editorが起動するとのことです。
SERVERのところをlocalhost:8080として「http://localhost:8080/HELM2MonomerService/hwe/」にアクセス!!

見つかりません!何故!
右往左往しているとhweディレクトリの中のexapmleにAPP.htm.というファイルがありました。 中身を見てみるとJSDrawディレクトリのjavascriptを読みに行っている感じです。

なんだかアプリっぽい。。。
URLを「http://localhost:8080/HELM2MonomerService/hwe/examples/App.htm」に変更してアクセス!

デモ版と同じ画面が立ち上がりました!あってた!嬉しい!
HELM Web editorの使い方
インストール完了したので使ってみましょう!
HELMEditorよりもHELM Web-editorの方が画面がスッキリしているので、Web-editorで遊んでみたいと思います。
といっても使い方は簡単!モノマーのブロックを繋いでお絵描きしていくだけです。
画面の説明
まずは全体の画面。3分割されていて以下のようになっています。
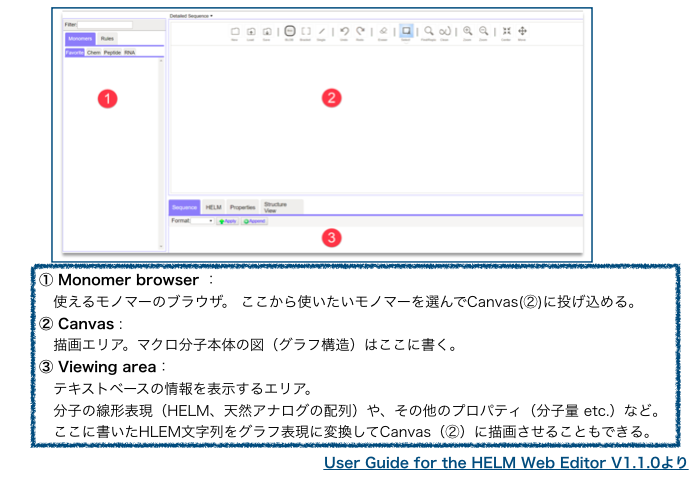
2次元構造は②Canvasに表示されます。①monomer browserでモノマーを選んで投げ込んでいけます。 ③Viewing areaはテキストベースの情報を見る箇所で、②と③の間で、グラフ構造とHELMを相互に変換する することも可能です。
monomer browser
monomer browserはSimple Polymer Type毎のタブに分かれています。 モノマーが略語で表されていますが、カーソルをあわせるだけで構造が表示されます。

RNAタブはこんな感じ。尚、Simple Polymer TypeのRNAはヌクレオチドポリマー全てを含むのでDNAも含みます。
ヌクレオチドR(A)PのAが「丸括弧( )」で囲まれているのは塩基を分岐として扱うことを反映しています。
構造の定まっていない曖昧なモノマーのための表現や、一発変換のRulesといったものも用意されてます。
デフォルトで用意されているルールはRNAポリマーのAをU、G、Tに変換するもののようです。 センス鎖-アンチセンス鎖を一発変換できれば便利そうと思いましたが、5'-3'配列順序も反転させないといけないので難しいのかもしれませんね。
Canvas
Canvasに用意されているボタンはアイコンを見ると使い方がだいたいイメージできるようになっています。

見慣れないのはBLOBですが、これは未知の構造/配列からなるSimple Polymerとして利用できます。
試しにお絵描きして見ます。始まりはいつも開始コドン!!!

できました!
モノマー間をSingleアイコンでつないでいますが、
singleを選択しなくても2つのブロックの中の文字をドラッグしながらつなぐだけで結合が作られます。
ついでにDNAのセンス鎖に変えてみます。

簡単!
Viewing area
Viewing areaには①Sequence、②HELM、③Properties、④Structure Viewの4つのタブがあります。
先にお絵描きした状態で表示される情報はこんな感じ。。。

一部重なってしまっていますが④Structure Viewで配列を構造に変換、確認できるのはちょっと感動です。
Propertiesでは分子量、組成式に加えてExtinction Coefficient(モル吸光係数?)も表示されています。
以上はCanvasに描いたグラフ構造の情報の確認ですが、反対にViewing areaに書いた配列をCanvasに反映させることも可能です。
終止コドンを加えてみます。開始即終了!!

できました!
ValidateでHELMが正しいかどうか確認できるのは手厚いですね!書き方のテストにも良さそうです。
簡単な描画画面の説明は以上です。
Monomer Library
HELM Web editorで利用可能なmonomer browserのMonomerやRulesといったデータの元を参照することも可能です。このデータを編集して表現を拡張することが意図されているようです。
グラフ描画の上のほうにカーソルをあわせると、下図のようなバナーが表示されます。
ここで画面を切り替えてMonomer LibraryやRulesetの確認、編集、追加といったことが可能になります。
Monomer Library画面へと切り替えてみましょう!図のようにデフォルトで入っているMonomer Listが表示されます。

SymbolやNameをつかってライブラリを検索できます。
登録されているチロシン(Y)の関連構造をみてみるために「Symbol: Y」で検索してみました。

天然のチロシン以外にD体やN-メチル体、ニトロチロシンが登録されています。 鉛筆マークをクリックするとmonomer情報を編集することができ、プラスマークでは新規monomerを追加できるようになっています。
・・・が、どうもうまくいかないようです。編集、追加ともにエラーが出てしまいました。

GitHubのissue #220 に同じ状況が報告されており、まだ解決していないようでした。
残念。。。*7
HELM editorの使い方
HELM Web editorではmonomerの追加がうまくいかなかったので、Web版ではないHELM Editorも試して見ることとしました。
画面構成と使い方
基本的な使い方は同じですがアイコンの見た目や配置が異なる箇所があります。
ざっと画面をみていきます。

- ① Web版のMonomer browserに相当
- ② Web版のCanvasに相当
- ③ Web版のViewing areaに相当。ただしApply、Appendといった機能はない
- ④ 線形表記をここに書いてLoadするとグラフ表現にできる。Web版のApplyに相当。
Web版のViewing areaで便利だったSequenceからHELMへの変換はToolsのConvert Notation機能で実行できます。

またグラフ表現上でクリックすることで構造の情報確認が可能です。

さらに面白い機能として、モノマーを編集して構造修飾を加えることができます!

上の図では塩基のウラシルを5-フルオロウラシルに編集しています。
編集してSaveすると、グラフの表記が5fUに変化し、
Sequence viewのUもイタリック(modified)に変わっています。
これは便利ですね!・・・配列に突如組み込まれるテガフール!
新しいモノマーの追加
さて、気になる新しいモノマーの追加です。デスクトップ版ではうまくライブラリを拡張できるでしょうか??
Web版のMonomer Libraryに相当するのはMonomer Managerという機能です。
ここでライブラリにない新しいモノマーの情報を入れてregisterとすることでライブラリに追加できます。

上の図では3-Fluoro-L-tyrosineを「Monomer ID FlY」として追加登録しました。
登録後、Monomer browserのOtherから新しいモノマーFlYが選べるようになっていることを確認できました!*8
Web版でうまくいかなかったモノマーの追加がうまくいったようです!!
これでオリジナル分子を自由にかけますね!
まとめ
以上、今回はHELMの利用をめざしてツールの導入と簡単な使い方を調べてみました。 ツールがあれば自分でHELMを書いたり、グラフ表現との変換が容易にできるのでいろいろと遊ぶことができそうです。
HELMエディタとしてデスクトップ版のHELM EditorとHELM Web editorの両方を試してみました。
Web editorはデスクトップ版よりも画面がスッキリしていて、よりシュッとした(?)インターフェースといった印象でした。 CanvasやViewing areaの使い勝手が良い一方で、Monomer Libraryの追加機能に不具合があるという問題点がありました。
デスクトップ版 HELM Editorは見た目がちょっと旧い感じはありますが、新しいモノマーが追加可能、構造の編集が容易という点がWeb版よりも優れている印象です。
特殊ペプチドや核酸アナログといった構造修飾、化学変換を組み入れていくには現在のところデスクトップ版を使う方が良いかもしれません。
マニュアルの図を貼り付けたりしていたらまた長い記事になってしまいました。
「途中のWeb版の長々としたインストールの説明必要だったのか?」というツッコミをいただきそうですが、 Tomcatのライオンキングみたいなロゴが気に入って使いたかっただけです。・・・すみません。
これでHELMの書き方は心配ないさ~~
適当に調べたことを書いているので誤り等ご指摘いただければ幸いです。
*1:但し、HELM toolkitが利用しているMarvinBeansはChemAxonのもので、HELMのためにロイヤリティフリーで提供されていますが、他の目的の利用についてはライセンスが必要となるそうです。
*2:https://pistoiaalliance.atlassian.net/wiki/spaces/PUB/pages/13795362/HELM+Notation
*3:インストールはこちらのページを参考にさせていただきました。
*4:User Guide for the HELM Web Editor V1.1.0より
*5:User Guide for the HELM Web Editor V1.1.0より
*6:User Guide for the HELM Web Editor V1.1.0より
*7:MonomerLib2.0.dbというそれっぽいファイルがあったのでパーミッションを変えて書き込みできるようにしてもダメでした、、、
*8:登録したモノマーの削除方法がわからないので試しに登録したFYも残っています。。。
マクロ分子の線形表記 HELMについて調べた話
先日@iwatobipen先生のブログ記事「Create macrocyclic mol object from sequence」を読み、RDKitでペプチドが取り扱えることに感動しました!*1
配列をそのまま原子レベルのオブジェクトに変換できるってすごい!
で、「もうちょっとこの辺りの話知りたいなー」となったので、「そもそも生物っぽい分子の表現ってどうなんですかね?」ということで調べてみることにしました。
とりあげるのはマクロ分子の表現方法、HELM!
こちらのレビュー(J. Cheminform. 2020(12)56)の中で取り上げられていました。*2
歴史やら表記のルールやら、だらだら書いていきますよ!
HELM?
HELMはHierarchical Editing Language for macromolelucesの略です。 雑にまとめると、「マクロ分子を原子レベルかつ簡潔に表現できる線形表記」といった感じで、 複雑な生物学的分子を単一の表記で記載することができます。
ペプチドやタンパク質、オリゴヌクレオチドといった生物学的な分子の線形表記というと、まずは配列、 アミノ酸や塩基といった(限られた)ユニットを表す文字の組み合わせが思い浮かびます。
対してHELEMはSMILESをベースとしており、配列だけではなく細かな化学構造も表すことができるため、 構造修飾や特殊なユニットといった、より豊かな表現が可能です。
つまり抗体+低分子のADCや修飾核酸医薬といった複合的な構造もきっちりと表現できます。
また、その表現力により表記とマクロ分子の構造とを1対1で対応させることができるので、 データベースへの格納といった厳密さが必要となる場面でも利用できます。
まさにNew Modality時代に必須の表現!!デファクトスタンダードとなるポテンシャルを持つ表現なのだ!!
・・・知らんけど。

歴史
元を辿ると2008年Pfizer社内でアルゴリズムの開発が始まったHELMですが、 2010年前後からPistoia Allianceという非営利のコンソーシアムの支援を受けてさらに開発が進みました。*4
2012年にオープンなものとしてリリースされ無料で利用できるようになりました。 同年、Pfizerより論文も発表されています。*5
HELM: A Hierarchical Notation Language for Complex Biomolecule Structure Representation
Zhang, T. et. al., J. Chem. Inf. Model. 2012, 52, 10, 2796–2806
2014年にはChEMBLがHELMの採用を公表し、2015年にはChemAxonがHELM toolkitのMarvinBeans 5.0ライセンスを無料化、 また、RDKitでもHELMが利用できるようになったりと、データベース・ツールともにHELMへのアクセスが広がり、 順調にデファクトスタンダードとしての地位を築きつつあります。*6
Pistoia Alliance

Pistoia Allianceはグローバル製薬企業(AstraZeneca、GSK、Novartis、Pfizer)の代表によって2007年構想、2009年設立された組織で、 2020年現在、100を超える企業・団体がメンバーとなっています。日本からは武田薬品が参加しているみたい。*8
組織の目的はR&Dイノベーションを妨げる共通の課題を協力して解決し、効率をあげることです。 具体的にはデータの収集、アクセス、共有方法といった、R&Dに必須でありながら、各社それぞればらばらに取り組んでいた課題があげられます。
規制当局と協力して新しい標準化を取り入れたり、法的な枠組みを提供することで、各社が競争する前の「場」をまずは整備しましょう、ということみたいです。
ちなみにPistoiaは会合が開かれたイタリアの都市の名前だそうです。ミニマルなデザインのロゴにトスカーナの風を感じますね!・・・知らんけど。
脱線しますが、個人的にとても良いな、と思ったのはPistoia Alliance Chemical Safety Libraryという取り組みです。
実際のラボ実験の経験にもとづき、危険な化学実験のリスクについて情報共有を行おうとするもので、CASと協力してオープンアクセスのプラットフォームChemical Safety Library の公開に至っています。*9
企業間の垣根を超えてノウハウを共有し、安全性の向上をめざしていこうというのはすごく良いですよね。推せるぞ!Pistoia!
HELM表記の構造と表現方法
背景がわかったところで具体的なHELMの中身について見ていきましょう。 Pistoia Allianceホームページの図表が非常に分かりやすいので、先のPfizerの論文を参照しつつ眺めていきます。
以下、特段の記載の限り図表はこちらのURL を引用させていただいています。
HELMの階層構造
Hierarchical Editing Language for macromolelucesという名前が示す通り、HELMは階層構造を持った表現です。
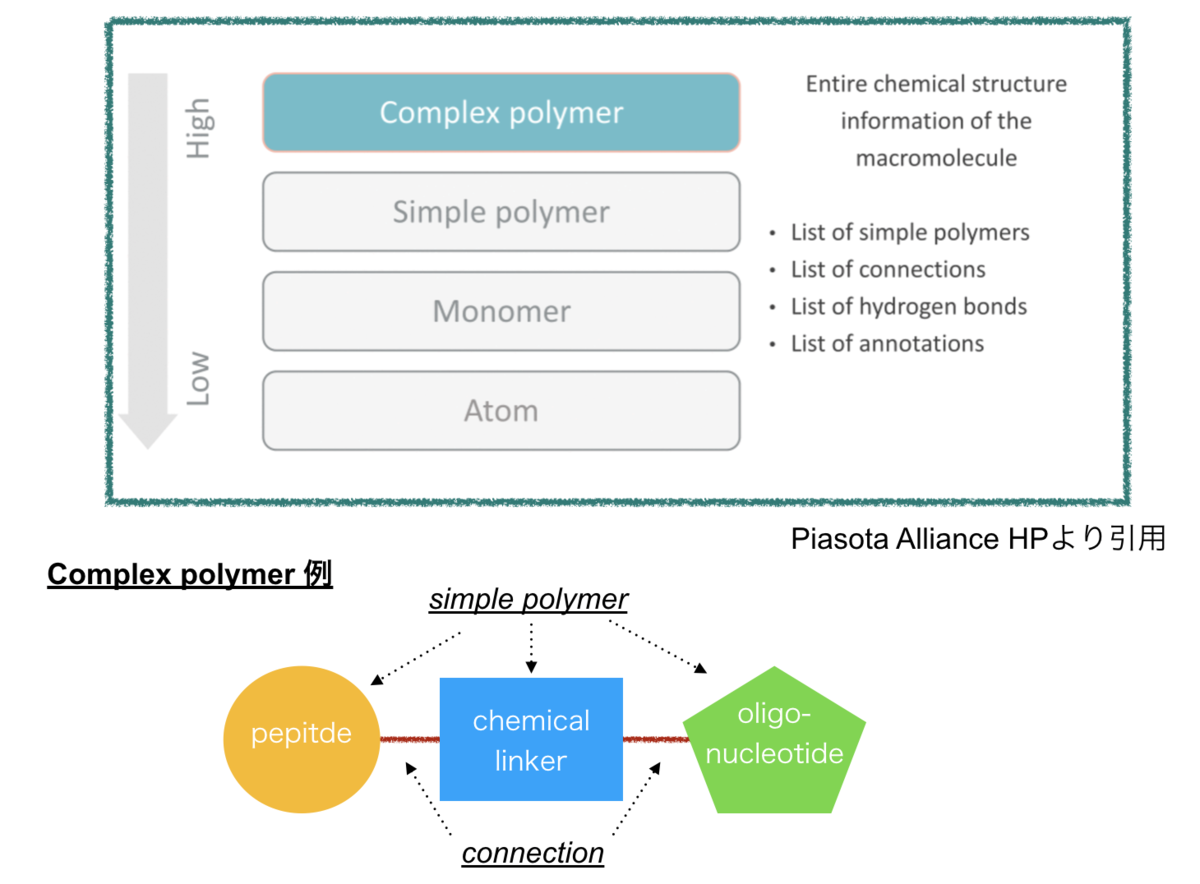
① Complex polymer、② Simple polymer、③ Monomer、④ Atomの4階層で構成されています。
階層構造であることにより、文脈に応じて異なるレベルで構造の解析と描画を行うことができます。 また、高次の階層は低次の階層の表現をもちいて表すことができます。
Complex polymer
では、まず、一番高次のComplex Polymerから。この階層はマクロ分子全体の化学構造の情報を表現します。
下図の通り、構成要素であるsimple polymerとそれらをつなぐ接続(connection)によって表すことができます。
Simple polymer
Simple polymerは同じpolymerタイプのmonomerから構成されています。
複雑さを避けるため単一の直鎖(single linear chain)として定義されています。 従って分岐(branching)や環状(cyclization)といった構造はこの階層では扱いません。
また、特定のpolymerタイプにはmonomer間の接続(connection)のルールが明示的に定義されており、 接続の位置とルールによってmonomer配列の方向性が表せるようになっています (ex. ペプチド:N末端→C末端)。

Monomer
Monomerは原子(atom)と結合(bond)から構成されており、MolfileやSMILESといったフォーマットで表すことができます。 ペプチドにおけるアミノ酸の表記(ex. A, G)と同様に、各monomerには単一のID(unique ID)が与えられており、 IDを使ってsimple polymerを表すことができます。
また、monomerの定義の中には接続の位置(attachment point)も含まれています。
monomerはsimple polymerのビルディングブロックとしても、複数のpolymer間をつなぐ接続要素としても用いることができます。

Atom

以上がHELMの階層構造でした。
各階層のプロパティと表記のルール
続いて、各階層のより具体的な表記方法、特徴を見ていきます。最終的にこんな感じの表記が理解できるようになりたい!
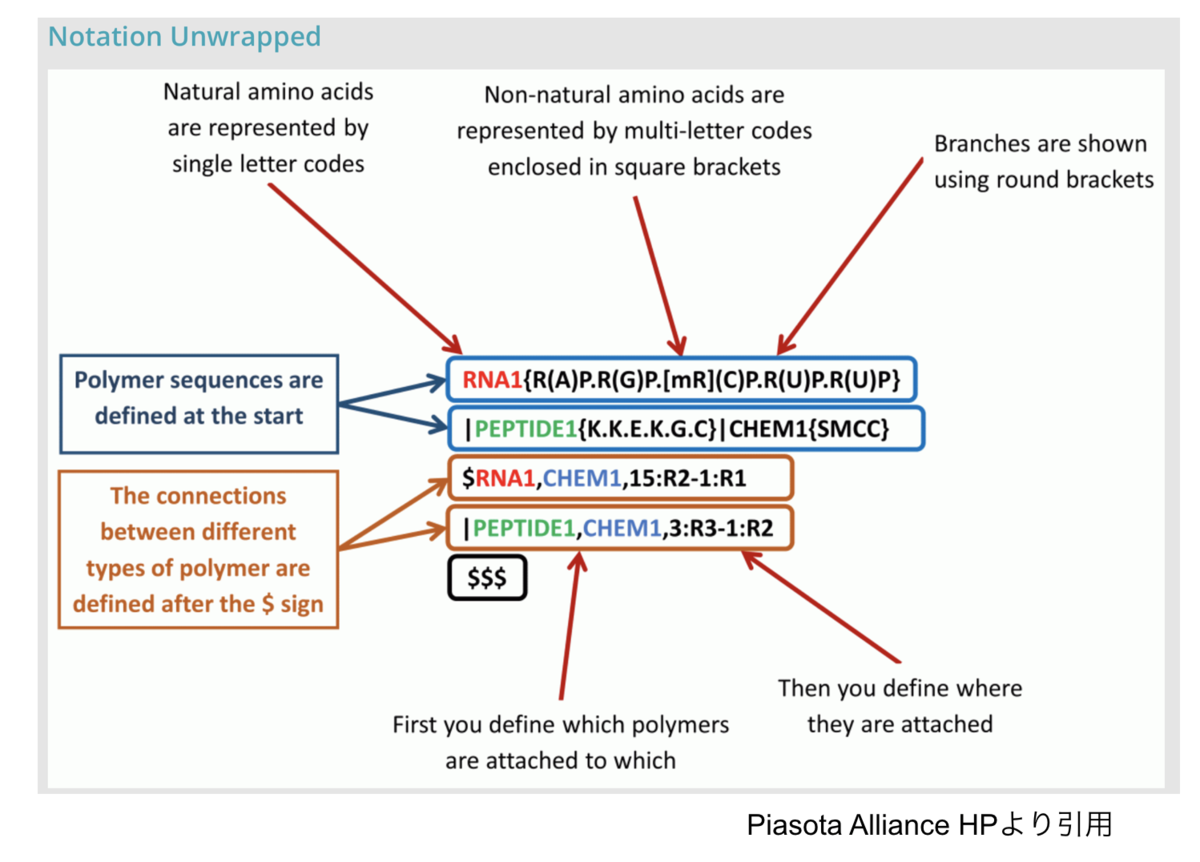
Monomer definition
HELMの表現の簡潔さはMonomerを単一のIDで表せることによるところが大きいと思われます。
従って、各Monomerがしっかりと定義されていることが重要となります。例えばChEMBLではHELM表現と一緒にMonomerの辞書(monomer library)も提供されていたりします。
ではそのMonomerがどのようなプロパティをもっているのか?先の図を拡大して見てみましょう。

プロパティの① ~ ③はこんな感じ。Attachment pointがちょっと細かい。

後半④ ~ ⑦はこんな感じ。

文字ばっかりの説明になってしまいました。
イメージを掴むために論文からFig.2を引用させていただきましょう。
ペプチド鎖のHELM表記の中で、monomerはこんな感じででてきます。
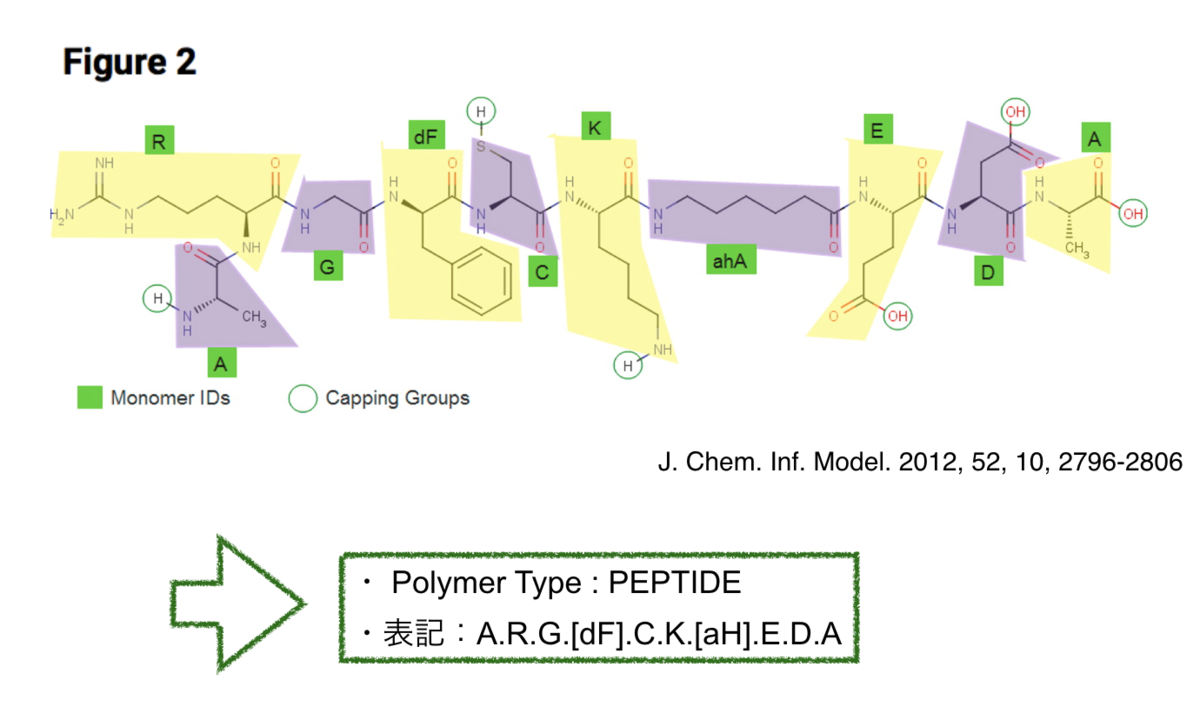
Attachment pointにおけるCapping groupや、 非天然のアミノ酸とNatural analogの表記の比較がイメージしやすいですね。
Simple Polymer の記載の仕方
Simple polymerの表記はmonomerの定義に基づいています。
特にプロパティの⑥ monomer typeと ③ attachment point が大事で、タイプによってR1、R2、R3の意味合いが変わってきます。

backboneであればR1、R2は別のbackbone monomerと接続して主鎖が伸びる場所で、 branchingであればR1はbackboneのR3のと接続する場所となります。
先のアラニンのmonomer定義でプロパティのSMILES表記は以下のようになっていました。伸張の方向性(N末からC末)からattachment pointがR1、R2となっているのが分かりやすいですね。
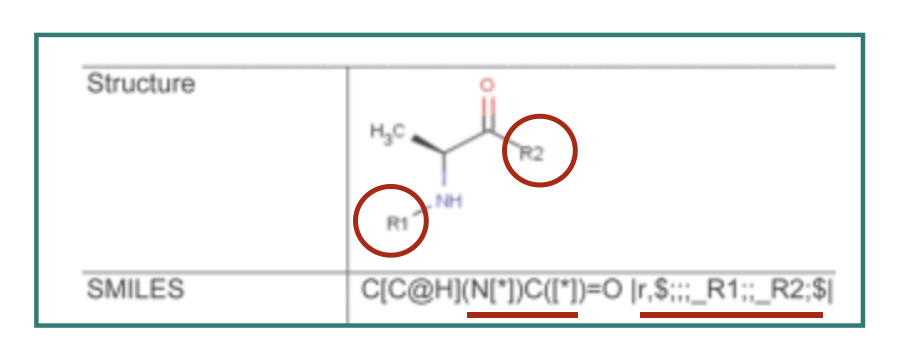
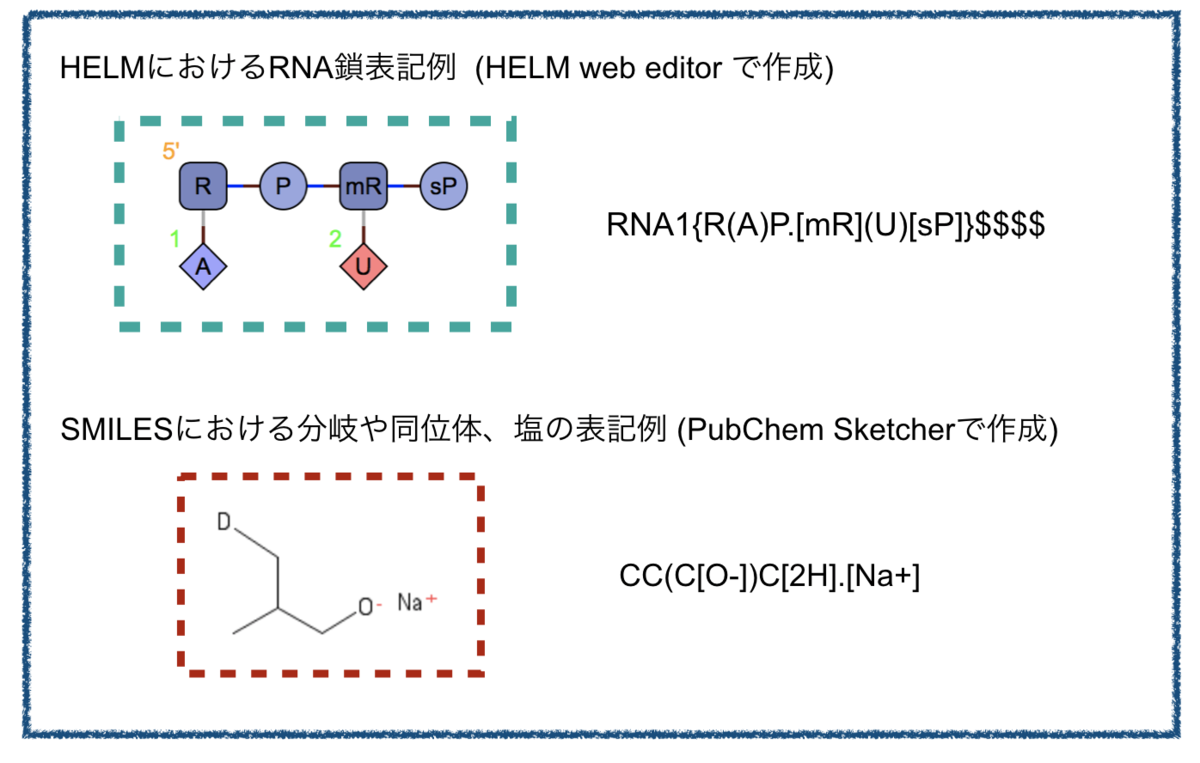
で、具体的な線形表記のルールですが、SMILESと非常に類似しています。こんな感じ、、、

適当に比較例をつくってみました。分岐や特殊なユニットの表し方といった文法が似てますね!
Simple Polymer Type の分類
ざっくりこんなタイプにわかれます。
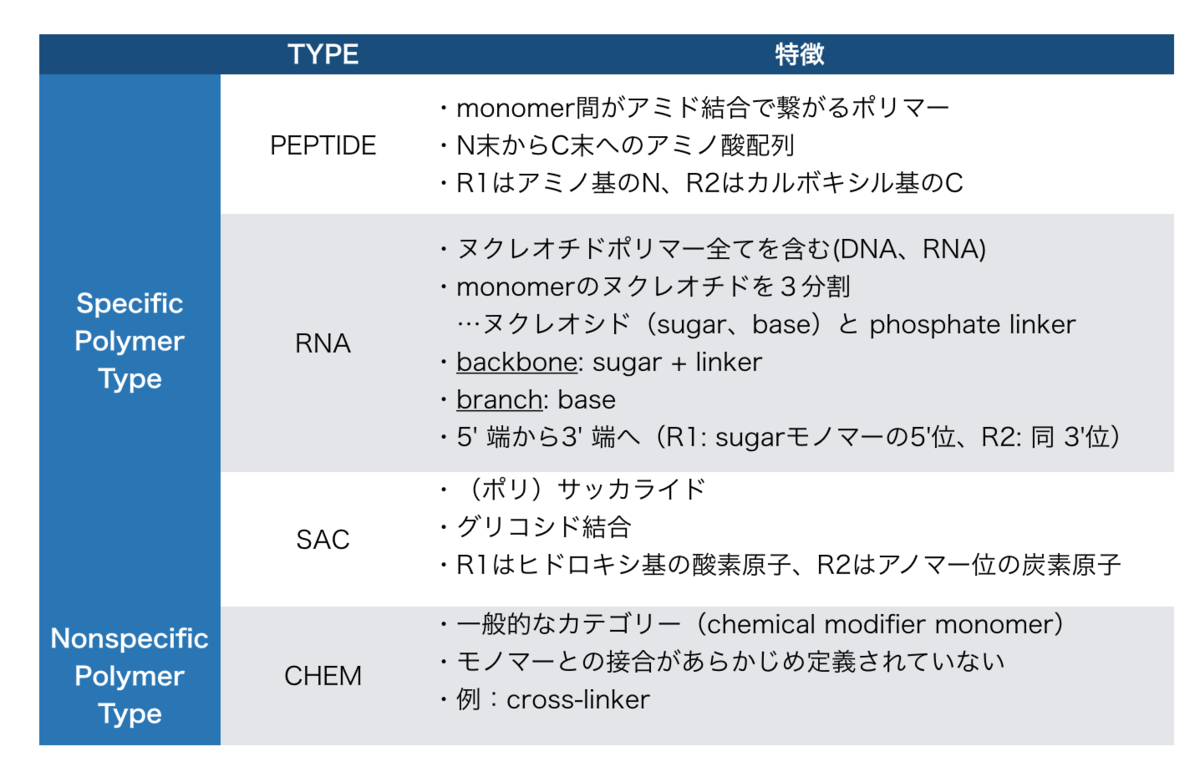
RNA TYPEのmonomerを3つの部分(sugar、base、linker)に分けて定義しているのは、 まとめて定義すると組み合わせごとに別の定義が必要になり、膨大な数になってしまうからだそうです。
またNonespecificなCHEM TYPEでは、モノマー間の接続が定義されていないので、 モノマー1つずつを別々のsimple CHEM polymerとして扱います。
従ってCHEMでは複数モノマーの表現がcomplex polymer notationの範疇で取り扱われる事になります。
Complex Polymer Notation Specification
複数のsimple polymerの組み合わせからなるcomplex polymerには、 Conjugated Polymers (ex. protein-drug conjugate)、 Cross-linked polymers (ex. antisense oligonucleoide dimers)、 Polymer mixtures (ex. siRNA) といったものがあります。
表記の特徴はこんな感じ・・・

各セクションは以下のようになっています。

また、simple polymerは一本鎖なので、環状構造(Cycles)や分岐鎖(Branches)はsimple polymer間を接続する事で表現されます。
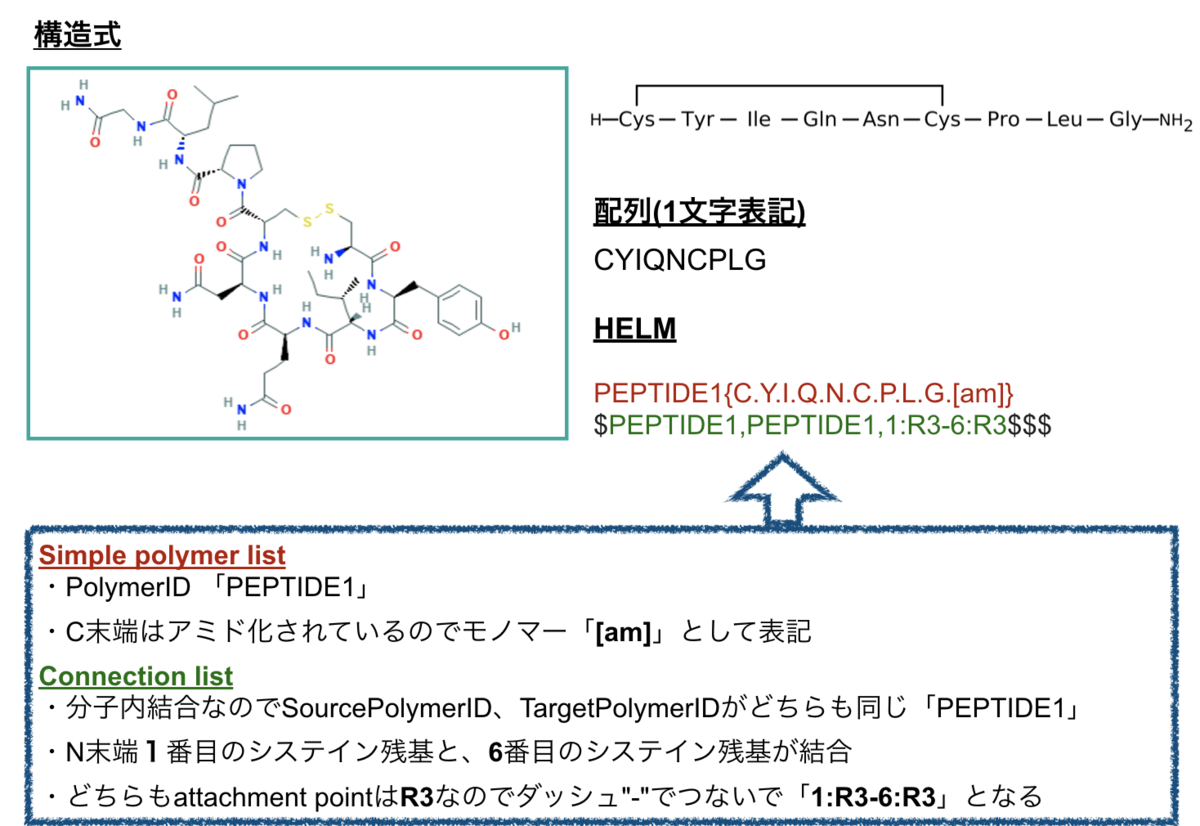
環状構造の具体例として、ペプチドホルモンオキシトシンのPubChem上の表記を見てみましょう。 2つのシステイン残基がジスルフィド結合で繋がれています。
以上でHELMの各階層の表記方法の説明はおしまいです。
HELMの表記例
表記方法が大体わかったので例をもう少し見て見ましょう。
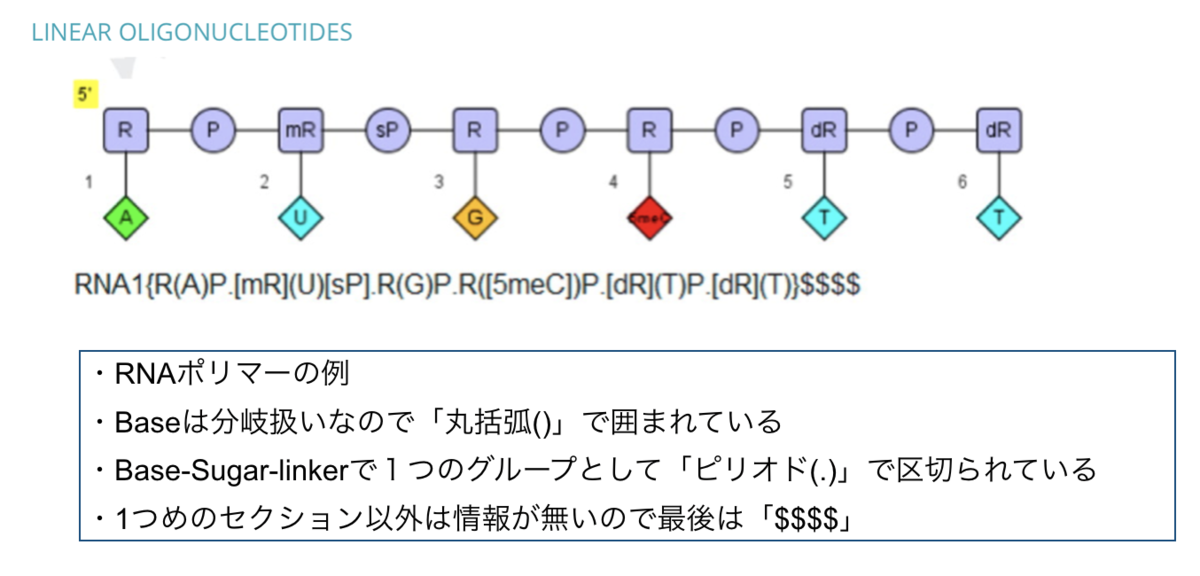
以下はいずれもPistoia Alliance HPに記載の例です。Complex polymerのグラフ表現とHELM表記とが並んでいます。
グラフは見慣れない図ですが、monomerをベースにした表現で、HELM web editor を使って同様の図を作成できます。*11
まずは直鎖のオリゴヌクレオチド…。塩基も糖もリン酸も修飾が入ったゴリゴリした例です。
続いて環状ペプチド。HELM表記は先にみたオキシトシンと同様に解釈できます。 monomerを1文字表記したグラフ表現はPubChemで見た構造式と比べてより簡潔な表現になっているように見えます。
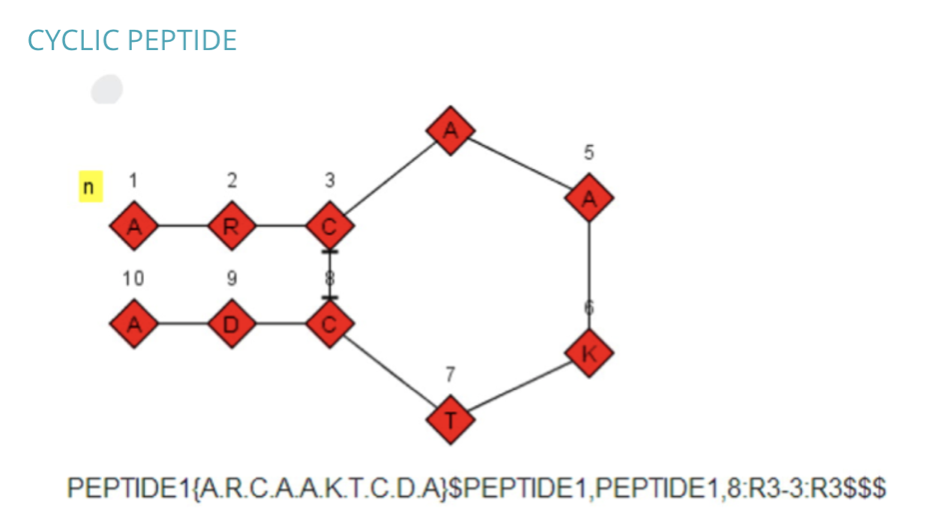
・・・赤色背景に黒字はColor Blind Friendlyじゃ無いので読むのがつらいです。。。カラーユニバーサルデザインでお願いします。。。
つぎはsiRNA、塩基対で水素結合を形成し2本鎖となっています。Hydrogen bond list、Attribute listの参考例としてどうぞー
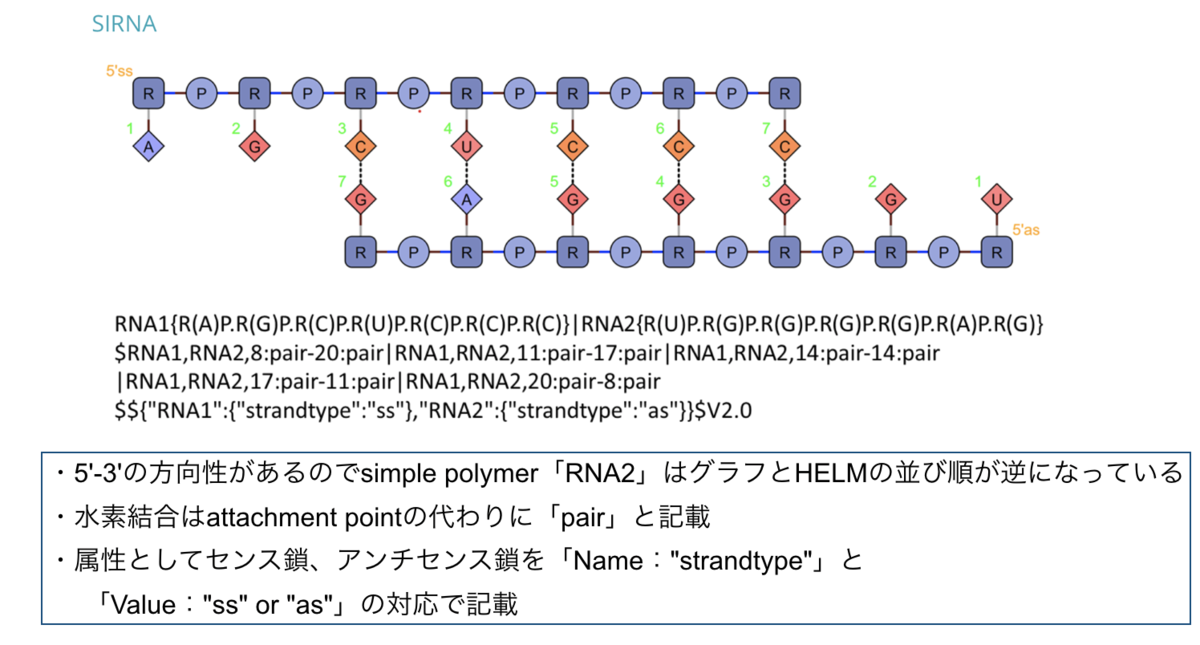
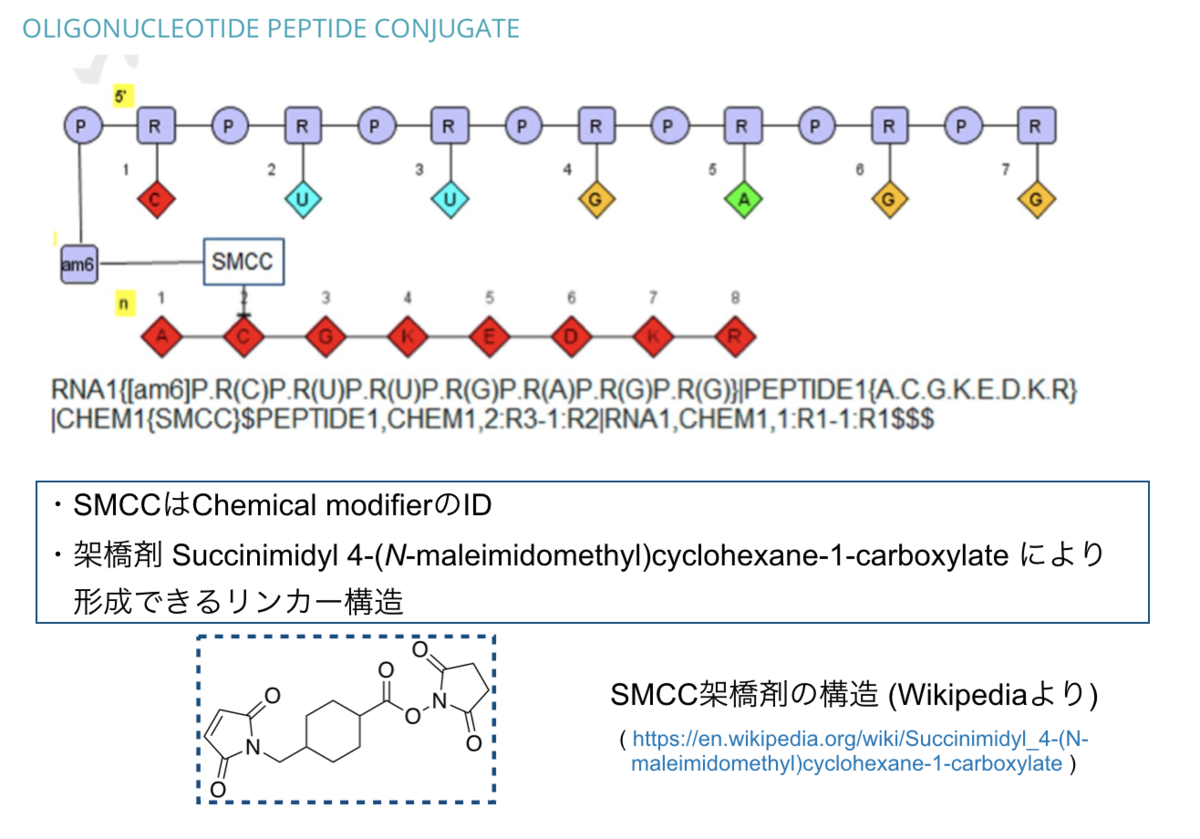
最後はオリゴヌクレオチドとペプチドのコンジュゲートです。
異なるPolymer typeの組み合わせの例。Non-specific polymerのCHEMも使われています。
以上、具体例でした。
HELMのバージョン
ここまでHELMの階層構造と表記方法について見てきましたが、HELMのバージョンについても触れておきます。
現在2つのバージョンがあり、新しいHELM2ではHELM1よりもさらに表現の幅が広がっています。*12
HELM1では自由にmonomerの定義を加えていく事で非天然のモノマーを含めていくことができました。 HELM2ではさらに定義可能な範囲が広がり、混合物のような曖昧さのある構造も表現可能となっています。

ChEMBLとRDKitにおけるHELM
オープンソース化されたマクロ分子の表記HELM。オープンなものとしてやっぱりChEMBLとRDKitは外せないですよね!
ChEMBLでは2015年リリースのChEMBL 20からHELMを含めるようになったようです。*13 ちなみに2020年10月現在のバージョンはChEMBL 27です。
PubChemでも検索してみたオキシトシンを調べてみましょう。
疲れた心と体に幸せホルモンを!!*14
ID CHEMBL395429でヒットしました。 Compound Report Cardをスクロールしていくと「HELM Notation」という項目があります。

PubChemと比較すると、わずかに異なるHELM表記となっていました。ChEMBLではconnection listのSourceとTargetが反対となっています(「6:R3-1:R3」)。
分子内ジスルフィド結合なのでどちらでも良いですが、個人的にはPubChemの数字が小さい方を先にする書き方の方が好きです。
また、monomerの定義はXML形式でダウンロードすることもできます。サイズ 3.9MBもある。。。
続いてRDKit!
現在の所、ペプチドだけサポートされているみたいです。まずはHELMからMolオブジェクトを作ってみましょう!
ライブラリをインポート・・・
from rdkit import rdBase, Chem from rdkit.Chem import Draw, rdDepictor from rdkit.Chem.Draw import IPythonConsole print('rdkit version: ', rdBase.rdkitVersion) # rdkit version: 2020.03.2
SMILESからMolオブジェクトをつくるときと同じノリでMolFromHELM( )とすればOKです!
Oxytocin_mol = Chem.MolFromHELM('PEPTIDE1{C.Y.I.Q.N.C.P.L.G.[am]}$PEPTIDE1,PEPTIDE1,1:R3-6:R3$$$')
Oxytocin_mol
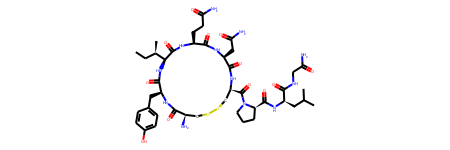
Molオブジェクトが無事生成されました! でももっと可愛く! *16
rdDepictor.SetPreferCoordGen(True)
Oxytocin_mol
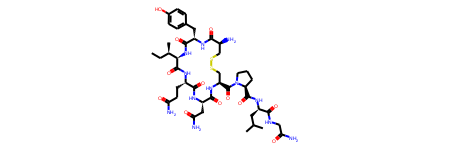
CoordGen良い!! 幸せホルモン分泌された!
逆にMolオブジェクトからHELM表記を生成してみます。こっちもいつものノリでMolToHELM( )とすればOKです!
Oxytocin_helm = Chem.MolToHELM(Oxytocin_mol) print("Generated HELM Notation: ", Oxytocin_helm)
「Generated HELM Notation: PEPTIDE1{C.Y.I.Q.N.C.P.L.G.[am]}$PEPTIDE1,PEPTIDE1,1:R3-6:R3$$$」と出力されました。
バッチリっすね! もうMolToHELMキャンペーンしたいくらい!
まとめ
以上、マクロ分子の線形表記 HELMついてでした。相変わらず大事なポイントを絞れないので無駄に長くなってしまいました。。。 まぁ、なんとなく表記の読み方もわかるようになったのでいいや。
さてHELMですが、原子数がべらぼうに多い巨大な分子でも簡潔に線形表記できてしまうところが素敵と思いました。
J.Cheminformのレビューでは、HELMのような表現は生物と化学の融合地点としてのBiocheminformatics的表現といったことが書かれていました(雰囲気意訳)。
冒頭にも書きましたが、化学修飾された生物学的分子を表現できることで、技術発展が著しいモダリティ分野をインフォマティクスの観点からもっと取り扱えるようになりそうです。
っていうか、もうできてるのか!知らなかったの私だけだったのか!!
無知を晒しついでの感想ですが、私はケモインフォを齧り始めて、「コンピューターに分子を認識させるのってこんなに難しいことだったのか!」とびっくりしました。
正直SMILESやら今回のHELMやら「なぜそれで一意に構造に対応させられるか?」といったことや、「どうやってこのルールに絞り込んだのか?」みたいなことはさっぱり分かりません。
ですが賢い人々が考えた表記方法を調べてみると新しい言葉を習う感じの面白さがあります。
ロゴスはロジックだから!・・・知らんけど!
適当に調べたことを書いているので色々と間違いがあると思います。ご指摘いただけると幸いです。
*1:関連するレビューを読んだりRDKit UGMをみたりしたところだったので、タイミングが良すぎてとても突き刺さりました。いや、本当に。
*2: こっちの記事でその他の表現とまとめているのでよろしければどうぞー
*3:図はHELM wiki、J. Cheminform. 2020(12)56のFigureを元に作成
*4:1. Whit Is HELM?(Scilligence blog記載より) 2. Hierarchical editing language for macromolecules (Wikipedia)
*5:J. Chem. Inf. Model. 2012(52)2796
*6:The Pistoia Alliance HELM project announces free MarvinBeans 5.0 licenses and integration of HELM with the RDKit cheminformatics suite(Pistoia Alliance HPより)
*7:https://www.pistoiaalliance.org より
*8:ABOUT THE PISTOIA ALLIANCE (Pistoia Alliance HPより)
*9:Oct. 19th 2020 CAS Release情報より
*10: https://pubchem.ncbi.nlm.nih.gov/compound/439302
*14:突然漂う不穏な空気
*15:https://www.ebi.ac.uk/chembl/compound_report_card/CHEMBL395429/
*16:化学の新しいカタチ「RDKitと二次元構造式:CoordGenで大員環化合物を綺麗に描画」を参考
オープンソースで逆合成解析 ~AiZynthFinder~ part 3. CLI
オープンソース自動逆合成解析ツール、AiZinthFinder。インターフェースとしてGUIとコマンドラインの2種類が用意されています。
Command Line Interface (CLI)の利点はバッチ処理ができることです。ポチッとやって多数の分子を処理できたら楽そうですね!
ツールは以下のGitHubに、
ドキュメントはこちらにあります。
課題設定
逆合成解析のバッチ処理の利用として、AI-drivenな創薬のDMTA探索サイクルが想定されているのだと思います。(生成モデルでデザインされた化合物をそのまま逆合成解析、合成ロボットに流す、みたいな?)
ですが、私はどうして良いのかわからないので、もう少し簡単な課題を設定したいと思います。
つまるところ、効率よく多種類の化合物を手に入れたいという課題です。そのためにはちょうど良い共通中間体を設定し、できるだけ少ない反応の数で異なる目的物が得られるようにすればOKです。
もう少し遡ると、化合物群に共通して必要となる試薬を確保する必要がある、ってなことになりそうです。
と、いうわけで、今回の目標は、「論文記載の化合物群をAiZynthFInderのCLIでまとめて逆合成解析し、それらの合成に必要な共通の試薬をみつけよう!」です。
データの準備
AiZynthiFinderのCLIで解析を行うには「SMILESが1行1化合物分格納されたテキストファイル」必要になります。早速準備していきましょう。
対象のデータとしては、py4chemoinformatics 第8章で用意してくださっているOrexin Receptorアンタゴニストを利用させていただきたいと思います。
Table 1から下記のMerckの文献3つを選び、ChEMBLから化合物をSDFでダウンロードしてきました。
| Doc ID | Journal |
|---|---|
| CHEMBL3098111 | Bioorg. Med. Chem. Lett. (2013) 23:6620-6624 |
| CHEMBL3867477 | Bioorg Med Chem Lett (2016) 26:5809-5814 |
| CHEMBL3352684 | Bioorg. Med. Chem. Lett. (2014) 24:4884-4890 |
RDKitでSDFをテキストファイルに変換していきたいと思います。
まずはどんな化合物たちか見てみます。
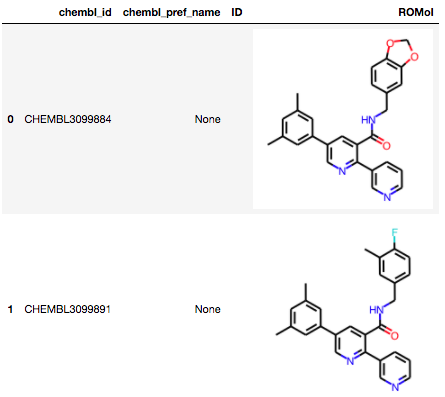
CHEMBL3098111はこんな化合物群、、、
from rdkit import Chem from rdkit.Chem import AllChem, PandasTools df_8111 = PandasTools.LoadSDF('./data_oxr/CHEMBL3098111.sdf') df_8111.head(2)
CHEMBL3867477はこんな感じ、、、
df_7477 = PandasTools.LoadSDF('./data_oxr/CHEMBL3867477.sdf') df_7477.head(2)
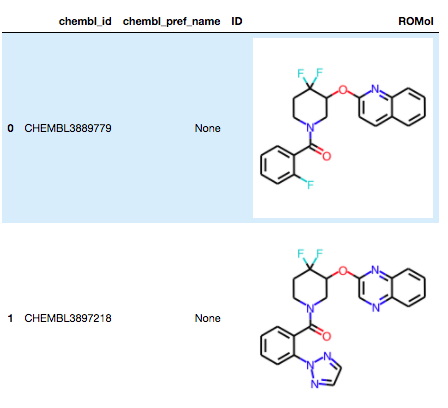
中央に位置する環がピリジンだったり4,4-ジフルオロピペリジンだったりするみたいですね。
では、テキストファイルに変換する関数をつくってみます。
一部、塩酸塩が含まれているようでしたので、SaltRemoverを使って脱塩処理をします。*1
また、重複した化合物を除くためにset()でリストからユニークなSMILESのみを残します。
こんな感じに書いてみました。
# SaltRemoverで脱塩処理を行うための設定 from rdkit.Chem import SaltRemover remover = SaltRemover.SaltRemover(defnData="[Cl]") def desalt_smis(sdf_data): # 空のリストを用意 desalted = [] Suppl = Chem.ForwardSDMolSupplier(sdf_data) for mol in Suppl: # 塩酸塩の脱塩処理 res = remover.StripMol(mol) # SMILESに変換 smi = Chem.MolToSmiles(res) desalted.append(smi) # unqueなSMILESのみ保持する unique_smi = set(desalted) return desalted
3つのSDFに適用してテキストファイルを作成します。「data_oxr」というディレクトリにSDFを保存したのでos.pathでパスを指定しました。
CHEMBL_IDs = ['CHEMBL3098111', 'CHEMBL3352684', 'CHEMBL3867477'] import os for chembl_id in CHEMBL_IDs: # SDFデータのパスを指定 sdf_file = os.path.join("data_oxr", chembl_id + '.sdf') # 作成した関数でSMILESに変換 chembl_smis = desalt_smis(sdf_file) # 出力テキストファイルを指定 txt_path = os.path.join("data_oxr", chembl_id + '.txt') with open(txt_path, mode='w') as f: for smi in chembl_smis: f.write(smi + '\n')
1行1化合物とするため、改行\nでつないでいます。
が、もっと簡単の方法があるらしい!
その名もSmilesWriter! @iwatobipen先生がこちらの記事で紹介してくださっていました。
折角なので使ってみます。SDFを入力としてテキストファイルを出力する関数を書き直してみます。
from rdkit.Chem.rdmolfiles import SmilesWriter def desalt_smi_write(sdf_data, text_file): # 空のリストを用意 desalted = [] Suppl = Chem.ForwardSDMolSupplier(sdf_data) for mol in Suppl: # 塩酸塩の脱塩処理 res = remover.StripMol(mol) desalted.append(res) # SDWriterで書き出し writer = SmilesWriter(text_file) for mol in desalted: writer.write(mol) writer.close return None for chembl_id in CHEMBL_IDs: # SDFデータのパスを指定 sdf_file = os.path.join("data_oxr", chembl_id + '.sdf') # 出力テキストファイルを指定 txt_path = os.path.join("data_oxr", chembl_id + '.txt') # SDFからテキストに変換する関数 desalt_smi_write(sdf_file, txt_path)
できました!3つとも無事テキストファイルとして出力されました。
今回は短いのであまりご利益を感じられないかもしれませんが、SMILESに変換してリストに格納する操作がいらなかったり、プロパティをもたせて書き出したりできるようなので便利そうです。
但し、SmilesWriterで書き出したテキストファイルは、各行のSMILESのあとに「空白 + インデックス」も追加されていました。
処理されるうえでは無視されるものかもしれませんが、AiZynthFinderで問題が起きないかわからないので、今回は無難に先に出力したファイルを利用することとします。
AiZynthFinder コマンドライン
データを格納したディレクトリ「data_oxr」に以下の4つも合わせて入れて起きます。
- config.yml
- zinc_stock_17_04_20.hdf5
- full_uspto_03_05_19_rollout_policy.hdf5
- full_uspto_03_05_19_unique_templates.hdf5
これらはデータと同じディレクトリにある必要はありませんが、configのパスを直すのが面倒なので入れてしまいました。・・・無精ですみません。
コマンド自体は簡単!ターミナルで作業ディレクトリを移動し、以下を実行するだけ*2。
export KMP_DUPLICATE_LIB_OK=TRUE aizynthcli --config config.yml --smiles CHEMBL3098111.txt
最初の1行は「OMP: Error #15」を回避するためのものなので、他の環境では必要ないかもしれません*3。
で、こんな感じでターミナルに出力されていきます。
Loading policy model from full_uspto_03_05_19_rollout_policy.hdf5 and templates from full_uspto_03_05_19_unique_templates.hdf5 to full_uspto Loading stock from zinc_stock_17_04_20.hdf5 to zinc Selected stocks: zinc Compounds in stock: 17422831 Selected policy is full_uspto Done with Cc1cc(C)cc(-c2cnc(-c3cccnc3)c(C(=O)NCc3ccc4c(c3)OCO4)c2)c1 in 70.4 s Done with Cc1cc(C)cc(-c2cnc(-c3cccnc3)c(C(=O)NCc3ccc(F)c(C)c3)c2)c1 in 69.8 s Done with COc1ccc(CNC(=O)c2cc(-c3cccc(Cl)c3)cnc2-c2cccnc2)cc1OC in 75.0 s
分子にもよりますが1化合物2分前後なので、10化合物で20~30分くらいをみておけば良いでしょうか? Macがウィンウィン唸るのを放っておくと、「output.hdf5」というhdf5ファイルが出力されます。
ファイル名が重なると上書きされてしまいそうなので、それぞれChEMBL IDを後ろにつけて別のフォルダに保存しておきました。
では、解析結果を見てみましょう!
バッチ処理結果解析
CHEMBL3098111
出力はhdf5なので、PandasのDataFrameに読み込むことができます。*4
import pandas as pd data_8111 = pd.read_hdf('./data_oxr/output_files/output_CHEMBL3098111.hdf5', "table")
こんな感じ
data_8111.head()
| target | search_time | number_of_nodes | max_transforms | max_children | top_score | is_solved | number_of_steps | number_of_precursors | number_of_precursors_in_stock | precursors_in_stock | precursors_not_in_stock | top_scores | trees | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 0 | Cc1cc(C)cc(-c2cnc(-c3cccnc3)c(C(=O)NCc3ccc4c(c... | 70.419213 | 545 | 7 | 19 | 0.986553 | True | 3 | 4 | 4 | OB(O)c1cccnc1, Cc1cc(C)cc(B(O)O)c1, NCc1ccc2c(... | 0.9866, 0.9866, 0.9866, 0.9866, 0.9866, 0.9866... | [{'type': 'mol', 'smiles': 'Cc1cc(C)cc(-c2cnc(... | |
| 1 | Cc1cc(C)cc(-c2cnc(-c3cccnc3)c(C(=O)NCc3ccc(F)c... | 69.782567 | 592 | 7 | 25 | 0.986553 | True | 3 | 4 | 4 | OB(O)c1cccnc1, Cc1cc(C)cc(B(O)O)c1, Cc1cc(CN)c... | 0.9866, 0.9866, 0.9866, 0.9866, 0.9750 | [{'type': 'mol', 'smiles': 'Cc1cc(C)cc(-c2cnc(... | |
| 2 | COc1ccc(CNC(=O)c2cc(-c3cccc(Cl)c3)cnc2-c2cccnc... | 74.974893 | 585 | 7 | 23 | 0.986553 | True | 3 | 4 | 4 | OB(O)c1cccc(Cl)c1, OB(O)c1cccnc1, COc1ccc(CN)c... | 0.9866, 0.9866, 0.9866, 0.9866, 0.9750 | [{'type': 'mol', 'smiles': 'COc1ccc(CNC(=O)c2c... | |
| 3 | Cc1cc(C)cc(-c2cnc(-c3cccnc3)c(C(=O)NCc3ccc(N(C... | 71.364885 | 590 | 7 | 22 | 0.986553 | True | 3 | 4 | 4 | OB(O)c1cccnc1, Cc1cc(C)cc(B(O)O)c1, CN(C)c1ccc... | 0.9866, 0.9866, 0.9866, 0.9866, 0.9866 | [{'type': 'mol', 'smiles': 'Cc1cc(C)cc(-c2cnc(... | |
| 4 | COc1ccc(CNC(=O)c2cc(-c3cc(F)cc(F)c3)cnc2-c2ccc... | 63.741923 | 587 | 7 | 22 | 0.986553 | True | 3 | 4 | 4 | OB(O)c1cc(F)cc(F)c1, OB(O)c1cccnc1, COc1ccc(CN... | 0.9866, 0.9866, 0.9866, 0.9750, 0.9634 | [{'type': 'mol', 'smiles': 'COc1ccc(CNC(=O)c2c... |
どのような合成ルートが提案されたか、画像ファイルとして出力することができます。
ドキュメントをコピペ・・・・
from aizynthfinder.analysis import ReactionTree # 全化合物について全てのツリーのリストを取得 all_trees = data_8111.trees.values # 最初の化合物についての情報を取り出す trees_for_first_target = all_trees[0] # 各ツリーを画像ファイル(png)として書き出していく for itree, tree in enumerate(trees_for_first_target): imagefile = f"route{itree:03d}.png" ReactionTree.from_dict(tree).to_image().save(imagefile)
実行すると最初のターゲット化合物について11個の合成ルートがそれぞれpngファイルとしてフォルダに出力されました。
こんな感じです。
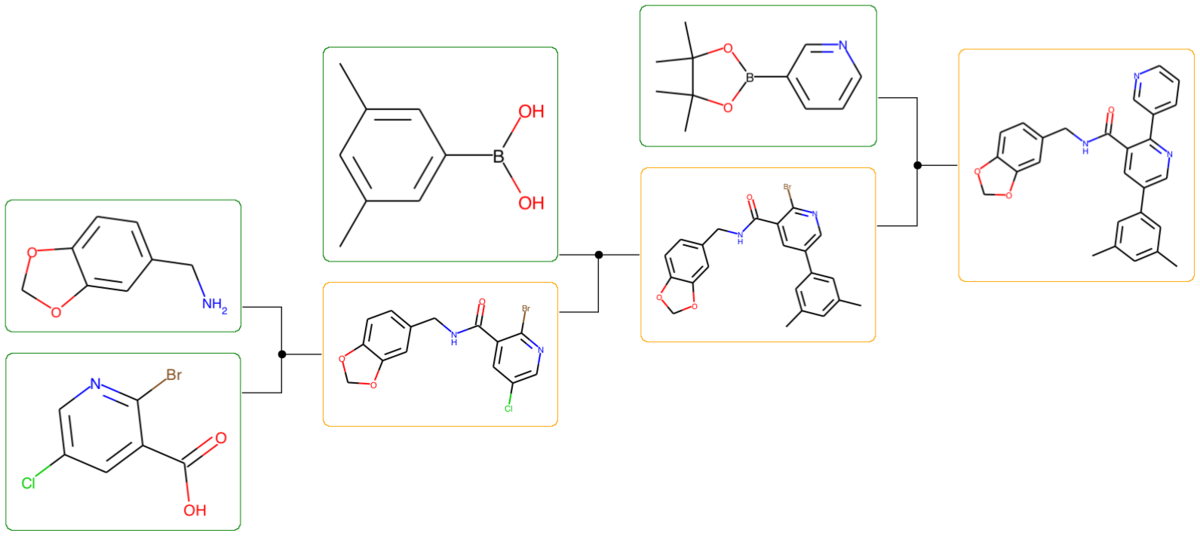
鈴木-宮浦クロスカップリングが2回使われています。ClとBrがあって先にClを反応させるルートになっています。反応性としては逆になってしまいそうです。
では、最初にもどって課題「論文記載の化合物群の合成に必要な共通の試薬」を検証してみたいと思います。
逆合成解析を進めた終点、試薬はDataFrameのprecursors_in_stockというcolumnに格納されています。
一つ目の例を取り出してみます。
precursors_8111 = data_8111['precursors_in_stock'] print(type(precursors_8111[0])) print(precursors_8111[0]) # <class 'str'> # OB(O)c1cccnc1, Cc1cc(C)cc(B(O)O)c1, NCc1ccc2c(c1)OCO2, O=C(O)c1cc(Br)cnc1Cl
str型で一つの文字列中に複数のSMILESが「カンマ+空白」(, )で区切られて格納されています。
構造式に起こしてみましょう。文字列をsplitで区切って、SMILES一つずつに分解した後、Molオブジェクトに変換しています。
precursor_mols = [] l = precursors_8111[0].split(', ') for smi in l: m = Chem.MolFromSmiles(smi) precursor_mols.append(m) Chem.Draw.MolsToGridImage(precursor_mols, molsPerRow=4)

合成ルートと見比べてみてきちんと目的の原料が取り出せているようです。
では、全ての化合物についてそれぞれで提案されているルートの出発原料を取り出し、集計してみます。
集計結果で出現頻度が多くなった原料は、複数の化合物の合成に必要となる試薬なので、優先順位高く確保する必要がありそうです。
具体的な処理としては、原料のSMILESをリストとしたのち、Python標準ライブラリcollectionsのcounterを使って出現回数に基づく処理を行います。*5
# 全化合物の全ての原料のSMILESを格納するためのリストを用意 precursors_list_8111 = [] for i in range(len(precursors_8111)): # 原料のSMILES 一つずつを要素としたリスト l = precursors_8111[i].split(', ') # extendでリストの要素を追加 precursors_list_8111.extend(l) import collections # Counterで各要素の出現頻度を数える c_8111 = collections.Counter(precursors_list_8111) # most_common()で出現頻度で並べ替え mc_8111 = c_8111.most_common() print(mc_8111) # [('OB(O)c1cccnc1', 17), ('O=C(O)c1cc(Cl)cnc1Cl', 10), ('Cc1cc(C)cc(B(O)O)c1', 9), ('COc1ccc(CN)cc1OC', 8), ('O=C(O)c1cc(Br)cnc1Cl', 6), ('OB(O)c1cc(Cl)cc(Cl)c1', 2), ('NCc1ccc2c(c1)OCO2', 1), ('Cc1cc(CN)ccc1F', 1), ('OB(O)c1cccc(Cl)c1', 1), ('CN(C)c1ccc(CN)cc1', 1), ('OB(O)c1cc(F)cc(F)c1', 1), ('CNCc1ccc2nccnc2c1', 1), ('CNCc1ccc2c(c1)OCO2', 1), ('COc1ccc(CN)cc1', 1), ('Cc1cc(C)cc(B2OC(C)(C)C(C)(C)O2)c1', 1), ('CN1CCc2cc(CN)ccc21', 1), ('COc1cccc(CN)c1', 1), ('NCc1ccc(C(F)(F)F)c(F)c1', 1), ('Cc1cc(F)cc(B(O)O)c1', 1), ('COC(=O)c1cc(Cl)cnc1Cl', 1), ('Nc1cc(C(F)(F)F)cc(C(F)(F)F)c1', 1), ('OB(O)c1cc(F)cc(Cl)c1', 1)]
(要素, 出現回数)という形のタプルが出現回数順に並んでいます。
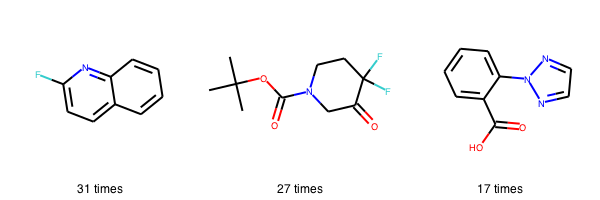
では、マストバイアイテム3つを見てみましょう!
must_buy_reagents = [] occurence = [] for i in range(3): smi = mc_8111[i][0] m = Chem.MolFromSmiles(smi) must_buy_reagents.append(m) occurence.append(str(mc_8111[i][1]) + ' times') Chem.Draw.MolsToGridImage(must_buy_reagents, legends=occurence)
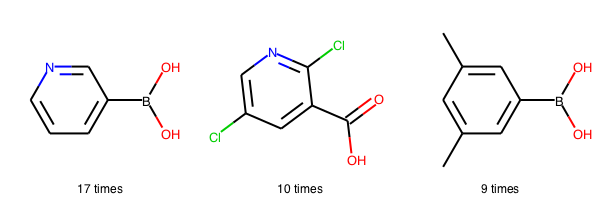
図の下、数字はこれらの試薬が出現した回数です。全17化合物ですので、3-ピリジンボロン酸はすべてに共通する試薬のようです。
とりあえずこの3つを購入してカップリングで骨格を作っておけば、様々なフラグメントとアミド結合をつくることで多種類の化合物を合成することができそうです。
カップリングの選択性は・・・頑張る!
CHEMBL3867477
CHEMBL3352684はCHEMBL3098111と構造が似ていたので割愛し、CHEMBL3867477の結果を見てみます。
まずは、提案ルート例から。コードは同じなので割愛します。
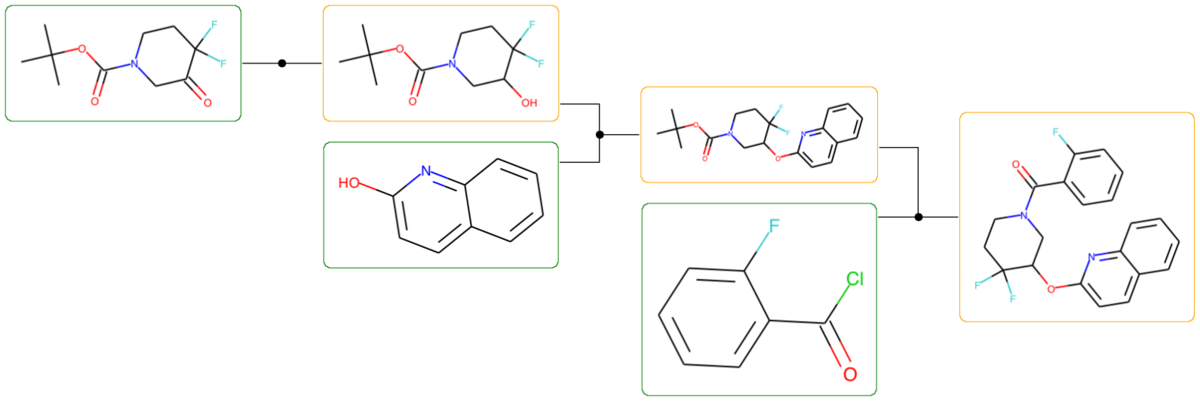
で、出現頻度の高い上位3試薬はこちら、、、
例に挙げた合成ルートでは、アルコールとフェノールの光延反応という感じですが、頻度の多い試薬を見るとフッ素原子を脱離基として求核置換のルートが多く提案されているようですね。
全49化合物ということを考えると、これらの試薬は半数前後で必要となる試薬です。これは買い、でしょうか???
まとめ
今回はAiZynthFinderのコマンドラインでバッチ処理を行ってみました。複数の化合物の解析結果、共通する原料・試薬があれば、多種類の化合物を作る上で重要となりそう、ということで検証してみました。
論文というストーリーのある化合物群なので、互いに類似した化合物群があげられている、という前提はありますが、頻度の高い重要そうな試薬が結構いい感じでみつかったかな、という印象です。
さて、なぜ今回こんな解析をしてみたか?
ここで思い出話を・・・
私は学生の頃、構造活性相関のようなテーマを行なっていました。
ダメな学生だったので指導教官から何度か急かされていたものの、ノラリクラリとかわしつつのんびり実験をしていました。
ある土曜の午後でした。TLCを焼いていると、指導教官が無言で足早に近づいてきます。
「早くテーブルの化合物作って!!!!」
温厚な指導教官も流石に堪忍袋の緒が切れたようでした。
やべぇやべぇ。至急、合成しなければ。。。
と、いうわけで多種類の化合物を急いで作らないといけないシチュエーションでした。
まずは共通の試薬を確保せねば!と、・・・必死でラボの試薬庫と冷蔵庫あさりました。
こんな時、今回みたいに重要な試薬がわかったら何を探せば良いか分かって楽だったかもしれませんね!
まあ、ちゃんと実験してればお叱り受けなかったですけどね!!
今回も色々と間違いが多いと思うので、ご指摘いただければ幸いです。
ではでは!
*2:aizynthfinder documentation参照
*3:前記事に記載したので省略します
オープンソースで逆合成解析 ~AiZynthFinder~ part 2. 遊んだ
オープンソースの自動逆合成解析ツール、AiZynthFInder。前回は背景を知るために文献を読んでみました。今回は実際に遊んでみたいと思います。
ツールは以下のGitHubに、
ドキュメントはこちらにあります。
インストール
GitHubのREADMEそのままにインストールしていきます。
Prerequisites
サポートされているOSはLinux、Windows、Macです。ツールの開発はLinuxで、Windows 10、macOS Catalinaではテスト済みとのこと。
ちなみに私のPCはmacOS High Serraです。後ほどいくつか躓いた点を書きますが、他のOSでは当てはまらないかもしれません。
あと、Python 3.6か3.7のanacondaかminicondaのインストールが必要です。
Installation
まずcondaのパッケージを3つインストール。
RDkitユーザーおなじみの仮想環境 my-rdkit-env に入れてみます。アクティベート! !
conda activate my-rdkit-env # お好みの環境でどうぞ conda install -c rdkit "rdkit=>2019.09.1" -y conda install -c anaconda tensorflow>=2.1.0 -y conda install graphviz -y
ついでAiZynthFinderパッケージをインストール
python -m pip install https://github.com/MolecularAI/aizynthfinder/archive/v1.0.0.tar.gz
簡単! でもこれでも失敗するんですよ、私は。
インストールの注意点
起動するとこんな感じになる予定でしたが、私はSMILESを入力しても構造式が描画されませんでした。
RDKitのモルオブジェクトがある、みたいな表示はされましたが、、、
READMEを見直すと、Graphvizに問題がありそうでした。インストールの際に代わりに-c anacondaを使ってくださいとのことだったので、こんな感じでやってみたら解決しました。
conda install -c anaconda graphviz -y
違いがわからない…
他に必要なもの
ツールを使うには以下の3つ用意する必要があります(ファイル的には4つ)。
- stock file : 逆合成の終点となる前駆体(購入可能な試薬)のリスト
- 訓練済みの rollout policy network : Kerasモデルと、ユニークなテンプレートのリスト(計2ファイル)
- configuration file
ありがたいことに1と2はfigshare からダウンロードできるようにしてくださっています。
以下の3つのファイルがダウンロードできます。結構大きいのでちょっと時間がかかります...
- zinc_stock_17_04_20.hdf5 (約650MB)
- full_uspto_03_05_19_rollout_policy.hdf5 (約300MB)
- full_uspto_03_05_19_unique_templates.hdf5 (約45MB)
ファイル名からstockはZINC、rollout policy model と template はUSPTOをベースとしてそうな感じがわかりますね。
で、figshareからはダウンロードした場合はconfiguration fileは自分で用意する必要があります。
ドキュメンテーションのConfiguration file を参考に、こんな感じのテキストファイルを書いて「config.yml」として保存しました。
policy:
files:
full_uspto:
- full_uspto_03_05_19_rollout_policy.hdf5
- full_uspto_03_05_19_unique_templates.hdf5
stock:
files:
zinc: zinc_stock_17_04_20.hdf5
面倒!! という方のためにもっと簡単な方法も用意されています。
次のコマンドで指定したフォルダにデータのダウンロードとconfigファイルの作成まで行ってくれます。「my_folder」のところはお好きなフォルダ名に変えてください。
download_public_data my_folder
著者親切すぎる・・・。
さて、ファイルが4つとも用意できたら、これらを全部作業ディレクトリに入れます。ディレクトリを移動して起動だ!!
・・・の前に、configの注意点を少し。
configの注意点
configuration fileはインターフェースにpolicy modelとstockへのファイルパスと、探索の詳細なパラメータを追加で指定するためのものです。
ですので、ファイルを作業ディレクトリ直下やパスの通っていない場所に置いておくとエラーが出てしまいます。
私は最初、作業ディレクトリ内に「data」というフォルダを作ってそこに「~.hdf5」ファイルを入れていました。この状態で先の「config.yml」を使うと以下のエラーが出ました。
OSError: SavedModel file does not exist at: full_uspto_03_05_19_rollout_policy.hdf5/{saved_model.pbtxt|saved_model.pb}
だよね! 見つかんないよね! 指定してないもんね!
相対パスに直したら動きました(「./data/」を追加)。
# config.yml
policy:
files:
full_uspto:
- ./data/full_uspto_03_05_19_rollout_policy.hdf5
- ./data/full_uspto_03_05_19_unique_templates.hdf5
stock:
files:
zinc: ./data/zinc_stock_17_04_20.hdf5
わかってみれば簡単でした。パス苦手・・・。
GUIアプリ
AiZynthFinderはJupyter Notebook上で使えるGUIと、コマンドベースのCUI両方が提供されています。
GUIの利点は何と言っても構造式を見ながら使えることでしょうか?
一方、CUIはBatchモードが使えるので複数の分子を一度に処理できるという利点があります。
ここは、コマンド苦手だからGUI一択で!
まずは作業ディレクトリに移動してJupyter notebookを起動します。
ちなみに、aizynthapp --config config_local.ymlとするとノートブックを自動で作ることもできるらしい。至れり尽くせり。。。
以下、2行をコピペしてCtrl+Enterでインターフェースが起動します。
from aizynthfinder.interfaces import AiZynthApp app = AiZynthApp("./config.yml")
出た!! さあ、逆合成開始だ!・・・と思いましたが、ここでもまた問題が! Kernelが死ぬ。。。
OMPとKernelの死(Mac限定?)
さて、インターフェースが起動したので早速検索を実行したところ、直ぐに画面が灰色になり「kernel die」と表示されnotebookが止まってしまいました。
・・・ YOU LOSE 的な?
ターミナルをみると以下のエラーが出てました。
OMP: Error #15: Initializing libiomp5.dylib, but found libiomp5.dylib already initialized. OMP: Hint This means that multiple copies of the OpenMP runtime have been linked into the program. That is dangerous, since it can degrade performance or cause incorrect results. The best thing to do is to ensure that only a single OpenMP runtime is linked into the process, e.g. by avoiding static linking of the OpenMP runtime in any library. As an unsafe, unsupported, undocumented workaround you can set the environment variable KMP_DUPLICATE_LIB_OK=TRUE to allow the program to continue to execute, but that may cause crashes or silently produce incorrect results. For more information, please see http://www.intel.com/software/products/support/.
どうもOpenMPというのがよろしくないらしい。原因がわからない。。。
ヒントに安全じゃないけど、と断りつつ回避方法も書いてあるのでそのままやってみます。Notebook上で環境変数を書き換えます、、、 *1
import os os.environ['KMP_DUPLICATE_LIB_OK']='TRUE'
パス苦手・・・。
この後、再度GUIを起動するコマンドをうつと今度は「kernel die」することなく検索できました!
I win !!!
もうちょっと調べるとmacOSでcondaを使っていることが原因な疑惑が出てきました。( stack overflowの記載より )
雰囲気こんな感じ・・・
- Condaは IntelのMKL optimizationsを使っていてそれにOpenMPが使われている。
- macOSはMKLではなく、独自のアルゴリズムを使っていてそこに既にOpenMPが使われいる。
- なのでOpenMPが重複することになり先のエラーが出る
解決方法として、MKLサポートなしでパッケージを再イントールすること(conda install nomkl)が書かれていました。試してませんが備忘録として・・・。
詳しい方ご存知でしたら教えていただけると嬉しいです。
逆合成してみた
やっと準備が整ったので、気を取り直して再度実行。
まとめると私の場合これを打ち込めばOK!
import os os.environ['KMP_DUPLICATE_LIB_OK']='TRUE' from aizynthfinder.interfaces import AiZynthApp app = AiZynthApp("./config.yml")
Favipiravir
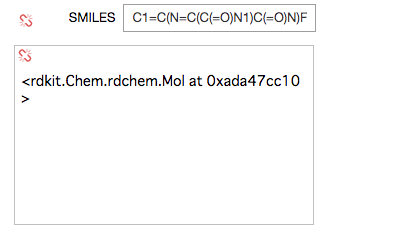
まずは小さなものから、ということで話題のアビガン(Favipiravir)!
PubChemによるとSMILESは「C1=C(N=C(C(=O)N1)C(=O)N)F」ということみたいです。
SMILESをコピペ + Enterで構造出ました!
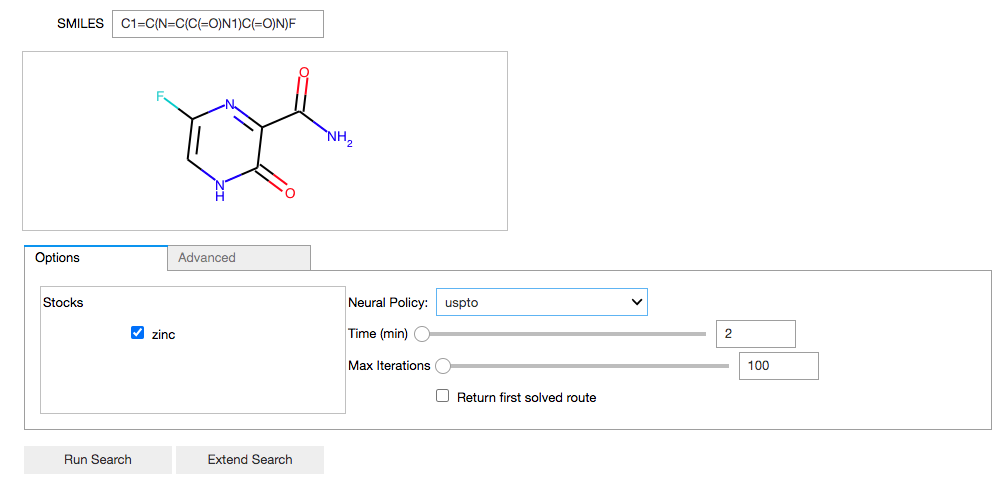
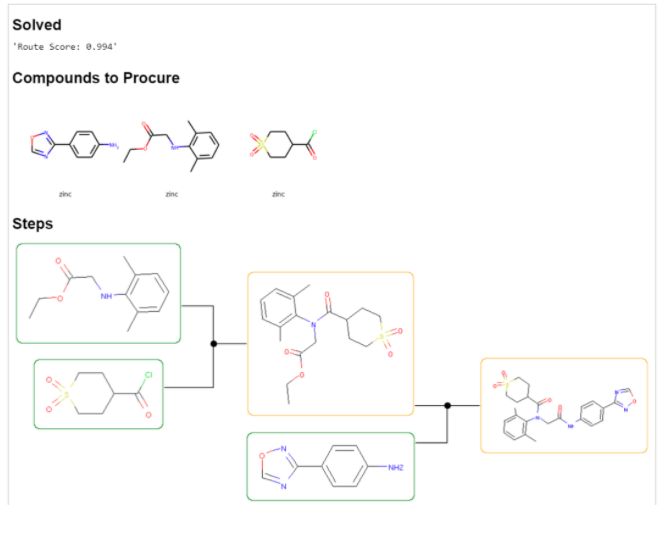
さあ、逆合成解析の実行だ! Run Search!!
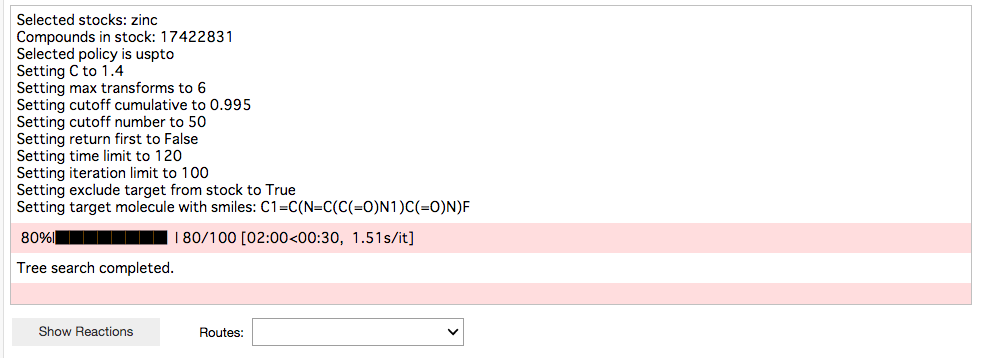
終わった。探索の最長時間 2分、最大イテレーション 100となっていたので、今回は2分経った時点で探索終了。イテレーションは80でした(80/100)。
どんな結果かみてみましょう。Show Reactionsをクリック!
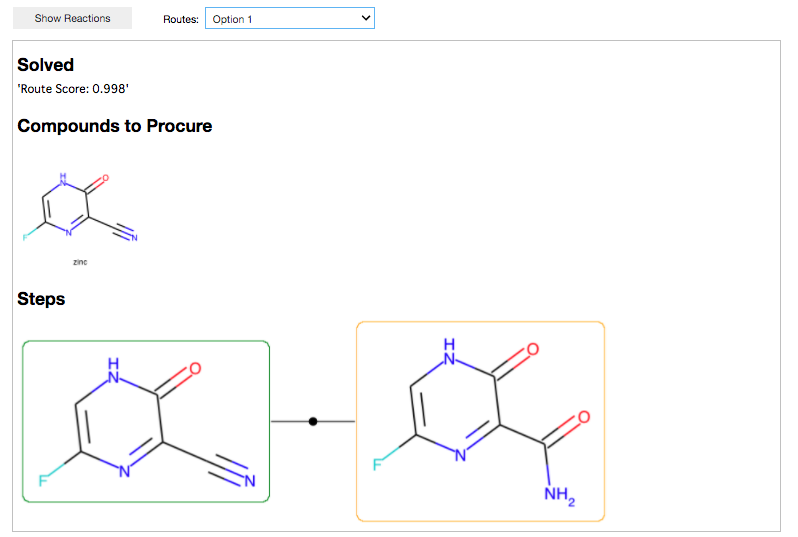
上位5つの結果から選択でき、上は最も良いスコアのルートです。
Compounds to ProcureがStock fileで指定したZincの中で見つかった手に入りそうな原料、Stepsが合成ルートです。
シアノ基の水和で1 stepのルート。ふむ、なるほど。。。
別のルートはどうでしょうか?
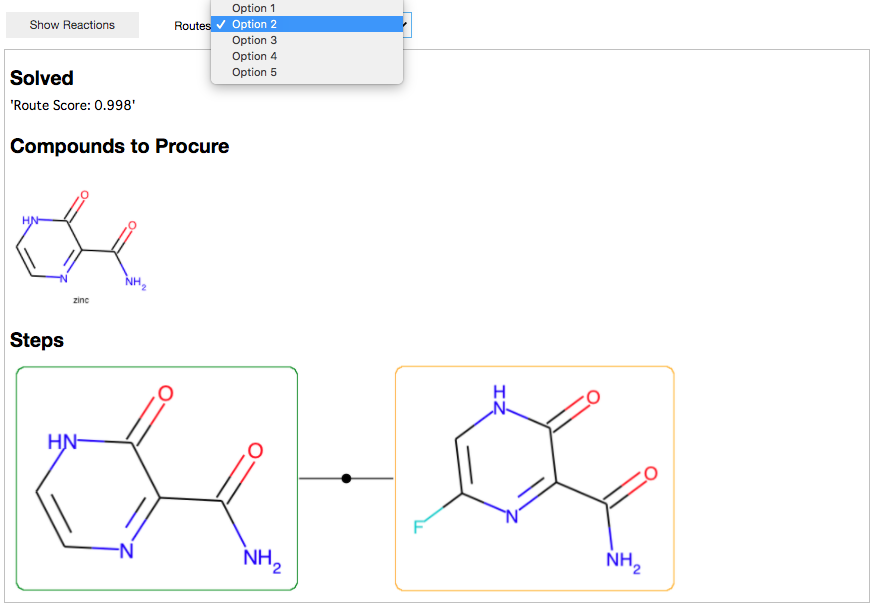
フッ素化・・・ミニスキ反応っぽいあれを使えばできる、、、のでしょうか?
3つめのルート。
N-oxideの転位で酸素を導入! ほーそういうのもあるのか!!
選択性の考慮は今後の課題と論文にありましたが、確かにこのルートでは酸化も転位も選択性が課題になりそうです。
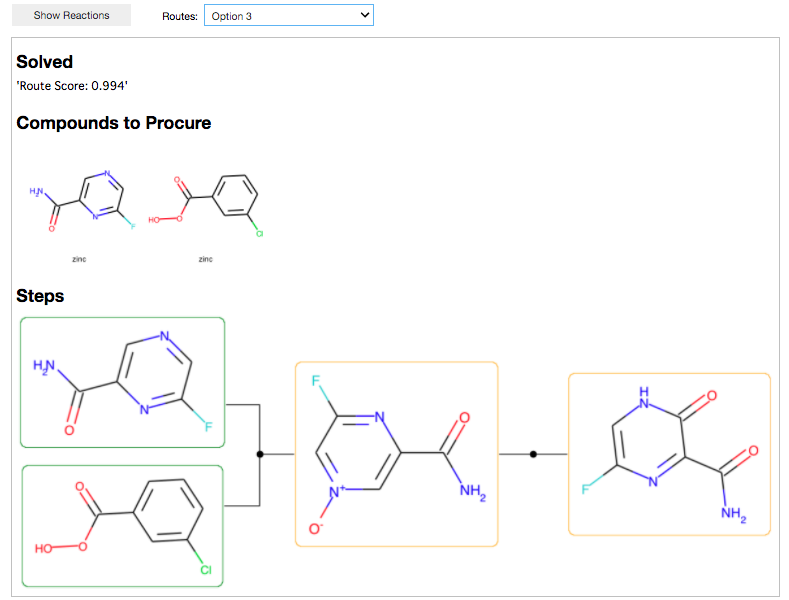
Remdesivir
もう少し大きな分子、ということで今度はレムデシビルを試してみます。
立体は考慮しないみたいですがとりあえずIsomeric SMILESは「CCC(CC)COC(=O)C@HNP@(OC[C@@H]1C@H(C#N)C2=CC=C3N2N=CN=C3N)O)O)OC4=CC=CC=C4」だそうです。
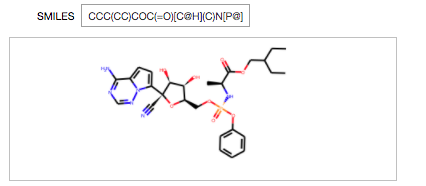
今回は探索時間2分でイテレーション78回でした。で、残念ながら解けなかったらしい(Not Solved, Route score: 0.715)。
最もスコアが良いものはこんな感じ、、、。
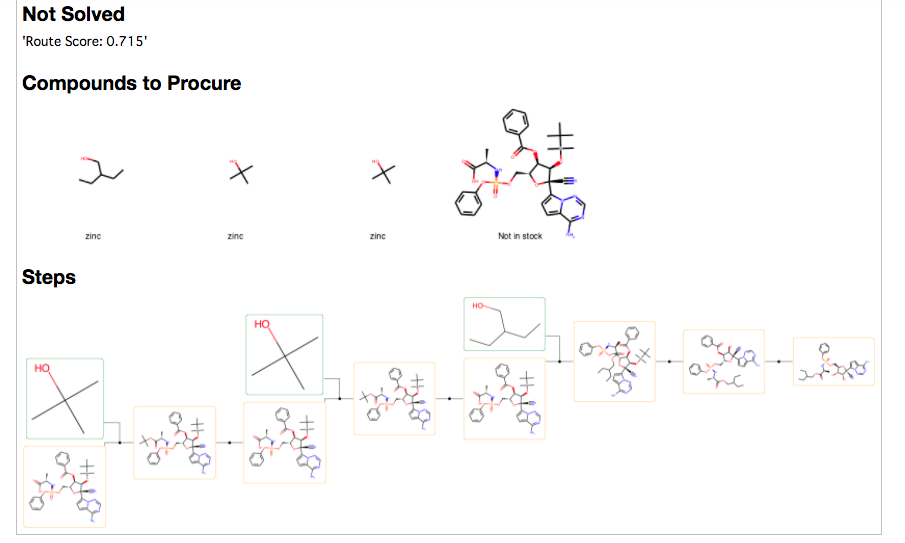
画像が小さいですが、Compounds to Procureの一番右の化合物はNot in stockとなっています。
Stock file内の構造まで分解できなかったみたいのでNot Solvedとなるようです。
ルートを見ると最初はtert-Butylエステルを作っては分解、作っては分解してます。
文献中に保護-脱保護の合成戦略はまだ組み入れられていないと記載されていましたが、確かに苦戦している様子が伝わってきます。
核酸アナログや糖って合成特殊な感じしますよね。ジョーンズ有機化学にも糖の合成しんどいみたいなこと書いてあった気がします。記憶の彼方ですが。。。
Ledipasvir
目標はもっと高く、大きな分子!ノーベル賞関連でC型肝炎治療薬、レジパスビルを逆合成!
SMILESも長い!! 「CC(C)C@@HN1CC2(CC2)C[C@H]1C3=NC=C(N3)C4=CC5=C(C=C4)C6=C(C5(F)F)C=C(C=C6)C7=CC8=C(C=C7)N=C(N8)[C@@H]9[C@H]1CCC@HN9C(=O)C@HC)NC(=O)OC)NC(=O)OC」
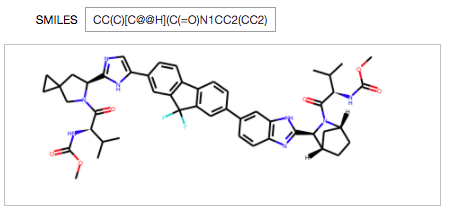
イテレーション3までしか進まずに探索が終了してしまいました。全くもってすごい分子ですよ。どうやって見つけたんだろう??
これもNot Solved(Route score: 0.534) で、分解の結果にZincのStock fileにないものが含まれていました。提案ルートはこんな感じ、、、

真ん中あたりで切って、左右ユニットに分かれたルート提案となっています。
構造の右半分ユニットのルートは図の上部、左半分ユニットは下部に分かれています。
どちらもイミダゾール環(右はベンゾイミダゾール)を構築しています。
なかなかヘテロ環、芳香環を切断-構築する逆合成って難しいのかな、と思っていたのですが、合成のプロからしたら普通なのでしょうか?
イミダゾールの特殊性みたいなものがあるのでしょうあ?ちょっとわからないので専門家の方コメントいただけると嬉しいです。
ここで気づいてしまいました。正解(実際の合成ルート)を知らず、妥当な合成を判断できない私が眺めていてもツールの性能は評価できない・・・・
答え合わせ?
というわけで合成ルートをググりました。巨人の肩の上に立つ!
実際の製造方法はわかりませんが一つのルートとして比較してみると面白いかもしれません。
Favipiravir
富士フィルム株式会社の特許から(JP2012180336)。以前Twitterで見かけて印象に残ったルートです。
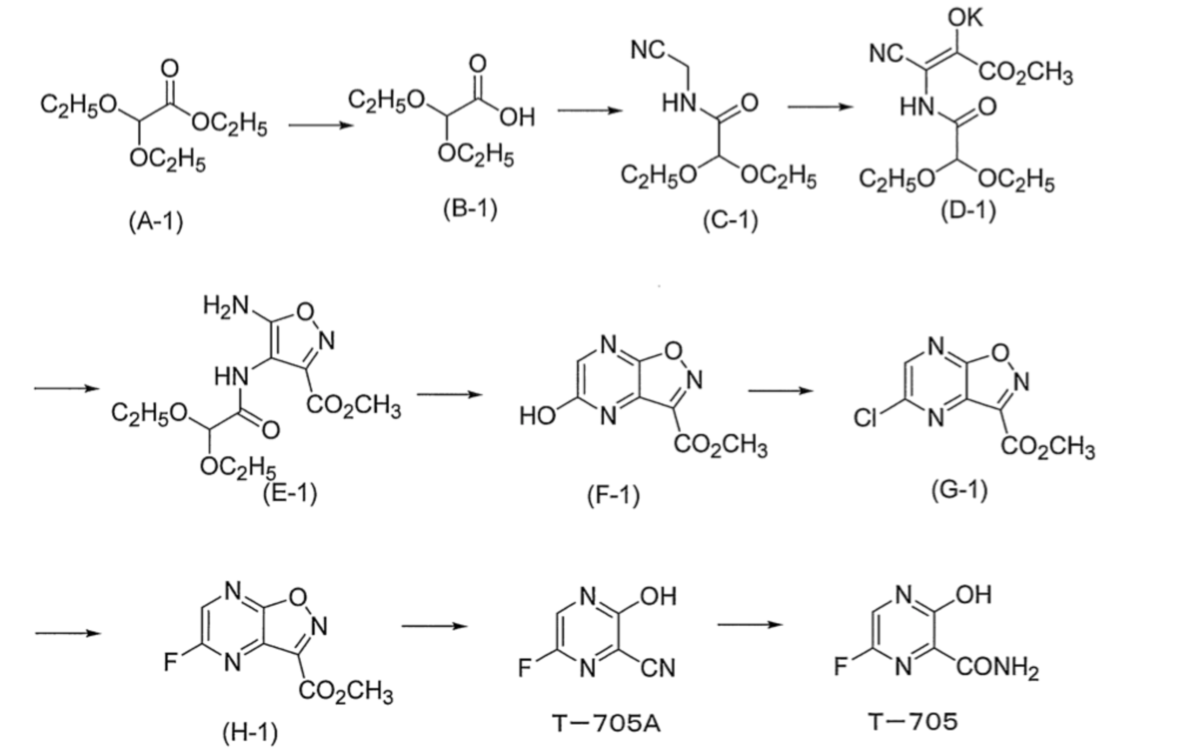
H1からT-705Aへは塩基性条件で加熱撹拌してます。加水分解、脱炭酸、ヘテロ環の開裂の3段階が起こってるんですかねー?お洒落!
・・・というか最終工程(T-705A → T-705)、AiZynthFinderが最初に予想したルートそのものです。えっ、凄い!!!
Remdesivir
New Drug Approvalsというサイトに合成ルート含めて色々と記載がありました。が、ちょっと長いのと、今回はAiZynthFinderの提案がうまくいってなかったので割愛させていただきます。
気になる方はサイト(New Drug Approcals: Remdesivir)をご覧ください。こちらによるとGilead Sciences社の特許に記載があるらしいです(WO2016069826)。
Ledipasvir
こちらも先のサイトに記載がありました。 リンクはこちら New Drug Approvals:Ledipasvir。
Gilead Sciences社の特許WO2013184702が引用されています。ルートの全体は上記サイトをみていただくとして、特許の一部を抜粋します。
右側ユニット・・・ベンゾイミダゾール巻いてます!っていうかそのまんま!
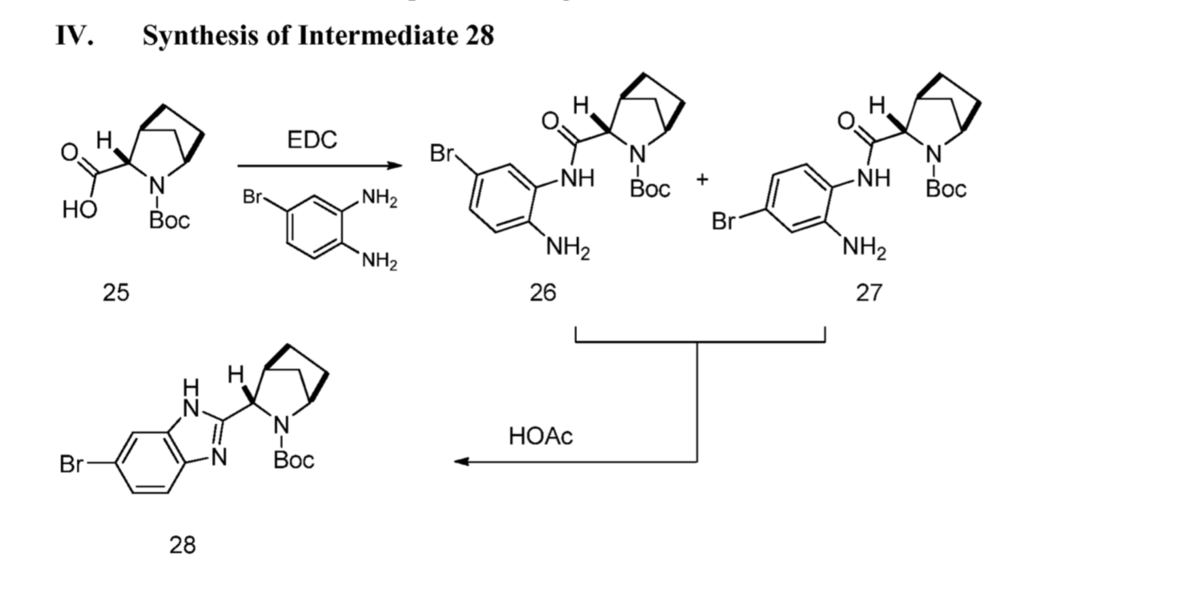
左側ユニット・・・こっちもイミダゾール巻いてます!保護基が特許はN-Boc基、AiZynthFInderはN-Cbz基という違いはありますがだいたい一緒!
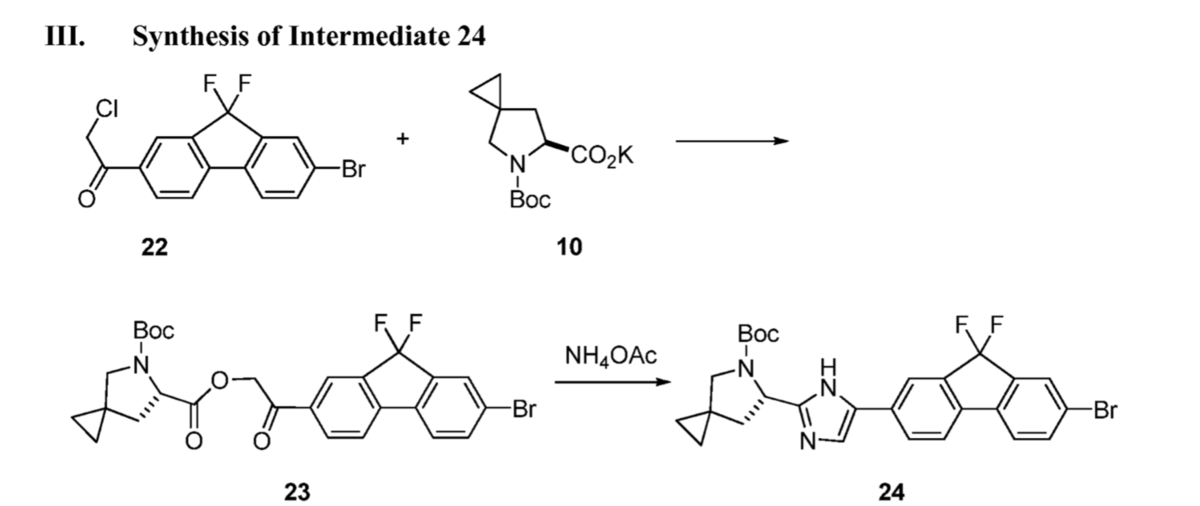
そんでもって右側と左側の連結は・・・ベンゾイミダゾールユニットのボリル化を経た鈴木-宮浦クロスカップリング!反応の選択一緒!
ここから特許では両端のBoc基を外し、2つともバリンメチルエステルとアミド化することでLedipasvirに導いています。
AiZynthFInderは、バリンユニットの導入のタイミングをのぞいて、ほぼ反応のコンセプトとしては同じルートを提案しているようです!
すごい!!!
まとめ
他にも機能はあるようですが、とりあえず今回はGUIで遊んでみました。これだけ親切設計なのに使えるまでに色々躓いてしまいました・・・。
実際に遊んで見るまでは、データから学習しているといっても限界があるのではないかな・・・と思ってましたが、AiZynthFInder予想以上に良い提案を出している、というのが私の感想です。
というか少なくとも私よりはるかに賢いです。。。ワタシ、チョットカナシイ。。。オベンキョウタイヘンダッタノニ。。。
しかし、これがオープンソースで公開されるというのは本当に凄いですね。私は遊ぶことしかできないので、みなさんぜひ使ってみてGitHubに改善を提案してください。合成の専門家からみたルートへのつっこみとか聞いてみたいです。
ではでは
オープンソースで逆合成解析 ~AiZynthFinder~ part 1. 文献メモ
有機合成の授業を受けた方なら習ったであろう逆合成、パズルみたいで楽しいって言ってる人もいましたが、私はこれがとても苦手でした・・・。全合成の研究発表見てはすげぇー、わけわかんねぇーて思ってました。
で、そんな私が知っていたら大喜びしたであろう素敵なツールがRDKit UGM 2020で紹介されていました!
その名もAiZynthFInder!!オープンソースの自動逆合成解析ツールです!!
これは遊ぶしかないですね!でもその前にどんなツールか背景をしらべましたよ。
AiZynthFinder ?
まずどんなツールなのか? 残念ながらUGMの発表をお聞きすることができなかったのですが、スライドをアップしてくださっていました。
スライドはこちら・・・ *1
ソフトウェアについての論文はChemRxivにあります。
文献1. Genheden S, Thakkar A, Chadimova V, et al (2020) AiZynthFinder: a fast, robust and flexible open-source software for retrosynthetic planning. ChemRxiv. Preprint.
使われているデータやアルゴリズムの背景についてはこっち。これもオープンアクセスです。ありがとうございます。
文献2. Thakkar A, Kogej T, Reymond J-L, et al (2019) Datasets and their influence on the development of computer assisted synthesis planning tools in the pharmaceutical domain. Chem Sci.
論文も読めるようにしてくださっているので、この記事では論文をとスライドを頼りにツールの理解を試みたいと思います。
背景
伝統的な逆合成解析は専門家の手作業 or (手作業で抽出された)ルールベースのエキスパートシステムでした。
最近、Deep Learningの発展に伴いデータドリブンな手法がでてきました。有名なのは2018年 Nature の論文("AlphaChem"?)。
化学同人の「化学」誌(2019年02月号)でも取り上げられていました。
で、逆合成解析のアルゴリズムは大きく2つに分かれるそうです。
- template-based approach
- template-free approach
template-base の"reaction template"は先のエキスパートシステムにおけるルールで、化学変換を表現するルールです。このルールを「既知の反応のデータベースから(自動的に)抽出しよう!」というのがDeep Learningにおけるアプローチ。
AlphaChem (で定着したんでしょうか?) もこちらの手法で、
- templateを優先する形でneural networkを訓練
- 合成経路の提案はMonte Carlo tree search アルゴリズムによる
- 2.の探索を導くポリシーとして、1.で学習したtemplateを使う
ってことみたいです。今回のAiZynthFinderもtemplate-basedで、アルゴリズムの大枠はAlphaChemと同じっぽい。間違ってたらごめんなさい。
また、もう一方のtemplate-freeのアプローチにおける典型的な例としては、化学反応を自然言語処理の問題として捉え、単語のセット(出発物質、reactant)を別の単語のセット(生成物、product)に変換する問題としてアプローチします。
アルゴリズム
概要
AiZynthFinderのアルゴリズムの概要は以下のようになっています。
- 目的: 入力の分子を購入可能な前駆体(precursor)にまで分解する
- 出力: 前駆体のリストで、それ以上はアルゴリズムで分解できないもの
- 探索: Monte Carlo tree search
- 終点: 設定した回数のイテレーションに達するか、時間制限に達した段階で探索ストップ
UCB?
ノードの選択で"upper confidence bound statistics"と書いてあったところを信頼区間上限としました。よくわからないですが、ググった感じ多腕バンディット問題っていうのと関係している感じでしょうか?
こんな雰囲気の話らしい…*2
- それぞれ得られる報酬の確率分布がわからない複数の選択肢がある状態で、限られた試行回数で報酬が最大となる選択肢を選びたい
- 探索と利用のバランスを取るアルゴリズムで報酬の最大化を目指す(Exploration-exploitation tradeoff)
- 探索 : 色々な選択肢を試す
- 利用 : 過去の結果から最適と思われる選択肢を試す
- 解法例
強化学習の文脈でよく取り上げられる問題なのでしょうか?
UCB(Upper Confidence Bound)法ではUCBスコアと呼ばれるスコアに基づいて次の選択肢を選びます。この中に、探索枝の選択をバンディット問題と捉えるUCT(Upper Confidence bounds on Trees)アルゴリズムというのもあるそうなので、多分これが関係してるのではないでしょうか??
よくわからないので詳しい方教えてください。
もう少し詳しく
話を戻して、 AiZynthFinderのアルゴリズムについてより詳しくはChemical Sicenceの文献を読んでね。と、いうことなのでそっちも見てみたいと思います。
大きく3つの点について議論されていました。
- 学習に用いたデータセット
- 反応テンプレートの抽出とサイズ、種類の影響
- スコア(解析がうまくいったかをどう評価するか?)
データセット
データセットとしては以下の4つが用いられています。
- USPTO dataset (patents, ~ Sep. 2016)
- Pistachio dataset (patents, ~Nov. 2017)
- Reaxys dataset (literature and patents)
- AstraZeneca ELN (industrial data)
抽出されたユニークな反応テンプレートがそれぞれどのデータセットに由来したものだったか、Fig. 1にベン図で示されています。
Reaxys datasetだけで見つかったテンプレートの割合がもっとも多く41.1%、ついで特許データ(USPTO and Pistachio)のみで見つかったものは32.5%とのことです。
抽出のルールにも依存した結果、とのことですが、Reaxysにも含まれていない特許情報があるかも、っていうのはちょっと面白かったです。あらかた全部カバーしているのかと思ってました。
AstraZenecaのin-houseデータにユニークなテンプレートも1.5%みつかったようですが、これは外部データベースに無い新規反応を社内で見つけているから、というよりも化合物の構造の多様性によるアーティファクトではないか、とのことです。
また、Fig.4 では各データセットごとに訓練したモデルについてそのパフォーマンスの比較が議論されています。
テンプレートの抽出
テンプレートの抽出は Coleyet al. (ACS Cent. Sci., 2017, 3 , 434-443) によるルール抽出の実装が応用されています。
元々は、反応中心に直近接した範囲のみを考慮するものでしたが、脱離基や保護基といった拡張した環境、反応性官能基(reactive speices)を考慮できないという問題点がありました。
その後、RDChiral (Coley et al. J. Chem. Inf. Model., 2019, 59 , 2529-2537 )で、有機合成でよく使われる約75種の官能基と保護基を含めるように修正されました。
これにより指定したグループについて、分子の拡張された環境をモデルが説明できるようになったそうです。
今後の課題として、ここではまだ保護、脱保護について合成戦略の観点を取り込むことができていないこと、指定していない新しい保護基や官能基について学習することができないこと、が挙げられています。
ネットワークとテンプレート
AiZynthFinderが参照しているAlphaChemでは3つのネットワークの訓練が行われていました。
- expansion policy : 与えられた化合物に適用されるテンプレートセットを予測
- rollout policy : より厳密で具体的なテンプレートセットの予測
- in-scope filter : 成功した(positive)反応にもとづいて訓練し、仮想的にうまくいかない(negative)反応のセットを列挙する
このうちAiZynthFinderでは 2. rollout policyに相当するネットワークだけに焦点をあてています。
入力に対してテンプレートを予測、そこから前駆体を導き、再帰的に当てはめていくことで逆合成の木(retrosynthetic tree)を生成していきます。
生成された逆合成ルートが妥当(valid)であるには、3つの条件をみたす必要があります。
- あたえられたcontextに対して予測される、データセットから抽出されたテンプレートがあること
- 予測されたテンプレートが「うまく(successfully)」適用できること
- 前駆体のセットが妥当なSMILESであること
逆合成解析の最終的な目標はウェットのラボでうまくいく合成ルートを提案することですが、この研究の主眼はまずin silico で何が予測できるかを見極めることにおかれています。それを踏まえた上で、先の条件の補足を見ていきます。
まず、「2. テンプレートの適用の成功(success)」について、これは「その反応が実際にうまくいくかどうか」ではありません。生成物(product)とテンプレートにマッチするサブグラフがあり、そのマッチするテンプレート使って前駆体/反応剤のセットが生成できること、を成功(success)としています。
3.のSMILESに関して、テンプレートベースのアプローチでは、生成物の全体の構造は保ったままで反応点(reactive site)だけが変更されます。したがって生成された前駆体のSMILESがvalidであれば、生成物と同じ構造の特徴を共有する前駆体のセットが得られたと推定できます。
ですが、ここにも実用上の問題があり、化学反応の選択性の問題などから、得られた前駆体が必ずしも実際の反応に適したものとは言えない、と指摘されています。
最後に、ここではin silicoでどこまでできるかを見る目的として、neural network policyの目標を、「適用可能なテンプレートの数を最大化すること」におかれています。また、ルートの終点は列挙された前駆体が購入可能か(commercially available)チェックすることで決定されています。
スコアと正確さ
先行研究では逆合成解析のパフォーマンス評価として正確さ(accuracy)が用いられていました。
正確さは「policy netoworkが反応テンプレートを正確に予測できるか」を反映します。
一方で、逆合成解析のタスクは「データセットにある一つの例(one to one)」ではなく、「適用可能な複数のテンプレートを予測(one product to many template)」することにあります。したがって、データセットに基づき1対1を評価する正確さでは、テンプレートの抽出源であるデータセットにオーバーフィッティングしてしまう可能性があります。
また、正確さはテンプレートの適用可能性をうまく説明できないことが指摘されています(Fig. 2)。例として、テンプレートの考慮する反応中心からの範囲(radius)を広げて特異度を上げていくにつれて、マッチするサブグラフを見つけるのに失敗して適用がうまくいかなくなり、モデルのパフォーマンスが下がる傾向にあるのに対し、正確さではその変化を捉えることができずミスリーディングな評価となってしまうそうです。
以上を踏まえて、著者らは正確さに加えて、policyの評価に適用可能なテンプレートの数を使うことを提案しています。もう少し全体論的な観点からモデルのパフォーマンスを評価しましょう、ということのようです。
比較検証したころ抽出したテンプレートのうち、種々の分子に適用可能なテンプレートの割合は非常に少なかったそうで、特異度(specificity)と一般化可能性(generalizablitity)のバランスを取るために、
- テンプレートは反応中心と、その一次の最近接したもの(the first degree nearest neighbors)を考慮したものにすること
- 種々の官能基と保護基をあわせて指定すること
といった提案がなされています。
テンプレートのサイズとパフォーマンス
評価方法についての議論を踏まえた上で、テンプレートライブラリーのサイズがパフォーマンスに与える影響を検証しています(Fig. 3)。
用いるテンプレートライブラリーが大きくなるほど正確さは低下する一方、「逆合成モデルがルートを生成できるか?」によるパフォーマンス評価は低下しませんでした。逆合成タスクは考えうる可能な出力を出すことに対し、正確さは"ground truth"(データセットにある実例)に基づくので、よりタスクに即した評価基準が重要になるかも、ということのようです。
テンプレートライブラリーのサイズの影響について以下が指摘されています。
- ライブラリサイズが大きくなるとテンプレートの化学的多様性が増加し、より合成ルートを見つけることができる
- 一方で、数が増えるにつれてノイズも増えパフォーマンスが低下する
反応テンプレートの数と、それらが表す反応の範囲(reaction space)にはバランスがあって、ライブラリを増やすについれてカバーできる範囲もふえるものの、逆に、適した反応テンプレートをライブラリの中から探し出すことが難しくなってしまい、増やしすぎるとパフォーマンスが落ちる可能性もあるよ。
ってな感じでしょうか?
以上が、アルゴリズムの背景でした。
オープンソース?
なんとなくソフトの裏で動いていることがわかってきたところで、もう一度ChemRxivの文献に戻ります。
イントロでは他のソフトウェアにも触れられています。比較のためにメモ・・・
逆合成解析のオープンソースソフトが限られている理由として、2点指摘されています。
- 反応データベースと、手作業によるルールの多くが商用であること
- 購入可能な前駆体のデータベース(ルート予測の終点判断によく使われる)も多くが商用
で、今回著者らがAiZynthFinderをオープンソースとして公開することにした理由としてIntroductionでこう書かれています。
...However, we believe that the scientific community would benefit from an open source implementation that provides algorithmic transparency and promotes reproducible research with sustainable software...(中略)...We envisage that by providing this tool free and open-source, other researchers can use it for benchmarking, contribute to a continuous development and, suggest synthetic routes for novel compounds.
素敵! ありがとうございます!
で、使えるように3つ提供するよ!っと
- 訓練済みのneural network policy
- 購入可能な前駆体のデータベースを作るためのツール
- 新規ユーザーの学習曲線を下げるための詳しいドキュメント
ツールは以下のGitHubで公開されています。
ドキュメントはこっち。
一旦まとめ
ぶっちゃけ理論の詳しいところはさっぱりわかりませんでしたが、これは楽しそうですね!
早速遊んでみたいですが、ちょっとここまでで力尽きたので一旦区切ります。
最後に、遊ぶのに覚えておかないといけなさそうな用語・・・
- policy : 反応データセットで訓練したNeural networkのモデル
- template : 反応データセットから抽出した反応テンプレート(ライブラリ)
- stock : 購入可能な前駆体のデータベース(ルート終点に関係)
RDKit UGM時間が合わずにほとんど見られなくて残念でした。でもDiscordの議論をみたらさっぱついていけかったので、これは聞けてもわかんなかっただろうな、ってなりました。残念なのは私の方でしたね!
色々と間違っていることが多そうなのでご指摘いただけると幸いです。
*1:最後のページにConfidentiality Noticeとか書いてあるんですけど良いのでしょうか?
*2:参考
1. 探索と利用のトレードオフとベイズ環境モデル 牧 野 貴 樹 計測と制御 第52巻 第2号 p154-161 https://www.jstage.jst.go.jp/article/sicejl/52/2/52_154/_pdf
2. 私のブックマーク 小宮山純平 Vol.31.No.5(2016/9)多腕バンディット問題 https://www.ai-gakkai.or.jp/my-bookmark_vol31-no5/
「分子の表現についてのレビューと実践ガイド」のメモ ~part 4 マクロ分子の表現~
レビューをDeepLに投げ込みながら読んでいるのでそのメモです。
ここまで3回で見て来たものは低分子に関するものでした。最後はマクロ分子の表現です。ケモインフォマティクスとバイオインフォマティクス の間の領域って感じのお話。
なお、順番は一部前後していますが、目次は論文そのままにしてます。また、 Creative Commons のもとで図表・内容を利用させていただいています。*1
- Representations for macromolecules
- Graphical representations for molecules and macromolecules
- AI applications within drug discovery using molecular representations
- Discussion
- Concolusions
- まとめにかえて
Representations for macromolecules
取り上げられているのはバイオポリマーやオリゴマー、合成ポリマーといったポリマー構造を持つものです。
ポリマーには以下の2種類があり、表現が難しくなっています。
- monodisperse:構成するモノマーが同じ鎖長
- polydisperse:確率的な (stochastic)性質があり鎖長が定義されない
またマクロ分子の表現はバイオインフォマティクスの要素が強くなります。
ケモインフォマティクスでは原子レベルで低分子を表現していたのに対し、ヌクレオチドやアミノ酸配列といった配列情報で表現するといった傾向があります。
Amino acid-based stuructures
ペプチド、タンパク質のビルディングブロックであるアミノ酸(AA)は1文字あるいは3文字の略記で表されます。
1文字表記の限界として、遺伝暗号に含まれる20種を表すのには十分ですが、 それ以外にも自然に存在するアミノ酸を表すには数が足りないということが挙げられています。
Peptides
ここでは大きく分けて2つの表記(CHUCKLES、HLEM)が紹介されています。
CHUCKLESはポリマーの配列からそのSMILESを導けるようにしたものです(逆方向の翻訳も可能)。 説明を読むとこんな感じ、、、

CHUCKLESのオリゴマーにおける応用例としてBIOPEP-UWMというデータベース、また拡張版としてCHORTLESが挙げられています。
さて、より広範なマクロ分子を表現できる表記として以下の2つが挙げられています。
- HELM (Hierarchical Editing Language for macromoleluces):SMILESベース
- SCSR (Self-Contained Sequence Representation):v3000 Molfile フォーマットベース
以下ではPfizer社により開発されたというHELMの詳細についてもう少し見てみます。イメージを掴むために先に図を引用・・・
構成要素として複数のタイプの構造を持つ組み合わせを表現するシステムを作ることを目的として開発されたそうです。

HELM2ではさらに発展してポリマー混合物や自由形式のアノテーションも可能となっており表現できる幅が増えているそうです。
HELMは多くの製薬会社で実装されており、様々なデータベースやパッケージでもサポートされているそうです。 ChEMBLやRDKitにもあるんですって!知らなかった。。。
で、HELMについて字数を割いて詳しく説明されているの何でかな?と思ったのですが、生物と化学の融合地点としてBiocheminformatics表現が広がることで、 ペプチド医薬品といった低分子と生物学的製剤の間にあるような分野の表現が発展することを重視しているようです。
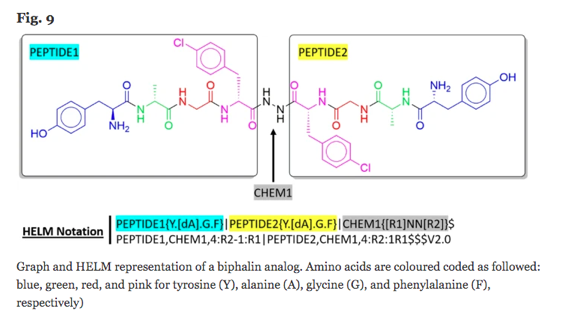
つまり、アトムベースのSMILESを配列ベースの表現に加えることで、天然のL-アミノ酸と非天然のD-アミノ酸の混ざったものといった表現の拡張ができる。非天然アミノ酸の組み入れはペプチドのバイオアベイラビリティの改善で重要なので、記述の幅が広がることでペプチド医薬の発展も記述できる、取り扱えるようになるというわけです。
なるほど!・・・それを踏まえた上で言わせてほしい! Fig. 9の図、フェニルアラニンに修飾(Cl)入ってるからFで表現するのは違うと思うで!!
Proteins
タンパク質(50以上のアミノ酸残基)については、PDBデータベースとそこで使われている表現が挙げられています。
PDBにおける原子は以下の情報で表されています。
1. 連続する番号
2. 特定の原子名
3. 対応する残基の名前と番号
4. 鎖(chain)を特定する1文字のコード
5. 空間座標 (x, y, z)
6. 占有率 (occupancy)
7. 温度因子 (temperature factor)
2008年にはProtein Line Notation(PLN)というものがBiochemfusionにより開発されPubChemにも実装されているそうです。
PLNでは残基の構造を擬原子(pseudo-atom)で表すことで、化学表現と配列フォーマット間の変換をロスなくできるようになっているそうです。
Key macromolecules
グリカンや合成ポリマーといったマクロ分子についても見ていきます。
Glycans
グリカン(olygosaccharide、polysaccharide)にもデータベースがありますが、monosaccharideベースの表記となっています。
ドッキングといった相互作用解析ではアトムベースの表記が必要となるので、 グリカンを医薬品探索におけるリガンドとして扱うにはmonosaccharideベースの表記では不十分です。 そこで、アトムベースの表記へと変換するツールの開発が行われています。
また、Web3 Unique Representation of Carbohydrate Structures (WURCS)という表現が開発されています。

WURCSは多くのデータベースに実装されていますが、ケモインフォマティクス のソフトウェアではほとんどサポートされていないそうです。
他のアプローチとして、ファーマコフォアに基づくものや言語モデルに基づくものといった研究もなされているようです。
Polymeric drugs
ポリマーの表現として最近、BigSMILES構文というものが開発されました。
以下のようなポリマーをエンコードすることができます。

まだカノニカルなものはなく、応用例もないようですがさらなる研究が行われているそうです。
Graphical representations for molecules and macromolecules
先のセクションで見たマクロ分子の表現は、データの格納とケモインフォマティクス的解析を意図したものでした。 ここでは、マクロ分子とその物理化学的特性の可視化表現をみていきます。
2Dと3Dをそれぞれ扱いますが、何はともあれ見た方が早いので図を引用しておきます。
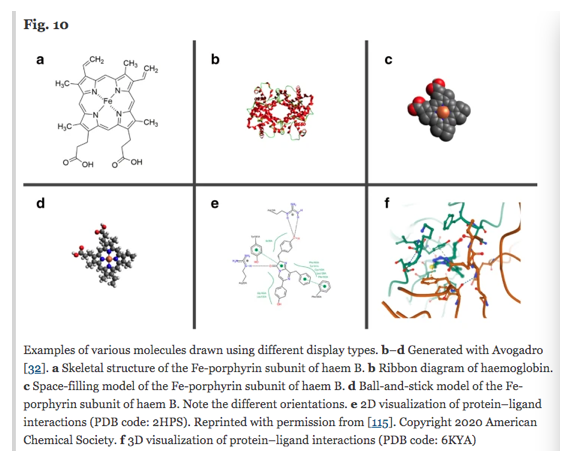
2D depictions
分子の2D描写として、骨格構造のラスタ図、ベクトル図がよく使われます (Fig. 10a)。
以下のような点が問題になることがあり、2008年にはIUPACがオススメの標準描画方法を出したりしています。

各ソフトウェアそれぞれより良い描画方法を目指してアルゴリズムの改善等試みられているそうです。
構造そのものの描画以外にも、反応や相互作用の研究といった点でも2Dの描画が使われます (Fig. 10e)。 ここでは分子の置かれた環境と振る舞いの表現が重視されています。
Fig. 10には描かれていませんが、以下の3つも特徴のある例として取り上げられています。

3D depictions
3D描画のソフトウェアの例としてAvogadro、PyMOL、VMDといったものがあります。
表現方法の例として以下が挙げられています。
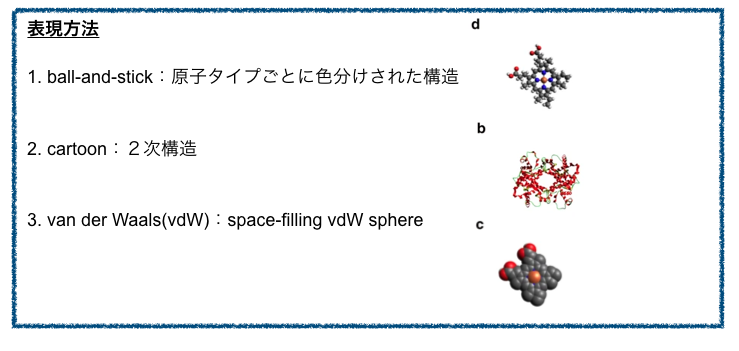
vdW球による可視化の応用例として、分子表面の描画による相互作用解析が挙げられています。
3D描画はドッキングや機構の研究で、2D描画は構造活性相関研究でよく使われているそうです。
以上がマクロ分子の表現でした。このあたりがAIにどのようにつながっていくのか、最後に応用例を見ていきたいと思います。
AI applications within drug discovery using molecular representations
Representations for macromolecules
マクロ分子の表現において人気のある応用はタンパク質構造の予測です。
また、グリカンの分野における研究例として、
- 擬受容体(pseudo-receptor)モデルを使ったバーチャルスクリーニング
- 機械学習によるグリカン-タンパク質相互作用の解析
- グリコシル化部位予測
が取り上げられています。
Graphical representations for molecules and macromolecules
可視化については、さらなる高速化の研究が行われており、最近ではバーチャルリアリティや3Dプリンティングといった技術と紐づいています。
また、増え続けるデータを処理するための構造マイニング技術として、Optical Character Recognition (OCR)が挙げられています。 機械学習や確率的パターン認識技術により、2D描画の標準的な化学表現への変換が行われていますが依然として課題はあります。
変換が難しくなるものとして、以下のような項目の処理があげられています。
- 画像の解像度
- 化学的略記のコンピュータによる解釈
- テキストに埋め込められた画像
- 複数の構造を含む図に埋め込まれた画像
- 反応パスウェイ中に埋め込まれた画像
- 骨格の式あるいはMarukush構造で表された画像
マクロ分子の表現については以上です。
さて、4回にわたって分子の表現方法について眺めて来ましたが、いよいよまとめの段階に入ってきました。残りはDiscussionとConclusionです。
Discussion
ここまで様々な表現方法をみてきましたが、著者らは医薬品探索における問題を解決するためには、複数の表現を同時に活用することが必要と指摘しています。
タンパク質の構造予測を例として以下のような流れをあげています。
- タンパク質の配列から始まり
- 粗い3Dモデルの作成
- 分子動力学法によるフォールディングメカニズムの理解
- 最終的な配置と構造予測
- ドッキング計算への応用
また、表現の選択に影響する要因として、2点あげられています。
- 表現を生成する手法の複雑さ
- オープンに利用可能なものか否か
・・・確かに。オープンソースツールで簡単に使えるものがあると遊んでみよう、ってなりますもんね!!
また、分子表現の変遷の歴史をたどってきたことを踏まえて、使われ続ける表現とそうでないものとの違い、要因を議論しています。
まず、変化の要因の一つとしてコンピューターテクノロジーの進化が挙げられています。
- ストレージの増強
- プロセッサーの質
- パラレルプログラミング
かつての線形表記、IUPAC-Dyson、WLNは当時の状況としては妥当だったものの、主に人間が操作するもので、計算機による取り扱いが難しかったので、計算機がより使われるようになると廃れていきました。
現在ではよりコンピューターが単純に扱える表現が使われる傾向にあります(molecular string representation)。また、より計算が必要となる詳細な表現も現在では使えるようになりました(e.g. hashed fingerprints)。
別の要因としては、ケモインフォマティクスコミュニティにおける受容があげられています。より人間が読める(human readable)表現の方が好まれ、結果として長く使われるようになったかも、、、らしいです。
最後に、分野の違いの及ぼす影響があげられています。
異なる分野(chemoinfomatics, bioinfomatics, AI)ではグループ内の歴史的、継続性の理由から表記の選択、使われ続けるか否かが変わってくるのではないか、ということです。
継続は力なりですね!(違うか)
Concolusions
分子は複雑な構造なので、その表現においては様々な特性とともに、低分子とマクロ分子のそれぞれの異なる性質といった点も表現しなければなりません。ケモインフォマティクス 、バイオインフォマティクスの発展とともに、医薬品探索のプロセスが加速し、分子の振る舞いについての理解も進んで来ました。このレビューで取り上げられているような様々な表記方法を学び、AI駆動の医薬品探索の裏側を知ることでもっと有効活用できるようになると良いですね!
みたいな感じのことが書いてありました!
まとめにかえて
以上、懲りもせずに適当なメモを垂れ流してみました。ひたすらCreative Commonsにのっかっていくスタイル!
線形表現の歴史や、派生系、マクロ分子の表現等々、知らないことがたくさんあって個人的にはとても勉強になる文献でした。 途中謎の自作の表や無意味な箇条書きが出て来たのは、私が文章では単語の関係性が理解できない読解力低いマンだからです。 human-readableですら私には遠いですわー。
さあ、これでAI創薬に飛び込む準備ができた!・・・のか?
知らんけど。。。
色々と間違いがありそうなのでご指摘いただけると幸いです。
「分子の表現についてのレビューと実践ガイド」のメモ ~part 3 化学反応の表現~
レビューをDeepLに投げ込みながら読んでいるのでそのメモです。
先の2回の記事では分子のグラフ表現と線形表現でした。単独の分子の表現方法を踏まえ、次のステップとして分子の変化、化学反応の表現方法へと話題がうつっています。知らない事が多かったので雑訳の羅列になってしまっていますがまあいつもの事ですね。
なお、順番は一部前後していますが、目次は論文そのままにしてます。また、 Creative Commons のもとで図表・内容を利用させていただいています。*1
- Representations for chemical reactions
- AI applications within drug discovery using molecular representations
- まとめ
Representations for chemical reactions
Harnessing reaction data for drug discovery
さて、化学反応は次のように理解できます。
- 一群の分子のセットを
- 関係するもう一つの分子のセットへと
- 特定の条件のセットの下で
- 相互変換すること
あらためて言葉で記述されると、なるほどそうなるのかーという感じです。
で、以下のような図式でよく書かれるわけですが、これが機械可読性が低い(machine-readableでない)のでどうにかしたい!、というわけです。

機械で処理できよるようにするには、前回、前々回見てきた分子の表現を応用する事ができます。(但し、各表現のもつ限界もそのまま引き継いでしまいます!予め注意喚起!)
また、フォーマットについてはRXNとRDファイルがよく使われます。グラフ表現のCTfileのところで出てきたやつですね。
以下、様々な化学反応の表現表現を見ていきます。下図のようなものが取り上げられています (Fig. 6より引用)。
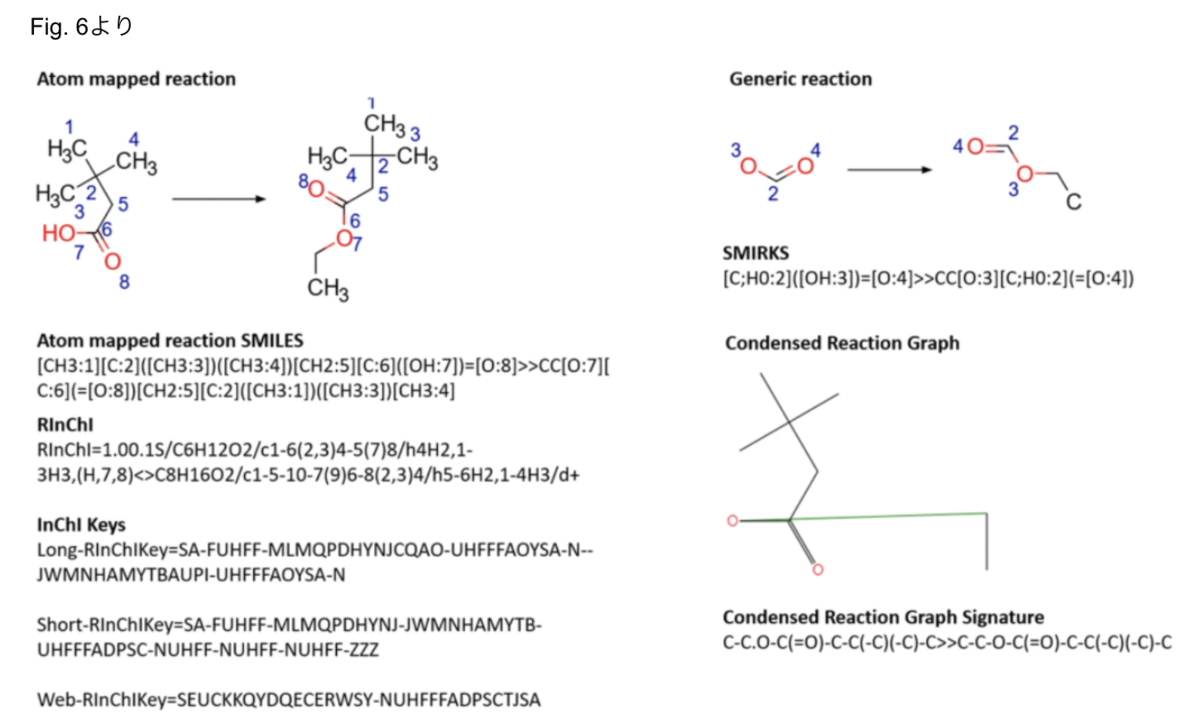
Reaction SMILES and SMIRKS
Reaction SMILESはSMILESで化学反応が扱えるように拡張されたものです。
記載の仕方は以下のような感じです。
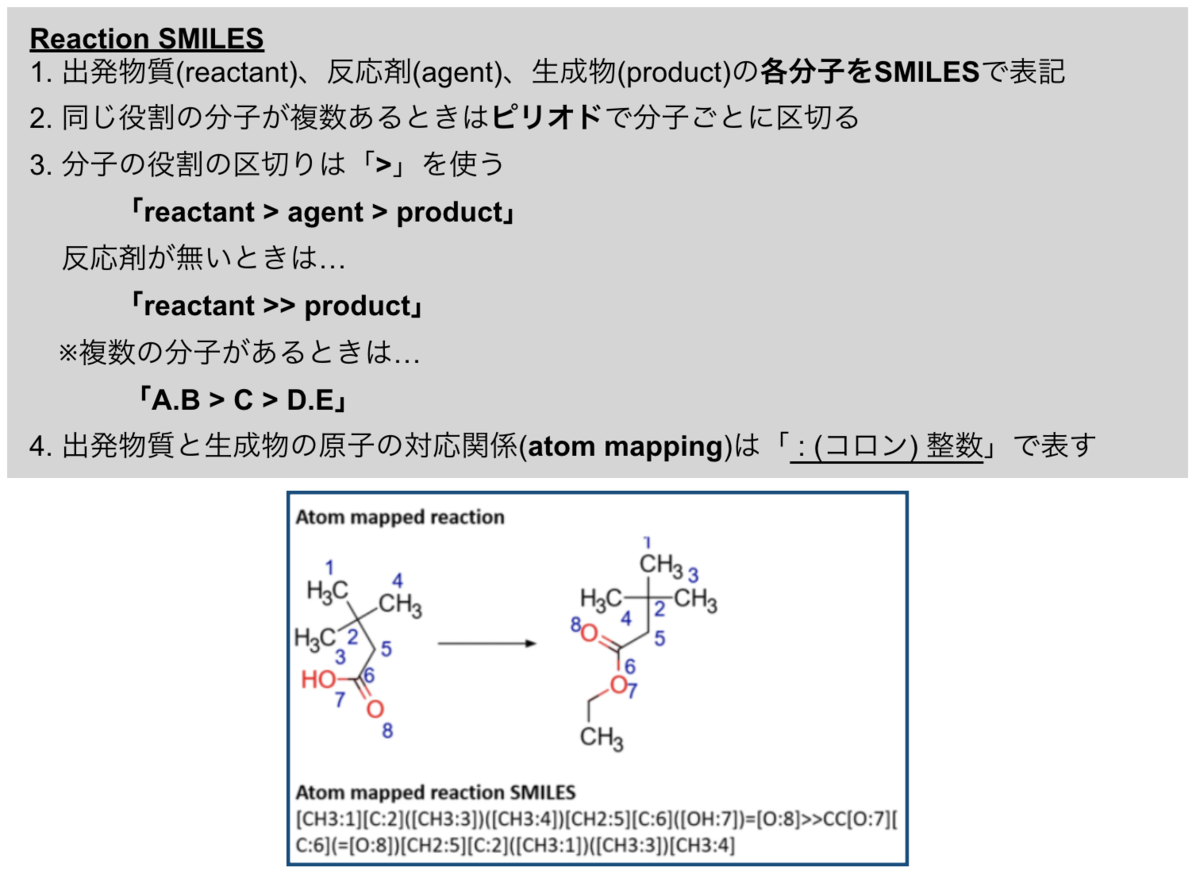
尚、Atom mappingは反応剤には使えません。
また、Reaction SMILESそのものにはテキストによる情報(反応中心や反応条件 etc.)を追記することはできませんが、ファイルレベルで、RXNやRDファイルフォーマットが取り扱えるメタデータへの記載という形で情報を追加することができます。
次にとりあげるSMIRKSは「SMILESとSMARTSのハイブリッド」とも言えるものです。
SMIRKSは反応における分子の変化のパターンを一般化して記述します。
(※ SMARTSでは分子のパターンと部分構造が一般化されて記述されていました。)
記述の特徴はこんな感じ…

また、図のようにSMIRKSはグラフ(reaction graph)に変換することもできます。
RInChI
RInChIはInChIを反応の表現に拡張したものです。
反応の「単一で、かつ順序の影響を受けない識別子 (unique & order invariant)」として導入されました。
Reaction SMILESは分子の順序が任意でしたが、RInChIでは同一の反応の表現が同じになることを意図されているので、反応の重複を見分ける事ができるという利点があります。
一方、文法がより複雑という課題があります。
カノニカルな表現を意図して導入されたInChIがSMILESと比べて人間にわかりづらいものだったこととそのまま対応する感じですかね?
RIhChIの記述はこんな感じ…
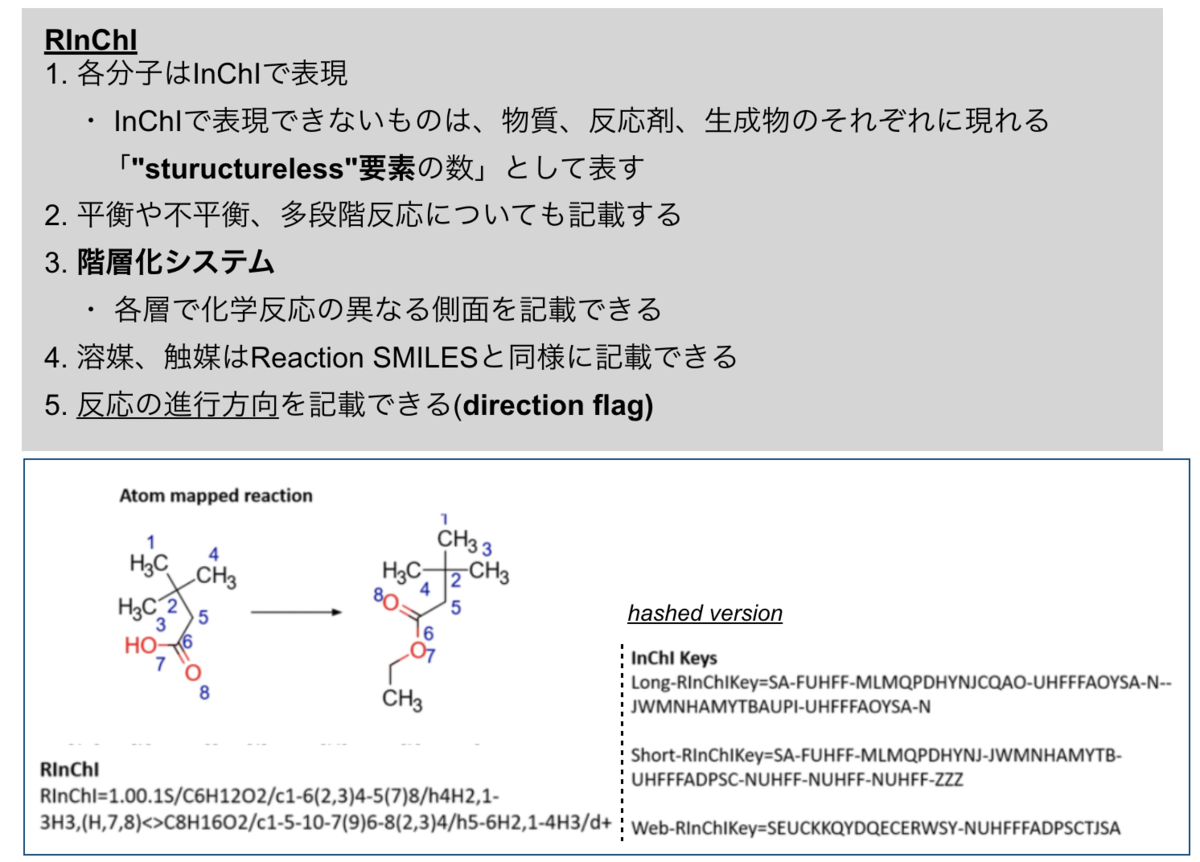
「5. 反応の進行方向」については、例えば同一の反応がわずかな条件の差で異なる結果になった時に、それぞれの条件での進行方向が記載できることで、細かな条件間の比較を行うことが容易になる、という利点があります。
更に、RInChIの拡張としてProcAuxInfoというものが提案されており、メタデータ(収率、温度、濃度 etc.)を格納できるようになっています。
また、ハッシュ化されたRInChI keyを使うと反応データをインデックス化して効率的に検索する事ができます。
難点としては、文法の複雑さ以外には、SMARTSやSMIRKSに相当するものがなく、 部分構造検索や一般化した化学反応の変化の記述が難しいということがあげられています。
Condensed graph of reaction (CGR)
CGRはVernekらにより開発された表現です。こんな感じ…
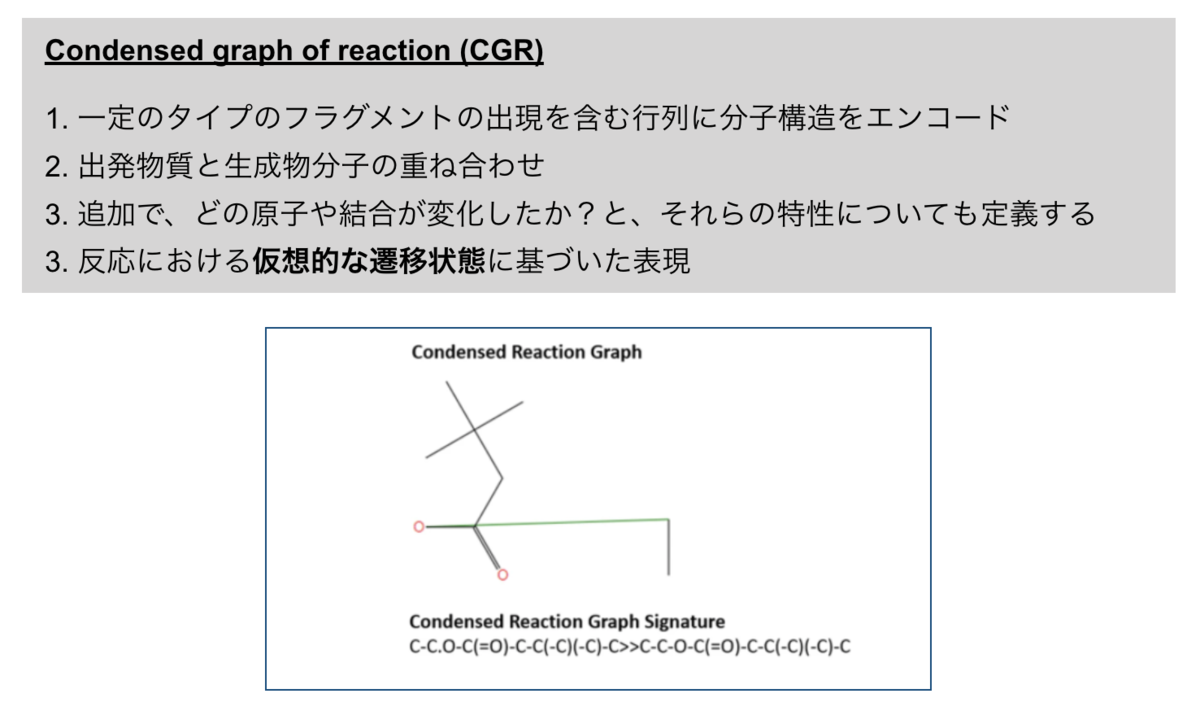
図では少し見づらいですが、エチル基はカルボキシ基の酸素原子と緑色の線で繋がれています。「エステル化の仮想的な遷移状態」っていうことでしょうか??
Bond electron matrices (BE-matrix)
Bond-electron matrixは反応を行列で表現しようとするものです。

原料のBE-matrixに反応のR-matrixを足す事で生成物のBE-matrixが得られます。
行列表現で表せる情報を増やそうとしている点が、他の表現とは異なるアプローチのようです。
Hierarchical organization of reaction of reactions through attribute and education (HORACE)
HORACEは機械学習のアルゴリズムを利用して化学反応の分類を行うもので、化学反応の階層的な表現ができます。

階層化された表現となっていることで、SMILESのような構造だけを表す反応表現よりも豊かな表現が可能となっています。
InfoChem CLASSIFY
InfoChem CLASSIFYはルールベースの合成計画法で使われるアプローチに発想を得た反応の表現です。

各原子について生成されたハッシュコードは全ての出発物質と一つの生成物について合計され、反応中心のユニークな表現として使われます。
また、ハッシュコードは反応の分類や、様々な合成計画ツールで反応中心を抽出するアプローチとしても使われています。
文献中では反応中心を見つけ出すアプローチとしてよく用いられるものとして、「出発物質と生成物の間の最大共通部分構造(MCS)をみつける事」があげられています。
また、MCSの問題点として、NP完全で非決定的な多項式時間がかかるという事指摘されてます。
Inform CLASSIFYはMCSに代わるアプローチになるかも、という事が言いたいのでしょうか?ちょっとよくわからなかったです。
ちなみに取り出した反応中心は必要とする特異度(specificity)のレベルに応じて、隣接する原子にも範囲を拡張できるそうです。
範囲と特異度の関係は以下のようになっています。
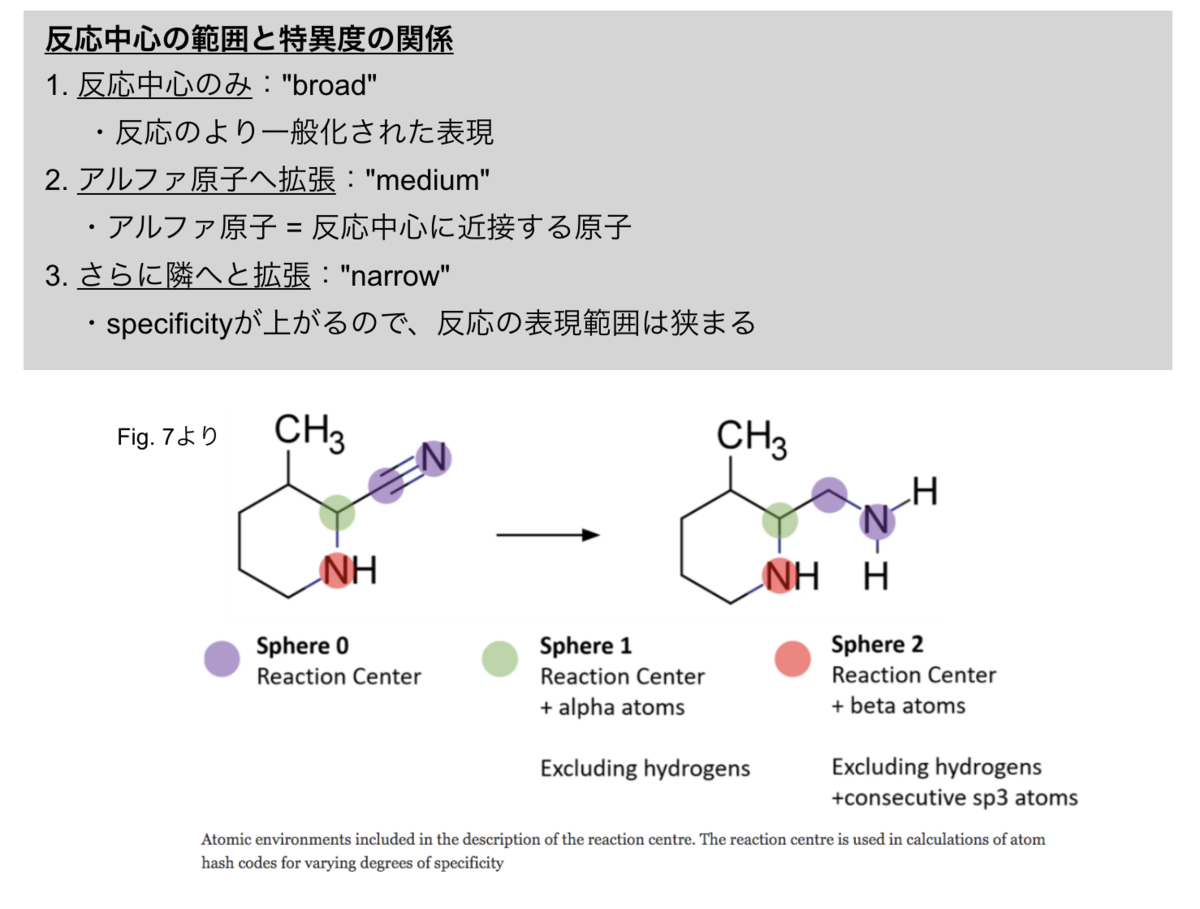
Reaction fingerprints
Reaction fingerprintは反応のベクトル表現です。反応中心で起こる構造の変化を表します。

こうして作成したベクトル表現は例えば、機械学習でモデルの構築に用いることができます。
reaction fingerprintは反応中心を特定する別のアプローチとして期待されていますが、「反応グラフ(reaction graph)への変換が難しい」という問題点があります。
また、立体化学の取り扱いはまだできません(研究は盛んに行われているそうです)。
以上が化学反応の表現でした。つづいてマクロ分子の表現になりますが、ここで一旦区切ります。
AI applications within drug discovery using molecular representations
今回も途中をすっ飛ばして化学反応表現の応用について触れた箇所に飛んでおきます。
Representations for chemical reactions
化学反応表現の応用の主なものは逆合成と反応予測です。
これらはAI駆動創薬において「ループを閉じる」ので重要となります。(Design-Make-Test-Analysisのループですかね?)
提案された構造が合成可能なものかを判定し、適切な逆合成により合成ルートを提案することが大事なんですって!
この辺りの話題についてより知りたい場合は以下の文献がオススメされていました。
- Mater AC, Coote ML (2019) Deep learning in chemistry. J Chem Inf Model 59:2545–2559
- Jensen KF, Coley CW, Eyke NS (2019) Autonomous discovery in the chemical sciences part I: Progress. Angew Chemie Int Ed
ACIEの文献はarxivにもあるっぽいです。
70ページある・・・長い・・・
まとめ
以上、化学反応の表現でした。私はreaction SMILESくらいしか聞いたことがなかったので、行列表現やベクトル表現に相当するものがあるというのはなるほど!という感じでした。
分子の表現と反応の表現の対応関係はこんな感じですかね?
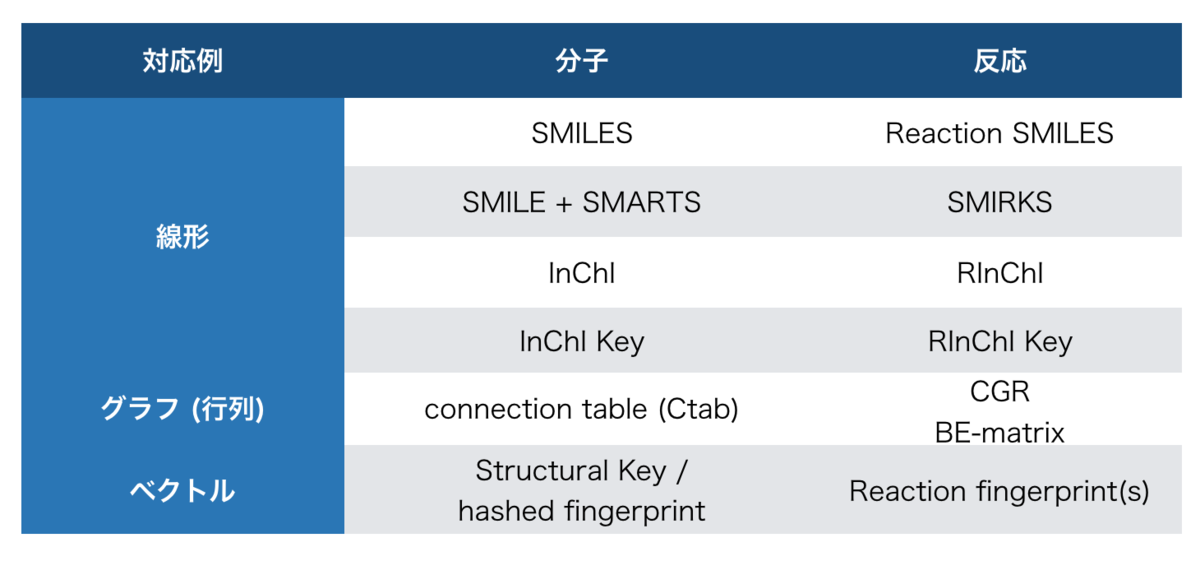
・・・HORACEとInfoChem CLASSIFYは対応するものがよくわからなかった。
また、「反応中心をどう見つけるか?」という点が議論に上がっていたのが興味深かったです。 確かに、複数の分子が記載されてる場合に、「どの分子を(目的物質に直接変化する)出発原料とするか?」は重要だし難しい問題だな、と思いました。
便利な試薬がたくさん開発されている分、複雑・高価な試薬が増えて、時と場合によっては原料よりも試薬の方が分子量が大きくて複雑な構造をしている、ということもあると思います。 そうすると、各々の分子だけからどれが原料かを判断するのは難しく、反応前後の差分、複数分子間の比較を取らないと分からない、というのも納得できます。
また、原料をどれに設定するかは、反応の収率の計算に直接影響するので、原料の設定次第で同じ反応に紐づいた数値データが変わってしまうことになります。 合成スキームとして書いてあったらすぐに気づくことができそうですが、機械可読な表現・数値に変換してしまうとこのあたりの微妙な差が埋もれて、 数字がひとり歩きしてしまいそうだな、と思ってしまいました。
(収率をb.r.s.m.で記載してたら良い反応に見えるけど、実際には・・・???みたいな問題とかもありますしねー・・・関係ないか。)
途中無意味な箇条書きを乱発しています。わかった気になるから!!わかんないけど・・・。
次回、最後!マクロ分子に続く!
色々と間違いがあると思います。ご指摘・ご教示いただければ幸いです。


