「分子の表現についてのレビューと実践ガイド」のメモ ~part 2 線形表現~
レビューをDeepLに投げ込みながら読んでいるのでそのメモです。
先の記事では分子のグラフ表現の箇所を取り上げました。続いて線形表記についてです。
なお、順番は一部前後していますが、目次は論文そのままにしてます。また、 Creative Commons のもとで図表・内容を利用させていただいています。*1
- Linear notations for small molecules
- AI applications within drug discovery using molecular representations
Linear notations for small molecules
線形表記(linear notation)は Ctab (connection table) をエンコードした文字列で、体系的なルールで解釈することができる分子の表現です。
線形表記には、行列と比べて以下のような利点があります。
- サイズが小さい(ディスク容量的にも、サイズ的にも)
- 操作が簡単 (Excelでコピペとか余裕)
Table 1 にアラニンを例に線形表記の例がのっています。

The IUPAC quest for a universal notation
線形表記はルールで解釈できると書きましたが、そのルールをどう設定するか?については紆余曲折があったようです。(現在も進行中?)
IUPAC(International Union of Pure and Applied Chemistry)の戦いの歴史が書いてありました。ざっとこんな感じ・・・

現在ではDyson、WLN両者ともに下火になっているそうです。次節、より現代使われている表記法へと話が続きます・・・
が、その前に、IUPACが望ましいとした特性や表記法の分類がなるほど感があったので載せときます。

また、表記法の形式による分類の考え方はこんな感じ…

この辺りを意識しながら以降の表記法の紹介を読んでいくと面白いかもしれません。
The advent of contemporary notations
Simplified Molecular Input Line Entry System
WLNよりも直感的な表記としてSMILES(Simplified Molecular Input Line Entry System)が1988年Weingingerらにより開発されました。
Daylight社のソフトウェアに組み込まれたことをきっかけに、現在でも同社が維持運営しているそうです。
SMILESには表記を生成するルールの違いや表される構造情報により複数の流派(?)があります。
生成のルールは以下のような感じです。次に引用したFig. 4と比較するとわかりやすいかもしれません。

色々とあるSMILESのうち、まずは生成ルールが異なる2種類(canonical smiles、randomized smiles)です。こんな感じ・・・

細かい変換ルール(Cとcの違いなど)は割愛します。
randomized SMILESは表記が毎回変わってしまい困ってしまいそうですが、逆にデータ拡張(data augmentation)に使えるという利点があるそうです。
他にも派生したSMILESが色々とあります。

一口にSMILESっていってもいろいろあるんですね。
続いてもう少し別の特徴がある線形表記SMARTSとInChIへと続きます。
SMILES Arbitrary Target Specification (SMARTS)
SMARTSは部分構造検索のために開発されたSMILESの拡張です。
- SMILESよりも利用できるシンボルが多く、より一般的な形で分子グラフを表現できる (→ コンピュータ科学における正規表現に類似)
- 原子が一つ異なる、あるいは、結合の位置が異なる分子の集合を表現できる
- 論理演算子 ("OR"、"NOT")も使える
- 同位体(isotope)や結合のタイプ(aromatic, aliphatic)を明示できる
- Recursive SMARTSでは原子の環境についての情報も含められる(ex. ortho, meta, para)
といった特徴があります。
SMILESは全てSMARTSとしても有効ですが、SMARTSがSMILESとして有効とは限りません。
International Chemistry Identifier
InChIは2006年 NISTにより標準的に自由に使える形式表現として導入されたものです(オープンソースのカノニカルな表記)。
様々な情報を含む多層構造で形成されており、下の図のような感じです。

InChIはSMILESと異なり元の分子グラフに戻すことができることは保証されていません。また、SMILESの方が人間には読みやすいという利点があります。
また、InChIのハッシュ化されたバージョンとしてInChIKeyがあります。
元のInChIの一義的な表現となるようになっていますが、InChIKeyが複数のInChIにマッピングされることがあります(InChIKey collision)。
Using chemical descriptors to represent molecules
ここまで見てきたのは原子に基づく(atom-based)ものです。
これに対して分子の特性をもとに表現するアプローチとして分子記述子(molecular descriptor)があります。
単純化するとこんな感じ…

すごいたくさん種類があるらしい(Dragonソフトウェアでは4885種類!)ですが、以下では大きく2つの分類(Structural Kyes, Hashed fingerprints)が紹介されています。
Structural Keys
構造キー(structural key)は特定の化学構造が有る(1)か、無い(0)かにしたがってエンコードします。
2つの例が挙げられています。

MACCSキーの注意点として、フラグメント166(or 960)種のセットに基づくといっても、ソフトごとに実装が違う場合があるので、異なるソフトでは同じ構造が異なるビット列に割り当てられる可能性がある、という点が指摘されています。
Hashed fingerprints
Chemical fingerprintはベクトルで、
- 各要素は物理化学的特性あるいは構造の特徴をエンコード
- それら要素がインデックス化(順序をつけられて)されて並べられて
います。
先の構造キーでは表す特徴のパターンがあらかじめ定義されていましたが、ハッシュ化されたフィンガープリントでは、各特徴が分子そのものから生成されるという点が異なります。
ビット列の長さを決めた上で生成し、分子の持つパターンをハッシュ関数によってビットに割り当てます。
2種類例が挙げられています。

Chemoinfomaticsでフィンガープリントがよく用いられるのは、 「分子のグラフ構造を直接的に素早くベクトル表現へと変換できるので、数理モデル化の入力として使える」ということにあるようです。
また、柔軟性のある表現なので、物理化学的特性を整数(integer, ex. 水素結合の数)や浮動小数点(float, ex. 分子量)にエンコードできるというのも魅力のようです。
線形表現については以上でお終いです。
つづいて話は変わって化学反応をどう表現するか?という話題が取り上げれています。ちょうど良いのでここで区切ります。
途中に出てきたIUPACによる分類指針を参考にここまでの分子の表現をざっくりまとめると次のようになりますでしょうか?

SMILESの派生系などを全て無視して単純化しすぎな分類ですが・・・
AI applications within drug discovery using molecular representations
今回も途中をすっ飛ばして線形表現の応用について触れた箇所に飛んでおきます。
Linear notations for small molecules
線形表現(SMILES、molecular fingerprint)は分子の特性予測やQSARでよく利用されます。
SMARTSは部分構造を定義し、関連する化合物を選択したり取り除いたりする目的で使われます。
ディープラーニングと関係する内容としては、
- 自然言語処理(NLP)のツールを使ったde novo分子デザインの分野でSMILESが活躍
- randomized SMILESを使ったデータ拡張
- オートエンコーダーを使った潜在空間表現の学習による分子特性の予測
といったものが挙げられます。
ニューラルネットワークでは分子のベクトル表現を学習したうえで予測の利用しますが、 これは古典的な機械学習のアプローチでハッシュ化されたフィンガープリントを入力として使うことに類似しているので、 「学習されたフィンガープリント(lerned fingerprint)」とも呼ばれるそうです。
以上が線形表現の応用でした。
次回、化学反応の表現に続く!
途中浅い理解で雑な表にまとめたりしているので、色々と間違いがあると思います。ご指摘・ご教示いただければ幸いです
「分子の表現についてのレビューと実践ガイド」のメモ ~part 1 グラフ表現~
HTSなどの技術の発展に伴い実験データが増加し、計算機による解析が盛んになるにつれて、コンピューターが処理可能かつ、様々な分野の科学者が理解できるような分子の表現(Molecular representation) が必要とされるようになってきました。
たくさん開発されてきた表現方法の中で、特に医薬品探索で使われる電子的な分子の表現 (electronic molecular representation)と巨大分子の表現(macromolecular representation)にフォーカスを当てて解説しちゃうよ!
・・・って感じのイントロのレビューを読みました。オープンアクセス万歳!!(CC by 4.0)
David, L., Thakkar, A., Mercado, R. et al. Molecular representations in AI-driven drug discovery: a review and practical guide. J Cheminform 12, 56 (2020).
知らないことがたくさんあったのと、「グラフ表現 → 線型表現」という説明の順が面白かった(1D → 2D の説明が多い気がする)ので備忘録を残します。
と言っても英語だと覚えられない自分のための雑メモですが・・・
長いので4回にわけて見ていきます。1回目はグラフ表現!
なお、内容は一部前後しますが、目次は論文そのままにしてます。
- Graph representations for small molecules
- Mathmatical definition of graph
- Graph traversal algorithms
- Advantages of graph representations
- Limitations of molecular graph representations
- Connection tables and the MDL file formats
- AI applications within drug discovery using molecular representations
Graph representations for small molecules
まずは分子グラフ(Molecular Graphs)をきちんと理解しましょう。

分子グラフは2次元のオブジェクトですが、3Dの情報(ex.原子座標、結合角、不斉点)を表すこともできます。 また、簡単に可視化できるソフトも多くあります。
注意点として、ノード間の関係性は属性(attribute)としてノード and/or エッジにエンコードしておく必要があります。
理由は、グラフのノードは数学的なオブジェクトであって(正式には)空間の位置を持っておらず、対ごとの関係性(pairwise)しか保持していないからです。
Mathmatical definition of graph
グラフはタプルで定義されます。

上は抽象的な数学の概念なので、コンピューターで扱えるように線形データ構造(linear data structure)にマッピングします。

一般的な表現方法は隣接行列(adjacency matrixあるいはconnectivity matrix)です。

原子や結合の種類は特徴行列(feature matrix)の形式で表すことができます。 下の図ではone-hotエンコーディングで表していますが、要素の値を整数(integer)として、よりサイズの小さい行列とすることもできます。

Graph traversal algorithms
非線形データ構造(non-linear data structure)のグラフを線形の行列表現にするにあたって、ノードに番号をつける必要が生じますが、この番号の付け方によって表現が変わってしまいます。
一つの分子を一貫して同じ行列表現で表すために、番号をつけるアルゴリズム(graph traversal algorithm)が重要となります。
実際、ソフトウェアごとのグラフ検索における分岐の切り方の違いの影響で、得られる行列表現が変わってしまうことがあります。
ただし、表現の一貫性を重視しないような場合、例えばディープラーニングへの応用で、よりノイズの多いデータが欲しいといった場合には、ランダムサーチを用いることもできます。
Fig. 2ではよく使われるグラフ検索アルゴリズムが図示されています。

Advantages of graph representations
分子をグラフで表現することの利点は、
- 3Dの情報を自然に埋め込むことができること
- サブグラフが解釈可能であること
です。
利点①は特徴行列にあわらされている通りです。
利点②はサブグラフを部分構造として解釈できるということを意味するのでしょうか???比較として、文字列表現であるSMILESの一部(substirngs)を取り出しても、妥当性のあるグラフとなるとは限らない、ということをもとに理解すれば良いようです。
Limitations of molecular graph representations
Breakdown of graph model
反対に欠点として「グラフでは表せない分子」の例が挙げられています。
欠点①は、原子価結合法(valence bond theory)で説明できないもので、原子間のペアワイズな関係性だけ表すのが難しい、というのがその理由です。
①の解決策の一例としてhypergraphが紹介されています。
Hypergraphでは、エッジを原子のタプルではなく、少なくとも2つの原子からなる一群のセット(hyperedge)として表すので、多価結合(multi-valent bond)を表すことができます。
欠点②は、結合が切断されたりで生じたりする分子は、静的なグラフ表現1つでは表し切れない、表現してもあまり意味がない、といったことのようです。
Challenges of working directly with the graph representation
またその他の欠点として、「グラフ表現が(メモリ的にも、文字通りサイズ的にも)コンパクトではない」ということが指摘されています。
行列にしろ図にしろ、グラフは(1D)線形表現と比べて
- 検索が困難
- 必要なメモリが大きい
ため、多数の分子を扱う時に問題となるようです。
Connection tables and the MDL file formats
続いて分子のグラフ表現と関係の深いフォーマットとして、connection table*1とMDLファイルフォーマット*2があげられています。
## connection tables
connection table(Ctab)は以下の図のようなもので、6つの部分に分かれます。

connection tableは化学構造情報の取り扱いにおける標準的なフォーマットの一つで、Molファイルフォーマットの下地になっています。
atom block部分の注意点として、より実用的には水素原子を暗に (implicit) 取り扱うということがあります。
具体的には、
- 水素原子をheavy atomのプロパティとして取り扱う
- 利点はatom blockとbond blockのサイズを小さくすることができること
- 水素原子は必要に応じて原子価モデル(valence model)のルールに基づいて再計算できる
- 再計算のため、価数の情報を明示しておく必要がある
という感じです。
The Molfile format
MDLにより開発された、Molファイルフォーマットを含む一連のファイルをCTファイル(CTfiles、chemital table files)と呼びます。
CTファイルでは分子の構造を表現するのにconnection tableを使用します。つまり、connection table自体はファイルフォーマットではない、ということに注意してください。
Molファイルは拡張が可能で、SDファイル(structure/data file)では複数の分子の構造情報とその他のプロパティのデータを含むことができるようになっています。

以上が分子のグラフによる表現とそれを格納するためのファイル形式でした。
論文では続いて線形表記が紹介されていますが、長くなったので一度表現の紹介は中断します。
AI applications within drug discovery using molecular representations
論文の順序とは前後してしまいますが、この論文の主題は「医薬品探索におけるAIの利用と分子の表現」ですので、 忘れないうちにグラフ表現の応用可能性について触れられた箇所をご紹介しておきたいと思います。
Graph representations for small molecules
この文献で取り上げられた分子の表現は、それ自体がディープラーニングの表現学習(representation learning)に使われています。
表現学習の考え方は
- 与えられたオブジェクト(e.g. 分子)の内部表現(e.g. ベクトル)を学習し
- その表現を予想タスクに用いる
というものです。
表現学習においては、何を入力とするかが大事で
- 入力に適した分子の表現をまずみつけること
- その表現が問題を解くのに必要とされる情報を十分に有している事
が最初のポイントとなります。
古典的な機械学習(RFやSVM etc.)とは異なり、ディープニューラルネットワーク(DNN)を使ったアプローチでは、基本的に表現学習を行っています。
最近の医薬品探索の分野ではグラフニューラルネットワークの発達とともに、分子のグラフ表現が利用される流れが来ています。
以下のような応用例があります。
- 分子の特性の予測 (property prediction)
- 分子グラフの生成 (molecular graph generation, de novo design)
- 合成ルートの提案 (synthesis prediction)
やり方の流れとしては大きく以下の2段階となっています。
- グラフ表現学習の実施
- グラフ表現全体から
- グラフネットワークを使って
- グラフの埋め込み(graph embedding)を取得
- 学習した表現を入力として予測モデルを構築
- RFやDNNなど
- ここは古典的なフィンガープリントを使うときと一緒
以前はよりコンパクトな線形表現が好まれていたそうですが、 最近はちょっとずつ変わってきていて、(メモリを食う)グラフ表現も使われるようなってきているそうです。
分子グラフの他の利用としては、原子レベルのシミュレーションの入力(e.g. 分子動力学)が挙げられていました。 力場を使ったエネルギー計算に原子配置や結合の情報が必要となるため応用されているとのことです。
以上、グラフ表現のAIへの応用でした。
関連する内容として、文献では以下のレビューがオススメされていました。
- 化学と医薬品探索におけるディープラーニングの応用
- Mater AC, Coote ML (2019) Deep learning in chemistry. J Chem Inf Model 59:2545–2559
- Chen H, Engkvist O, Wang Y, Olivecrona M, Blaschke T (2018) The rise of deep learning in drug discovery. Drug Discov Today. 23(6):1241–1250
- 生成モデル
- Sanchez-Lengeling B, Aspuru-Guzik A (2018) Inverse molecular design using machine learning: generative models for matter engineering. Science. 361(6400):360–365
では、次回線形表現に続く!
途中浅い理解で雑な表にまとめたりしているので、色々と間違いがあると思います。 ご指摘・ご教示いただければ幸いです。特にハイパーグラフはなんのことやら分からなかった・・・
TeachOpenCADD トピック11(パートC)〜オンラインAPI/サービスを使った構造に基づくCADD〜
こちらはTeachOpenCADDの試訳です。TeachOpenCADD GitHubレポジトリのトークトリアル11をもととしておりCC BY 4.0のライセンスに従っています。
トークトリアル11は論文発表時のTeachOpenCADDには含まれておらず新しく追加されたものです。
4つのノートブックに分かれており、ここではその4つ目を訳出します。
パートCではパートBで行ったドッキング結果を解析しますが、パートBで使われていたオンラインサービスはすでに利用できなくなっていたため、ローカル環境ドッキングを行いました(実施方法はこちらの記事に記載いたしました)。
AutoDockVinaの2つの出力ファイルlog.txt、out.pdbqtをそれぞれファイル名を変更してlocal_vina.out、local_result.pdbqtとしてdataフォルダに格納し解析を行っています。
~~ 追記 ~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~
以下のページに翻訳したノートブックを保存しています。よろしければご利用ください。
~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~
以下、訳。
トークトリアル 11 (パートC)
オンラインAPI/サービスを使った構造に基づくCADD
Developed at AG Volkamer, Charité
Dr. Jaime Rodríguez-Guerra, Dominique Sydow
このトークトリアルの目的
これは「オンラインWebサービス」についてのトークトリアルのパートCです。:
- 11a. キナーゼ阻害剤の候補をKLIFとPubChemで検索
- 11b. 11aで取得した候補化合物をターゲットタンパク質に対してドッキング
- 11c. 結果を評価し既知のデータと比較
入力構造を取得し、ドッキングが終わったので、結果が役に立つものかどうか評価します。
学習の目標
理論
- タンパク質ーリガンド相互作用
- ドッキングの偽陽性
実践
- 結果の可視化
- 自動化した解析の実行
レファレンス
- タンパク質ーリガンド相互作用に関するレビュー(Int. J. Mol. Sci. (2016), 17, 144)
- PDBデータベースにおける非共有性結合相互作用の系統的な解析 (M. Med. Chem. Commun. (2017), 8, 1970-1981)
- タンパク質ーリガンド相互作用が医薬品の作用にとって鍵となることについての次の書籍の中の章(Klebe G. (eds) Drug Design. Springer, Berlin, Heidelberg. DOI: 10.1007/978-3-642-17907-5_4)
- Notebookのためのインタラクティブな可視化ソフト、NGLView (Bioinformatics (2018), 34, 1241–124)
- 自動的にタンパク質ーリガンド相互作用を見つけるためのPLIP(Nucl. Acids Res. (2015), 43, W1, W443-W447)
理論
タンパク質ーリガンド相互作用
リガンドの結合は、リガンドとタンパク質ポケットの表面との間、あるいはタンパク質ータンパク質境界面における非共有結合性相互作用によって主に支配されています。このプロセスは、静電的相補性と形状相補性、誘導適合(inducued fitting)、脱溶媒和過程、その他に応じて変化するものです。
文献からいくつか引用します。
José L. Medina-Franco, Oscar Méndez-Lucio, Karina Martinez-Mayorgaから改変して引用します。
タンパク質ーリガンド相互作用(protein-ligand interactions, PLIs)とタンパク質ータンパク質相互作用(protein-protein interactions、PPIs)を理解することは、分子の認識における核となる部分で、多くの科学分野において重要な役割を果たします。PLIsとPPIsは医薬品探索において幅広い分野で実践的な応用があります。含まれるものをいくつかあげれば、ドッキング、構造に基づくデザイン、フラグメント化合物と低分子化合物、そして他の種類の化合物のバーチャルスクリーニング、複合体のクラスタリング、そしてアクティビティクリフの構造的な解釈といったものがあり、他にもあります。
もちろん、これらの相互作用はいくつかの方法によって合理的に説明することが可能で、ドッキング結果の系統的な解析への道を開きます。例えばMed. Chem. Commun., 2017,8, 1970-1981改変して引用します。
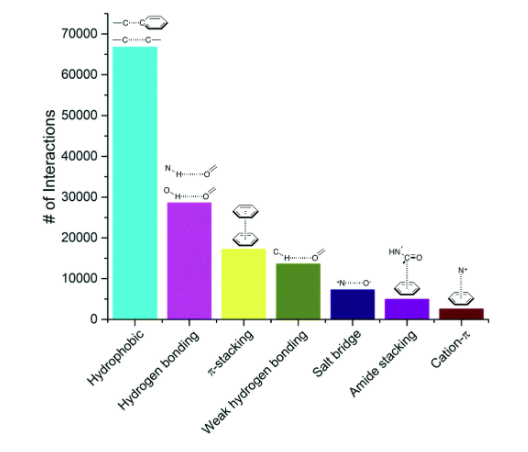
私たちはPDBから、タンパク質と複合体を形成している低分子化合物で、分解能2.5Å以下の全てのX線構造を抽出し、その結果11,016個の複合体のコレクションを得ました。リガンドとして考慮するためには、化合物は低分子である、医薬品化学の応用にとって興味深いものである、といったいくつかの判断基準を満たしていなければなりません(緩衝液や結晶作成用の混合物の一部は除外します)。このコレクションには750,873のリガンドータンパク質の原子のペアが含まれており、ここでは原子のペアは4Å以内の距離にある2つの原子として定義されています。最も頻度の高いトップ100のリガンドータンパク質原子ペアは7つの相互作用のタイプに分類することができました(下の図を参照してください)。最も頻度高くみられた相互作用の中には、疎水性相互作用(hydophobic contact)、水素結合そしてπ-スタッキングといった、リガンドデザインにおいてよく知られており、広く使われている相互作用がありました。それらに続いて、弱い水素結合、塩橋、アミドスタッキング、そしてカチオンーπ相互作用といったものがありました。
タンパク質ーリガンド相互作用を自動的に評価するプログラムはいくつかあります。最も人気のあるプログラムの一つはPLIPで、Webサーバーが公に利用可能で無料で使えるPythonライブラリがあるというのが人気の理由です。文献に付随するsupporting informationはタンパク質ーリガンド相互作用を簡潔に解説しており、非常に簡単に理解できるようになっています。導入としては、次のパラグラフで十分でしょう。
PLIPはタンパク質残基とリガンドの非共有結合性相互作用をみつけるためのルールベースのシステムを使っています。タンパク質とリガンドの接触している原子間の非共有結合性相互作用の特徴を見つけるために、特定の相互作用に寄与することができる化学官能基(例、水素結合のドナーに必要とされるもの)と相互作用の幾何学的特徴(例、距離と角度の閾値)についての文献に基づく情報を使います。各結合サイトについて、このアルゴリズムは最初に、特定の相互作用のパートナーとなりうるタンパク質とリガンドの原子、あるいは原子団を探します。ついで、相互作用を形成するタンパク質とリガンドの条件に適合したグループに対して幾何学的なルールが適用されます。
より詳細な情報が必要なら、PDFドキュメントをチェックしてください!
# "ファイル > ライブPDFプレビューを使うためにこのNotebookを信頼する"をクリックする必要があるかもしれません import requests from IPython.display import HTML pdf = "https://www.ncbi.nlm.nih.gov/pmc/articles/PMC4489249/bin/supp_gkv315_nar-00254-web-b-2015-File003.pdf" display(HTML(f""" <iframe src="https://docs.google.com/viewer?url={pdf}&embedded=true" frameborder="0" webkitallowfullscreen mozallowfullscreen allowfullscreen width="900" height="600"> </iframe> """))
訳注(2020/05)
ここにはPLIPの文献PDFが表示されていますが、サイズの問題で反映させていません。PubMedのこちらのページでSUPPLEMENTARY DATAをご確認ください。
訳注ここまで
偽陽性
ドッキングソフトウェアは最終的な答えをあたえるものではありません!全ての結果は厳密に検証しなければなりません。例えば、このレビューL.Yang, J.Zhang, X.Che, Y.Q.Gao, in Methods in Enzymology, 2016 では次の指摘がなされています。
タンパク質ーリガンド相互作用の研究においては、リガンドの自由度も重要な役割を果たす可能性があります。しかしながら、リガンド/タンパク質相互作用の研究においては、通常非常に限られた数のコンフォメーションしか考慮されません。一方で、リガンド/基質ータンパク質の相互作用を調べ、予測する上で、リガンドのコンフォメーションの集合を徹底的に調べることが極めて重要であることを示す例が増えています。さらに、構造ベースの医薬品デザインにおいて、テストされたリガンドのコンフォメーションが続いて行う計算の基礎となります。特に、リガンドは作用するタンパク質に結合する前と結合した後で、非常に異なるコンフォメーションをとることがあり、多くのリガンドが局所最小エネルギーのコンフォメーションではタンパク質に結合していないことを示す証拠があります。そのような場合、自由なリガンド単体のコンフォメーションの不適切な解析は、誤った結合エネルギー予測につながり、さらに酵素反応が含まれている場合には問題となる可能性があります。したがって、自由なリガンド単体の溶液中の構造を徹底的にサンプリングすることが必要で、それは適切な強化されたサンプリング手法と組み合わせたMDシミュレーションによって費用対効果良く実現することができます。
これはありうる失敗する点のうちの一つです。
実践
結果の可視化
nglviewを使いましょう!Webベースの分子ビューワーでJupyter Notebook上で実行することができます!また、ボックスから取り出したPDBQTファイルとも互換性があります(が、最初のモデルだけしか読み込みません。。。これをどうするかは後ほどとりあげます)。
nglviewをインストールするには次を実行してください。
!pip install nglview ipywidgets>=7.5 !nglview install !nglview enable
ノートブックでインタラクティブなGUIを作成するためにipywidgetsを使います。これにより、異なるリガンドをクリックし、クリックに応じてビューワーが更新されるようになります。特に、我々の小さなGUIには以下のことをさせたいと思います。
- Vinaの出力で報告されているように、ポーズと親和性のリストを表示する
- タンパク質構造をリボン表示、リガンドを Ball&Stick、そして周りの残基をlicorice(Stickのみ)で表示する
- 3Dの可視化がユーザーによるリストの異なるポーズの選択に応答するようにする
つまり、私たちは次のことをする必要があります。
- NGLビューワーが適切な描画をするよう呼び出す
- 結果のインタラクティブなテーブルを作成する(ヒント:
ipywidgets.Selectを使ってください) - NGLビューワーとコミュニケーションできるイベントハンドラーを書く。つまりユーザーが新しいエントリをクリックすると、ディスプレイのリガンドと周囲の残基を更新するようにします(リボン表示は更新する必要がない)。
import pandas as pd import time import nglview as nv # AppLayoutをつかうにはipywidgetsがv7.5+でなければなりません from ipywidgets import AppLayout, Layout, Select, Button
Vinaの作成したPDBQTファイルはいくつかモデルを含んでいますが、nglviewは最初のものしか解析しません。この問題を回避する方法は単純です。ENDMDL行が見つかる度にスプリットすることでファイルを各モデルに分割します。
def split_pdbqt(path): """ 複数のモデルのPDBQTを別々のファイルに分けます """ files = [] with open(path) as f: lines = [] i = 0 for line in f: lines.append(line) if line.strip() == 'ENDMDL': fn = f'data/results.{i}.pdbqt' with open(fn, 'w') as o: o.write(''.join(lines)) files.append(fn) i += 1 lines = [] return files
Vinaの出力は結果のテーブルを含む単純なテキストファイルです。テーブルの解析は比較的簡単です。必要であれば、単純な可視化のためにpandas.DataFrameを返します。
def parse_output(out): """ Vina出力ファイルからDataFrameを作成します """ with open(out) as f: data = [] for line in f: if line.startswith('-----+'): line = next(f) while line.split()[0].isdigit(): index, *floats = line.split() data.append([int(index)] + list(map(float, floats))) line = next(f) return pd.DataFrame.from_records(data, columns=['Mode', 'Affinity (kcal/mol)', 'RMSD (l.b.)', 'RMSD (u.b.)'], exclude=['Mode'])
Vinaの出力を解析
作成した関数をつかって次のテキストファイルを…
# !cat data/vina.out # T11bでオンラインの取得がうまくいかなかったためローカル環境で # AutoDock Vinaを実施した結果を用いています。 !cat data/local_vina.out
#################################################################
# If you used AutoDock Vina in your work, please cite: #
# #
# O. Trott, A. J. Olson, #
# AutoDock Vina: improving the speed and accuracy of docking #
# with a new scoring function, efficient optimization and #
# multithreading, Journal of Computational Chemistry 31 (2010) #
# 455-461 #
# #
# DOI 10.1002/jcc.21334 #
# #
# Please see http://vina.scripps.edu for more information. #
#################################################################
Detected 4 CPUs
Reading input ... done.
Setting up the scoring function ... done.
Analyzing the binding site ... done.
Using random seed: 267553816
Performing search ... done.
Refining results ... done.
mode | affinity | dist from best mode
| (kcal/mol) | rmsd l.b.| rmsd u.b.
-----+------------+----------+----------
1 -6.8 0.000 0.000
2 -6.8 5.072 7.461
3 -6.7 5.243 7.469
4 -6.6 4.035 6.317
5 -6.6 6.833 8.653
6 -6.6 8.876 10.836
7 -6.5 7.611 9.316
8 -6.5 1.094 2.109
9 -6.5 7.680 9.301
Writing output ... done.
綺麗にフォーマットされたpandas.DataFrameに変換することができます。
# parse_output("data/vina.out") parse_output("data/local_vina.out")
| Affinity (kcal/mol) | RMSD (l.b.) | RMSD (u.b.) | |
|---|---|---|---|
| 0 | -6.8 | 0.000 | 0.000 |
| 1 | -6.8 | 5.072 | 7.461 |
| 2 | -6.7 | 5.243 | 7.469 |
| 3 | -6.6 | 4.035 | 6.317 |
| 4 | -6.6 | 6.833 | 8.653 |
| 5 | -6.6 | 8.876 | 10.836 |
| 6 | -6.5 | 7.611 | 9.316 |
| 7 | -6.5 | 1.094 | 2.109 |
| 8 | -6.5 | 7.680 | 9.301 |
Vinaの出力には3つの列があります。
- 予測された結合親和性
- 対称性修正アルゴリズム(symmetry-corrected algorithm)を使った最良の解(親和性最小値)に対するRMSD
- 対称性修正(symmetry correction)を用いていない最良の解(親和性最小値)に対するRMSD
私たちは親和性にだけ興味があるので、次のセルではpandas.DataFrameのために列を一つだけ取得しています。
それでは、NGLビューワーのインスタンスを作成します。各タンパク質ーポーズのペアについて新しいインスタンスを作成する代わりに、同じ描画面を通して再利用し、必要なリガンドを隠すか表示します。最初に全てを読み込み、リガンドをそれぞれの親和性でラベルづけします。ビューワーに読み込まれた各分子を「component」とよびます。最初にタンパク質を読み込むので、component_0となります。リガンドはcomponent_1から始まり続いていきます。
import time def create_viewer(protein, ligands, affinities): """ タンパク質と親和性のラベルをつけた全てのリガンドをもつnglview widgetを作成します """ viewer = nv.NGLWidget() viewer.add_component(protein, ext="mol2") # 親和性のラベルを有する分子(@0)の最初の原子を選択します label_kwargs = dict(labelType="text", sele="@0", showBackground=True, backgroundColor="black") for ligand, affinity in zip(ligands, affinities): ngl_ligand = viewer.add_component(ligand, ext="pdbqt") ngl_ligand.add_label(labelText=[str(affinity)], **label_kwargs) return viewer
そして最後に、次の下のセルで実際のGUIを作成します!
ipywidgets.AppLayoutレイアウトを使って水平に配置された2つのwidgetsからなります。
- セレクター(
ipywidgets.Select) - NGLビューワーそのもの
ユーザーがセレクターで新しいエントリーをクリックすると、_on_selection_changeが呼ばれ以下のことを行います。
- 新しい値が前のものと異なるかを確認します。異なる場合、次に…
- 全てのリガンドを隠しビューをリセットします
- 選択したものを表示し、500msの格好いいアニメーションとともにカメラの中心に合わせます
- 新しいポーズの重心の5Å内にある残基のリストを更新するためにNGLビューワーにJavaScriptを実行します。
# Python widgetからは隠されているので、 # リガンドの周りの残基を更新するにはJavaScriptのコードが必要です。 # http://nglviewer.org/ngl/api/manual/snippets.html に基づきます _RESIDUES_AROUND = """ var protein = this.stage.compList[0]; var ligand_center = this.stage.compList[{index}].structure.atomCenter(); var around = protein.structure.getAtomSetWithinPoint(ligand_center, {radius}); var around_complete = protein.structure.getAtomSetWithinGroup(around); var last_repr = protein.reprList[protein.reprList.length-1]; protein.removeRepresentation(last_repr); protein.addRepresentation("licorice", {{sele: around_complete.toSeleString()}}); """ def show_docking(protein, ligands, vina_output): # 複数のPDBQTリガンドファイルを別々のファイルに分割する ligands_files = split_pdbqt(ligands) # 親和性('Affinity')の取得(DataFrameのこの列だけが必要です) affinities = parse_output(vina_output)['Affinity (kcal/mol)'] # viewer widgetの作成 viewer = create_viewer(protein, ligands_files, affinities) # selection widgetの作成 # オプションは(text, value)のタプルのリストです。select上でクリックすると、 # `.observe(...)`に登録されている呼び出し可能なもの(callable)にvalueが渡されます。 selector = Select(options=[(f"#{i} {aff} kcal/mol", i) for (i, aff) in enumerate(affinities, 1)], description="", rows=len(ligands_files), layout=Layout(width="auto")) # GUI要素の配置 # selectionボックスを左、viewerをウィンドウの残りの部分を占めるようにします。 display(AppLayout(left_sidebar=selector, center=viewer, pane_widths=[1, 6, 1])) # これがイベントハンドラーです。ユーザーがselectionボックスでクリックした時に動作を取り出します。 # viewerの変数を「見る」ことができるようにここで定義する必要があります。 def _on_selection_change(change): # ユーザーが異なるエントリーでクリックした時だけ更新する if change['name'] == 'value' and (change['new'] != change['old']): viewer.hide(list(range(1,len(ligands_files) + 1))) # 全てのリガンドを隠す(components 1-n) component = getattr(viewer, f"component_{change['new']}") component.show() # 選択した一つを表示 component.center(500) # viewをズームする # リガンド周りの残基を表示するためにJSコードを呼ぶ viewer._execute_js_code(_RESIDUES_AROUND.format(index=change['new'], radius=5)) # イベントハンドラーの登録 selector.observe(_on_selection_change) # 最初の解に焦点をあてるために手動でイベントを起こす _on_selection_change({'name': 'value', 'new': 1, 'old': None}) return viewer
上の関数ではPythonの応用をいくつか行なっています。もし興味があれば、下のヘッダーをクリックしてより詳細について見ることができます。
最初に、JavaScriptとPythonのコードを混ぜていることに気づいたかもしれません!これは
NGLViewer.Viewer._execute_js_codeで提供されているように、ipywidgetsの基盤のおかげで可能になっています。ここで(JavaScriptのコードを含む)文字列を渡しておくと、後でNGL widgetのスコープで実行されます(thisはインタラクティブな描画面を差します)。パラメーター化するために、_on_selection_changeを呼ぶ度にフォーマット化されるテンプレート・プレースホルダーを追加しました。この関数
_on_selection_changeがユーザーのインタラクション(selectionボックス上でのクリック)をPythonの世界と繋げる糊の役割を果たします。selectionボックス上でクリックする度ごとに呼ばれ、引数changeを一つもちます。イベントのタイプと新旧両方の値を保持する辞書です(したがって比較し、それを取りあつかうことができます)。しかしながら、vewerを関数に含める必要もあります…そして、追加の値を渡すことはできません!関数で
viewerを使えるようにする一つの方法はshow_docking関数にその定義をネストすることです。そうすることで、Notebookの(global)scopeの中を探す必要なく外側の領域にアクセすることができるようになります。 このポストでscopeとclosureについて情報をもっと見ることができます。
ドッキング結果を見る
# viewer = show_docking("data/protein.mol2", "data/results.pdbqt", "data/vina.out") viewer = show_docking("data/protein.mol2", "data/local_result.pdbqt", "data/local_vina.out")
訳注(2020/05)
以下は画像ですが、ノートブック上ではインタラクティブなビューワーが表示されます。
訳注ここまで
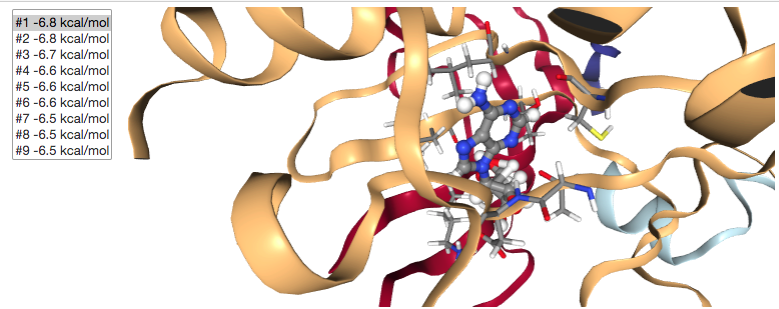
viewer.render_image(),
(Image(value=b'', width='99%'),)
# 静的な出力
viewer._display_image()
import time time.sleep(10)
タンパク質ーリガンド相互作用の解析
PLIPは自動化された解析のためのWebサーバー を提供していますが、残念ながらAPIはありません。標準のWeb UIを使っているかのようにHTML形式を使ってみることもできますが、ライブラリ自体がPython-3で使えるように準備されており、pipで簡単にインストールすることができるので、簡単のためローカルで使うことができます。
!conda install -y -c openbabel openbabel lxml !pip install https://github.com/ssalentin/plip/archive/stable.zip --no-deps
from plip.modules.preparation import PDBComplex from plip.modules.report import BindingSiteReport from glob import glob import os import pandas as pd
PILIPはPDBファイルだけを受け入れますが、PDBQTファイルはPDBの拡張された記法なので、相互の変換は列とレコードのうち正しい部分を選択するだけの問題です。この場合、ATOM(タンパク質原子)とHETATM(リガンド原子)レコードにだけ興味があるので、追加のフィールド(66列以降)を廃棄します。ついで、単に連結を行うだけでそれらを合わせてまとめることができます。
def pdbqt_to_pdbblock(pdbqt): lines = [] with open(pdbqt) as f: for line in f: if line[:6] in ('ATOM ', 'HETATM'): lines.append(line[:67].strip()) return '\n'.join(lines) def create_protein_ligand_pdbs(protein_pdbqt, ligands_pdbqt): ligands_pdbqt = sorted(list(glob(ligands_pdbqt)), key=lambda s: int(s.split('.')[-2])) protein_pdbblock = pdbqt_to_pdbblock(protein_pdbqt) for i, ligand_pdbqt in enumerate(ligands_pdbqt, 1): ligand_pdbblock = pdbqt_to_pdbblock(ligand_pdbqt) with open(f"data/docked_protein_ligand.{i}.pdb", 'w') as f: f.write(protein_pdbblock) f.write("COMPND UNNAMED\n") f.write(ligand_pdbblock)
ジャジャーン!できました!
create_protein_ligand_pdbs("data/protein.mol2.pdbqt", "data/results.*.pdbqt")
これでPDBファイルをPLIPに渡し、その魔法が使えるようになりました。BindingSiteReportクラスはPDBComplexの検出された各結合サイトを処理し、興味のあるフィールドをもつオブジェクトを作成します。
hydrophobichbondwaterbridgesaltbridgepistackingpicationhalogenmetal
これらのフィールドは(列名を保持する)<field>_featuresと(実際のレコードを保持する)<field>_infoに分けられます。オブジェクトを順に処理してgetattr()で正しい属性(attribute)の名前を取得すれば、概要をよくみるためにpandas.DataFrameに渡す辞書を構成することができます。以下をチェックしてください。
def analyze_interactions(pdbfile): protlig = PDBComplex() protlig.load_pdb(pdbfile) # pdbの読み込み for ligand in protlig.ligands: protlig.characterize_complex(ligand) # リガンドを見つけ相互作用を解析する sites = {} for key, site in sorted(protlig.interaction_sets.items()): binding_site = BindingSiteReport(site) # 相互作用に関するデータをあつめる # *_featuresと*_infoのタプルをpandas dfに変換します keys = "hydrophobic", "hbond", "waterbridge", "saltbridge", "pistacking", "pication", "halogen", "metal" interactions = {k: [getattr(binding_site, k+"_features")] + getattr(binding_site, k+"_info") for k in keys} sites[key] = interactions return sites
interactions_by_site = analyze_interactions("data/docked_protein_ligand.1.pdb")
interactions_by_site
{'A:A:1': {'hydrophobic': [('RESNR',
'RESTYPE',
'RESCHAIN',
'RESNR_LIG',
'RESTYPE_LIG',
'RESCHAIN_LIG',
'DIST',
'LIGCARBONIDX',
'PROTCARBONIDX',
'LIGCOO',
'PROTCOO')],
'hbond': [('RESNR',
'RESTYPE',
'RESCHAIN',
'RESNR_LIG',
'RESTYPE_LIG',
'RESCHAIN_LIG',
'SIDECHAIN',
'DIST_H-A',
'DIST_D-A',
'DON_ANGLE',
'PROTISDON',
'DONORIDX',
'DONORTYPE',
'ACCEPTORIDX',
'ACCEPTORTYPE',
'LIGCOO',
'PROTCOO'),
(45,
'LYS',
'A',
1,
'A',
'A',
True,
'2.64',
'3.42',
'133.60',
True,
431,
'N3+',
2953,
'O3',
(3.232, 17.45, 38.708),
(4.782, 15.554, 36.326)),
(150,
'ASP',
'A',
1,
'A',
'A',
False,
'3.07',
'3.43',
'102.37',
True,
1470,
'Nam',
2955,
'O3',
(1.743, 18.722, 37.254),
(0.523, 19.864, 34.258)),
(45,
'LYS',
'A',
1,
'A',
'A',
False,
'2.66',
'3.10',
'106.87',
True,
422,
'Nam',
2951,
'O3',
(5.392, 21.015, 40.948),
(7.994, 19.469, 41.639)),
(72,
'LEU',
'A',
1,
'A',
'A',
False,
'3.65',
'4.01',
'103.83',
True,
694,
'Nam',
2945,
'Npl',
(3.257, 24.961, 34.128),
(2.841, 27.919, 36.804)),
(61,
'MET',
'A',
1,
'A',
'A',
False,
'2.75',
'3.76',
'161.43',
False,
2945,
'Npl',
595,
'O2',
(3.257, 24.961, 34.128),
(2.892, 28.611, 33.325)),
(149,
'THR',
'A',
1,
'A',
'A',
True,
'2.02',
'2.89',
'147.03',
False,
2955,
'O3',
1467,
'O3',
(1.743, 18.722, 37.254),
(0.131, 21.066, 36.729)),
(150,
'ASP',
'A',
1,
'A',
'A',
True,
'2.70',
'3.64',
'162.21',
False,
2953,
'O3',
1478,
'O3',
(3.232, 17.45, 38.708),
(1.632, 16.073, 35.738))],
'waterbridge': [('RESNR',
'RESTYPE',
'RESCHAIN',
'RESNR_LIG',
'RESTYPE_LIG',
'RESCHAIN_LIG',
'DIST_A-W',
'DIST_D-W',
'DON_ANGLE',
'WATER_ANGLE',
'PROTISDON',
'DONOR_IDX',
'DONORTYPE',
'ACCEPTOR_IDX',
'ACCEPTORTYPE',
'WATER_IDX',
'LIGCOO',
'PROTCOO',
'WATERCOO')],
'saltbridge': [('RESNR',
'RESTYPE',
'RESCHAIN',
'RESNR_LIG',
'RESTYPE_LIG',
'RESCHAIN_LIG',
'DIST',
'PROTISPOS',
'LIG_GROUP',
'LIG_IDX_LIST',
'LIGCOO',
'PROTCOO')],
'pistacking': [('RESNR',
'RESTYPE',
'RESCHAIN',
'RESNR_LIG',
'RESTYPE_LIG',
'RESCHAIN_LIG',
'CENTDIST',
'ANGLE',
'OFFSET',
'TYPE',
'LIG_IDX_LIST',
'LIGCOO',
'PROTCOO')],
'pication': [('RESNR',
'RESTYPE',
'RESCHAIN',
'RESNR_LIG',
'RESTYPE_LIG',
'RESCHAIN_LIG',
'DIST',
'OFFSET',
'PROTCHARGED',
'LIG_GROUP',
'LIG_IDX_LIST',
'LIGCOO',
'PROTCOO')],
'halogen': [('RESNR',
'RESTYPE',
'RESCHAIN',
'RESNR_LIG',
'RESTYPE_LIG',
'RESCHAIN_LIG',
'SIDECHAIN',
'DIST',
'DON_ANGLE',
'ACC_ANGLE',
'DON_IDX',
'DONORTYPE',
'ACC_IDX',
'ACCEPTORTYPE',
'LIGCOO',
'PROTCOO')],
'metal': [('RESNR',
'RESTYPE',
'RESCHAIN',
'RESNR_LIG',
'RESTYPE_LIG',
'RESCHAIN_LIG',
'METAL_IDX',
'METAL_TYPE',
'TARGET_IDX',
'TARGET_TYPE',
'COORDINATION',
'DIST',
'LOCATION',
'RMS',
'GEOMETRY',
'COMPLEXNUM',
'METALCOO',
'TARGETCOO')]}}
この辞書は2つのレベルからなります。最初のレベルは見つかった結合サイトです。一つのリガンドだけをドッキングしているので、一つだけ見つかることが予想されます。各結合サイトについて、8個のリストを含むもう一つの辞書があり、リストは各特定の相互作用のためのものです。各リストは最初の列に列名を持ち、つづいて(該当するものがあれば)データが格納されています。これを踏まえて、次のようにPandas.DataFrameを構築することができます。
# オリジナルのノートブックと異なる解析結果を利用しているため、結合サイトの名前も異なっています。 # 結果に合わせてオリジナルのコード ["UNL:Z:1"] を['A:A:1']に変更しました。 # df = pd.DataFrame.from_records(interactions_by_site["UNL:Z:1"]["hbond"][1:], # データは列名の後に格納されています。 # columns=interactions_by_site["UNL:Z:1"]["hbond"][0]) # 列名は常に最初の要素です。 df = pd.DataFrame.from_records(interactions_by_site["A:A:1"]["hbond"][1:], columns=interactions_by_site["A:A:1"]["hbond"][0]) df
| RESNR | RESTYPE | RESCHAIN | RESNR_LIG | RESTYPE_LIG | RESCHAIN_LIG | SIDECHAIN | DIST_H-A | DIST_D-A | DON_ANGLE | PROTISDON | DONORIDX | DONORTYPE | ACCEPTORIDX | ACCEPTORTYPE | LIGCOO | PROTCOO | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 0 | 45 | LYS | A | 1 | A | A | True | 2.64 | 3.42 | 133.60 | True | 431 | N3+ | 2953 | O3 | (3.232, 17.45, 38.708) | (4.782, 15.554, 36.326) |
| 1 | 150 | ASP | A | 1 | A | A | False | 3.07 | 3.43 | 102.37 | True | 1470 | Nam | 2955 | O3 | (1.743, 18.722, 37.254) | (0.523, 19.864, 34.258) |
| 2 | 45 | LYS | A | 1 | A | A | False | 2.66 | 3.10 | 106.87 | True | 422 | Nam | 2951 | O3 | (5.392, 21.015, 40.948) | (7.994, 19.469, 41.639) |
| 3 | 72 | LEU | A | 1 | A | A | False | 3.65 | 4.01 | 103.83 | True | 694 | Nam | 2945 | Npl | (3.257, 24.961, 34.128) | (2.841, 27.919, 36.804) |
| 4 | 61 | MET | A | 1 | A | A | False | 2.75 | 3.76 | 161.43 | False | 2945 | Npl | 595 | O2 | (3.257, 24.961, 34.128) | (2.892, 28.611, 33.325) |
| 5 | 149 | THR | A | 1 | A | A | True | 2.02 | 2.89 | 147.03 | False | 2955 | O3 | 1467 | O3 | (1.743, 18.722, 37.254) | (0.131, 21.066, 36.729) |
| 6 | 150 | ASP | A | 1 | A | A | True | 2.70 | 3.64 | 162.21 | False | 2953 | O3 | 1478 | O3 | (3.232, 17.45, 38.708) | (1.632, 16.073, 35.738) |
さて、これらの相互作用をNGLビューワーで表しましょう。相互作用ポイント(pandas.DataFrameのLIGCOOとPROTCOO)の間に円柱(cylinder)を描き、color_mapに表されているように色分けします(RGBタプルを使います)。
color_map = {
'hydrophobic': [0.90, 0.10, 0.29],
'hbond': [0.26, 0.83, 0.96],
'waterbridge': [1.00, 0.88, 0.10],
'saltbridge': [0.67, 1.00, 0.76],
'pistacking': [0.75, 0.94, 0.27],
'pication': [0.27, 0.60, 0.56],
'halogen': [0.94, 0.20, 0.90],
'metal': [0.90, 0.75, 1.00],
}
def show_docking_and_interactions(protein, ligands, vina_output):
# 複数のPDBQTリガンドファイルを別々のファイルに分割します
ligands_files = split_pdbqt(ligands)
# 親和性('Affinity')を取得します( DataFrameのこの列だけが必要です)
affinities = parse_output(vina_output)['Affinity (kcal/mol)']
# selection widgetの作成
# オプションは(text, value)のタプルのリストです。select上でクリックすると、
# `.observe(...)`に登録されている呼び出し可能なもの(callable)にvalueが渡されます。
selector = Select(options=[(f"#{i} {aff} kcal/mol", i-1) for (i, aff) in enumerate(affinities, 1)],
description="", rows=len(ligands_files), layout=Layout(width="auto"))
# GUI要素の配置
# selectionボックスを左、viewerをウィンドウの残りの部分を占めるようにします。
app = AppLayout(left_sidebar=selector, center=None, pane_widths=[1, 6, 1], height="600px")
# これがイベントハンドラーです。ユーザーがselectionボックスでクリックした時に動作を取り出します。
# viewerの変数を「見る」ことができるようにここで定義する必要があります。
def _on_selection_change(change):
# ユーザーが異なるエントリーでクリックした時だけ更新する
if change['name'] == 'value' and (change['new'] != change['old']):
if app.center is not None:
app.center.close()
# NGLビューワー
app.center = viewer = nv.NGLWidget(height="600px", default=True, gui=True)
prot_component = viewer.add_component(protein, ext="pdbqt") # add protein
time.sleep(1)
lig_component = viewer.add_component(ligands_files[change['new']], ext="pdbqt") # add selected ligand
time.sleep(1)
lig_component.center(duration=500)
# 相互作用の追加
interactions_by_site = analyze_interactions(f"data/docked_protein_ligand.{change['new']+1}.pdb")
interacting_residues = []
for site, interactions in interactions_by_site.items():
for interaction_type, interaction_list in interactions.items():
color = color_map[interaction_type]
if len(interaction_list) == 1:
continue
df_interactions = pd.DataFrame.from_records(interaction_list[1:], columns=interaction_list[0])
for _, interaction in df_interactions.iterrows():
name = interaction_type
viewer.shape.add_cylinder(interaction['LIGCOO'], interaction['PROTCOO'], color, [0.1], name)
interacting_residues.append(interaction['RESNR'])
# 相互作用する残基の表示
res_sele = " or ".join([f"({r} and not _H)" for r in interacting_residues])
res_sele_nc = " or ".join([f"({r} and ((_O) or (_N) or (_S)))" for r in interacting_residues])
prot_component.add_ball_and_stick(sele=res_sele, colorScheme="chainindex", aspectRatio=1.5)
prot_component.add_ball_and_stick(sele=res_sele_nc, colorScheme="element", aspectRatio=1.5)
# イベントハンドラーの登録
selector.observe(_on_selection_change)
# 最初の解に焦点をあてるために手動でイベントを起こす
_on_selection_change({'name': 'value', 'new': 1, 'old': None})
return app
show_docking_and_interactions はほとんどshow_docking関数とと同じですが、各選択イベントについてPLIP相互作用も計算します。この場合、イベントハンドラーはもう少しだけ関与しています。相互作用を示す円筒を手動で消すことができないので、各選択イベントについてNGLビューワーを作り直す必要があります。ですが、今回はJavaScriptは含まれていません。純粋なPythonだけです。ですが、ビューワーのwidgetを初めから作り直し、分子を読み込み、相互作用を計算するにはより時間が必要となり、体感としてはそれほどダイナミックなものにはなりません。
ドッキングと相互作用の表示
# app = show_docking_and_interactions("data/protein.mol2.pdbqt", "data/results.pdbqt", "data/vina.out") app = show_docking_and_interactions("data/protein.mol2.pdbqt", "data/local_result.pdbqt", "data/local_vina.out") app
訳注(2020/05)
以下は画像ですが、ノートブック上ではインタラクティブなビューワーが表示されます。
訳注ここまで
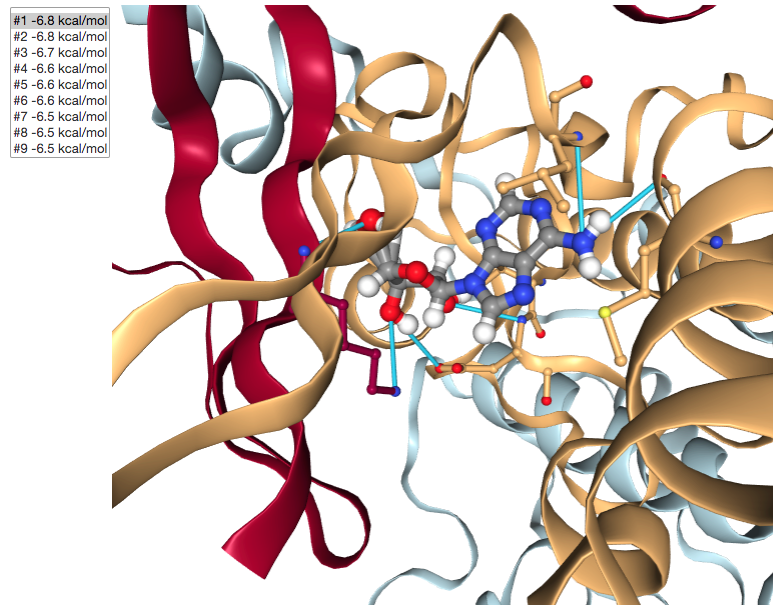
app.center.render_image(),
(Image(value=b'', width='99%'),)
# 静的なプレビュー
app.center._display_image()
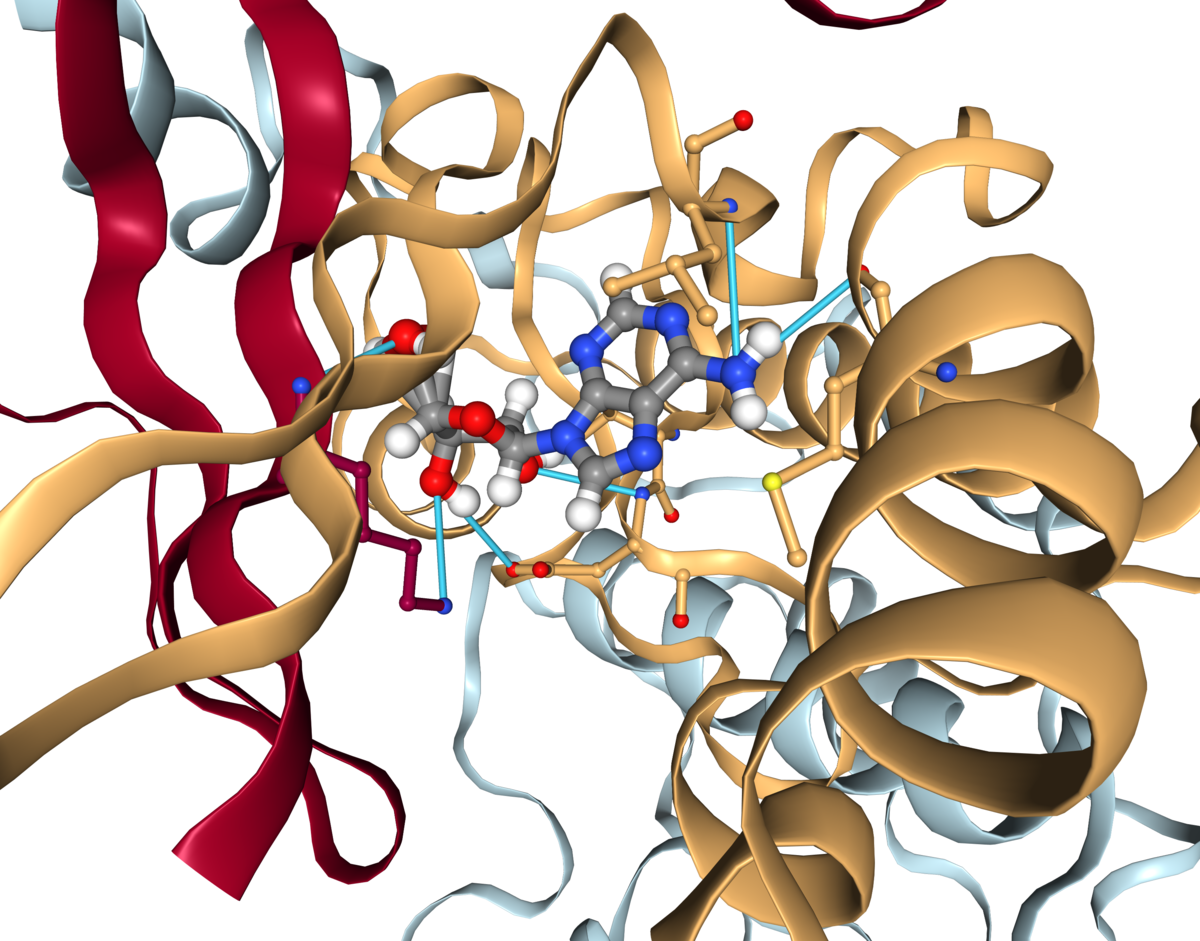
ディスカッション
このトークトリアルの最後のパートでは、Vinaによるドッキングの結果を可視化し解析する方法を学びました。他のパートと異なり、このNotebookはローカル環境で実行され、Webサーバーは呼びませんでした。しかしながら、とてもたくさんのWebテクノロジーを取り扱っており、主にNGLビューワーとipywidgetsレイアウトに基づいています。
はじめに、選択したタンパク質ーリガンドポーズとその周囲でインタラクティブにビューワーを更新するために(observe()で登録されているように)イベントハンドラーを使いました。次に、ビューワーで相互作用を描くためにPLIPの自動化された解析を使って可視化を拡張しました。
クイズ
- 観察したドッキング解析において最も重要だったのはどのような相互作用でしたか?文献との一致は見られましたか?またそれはなぜでしょうか?
- 疎水性相互作用と水素結合の間の主な違いは何でしょうか?またどの点で類似しているでしょうか?
- パートBで学んだ内容を応用してPLIPのWebサーバーを使う関数を描いてください。メインの
formエントリーとactionのエンドポイント、リクエストを投げるのに必要なinputタグの場所を見つけるためにHTMLソースコードをチェックする必要があります。
訳以上
追記
以上です。今回も色々と誤りがあると思います。ご指摘いただければ幸いです。
以下、今回のトークトリアルでよく分からなかった箇所とエラーについて調べた内容を記載します。
- 追記 1) AutoDockVinaの結果、RMSDのsymmetry correction に関して
RMSDについてsymmetry correctionを行ったか否かの2種類の記述があります。以下は、私の理解です。
リガンドの各原子を区別してラベルしている場合、分子に対称性があると、化学的には同じ構造でも原子のラベルとしては異なるという可能性があります(ex. ベンゼンの6つの炭素原子にラベルをつける場合)。ドッキングの結果の比較として、同じリガンドの異なる結合ポーズをRMSDで評価しようとする場合、各原子のラベルの対応を元に計算できます。ですが、対称性がある分子では、実際には重なりのよい(RMSDの小さい)ポーズでも、ラベルの付け方が複数通り可能なために原子同士が離れている(RMSDが大きくなる)と計算してしまいます。この問題を考慮するか否か、というのがsymmetry correctionを行うか否か、という違いだと思います。
- 追記2) PLIP実行時のエラーについて
PLIPでPDBを読み込む際に下記のエラーが出ました。
~/.pyenv/versions/anaconda3-5.3.1/envs/teachopencadd/lib/python3.6/site-packages/plip/modules/supplemental.py in read_pdb(pdbfname, as_string) in read_pdb(pdbfname, as_string)
396 if os.name != 'nt': # Resource module not available for Windows
397 maxsize = resource.getrlimit(resource.RLIMIT_STACK)[-1]
--> 398 resource.setrlimit(resource.RLIMIT_STACK, (min(2 ** 28, maxsize), maxsize))
399 sys.setrecursionlimit(10 ** 5) # increase Python recoursion limit
400 return readmol(pdbfname, as_string=as_string)
ValueError: current limit exceeds maximum limit
使用するシステムのリソースを設定する箇所でのエラーです。こちらはどうもPLIPの問題ではなく、Mac OSとPython 3.6の組み合わせで生じる問題のようです。Pythonにissue( 課題34602:python3 resource.setrlimit strange behaviour under macOS ) が出ていました。PLIPの該当箇所をコメントアウトしGitHubの修正履歴を参考に以下のように書き換えました。
def read_pdb(pdbfname, as_string=False):
"""Reads a given PDB file and returns a Pybel Molecule."""
pybel.ob.obErrorLog.StopLogging() # Suppress all OpenBabel warnings
"""エラー箇所
if os.name != 'nt': # Resource module not available for Windows
maxsize = resource.getrlimit(resource.RLIMIT_STACK)[-1]
resource.setrlimit(resource.RLIMIT_STACK, (min(2 ** 28, maxsize), maxsize))
"""
# 変更ここから
if sys.platform == 'darwin':
try:
import resource
except ImportError:
pass
else:
soft, hard = resource.getrlimit(resource.RLIMIT_STACK)
newsoft = min(hard, max(soft, 1024*2048))
resource.setrlimit(resource.RLIMIT_STACK, (newsoft, hard))
# 変更ここまで
sys.setrecursionlimit(10 ** 5) # increase Python recoursion limit
return readmol(pdbfname, as_string=as_string)
これでエラーが出なくなりました。
- 追記3) ビューワー上のリガンドに関して
ビューワーを画像にしてしまっているためわかりにくいですが、ドッキング結果のリガンドは結合におかしな点がみられます。ドッキング結果を出力したPDBファイルには原子の座標のみで、結合関係の情報が入っていないようでしたので、その辺りが影響しているのかもしれません。
終わりに
これでトークトリアル11含め全てのトークトリアル終了となります。長々とお付き合いいただきありがとうございました!
私は、CADDに関連する項目についてはぼんやりと用語を聞いたことがある程度だったので、各トピックごとに理論と実践の両方が扱われていて、まとめて勉強する良い機会となりました。一つの標的EGFRに絞って、色々なトピックでアプローチすることで、様々な視点から情報を捉えるというのは面白いやり方ですね。恐らく内容としては初歩の初歩で、CADDのほんの入り口に過ぎないのだと思いますが、全体の流れと道しるべ的な専門用語を拾えたので、何が話題になっているか程度には理解できるようになっているといいなと思います。ではでは!
オフラインでドッキングする話(T11パートBの備忘録)
T11パートBはオンラインWebサービスを用いたターゲットタンパク質に対するドッキングでした。残念ながら用いられていたSwissDock上のドッキング、OPAL共に現在では利用ができなくなっていました。そこで、趣旨とは異なってしまいますがオフラインでのドッキングを行って見たいと思います。
目標はこんな感じです。
| 用いるデータ | 概要 | 詳細 |
|---|---|---|
| 参照構造 | EGFR-リガンド共結晶構造 | PDB ID:3W33、但しKLIFSに登録されているデータ |
| ドッキングする化合物 | リガンドと構造類似の化合物群 | リガンド(ADP)をテンプレートとしてPubChemから取得 |
| やること | T11パートB | この記事 |
|---|---|---|
| 結合サイトの位置 | DoGSiteScorer | 同左 |
| タンパク質・リガンドの準備 | AutoDockTools | ODDT |
| ドッキング | OPAL上のAutoDockVina | PCにインストールしたAutoDockVina |
では、素人による適当解説やっていきますよ!
DogSiteScorerで良い感じのポケットを探す
Webサイトで遊ぶ
まずはドッキングを行うポケットの位置を決めます。ここはT11パートBでも実行できていたのでやり方を踏襲します。・・・が、やっぱりコードだけをみてもよくわからないのでWebサイトではどのような結果になるか見に行きたいと思います。
DoGSiteはProteinsPlusというサイトで提供されているソフトです*1。ProteinsPlusはハンブルク大学Computational Molecular Design Group(AMD、Matthias Rarey教授)で開発されたツールを利用できるように公開されているWebサーバーで、ポケットを探索するDoGSite以外にもタンパク質の構造を取り扱うためのツールが多数組み込まれています*2。
トップページ格好良い・・・

PDB IDでの検索も可能なようですが、今回はT11に従いKLIFSから取得したデータを使うため、Upload Proten(PDB format)にPDBファイルを投げ込みGo!。すぐに結果がでてこんなページが出てきます。

素敵・・・。
右側のパネルから利用できるサービスを選べるようになっています。DoGSiteScorerに行きましょう。色々とオプションがありますが今回はデフォルトのまま計算して見ます。

1−2分で結果が出ます。今回は10個のポケットが見つかりました。

ポケットのサイズに関する情報(体積、表面積)に加えてスコア(Drug Score、Simple Score)も出ています。スコアが高ければ医薬品の標的とするポケットとして良いのだと思います(論文を読んでいません。すみません)。確かにトップのポケット(P_0)は共結晶構造中のリガンドの位置と一致しています。
ポケットの情報はテーブルをスクロールした下部にあるDownloadから取得できます(zipファイル)。ありがたく頂戴して中身を見て見ましょう。
ダウンロードした情報の確認
ダウンロードしたディレクトリには以下のファイルが含まれています。
- 「parameter.txt」・・・計算の設定
- 「ジョブ名_desc.txtファイル」・・・ポケットの情報(Webサイト上に表示されているテーブル)。
- 「PocXlsDescriptors.txt」・・・ポケット情報の省略語の説明
- 「pockets」・・・各ポケットの座標等の情報のファイルを含むディレクトリ(.ccp4.gz、ポケットごとに別のファイル(今回は10個))
- 「residues」・・・各ポケットの残基等の情報のファイルを含むディレクトリ(.pdb、ポケットごとに別のファイル(今回は10個))
Webサイト上で画像表示されていたもののベースになる情報がそっくり取得できるようです。
ポケット情報のテキストファイル(2)はタブ区切りのテーブルのようでした。Pandasの出番だすな!
import pandas as pd df = pd.read_table('./DogSiteResults/Job_desc.txt') # ファイルの名前は長いので変えています print(df.shape) df.head() # (10, 50)
| name | lig_cov | poc_cov | lig_name | volume | enclosure | surface | depth | surf/vol | lid/hull | ... | MET | PHE | PRO | SER | THR | TRP | TYR | VAL | simpleScore | drugScore | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 0 | P_0 | 0.0 | 0.0 | NaN | 1268.67 | 0.13 | 1422.18 | 26.50 | 1.121001 | - | ... | 2 | 3 | 2 | 1 | 2 | 1 | 0 | 4 | 0.64 | 0.817267 |
| 1 | P_1 | 0.0 | 0.0 | NaN | 371.84 | 0.17 | 681.06 | 13.45 | 1.831594 | - | ... | 2 | 1 | 3 | 0 | 1 | 0 | 1 | 2 | 0.23 | 0.642846 |
| 2 | P_2 | 0.0 | 0.0 | NaN | 338.56 | 0.07 | 524.19 | 13.65 | 1.548293 | - | ... | 1 | 1 | 1 | 3 | 1 | 2 | 1 | 2 | 0.18 | 0.625701 |
| 3 | P_3 | 0.0 | 0.0 | NaN | 234.94 | 0.10 | 473.53 | 10.68 | 2.015536 | - | ... | 0 | 2 | 2 | 1 | 0 | 0 | 1 | 1 | 0.11 | 0.421320 |
| 4 | P_4 | 0.0 | 0.0 | NaN | 207.36 | 0.12 | 353.27 | 11.60 | 1.703655 | - | ... | 1 | 1 | 0 | 0 | 0 | 0 | 1 | 2 | 0.04 | 0.444417 |
5 rows × 50 columns
ポケットにどんな残基がいくつ含まれているかも含め、たくさんの情報が入っているようです。
具体的なポケットをPyMolで確認してみます。

ccp4ファイルはObjectMapCCp4として認識され、立方体が読み込まれました。ポケットを単位胞のように認識しているのでしょうか?・・・不勉強なのでわかりません。すみません。
pdbファイルのHEADERにはポケットの中心座標の情報も記載されています。例えば今回のポケットP_0では・・・
- HEADER Geometric pocket center at 0.63 18.01 37.64 with max radius 22.90
でした。この情報があればドッキング計算でポケットの位置を指定できそうです。
APIで実行
同じ作業をAPIで行ってみます。といってもT11パートBのコードを切りはりするだけですが・・・。
PDBファイルをアップロードするの複雑なのでそのままPDB IDを使ってやってみます(PDB ID: 3W33)。ProteinPlusはREST APIのドキュメンテーションもあり、DogSiteScorerはこちらです。POSTでジョブを作成し、GETで結果を取得します(json形式)。
import requests r = requests.post("https://proteins.plus/api/dogsite_rest", json={ "dogsite": { "pdbCode": "3W33", "analysisDetail": "1", "bindingSitePredictionGranularity": "1", "ligand": "", "chain": "" } }, headers= {'Content-type': 'application/json', 'Accept': 'application/json'} ) r.raise_for_status() # エラーに備える print(r.status_code) # 200
HTTP ステータス:200なのでうまくいっているようです。ドキュメンテーションには各ステータスコードの場合に得られる結果も記載されています。

実際にtext属性を確認してみると同じ結果となっています。
print(r.text) # {"status_code":200,"location":"https://proteins.plus/api/dogsite_rest/ジョブのID","message":"Job already exists"}
ジョブがうまく投げられたので計算結果を取得します。locationがジョブのIDなのでこちらを使います。
job_location = r.json()['location'] result = requests.get(job_location) print(result.status_code) # 200
こちらもうまくいったようです。結果は以下となっているそうです。

このうちresiduesの一番最初(インデックス0)が最もスコアの良いポケットだそうです。Webで結果をダウンロードした時と同じですね。
residuesはPDBファイル形式となっています。今欲しい情報は結合ポケットの中心の座標と半径で、こちらはHEADERのGeometric pocket center ~という行に含まれていました。こちらを取得します。
# PDBの取り出し best_pocket_pdb = requests.get(result.json()['residues'][0]).text # PDBから目的の情報を含む行を見つける # スペースで区切ったインデックス5,6,7([5:8])が中心の座標(x,y,z) # 最後(インデックス[-1])が半径(radius) for line in best_pocket_pdb.splitlines(): line = line.strip() if line.startswith('HEADER') and 'Geometric pocket center at' in line: fields = line.split() center = [float(x) for x in fields[5:8]] radius = float(fields[-1]) print("center: ", center) print("radius: ", radius) # center: [20.37, 29.52, 10.77] # radius: 19.95
先ほどWebインターフェースで実行した場合と座標、半径が異なっています。KLIFSから取得したファイルとPDB IDを指定して行った違いのためでしょうか?同じ構造でも情報の取得減に応じて座標が異なる可能性があるというのは要注意そうです。
以降ではT11でKLIFSから取得したファイルを使うので、先にWebで得たポケットの情報を使うことにしたいと思います。
ドッキングの実行
ドッキングソフトの取得
結合ポケットの情報がわかったので実際のドッキングに進みたいと思います。ドッキングはAutoDockVinaで行いますが、AutoDockVinaを使うためにはタンパク質とリガンドをそれぞれ専用の形式(PDBQT)で準備する必要があります*3。T11パートAではAutoDockToolsを修正したものが使われていましたが、今回は別のツールODDT(Open Drug Dicovery Toolkit)を利用したいと思います。
ODDTは様々なオープンソースのツールの入出力方法を統合してもっと便利に使えるようにしましょう、という素敵なツールです。ツール多すぎて無理・・・って感じの私にはとてもありがたいです。詳細は以下をご参照ください。
- Wójcikowski, M., Zielenkiewicz, P., and Siedlecki, P. Open Drug Discovery Toolkit (ODDT): a new open-source player in the drug discovery field Journal of Cheminformatics 2015(7)26
- 文献URL
- ドキュメント
- GitHub
インストールはpipかcondaでできます。
# pipでインストールする場合 pip install oddt # condaでインストールする場合 conda install -c oddt oddt
AutoDockVina自体はこちらのページのDownloadから取得できます。計算化学.COMさんのこちらのページ にインストールと使用方法が詳しく書かれいるのでご参照ください*4。
タンパク質の準備
ではまずタンパク質だけを含むファイル(mol2)をODDTで読み込んでみます。ODDTではタンパク質の変数名をproteinとするようにとドキュメントに書かれていたので合わせておきます。*5
import oddt from oddt import toolkit protein = next(oddt.toolkit.readfile('mol2', "data/protein.mol2")) protein.protein = True type(protein) # oddt.toolkits.ob.Molecule
タンパク質の情報を読み込みオブジェクトの生成ができたので、こちらをpdbqtファイルに変換します。ODDTのdockingモジュールからAutodockVina.write_vina_pdbqt(molオブジェクト, 出力先のディレクトリ)を実行します。タンパク質の場合は引数flexible=Falseとしておく必要があります。
from oddt import docking oddt.docking.AutodockVina.write_vina_pdbqt(protein, "data", flexible=False) # 'data/3w33_A.pdbqt'
タンパク質の情報(PDB ID:3w33のChain A)を反映したファイル名で出力されました。変数proteinの中身をこちらに切り替えておきます。
protein = next(oddt.toolkit.readfile('pdbqt', "data/3w33_A.pdbqt")) protein.protein = True print(type(protein)) # oddt.toolkits.ob.Molecule
実際にはODDTはAutoDockVinaのドッキングを行う際にpdbqtへの変換も同時に行うので予め変換しておく必要はありませんが、ここでは使い方の練習ということでご容赦ください。
リガンドの準備
リガンドはT11パートAで取得したPubChem化合物を使います。SMILESで保存しているので、RDKitで3次元構造におこします。AllChem.EmbedMolecule()で3次元にしますが、性能が良いというETKDGv2()を指定して構造を生成します。*6
# SMILESファイルの読み込み SMILES_FILE = "data/similar_smiles.txt" with open(SMILES_FILE) as f: smiles = [line.strip() for line in f] # 必要なものをインポート from rdkit import Chem from rdkit.Chem import AllChem # 3次元構造を生成してリストに格納 mols = [] for s in smiles: mh = Chem.AddHs(Chem.MolFromSmiles(s)) AllChem.EmbedMolecule(mh, AllChem.ETKDGv2()) mols.append(mh) # 描画で確認 from rdkit.Chem import Draw Draw.MolsToGridImage(mols, molsPerRow=5)

立体感でてますね!SDFに書き出しておきます。
writer = Chem.SDWriter("data/similar_ligands.sdf") for mol in mols: writer.write(mol) writer.close()
ODDTでリガンドを読み込みPDBQTファイルに変換します。
# SDFの読み込み ligands = list(oddt.toolkit.readfile('sdf', 'data/similar_ligands.sdf')) # PDBQTに一つずつ変換 ligand_id = 0 for ligand in ligands: file_name = "ligand_" + str(ligand_id) oddt.docking.AutodockVina.write_vina_pdbqt(ligand, "data", name_id=file_name) ligand_id += 1
dataディレクトリの中に10個のリガンドpdbqtファイルができました。次のドッキングでは一番最初のリガンドを使って試そうと思うので読み込んでおきます。
ligand = next(oddt.toolkit.readfile('pdbqt', 'data/ligand_0.pdbqt'))
ドッキングの実行
結合ポケットの情報、タンパク質、リガンド構造の準備が完了したのでドッキングを実行します。ドッキングを行う空間はDoGSiteScorerのWeb版で実行しPyMolでも確認した以下のポケットです。
- 中心座標・・・ [0.63, 18.01, 37.64]
- サイズ・・・ 22.90 → 23にします。
from oddt.docking import AutodockVina # パラメーターの設定 DockingParams = AutodockVina.autodock_vina(protein=protein, size = (23, 23, 23), center = [0.63, 18.01, 37.64], exhaustiveness = 8, num_modes = 9, energy_range = 3, n_cpu = 3, executable='../../../../autodock_vina_1_1_2_mac/bin/vina')
引数executableはインストールしたAutodockVinaのlocalのpathを指定します。ここでは相対パスで指定しています。パラメーターの設定が完了したのでリガンドを渡してドッキングを実行します。
DockingResult = DockingParams.dock(ligand) len(DockingResult) # 9
9個の結果が生成されました。それぞれODDTのMolオブジェクトで、スコアはdataメソッドに格納されています。to_dict()で辞書に変換することができるので、辞書を経由してDataFrameに変換してみます。
data_list = [mol.data.to_dict() for mol in DockingResult] data_df = pd.DataFrame(data_list) data_df = data_df[['vina_affinity', 'vina_rmsd_lb', 'vina_rmsd_ub','vina_rmsd_input', 'vina_rmsd_input_min']] data_df
| vina_affinity | vina_rmsd_lb | vina_rmsd_ub | vina_rmsd_input | vina_rmsd_input_min | |
|---|---|---|---|---|---|
| 0 | -6.8 | 0.000 | 0.000 | 43.380367 | 43.380367 |
| 1 | -6.8 | 2.927 | 5.787 | 43.486763 | 43.486763 |
| 2 | -6.6 | 11.173 | 12.829 | 37.1132 | 37.1132 |
| 3 | -6.6 | 2.950 | 4.507 | 43.876957 | 43.876957 |
| 4 | -6.5 | 3.989 | 6.204 | 43.624863 | 43.624863 |
| 5 | -6.5 | 3.524 | 5.691 | 44.41204 | 44.41204 |
| 6 | -6.4 | 4.251 | 6.471 | 45.132126 | 45.132126 |
| 7 | -6.4 | 7.300 | 8.536 | 43.149113 | 43.149113 |
| 8 | -6.3 | 8.683 | 9.959 | 42.101715 | 42.101715 |
ここではvinaの出力に関係する列のみを取り出しています。vina_affinityとなっている列が親和性で最も良いもので-6.8となっています。DataFrameをcsvファイルに書き出しておきます。またドッキング結果のリガンドの情報もSDFに書き出しておきます。
# csv作成 data_df.to_csv("data/AutoDockVina_DockingResult.csv") # sdf作成 ODDTsdfWriter = oddt.toolkit.Outputfile('sdf', 'data/ODDT_DockingResult_ligand_0.sdf') for mol in DockingResult: ODDTsdfWriter.write(mol) ODDTsdfWriter.close()
引数のsdfの部分を変えればmol2等別の形式で出力することも可能です。
ODDTの出力におけるエラー
ODDTは非常に扱いやすいのですが、問題点としてAutoDockVinaでドッキング後のリガンドを出力すると原子、結合の帰属がおかしなことになっているということがあります。まず出力前の構造をJupyterNotebookで表示させたのちに、PDBQTで出力したリガンドをPyMolに表示させてみます。
DockingResult[0]

このように、骨格全体の座標としては保たれているものの、原子のタイプ、結合関係が壊れてしまっています。残念ながら出力のファイルタイプを変えてもこのエラーは修正されませんでした。PyMolで眺めて遊びたかったのに・・・
コマンドラインでAutoDockVinaを使う
ODDTで惜しいところまで行きましたが、私の能力では望みの構造の取得にまで到達できませんでした。残念・・・と思っていたらAutoDockVinaの使用方法のページ にコマンドラインで実行する方法が書いてありました。
コマンドはすごい苦手、というかさっぱりわからないのですが、ここまできたらやるしかない。
用意するものは3つ
- タンパク質のPDBQTファイル(ODDTで作成済み。flexible = Falseで作成していないと動かない)
- リガンドのPDBQTファイル(ODDTで作成済み)
- パラメーターを書いたconfigurationファイル(config.txt)
です。
config.txtの中身は以下のような感じ・・・
receptor = 3w33_A.pdbqt center_x = 0.63 center_y = 18.01 center_z = 37.64 size_x = 23 size_y = 23 size_z = 23 num_modes = 9
receptorにタンパク質のpdbqtファイル名、center_x,y,zはグリッドの中心座標、size_x,y,zはグリッドのサイズです。num_modesは最大いくつの結合ポーズを出すかを指定します。
ファイルの準備ができたら
- ターミナルでディレクトリに移動
- AutoDockVinaのインストール箇所へのPATHを通す。(
export PATH=$PATH:~~~~~) - コマンド実行!
vina --config conf.txt --ligand ligand_0.pdbqt --out out.pdbqt --log log.txt

と、出て結合ポーズを含むout.pdbqtとスコアなどの情報を含むlog.txtが出来ました!!早い!
PyMolで結果を眺めてみます。折角なので元の共結晶のリガンドを含むファイルと重ねてみます。

エモい!よくわからないけど!
スコアが良いものから悪いものへと順番に流れています。スコアの良いものは画面の右、悪いものは左側という印象でした。詳しい解析はT11パートCで扱われるはず・・・。
ちなみにAutoDockVinaは複数のリガンドをドッキングするためのシェルクリプトも用意してくださっています。こちらのページ からダウンロード出来ます。
chmodで実行権限を設定して実行すればまとめて処理できます。すごい便利ですね。食わず嫌いせずにシェルスクリプト勉強しないと・・・。
まとめ
以上、今回はドッキングを行ってみました。オンラインサービスは自分で環境を準備しなくてもよく、パワーのある計算機を使えるので素敵ですが、いつ止まるかわからないという不安があります。お手元のPCでも試せると便利かもしれません。心置きなく適当なことできますしね!
ODDTを導入したものの結果をうまく出力できなかったり、と相変わらずグダグダになってしまいましたが、T11パートBのオンラインではできなかったことをボンヤリとオフラインで実行できたので良し、ということにしていただければ幸いです。
色々と間違っていることがあると思うのでご指摘いただければ幸いです。
*1: DogSite文献 J. Chem. Inf. Model. (2010), 50, 11, 2041-2052
Bioinformatics (2012), 28, 15, 2074–2075
*3:J. Comput. Chem. 2010(31)455-61
*4:ドッキングシミュレーションのやり方【AutoDock vina】
*5: タンパク質ファイルの読み込みが遅く、セルが [*] のまま進まないときはJupyter Notebookの拡張機能Variable Inspectorが原因の可能性があります。こちらの参考記事 【Jupyter Notebook】Jupyter notebookの動作が重い およびIssue と同じエラーかはわかりませんが、とりあえずnb_extensionsから外すと次のセルが実行できました。
TeachOpenCADD トピック11(パートB)〜オンラインAPI/サービスを使った構造に基づくCADD〜
こちらはTeachOpenCADDの試訳です。TeachOpenCADD GitHubレポジトリのトークトリアル11をもととしておりCC BY 4.0のライセンスに従っています。
トークトリアル11は論文発表時のTeachOpenCADDには含まれておらず新しく追加されたものです。
4つのノートブックに分かれており、ここではその3つ目を訳出します。
パートBで取り上げられているWebサーバー、サービスのうちドッキングに関するSwissDock、OPALはともに2020年5月現在サービスが提供されていないようで、実行できなくなっています。使えるものではなくなってしまっていますが、基本的な考え方や手法を知る上では役にたつかもしれません。
~~ 追記 ~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~
以下のページに翻訳したノートブックを保存しています。よろしければご利用ください。
~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~
以下、訳。
トークトリアル 11 (パート B)
オンラインAPI/サーバーを使った構造に基づくCADD
Developed at AG Volkamer, Charité
Dr. Jaime Rodríguez-Guerra, Dominique Sydow
このトークとリアルの目的
これは「オンラインWebサービス」についてのトークトリアルのパートBです。:
- 11a. キナーゼ阻害剤の候補をKLIFとPubChemで検索
- 11b. 11aで取得した候補化合物をターゲットタンパク質に対してドッキング
- 11c. 結果を評価し既知のデータと比較
入力構造を取得したあとで、ドッキングソフトウェアを使って良いタンパク質-リガンドポーズを見つけます。
学習の目標
理論
- ドッキングの基本
- 利用可能なソフトウェア
- 知られている限界
実践
- 構造の準備
- 結合サイトの推定
- ドッキング計算の実行
- 結果の保存
レファレンス
- Structure‐Based Virtual Screeningの章 Ch. 6.8 in "Applied Chemoinformatics: Achievements and Future Opportunities" (2018)
- ドッキングソフトウェアをベンチマークテストする方法 (J. Med. Chem. (2006), 49, 23, 6789–6801)
- ドッキングソフトウェアについてのレビュー (Biophysical Reviews (2017), 9, 2, 91–102)
- DoGSiteScorer:結合サイトを同定するプログラム。
- SwissDock:ドッキングプログラム
- OPAL Webサービス:必要に応じてリモートでソフトウェアを実行可能
- AutoDock Vina:ドッキングプログラム (J Comput Chem. (2010), 31, 455–461)
理論
ドッキング
Pagadala, Syed & Tuszynski, Biophysical Reviews 2017, 9, 2, 91–102 から引用して改変:
ドッキングは標的タンパク質の結合サイト内における低分子化合物の振る舞いを探索する手法です。適用されたソフトウェアは探索アルゴリズムを実行し、最小エネルギーに収束するまでリガンドの配座を再帰的に評価します。最後に、候補となるポーズをランク付けするために、親和性スコア関数ΔG (合計の単位はkcal/mol)を静電エネルギーとファンデルワールスエネルギーの合計として用います。生物学的な系におけるこれらの特定の相互作用のドライビングフォースは、結合サイトの表面とリガンドあるいは基質の形状と静電的な状態の相補性への志向です。
これらのスコア関数は計算が速くなるように合わされているので、他の分子モデリング手法と比較してしばしば精確性に劣ります。通常、これらは次に基づいています。
- 分子力学法原理
- 知識に基づくポテンシャル
- 形状と幾何学的な相補性
探索空間の次元を減らすために、いくつかの近似がよく適用されます。
- タンパク質の構造はほとんどが剛直なものと考えられますが、探索空間に近接するいくつかの側鎖は限られた配座セットの探索が許可されていることもあります(ロータマー)。
- リガンドはバーチャルスクリーニングの研究では剛直なものとして考えられますが、より詳細な計算においては結合の二面角を自由に探索することがよく許可されます。二つのオプションの折衷案は、とりうる配座のセットを予め定義しておくことです。
既存ソフトウェアの例
- 商用
- GOLD
- Schrödinger
- FlexX
- 無料(あるいはアカデミックフリー)
- AutoDock
- AutoDock Vina
- DOCK
- OpenEye
知られている限界
次の近似は計算においてアーティファクトを取り込む原因となりうります。
- タンパク質のほとんどを剛直したものとして扱うので、タンパク質-リガンド結合の動的、適応的な性質はほとんど探索されません。このため偽陽性につながります。リガンドが結合ポケットで適切なポーズを見つけたとしても、タンパク質が近接するエネルギー安定な配座を探索できないと、このポーズは保証されません。言い換えれば、リガンドが結合ポケットにとどまるかどうか確認するために短時間の分子動力学(MD)トラジェクトリを計算することが常に推奨されています。
- スコア関数は解く計算コストが低くなければなりません。良いポーズと悪いポーズを区別するのに十分な精度がある一方で、最も良い複数のポーズを並べ替えるには問題があることがあります。例えば、最も人気のあるドッキングプログラムは、実験で明らかとされているポーズを計算で見いだすことができる一方で、このポーズが提案されたポーズのセットの中で最も良いものであることは稀です。
- 計算コストを減らすために、ドッキング手順はタンパク質の一部分(通常、結合ポケットとして知られている場所の周辺)でのみ実行されます。CADDパイプラインにおいて、正しい結合ポケットを選択することはもう一つの課題となります。
- 計算の精確性を高めるために、適宜構造を削減する必要があります。アミノ酸とリガンドのプロトン化状態は正しい状態を得るにはこつがいることがあり、特に互変異性体(の可能性がある)の場合難しくなります。これは誤った結果を得る、もう一つの原因となります。
これらの限界にも関わらず、依然としてドッキング計算は全ての医薬品研究室で人気のある手法で、他の種類の分子シミュレーションとともに使われています。このトークトリアルのパートでは次の方法を学びます。
- パートAで取得したタンパク質とリガンドの準備(ローカルで実行)
- もっとも可能性が高い結合ポケットの推定(オンラインで実行)
- ドッキング計算の実行(オンラインで実行)
構造の準備
OPALWebサービスにあるVinaを使います。しかし、あらかじめ構造を準備することが必要となります。オススメのアプローチはAutoDockToolsにある準備のためのスクリプトを使うことです。これらのツールはツール自体でディストリビューションされており、Python2としか互換性がありませんが、私たちはタンパク質とリガンドの準備に必要な部分を含むPython 3で利用可能なフォークを準備しました。このトークトリアルで必要なことを行うには十分のはずです。
訳注(2020/05)
AutoDockToolsのライセンスは、非商用の科学研究で使用する場合は無料ですが、商用で使用するには正式な登録手続きが必要とのことです。正確な情報はソフトウェアのライセンスを確認してください。
訳注ここまで
結合ポケット予測
ドッキング計算は、合理的に狭い探索空間で実施すると最もパフォーマンスがよくなり、この空間は通常、一つの結合ポケットを含みます。最も良い結合ポケットを計算に基づいて予測するために、Protein.plusで無料でオンラインで利用できるDoGSiteScorerを使うことができます。
Proteins.plus DoGSiteScorer
- 役割: いくつかのCADDツールのためのインタラクティブなWenインターフェース
- Webサイト: http://proteins.plus
- API: RESTに基づくものがあります。
requestsをそのまま適用するだけで十分です。 - ドキュメンテーション: https://proteins.plus/help/index
- 文献:
自動化されたタンパク質活性部位の予測は、大規模なタンパク質機能予測と分類、そしてドラッガビリティ(druggability、医薬品の標的可能性)を推測する上で必須となります。ここで我々は、画像処理に端を発するガウシアン差分(Difference of Gaussian, DOG)の方法に基づきタンパク質の結合サイトを予測する、新規な構造に基づく手法、DoGSiteを提案します。既存の手法とは対照的に、DogSiteは予測したポケットをサブポケットへと分割し、活性部位のトポロジーの洗練された表現を明らかにします。DoGSiteはPDBBindとscPDBデータセットの92%以上で精確に結合ポケットを予測し、現在利用可能な手法のうち最もパフォーマンスの良いものと並ぶ性能があります。PDBBindデータセットの63%で、見つかったポケットはより小さなサブポケットへと細分化することができました。予測の87%で、共結晶化されたリガンドは正確に一つのサブポケットに含まれていました。さらに、リガンドとポケットの組み合わせの網羅する範囲を考慮に入れることで、より精確な予測性能の評価を導入しました。90%の事例で、DoGSiteは少なくともリガンドの半分を含むポケットを予測しました。70%の事例で、さらに各ポケット自体の1/4以上が共結晶化されたリガンドで覆われていました。サブポケットを考慮することで、適用範囲が広がり、後者の性能評価において成功確率83%となりました。
ドッキング
オンラインで無料で利用できるWebサービスが2、3あります。SwissDockとOPAL Webサービス(AutoDock Vinaを含みます)です。
SwissDock
- 役割: ドッキング計算を実行します
- Websサイト: http://www.swissdock.ch
- API: SOAPに基づくものがあります。正式なクライアントはありません。
suds-communityを使ってください。 - ドキュメンテーション: http://www.swissdock.ch/pages/soap_access
- 文献:
SwissDockはターゲットタンパク質と低分子化合物の間で生じうる分子間相互作用を予測するWebサービスです。 SwissDockはドッキングソフトウェアEADock DSSに基づいていますが、そのアルゴリズムは次のステップからなります。
1. ボックスの中(ローカルドッキング)あるいは全ての標的のくぼみの近傍(ブラインドドッキング)で、結合モードを数多く生成します。 2. 同時に、グリッド上でCHARMMエネルギーが予測されます。 3. 最も好ましいエネルギーの結合モードがFACTSで評価され、クラスター化されます。 4. 最も好ましいクラスターをオンラインで可視化し、自分のコンピュータにダウンロードすることができます。
OPAL Webサービス
- 役割: サービスとしてのCADD (CADD as a service)
- Webサイト: http://nbcr-222.ucsd.edu/opal2/dashboard
- API: SOAPに基づくものがあります。正式なクライアントはありません。
suds-communityを使ってください。 - ドキュメンテーション: http://nbcr-222.ucsd.edu/opal2/dashboard?command=docs (現在オフラインとなっています)
- 文献:
医学生物的なアプリケーションはますます複雑となってきており、しばしば多数のプロセッサとメモリを有する大規模な高性能計算資源を必要とします。アプリケーションのデプロイの複雑さと、クラスターコンピューティング、グリッドそしてクラウドコンピューティングにおける進歩は、医学生物学研究をサポートする新しい方式を必要としています。サービスとしての科学ソフトウェア(Scientific Software as a Service、SaaS)は、簡単な標準化されたWebインターフェースを通して、医学生物学的なアプリケーションに、大規模に実行可能で透明性のあるアクセスを可能とします。この目的のため、私たちは2007年8月にMEMEと名付けられたバイオインフォマティクスのアプリケーションをサポートするための運用Webサーバーを構築しました( http://ws.nbcr.net )。以来、このサーバーは成長を続け、AutoDockとAutoDock Vinaによるドッキング分析、PDB2PQRとAPBSを使った静電計算、そしてSMAPを使ったオフターゲット分析を含むようになりました。サーバー上のアプリケーションは全てOPALで動いています。OPALは、単純なXML環境設定ファイルを書くことで、科学的なコードに一切変更を加えることなく、科学アプリケーションをWebサービスとして簡単に使えるようにするツールキットです。OPALにより、我々の全てのアプリケーションにWebフォームに基づくアクセスとプログラムによるアクセスのどちらでもアクセスすることができるようになります。OPALツールキットは現在、国立生物医学計算資源(National Biomedical Computation Resource、NBCR)と、系列下の共同研究、サービスプロジェクトによる多数の人気のあるアプリケーションへのSOAPに基づくWebサービスアクセスをサポートしています。さらに、OPALはプログラムによるアクセスが可能なので、VisionやKepler、Nimrod/KそしてVisTrailsといった様々なワークフローツールを通して我々のアプリケーションにアクセスすることができます。2007年8月中旬から2009年の終わりにかけて、239,814個のジョブの実行に成功しました。2008年から2009年の間に、1日あたりの実行に成功したジョブは205から411へと2倍以上に増加しました。OPALで実現されているサービスモデルは様々なアプリケーションにとって有用です。Webサービスインターフェースによる他のアプリケーションとの相互運用を提供し、そしてアプリケーション開発者が科学的ツールとワークフローの開発に集中することができるようにします。Webサーバーは次で利用可能です( http://ws.nbcr.net )。
実践
パートAからファイルを取得
まず最初に、パート Aで取得したファイルを指定する変数をいくつか定義します。
PROTEIN: 標的タンパク質の構造を含む.mol2ファイルへのパス。リガンドやイオンは含まないCOMPLEX: 標的タンパク質の構造と元々のリガンドを結合サイトに含む.pdbファイルへのパスSMILES_FILE: PubChemで見つかった全ての類似化合物を表すSMILESを含む.txtファイルへのパスsmiles:SMILES_FILEに含まれるSMILES文字列のリスト
PROTEIN = "data/protein.mol2" COMPLEX = "data/complex.pdb" SMILES_FILE = "data/similar_smiles.txt" with open(SMILES_FILE) as f: smiles = [line.strip() for line in f]
SwissDockを利用する
SwissDockはSOAPインターフェースを使っているので、sudsをインストールする必要があります。
注: SwissDockのサーバーは最近機能していません。代わりに下のOPALヘ進んでください! !pip install suds-community
from suds.client import Client import zlib import string import requests def swissdock_client(): # 現在サーバーが落ちているようです。。。 # http://swissdock.vital-it.ch/soap/ は503利用不能を返します。 # 誤ったドメインを指定しているからでしょうか。。。修正しましょうか? SWISSDOCK_WSDL_URL = "http://www.swissdock.ch/soap/wsdl" r = requests.get("http://www.swissdock.ch/soap/wsdl") r.raise_for_status() WSDL = r.text.replace("http://swissdock.vital-it.ch/soap/", "http://www.swissdock.ch/soap/") with open("data/swissdock.wsdl", "w") as f: f.write(WSDL) HERE = _dh[0] return Client(f"file://{HERE}/data/swissdock.wsdl") def prepare_protein(client, protein): """ 与えられたPDBファイル(中身は文字列)に対してPSFとCRDを返す。 """ encoded_protein = zlib.compress(protein.encode('utf-8')) job_id = client.service.prepareTarget(target=encoded_protein) while True: result = client.service.isTargetPrepared(jobID=job_id) if result is None: raise ValueError("No such a job present") if result in (False, "false", 0): time.sleep(5) else: # 準備できました! break protein_files = client.service.getPreparedTarget(job_id) if protein_files is None or len(protein_files) != 2: raise ValueError("Could not prepare protein!") return protein_files def prepare_ligand(client, ligand): """ 与えられたMOL2ファイル(中身は文字列)に対してPDB、RTFとPARを返す。 リガンドはあらかじめプロトン化されていなければなりません! """ encoded_ligand = zlib.compress(ligand.encode('utf-8')) job_id = client.service.prepareLigand(ligand=encoded_ligand) while True: result = client.service.isLigandPrepared(jobID=job_id) if result is None: raise ValueError("No such a job present") if result in (False, "false", 0): time.sleep(5) else: # 準備できました! break ligand_files = client.service.getPreparedLigand(job_id) if ligand_files is None or len(ligand_files) != 3: raise ValueError("Could not prepare ligand!") return ligand_files def dock(client, protein, ligand, name=None): protein_psf, protein_crd = prepare_protein(client, protein) ligand_pdb, ligand_rtf, ligand_par = prepare_ligand(client, ligand) if name is None: name = "teachopencadd" + ''.join([random.choice(string.ascii_letters) for _ in range(5)]) job_id = client.service.startDocking( protein_psf, protein_crd, ligand_pdb, [ligand_rtf], [ligand_par], name) if job_id in (None, "None"): raise ValueError("Docking job could not be submitted") while not client.service.isDockingTerminated(job_id): time.sleep(5) all_files = client.service.getPredictedDockingAllFiles(job_id) with open('docking_results.zip', 'w') as f: f.write(all_files) target, docked = client.service.getPredictedDocking(job_id) client.service.forget(job_id) return target, docked def smiles_to_pdb(s, out='output.pdb'): m = Chem.AddHs(Chem.MolFromSmiles(s)) AllChem.EmbedMolecule(m) if out is None: return Chem.MolToPDBBlock(m) Chem.MolToPDBFile(m, out)
try: import Mol2Writer except ImportError: # RDKitのMol2 writer/readersを手に入れるための醜いハック import os working_dir = os.getcwd() os.chdir(_dh[0]) !wget https://raw.githubusercontent.com/rdkit/rdkit/60081d31f45fa8d5e8cef527589264c57dce7c65/rdkit/Chem/Mol2Writer.py > /dev/null os.chdir(working_dir) import Mol2Writer
def step_03_swissdock(protein_pdb, ligand_smiles): ligand = Chem.AddHs(Chem.MolFromSmiles(ligand_smiles)) AllChem.EmbedMolecule(ligand) ligand_mol2 = Mol2Writer.MolToCommonMol2Block(ligand) client = swissdock_client() return dock(client, protein_pdb, ligand_mol2)
現在SwissDockサーバーが利用できないので、下のセルは実行しないようにしています。実行したいのであれば、まずタンパク質のmol2ファイルをPDBに変換する必要があります。OpenBabelを使って行うことができます(パートCでインストールします)。ご自由に都合の良い
mol_to_pdb関数を定義してください。
# TODO: mol2からpdbに変換すること! # protein_pdbblock = mol2_to_pdb(PROTEIN, out=None) step_03_swissdock(protein_pdbblock, smiles[0]) ### OPAL Webサービスを使ったドッキングの実行
SwissDockは現在稼働していません。そこで手を変え別のWebサービスを利用しましょう。インターフェースは少々より原始的ですが、稼働するはずです。ですが、タンパク質とリガンドはローカル環境でAutoDockToolsを使って準備しなければなりません。Python3で利用可能なフォークを準備しましたが、テストは十分にできていません。ですが、ここでの目的には十分機能するように見えます。
次を実行してインストールすることができます:
!pip install https://github.com/jaimergp/autodocktools-prepare-py3k/archive/master.zip
ドッキング計算の手順は次のようになっています。
- AutoDockToolsでタンパク質とリガンドを準備します。(ローカル環境で実施)
- Proteins.plus' DoGSiteScorerを使って結合ポケットの可能性があるもののうち最も良いものを見つけます。この情報をVinaによる計算の設定を行うために使います。(幾何学的な中心と探索空間のサイズ)
- OPAL上でVinaの計算を実行します。
import time import os from io import StringIO
構造の準備
構造の準備はAutoDockTooksライブラリの適切な部分を実行するだけでできます。
MolKitで構造のファイルを読み込む- 準備のための正しい機能を適用する。タンパク質には
AD4ReceptorPreparationを、リガンドにはAD4LigandPreparationを適用します。
準備自体は次のような項目に注意して行います。
- タンパク質とリガンドに水素原子を付加する。
- タンパク質に属さないおかしな残基を取り除く。
- 原子タイプと部分電荷を割り当てる。
- リガンドの回転可能(torsionable)な分岐をみつける。
結果はPDBQTファイルとしてディスクに書き込まれます。
###################### # # 構造の準備 # ###################### import MolKit from AutoDockTools.MoleculePreparation import AD4ReceptorPreparation, AD4LigandPreparation def opal_prepare_protein(protein): """ AutoDockを使うにはPDBQTファイルが必要です。 """ mol = MolKit.Read(protein)[0] mol.buildBondsByDistance() RPO = AD4ReceptorPreparation(mol, outputfilename=protein+'.pdbqt') return protein + '.pdbqt' def opal_prepare_ligand(ligand): """ AutoDockを使うにはPDBQTファイルが必要です。 """ mol = MolKit.Read(ligand)[0] mol.buildBondsByDistance() RPO = AD4LigandPreparation(mol, outputfilename=ligand+'.pdbqt') return ligand + '.pdbqt'
結合サイトの予測
Proteins.plusのDoGSiteScorerは、PDBデータベースにあるタンパク質の処理が必要なだけならば、とても簡単に利用できるREST APIを提供しています(dogsite_scorer_submit_with_pdbidを参照してください)。ですが、我々はKLIFSから取得した構造を使っているので、公式のPDBに登録されている構造の位置と向きが、KLIFSのものと一致しているか保証することができません。両者を重ね合わせて得られた変換行列を、取得したポケットに適用することもできますが、標準Webインターフェースで提供されている方法と同様に、単純にPDBファイルをProteins.plusにアップロードするだけの方がより簡単でしょう。
しかし、REST APIはそのようなオプションを提供していないので、我々はこの処理をリバースエンジニアリングする必要があります。適切なHTTPリクエストの場所をみつけるためには、Chromデベロッパーツールのネットワークタブを開き、通常のWebサイトを利用した際にどのHTTPリクエストが実行されているか書き留めておく必要があります。ここで有効なHTTPリクエストを手に入れるためのポイントは、信頼性トークン(Authenticity token)とHTTPヘッダーです。
この調査の結果は統合して整理しdogsite_scorer_submit_with_custom_pdb 関数に反映させています。技術的な詳細について興味があれば、関数のコメントを読んでください。
このアプローチを行うには、結合サイトを予測するためにHTMLコードを構文解析するため、
BeautifulSoupも必要となります。
!pip install BeautifulSoup4
この方法を使えば、最も可能性の高い結合ポケットの幾何学的な中心とそのサイズを取得することができます。Vinaの計算を設定するにはどちらの値も必要となります。
###################### # # 結合ポケット予測 # ###################### from bs4 import BeautifulSoup import requests def dogsite_scorer_submit_with_pdbid(pdbid, chain): """ これは公式APIですが、PDBコードしか使えません。 パラメーター ---------------- pdbid : str 4文字のPDB ID chain : str 解析するタンパク質の鎖(chain) 戻り値 ---------------- str ジョブが実行中か完了したかについて更新情報を受け取るために用いるURL """ # Proteins.plusにジョブを投げる r = requests.post("https://proteins.plus/api/dogsite_rest", json={ "dogsite": { "pdbCode": pdbid, "analysisDetail": "1", "bindingSitePredictionGranularity": "1", "ligand": "", "chain": chain } }, headers= {'Content-type': 'application/json', 'Accept': 'application/json'} ) r.raise_for_status() # 計算の更新情報を取得するためには"location"を問い合わせる必要があります。 return r.json()['location'] def dogsite_scorer_submit_with_custom_pdb(pdbfile): """ カスタムしたPDBをアップロードするために、実際のHTMLフロントエンドを模倣する必要があります。 1. HTMLメタヘッダーからCSRF(クロスサイトリクエストフォージェリ)トークンを取得します。 2. アップロードするためにファイルをPOSTします。 3. 返ってくるHTMLページはURL IDを含んでおり、次に内部の省略されたジョブIDを取得するために利用できます。 (Webインターフェースを使っている時と同様に)Webサーバーがフロントエンドで実行する非同期の呼び出しを真似することで、 返ってきたHTMLページのURL IDを使ってパブリックなジョブのAPI IDを取得することができます。 4. パブリックジョブIDを手に入れたらREST APIの利用に切り替えることができます。 """ # プロセスの間、途中のcookieを保持するために`session`を使う必要があります。 session = requests.Session() r = session.get("https://proteins.plus/") r.raise_for_status() # ホームページには我々のリクエストを有効にするのに必要なCSRFトークンが含まれています。 # そうでないと安全ではありません!リクエストを行なっている間、これを使わなければいけないので、 # session HTTPヘッダーの一部として設定しておくのが最善の方法です。 html = BeautifulSoup(r.text) token = html.find('input', {'name': 'authenticity_token'}).attrs['value'] session.headers['X-CSRF-Token'] = token # 1. ファイルをアップロード with open(pdbfile, 'rb') as f: r = session.post("https://proteins.plus", files={'pdb_file[pathvar]': f}) r.raise_for_status() # REST APIがファイルのアップロードをサポートしているのであれば、すでにパブリックIDを手に入れていることになりますが、 # それまでは応急処置としてこの方法で済ませておく必要があります。 # 2. 内部のlocation IDの取得 html = BeautifulSoup(r.text) pdb_id = html.find('input', {'name': "dogsite[pdbCode]"}).attrs['value'] # 3. 内部のジョブIDの取得 session.headers['Referer'] = "https://proteins.plus" + pdb_id r = session.post(f"https://proteins.plus/{pdb_id}/dogsites", json={"dogsite": {"pdbCode": pdb_id}}, headers= {'Content-type': 'application/json', 'Accept': 'application/json'} ) r.raise_for_status() job_id = r.json()['job_id'] time.sleep(3) # サーバーがリクエストを処理できるように、処理を続ける前に少し待ちます。 # 4. パブリックなジョブIDの取得 while True: r = session.get(f"https://proteins.plus/{pdb_id}/dogsites/{job_id}?_={round(time.time())}", headers= { 'Accept': 'application/json, text/javascript, */*', 'Sec-Fetch-Mode': 'cors', 'Sec-Fetch-Site': 'same-origin', # どうもこの下の1行が大事なようです。 # これ無しではエラー406が投げられます。 'X-Requested-With': 'XMLHttpRequest'} ) r.raise_for_status() if 'Calculation in progress...' in r.text: # まだ完了していません。 time.sleep(5) continue if 'Error during DogSiteScorer calculation' in r.text: # 形式のおかしなファイル? raise ValueError('Could not run DoGSiteScorer!') break results_id = None for lines in r.text.splitlines(): for line in lines.split('\\n'): if 'results/dogsite' in line: results_id = line.split('/')[3] break if results_id is None: raise ValueError(r.text) return f"https://proteins.plus/api/dogsite_rest/{results_id}" def dogsite_scorer_guess_binding_site(protein): """ タンパク質の最も可能性が高い結合サイトを取得するためにProteins.plusのDoGSiteScorerを使います。 """ if len(protein) == 4: # PDBコード job_location = dogsite_scorer_submit_with_pdbid(protein) elif protein.endswith('.pdb'): job_location = dogsite_scorer_submit_with_custom_pdb(protein) else: raise ValueError("`protein` must be a PDB ID or a path to a .pdb file!") # 計算が完了したか確認します。 while True: result = requests.get(job_location) result.raise_for_status() # 失敗した場合、ここで止まります。 if result.status_code == 202: # まだ実行中です。 time.sleep(5) continue break # residuesファイルは幾何学的中心と半径をPDBファイルのコメントとして保持しています。 # 最初のファイル(residues[0])が最もスコアの良いポケットです。 pdb_residues = requests.get(result.json()['residues'][0]).text for line in pdb_residues.splitlines(): line = line.strip() if line.startswith('HEADER') and 'Geometric pocket center at' in line: fields = line.split() center = [float(x) for x in fields[5:8]] radius = float(fields[-1]) break return center, radius # これがVinaの計算に必要となるものです。
OPAL上でVinaを実行
(1)タンパク質とリガンドを準備し、(2)探索空間の推定が完了したら、OPAL Webサーバーに実際の計算をサブミットできます。以下の処理を行います。
sudsを使ってSOAPクライアントを初期化する- SOAP XMLリクエストに加えられるように、ファイルを
base64文字列 としてエンコードする - ジョブリクエストをサブミットし、ジョブIDを受け取る
- 計算が完了したかどうか確認するため、サーバーにジョブIDを使って問い合わせる
requestsで関連するファイルをダウンロードする
上記の手順はOPAL Webサイトにはあまり記述されておらず(実際、ドキュメンテーションは利用できなくなってきます))、これらのサーバーに依存しているUCSF Chimeraモジュールのソースコードの中身を調査することでわかりました。
通常、計算には5−15分かかるので、4番目のステップのためにJupyterの便利な機能を使ってリアルタイムに出力ファイルの中身を更新します(iprint()関数を参照してください)。こうすることで、計算がうまく進んでいるかどうか盲目的に信じる代わり進行度合いを確認できます!
###################### # # 計算の実行 # ###################### from suds.client import Client from IPython.display import display, clear_output, HTML from rdkit import Chem from rdkit.Chem import AllChem VINA_CONFIG = """ center_x = {center[0]} center_y = {center[1]} center_z = {center[2]} size_x = {size[0]} size_y = {size[1]} size_z = {size[2]} """ def opal_run_docking(protein, ligand, center, size, stream_output=True): """ OPAL Webサービスに接続しジョブをサブミットする """ client = Client("http://nbcr-222.ucsd.edu/opal2/services/vina_1.1.2?wsdl") files = 'receptor.pdbqt', 'ligand.pdbqt', 'vina.conf' with open(protein) as f: protein_contents = f.read() with open(ligand) as f: ligand_contents = f.read() file_map = [ {'name': 'receptor.pdbqt', 'contents': base64ify(protein_contents)}, {'name': 'ligand.pdbqt', 'contents': base64ify(ligand_contents)}, {'name': 'vina.conf', 'contents': base64ify(VINA_CONFIG.format(center=center, size=size))}, {'name': 'results.pdbqt', 'contents': ''}, ] cli_args = "--receptor receptor.pdbqt --ligand ligand.pdbqt --config vina.conf --out results.pdbqt" response = client.service.launchJob(cli_args, inputFile=file_map) job_id = response.jobID url = f"http://nbcr-222.ucsd.edu/opal-jobs/{job_id}" message = "Waiting for job " + url while True: r = requests.get(url + "/vina.out") try: r.raise_for_status() except: # 最初のチェック段階では出力ファイルはまだ出来ていないかもしれません。 iprint(message) else: iprint(f"{message}\n{r.text}") if client.service.queryStatus(job_id).code == 2: time.sleep(10) continue print('\nFinished!') break output_response = client.service.getOutputs(job_id) output_files = { 'stdout.txt': requests.get(output_response.stdOut).text, 'stderr.txt': requests.get(output_response.stdErr).text, } for f in output_response.outputFile: if f.name in files: continue r = requests.get(f.url) r.encoding = 'utf-8' r.raise_for_status() contents = r.text output_files[f.name] = contents time.sleep(0.1) return output_files ###################### # # ユーティリティー # ###################### import base64 def base64ify(bytes_or_str): """ Py2k base64encodeの動きを真似する """ if isinstance(bytes_or_str, str): input_bytes = bytes_or_str.encode('utf8') else: input_bytes = bytes_or_str output_bytes = base64.urlsafe_b64encode(input_bytes) return output_bytes.decode('ascii') def iprint(s): """ この関数を使って出力をプリントし、その前のものを上書きすることで、 継続的に更新されているようにみえるようにすることができます:) """ clear_output(wait=True) s = s.replace("\n", "<br />") display(HTML(f'<pre>{s}</pre>'))
全部まとめる
必要な関数をすべて定義し終わったので、パイプラインを作成することができます。
def step_03_opal(protein, smiles, pdbcomplex): """ タンパク質の構造とSMILES文字列のリストを与え、以下の手順を実施します・ ステップ: 1. AutoDock Vinaのためのタンパク質を準備(ローカルで実行) 2. DoGSiteScorerを使って最も可能性の高い結合サイトを見つける 3. 各リガンドについてRDKitを使って3DのPDBファイルを書き出し、 OPALでAutoDockを実行する すべて行うのに大体5-15分かかります。 結果は出力ファイルを含む辞書です。我々が主に興味があるのは結果の result['results.pdbqt']です。 """ prepared_protein = opal_prepare_protein(protein) center, radius = dogsite_scorer_guess_binding_site(pdbcomplex) size = [radius] * 3 # Vinaは立方体のボックス以外に対応していますが、単純のため立方体を使います for i, smile in enumerate(smiles): smiles_to_pdb(smile, f'data/ligand{i}.pdb') prepared_ligand = opal_prepare_ligand(f'data/ligand{i}.pdb') result = opal_run_docking(prepared_protein, prepared_ligand, center, size) return result
実行します!マジックコマンド%timeでどれくらい時間がかかったか測ります。
# `smiles`の最初のリガンドだけを処理します。 %time result = step_03_opal(PROTEIN, smiles[:1], COMPLEX)
訳注(2020/05)
上記のセルはエラーを返しました。OPALがサービスを停止しているのか現在実行できなくなっています。
NBCRと関係するWebサイト、Webサービスが2020年4月30日をもって終了したようです(NBCRリンク )。
訳注ここまで
出力結果を理解する
resultは辞書で、出力ファイルとその中身のテキストに相当するいくつかのkeyをもちます。我々が主に興味があるのは次です。
- ドッキングされたリガンドを含む
results.pdbqt。複数のモデルをふくむ修正されたPDBファイルです。タンパク質のグリッドを保持しているので、もともとのタンパク質の構造と一緒に各リガンドのモデルを開くだけで大丈夫です。 vina.outは上記で見たテキストの出力です。テーブルのような情報を見ることができます。
あとで取り出せるようにディスクに保存しておきます。
with open('data/results.pdbqt', 'w') as f:
f.write(result['results.pdbqt'])
with open('data/vina.out', 'w') as f:
f.write(result['vina.out'])
ドッキング結果の可視化
計算を実行しファイルをダウンロードしたら、可視化しましょう!やり方はパートCを見てください。
ディスカッション
OPALはドッキング計算を行うVinaを無料で提供していますが、サブミットする前に入力ファイルをローカル環境で準備する必要があります。準備の中には、探索空間の定義も含まれており、通常既知の結合サイトの周囲を指定します。視覚的に推定する代わりに、Proteins.plusのDoGSiteScorerサーバーを使って、最も可能性が高い結合サイトの中心と半径を計算しました。これら2つのサービスは異なるコミュニケーションインターフェースを使っていました。
Protein.PlusはRESTを使っていますが、KLIFSとは異なりswagger.jsonの定義を提供していないので、自分のリクエストを手動で構築しなければなりませんでした。幸い、Webサービスが単純だったので2、3個のリクエストが必要なだけ、、、と思っていました!!現在のAPIはカスタマイズしたPDBのアップロードを許可していないので、通常のブラウザーを使っているふりをするために多少のGET、POSTリクエストを使う必要がありました。正しいリクエストを推測するうえで、Chromデベロッパーツールのネットワークタブはとても便利でした。Webサービスのリバースエンジニアリングが必要になったら、便利なツールの一つです!
OPALはXMLベースの標準SOAPで構築されており、JSONベースのRESTよりもちょと扱いにくですが、sudsを使うことでとても簡単になりました。sudsは利用できるメソッドを教えてくれますが、これらはあまりきちんと説明されていません。幸い、UCSF Chimeraのモジュールにコードの例がいくつか散らばっていました。ジョブIDを手に入れたらOPALにサブミットされたジョブはパブリックなものとなり、ファイルはリアルタイムに更新されるので、サーバーにN秒毎に問い合わせを行い、動的にdisplay()関数で表示されるHTMLを更新することで、計算の出力をライブでプレビューすることができました。この技は自分のマシンで行なっている計算を問い合わせるのにも使うことができます。ファイルの中身を読み、現在の出力を消して新しい出力をHTML()オブジェクトとして表示するだけです(ipirnt()関数を参照してください)。
クイズ
- リモートサーバーでドッキングが成功したかどうかどうすればわかりますか?
- なぜAutoDock Vinaの入力ファイルをローカル環境で準備する必要がありましたか?
- 応用:MCule'sドッキングサーバーをJupyter Notebookから使うための正しいHTTPリクエストを見つけることはできますか?
訳以上
追記
今回も訳出、用語の選択に誤りがあるかもしれません。ご指摘いただければ幸いです。
残念ながらノートブックの実行はできませんでした。オンラインサービスを使うとなるとサービスが停止したり安定しないといった問題もあるんですね。
パートBの内容はアプリケーションをプログラム上で扱うためのコードばかりで私は全くついていけませんでした。文章から理解する能力がないので絵で説明してほしいです・・・。
KLIFSで遊んだ話(T11パートAの備忘録)
トークトリアル11イントロに引き続きトークトリアル11パートAも耳慣れない言葉が多く内容のフォローが難しかったです。
と、いうわけで今回も素人による適当解説をしますよ!!
KLIFS
今回はKLIFS(Kinase-Ligand Interaction Fingerprints and Structures)について調べてみます。トークトリアルの説明は文字のみで、データの取得に用いただけだったのですが、実際にWebページ にアクセスしてみるととても素敵な感じでした!

KLIFS Webページで遊ぶ
折角なのでT11パートAでコードで実施していた内容をWebページ上でも行なってみます。T11 パートAの内容は以下でした。
- Kinaseファミリー 「EGFR」 を検索
- PDB ID 3w33 を選択
- 描画
まずは検索から。上タブからSearchページに移動します。タンパク質やリガンドだけでなく色々な検索方法が用意されています。興味深いところでは結合ポケットのなかの水分子や、リガンド-キナーゼ相互作用パターンによる検索といったものまであります。目移りしてしまいますが、まずはT11通りにKinaseファミリーで検索します。
Search by classificationにて「Family:EGFR」「Organism:Human」として検索しました。

ヒットした構造(355エントリー)がテーブルとして表示されました。下図に化合物2次元構造がありますが、Orthosteric ligandの列の化合物の名称にマウスオーバーするだけで描画されます。レスポンスも早くてすごい!

またテーブル左上Plot results on Kinomeをクリックするとキノーム(ゲノム上のプロテインキナーゼ一式)の中で、どの位置に相当するか図示されます。

検索結果のテーブルはExcel、csv形式でダウンロードすることも可能で、またDownload Structuresで構造をダウンロードすることもできるようです(スクロールしていくとボタンがあります。)
「PDB ID: 3W33」の詳細を見るためにDetails列のShowをクリックして見ます。こんな感じ

エントリーの詳細な情報に加えて3Dの描画やリガンドの活性値情報もあります。ChEMBLから取得されているようで、EGFR以外のキナーゼに対する活性も合わせてみることができるので、選択性やオフターゲットの雰囲気もつかめそうです。
スクロールするともっと面白い項目もあります。

Kinase-ligand interaction patternは、リガンドがタンパク質のどの残基とどのような相互作用をしているか一目でわかるようになっています。私は結晶構造の描画をみても「格好いい!! で、どこが大事なんですかね???」となってしまうので、こういう見所(?)テーブルがあるのは非常に良いなと思います。また、一番下にひっそりとInteraction pattern searchなるものがあり、類似の相互作用パターンをもつ複合体をKLIFSから探してくれます。エモい・・・
KLIFSの背景
KLIFSのWebページの充実ぶりすごいですね。ちょっと背景を知りたいので論文を参照してみたいと思います。オンラインサービスに関する文献 Nucleic Acids Res. (2016), 44, 6, D365–D371 はオープンアクセスでした。
T11 パートAにも説明がありましたが、KLIFSはPDBからキナーゼの結晶構造を抜き出し、リガンドとの相互作用に関して分析処理を加えたデータベースです。特徴としてキナーゼの触媒ドメインに着目していることが挙げられます。
内部に踏み込む前にまずはキナーゼの構造について一般的な内容から
キナーゼの構造の特徴
こちらの日本語レビュー「 キナーゼを標的とした構造生物学および創薬の現状」が非常に分かりやすかったです。 *1 ざっくりと理解したことを紹介します。
キナーゼは基質をリン酸化するタンパク質ですが、リン酸基のソースはATPです。つまり共通して触媒キナーゼドメインの結合サイトにATPが結合します。
同じものが結合するならポケットも似ているだろ、ということでキナーゼ触媒ドメインはお互いにある程度似た構造をしています。
多くのキナーゼ阻害剤はこのATP結合領域に結合します。なので、異なるキナーゼ間で構造が似ていると、標的以外のキナーゼにも結合して阻害する可能性あります。
OpenTeachCADDで度々出てくる選択性の問題ですね。
よく保存された構造をもつキナーゼの結合ポケットですが、その特徴的な部分には名前がつけられています。例えば・・・
- Hinge : ATPのアデニンと相互作用する残基部位
- Gate Keeper : ATP結合ポケットの一番奥の残基部位。ATP結合サイトの立体構造への影響大
- DFG motif : Asp-Phe-Glyの1文字表記。側鎖の動きが大きくDFG-in構造、DFG-out構造とよばれるコンフォメーションの変化がある
などなど。また、阻害剤にも結合するサイト・阻害様式によってType I ~ IV、共有結合型などの分類があるそうです。
こういうの格好いいですよね!共通の情報が整理されて見通し良くなってく感じが分野の発展の歴史を見ているようで楽しいです。
まとめると「医薬品としてキナーゼ阻害剤は大事だが選択性が課題となる。背景には結合サイトにおける保存されたキナーゼの構造的特徴があり、特徴に従い分類、キナーゼー阻害剤相互作用の細かな差異を分析することで課題解決の有用な情報となる。」・・・ってことですかね?
つまり、そういう情報まとめたKLIFSは素敵なリソース!!
KLIFS文献参照
やっとKLIFSの文献に戻ってFigure 2. を引用します。こちらはKLIFSがどのように複合体のアノテーションを行なっているかを説明した図です。

先に見た単語がちらほら見えますね。この図で面白いのはキナーゼーリガンド相互作用をフィンガープリントして表現しているFig.2 A)下部だと思います。各アミノ酸残基について、疎水性相互作用や水素結合といった7種類の相互作用がリガンドとの間にみられるか?というのを、0、1のビットで表記しています(kinase-ligand interaction finger print, IFP)。
KLIFSは触媒ドメインを含む85残基を解析の対象としています。したがって、各複合体のキナーゼーリガンド相互作用の情報は「85残基x相互作用7種=595ビット」のフィンガープリントにエンコーディングされることになります(?あってます?)。これだけ情報が圧縮されてるから「類似の相互作用の複合体は他にあるのか?」といった検索が容易になるんですね。なるほど。
さて、KLIFSでは相互作用のフィンガープリント化と結合ポケットの部位の分類(3 major pockets + 12 subpockets)を行なったことで、各複合体のリガンドの結合ポーズ(binding mode)の分類が可能になったそうです。
また、興味深いことにリガンドに加えて水分子についても解析が行われています。下に引用した図のように、保存された水分子のクラスター位置が見つかっています。こちらもWebページ上で確認、検索することが可能になっています。

以上、非常にざっくりですがKLIFSで行われていることの雰囲気です。分類と解析とが体系化されることで自動化が可能になり、データベースの継続的なアップデートに繋がっているようです。
KLIFSのAPI
KLIFSのWebページについてはだいたいわかったので、T11パートAではAPIを使ってどのように情報が取り出されていたのかを見ていきたいと思います。
取り上げるのは途中で定義された以下の3つの関数です。
_all_kinase_families(): KLIFSデータベースに含まれているキナーゼファミリーの名前を取得_kinases_from_family(family, species="HUMAN"): 指定したキナーゼファミリーに含まれるキナーゼの名前を取得_protein_and_ligand_structure(*kinase_ids): 指定したKLIFSのキナーゼIDに紐づいたリガンド-キナーゼ複合体構造 (PDB)、タンパク質のみの構造(MOL2)、リガンド(SMILESリスト)を取得
REST?SWAGGER?
コードの中身を見る前にまずはよくわからない単語たち・・・
- REST(Representational State Transfer): Web APIの仕様を決める上での基本的な考え方。リソースのURIと、アクセスするメソッド(HTTPのGET、POSTなどの)の組み合わせの設計方針。*2
- SWAGGER: REST APIを定義、仕様を管理するためのフレームワーク。OpenAPI仕様をもとにした実装
・・・わからない。KLIFSのAPI をみてみます。

たとえばKinase groupsについて情報が欲しければ、リソースのURI(Uniform Resource Identifier)「~/kinase_groups」に対して、GETメソッドを使えば良いということのようですね。
また、今回のトークトリアルでは、このSwaggerサービスを使うためのクライアントの生成をライブラリBravadoを使っておこなっています。Bravadoのドキュメンテーションはこちらです。
bravadoによるSwagger用のクライアントの作成
それではbravadoを使って、KLIFS APIのURLからクライアントを作成します。URLからわかるようにjson形式となっているようです。
from bravado.client import SwaggerClient # URLを定義 KLIFS_API_DEFINITIONS = "http://klifs.vu-compmedchem.nl/swagger/swagger.json" # SwaggerClient : A client for accessing a Swagger-documented RESTful service KLIFS_CLIENT = SwaggerClient.from_url(KLIFS_API_DEFINITIONS, config={'validate_responses': False})
作成したSwaggerClientの属性をdir()で確認して見ます。
print(dir(KLIFS_CLIENT)) # ['Information', 'Interactions', 'Ligands', 'Structures']
これらが利用可能なリソースで、KLIFSのAPIに書かれている4種類と同じものがきちんと入っているようです。

関数_all_kinase_families()の中身
こちらはKLIFSからKinaseファミリーを取得する関数です。こちらが何をしているか順番に見ていきます。
def _all_kinase_families(): return KLIFS_CLIENT.Information.get_kinase_families().response().result
まず、リソースとしてinformationを選択し、そのkinase_familyに対してGETで情報を取得します(get_kinase_families())。Bravadoのドキュメンテーションによると、メソッドを実行することで戻り値としてHttPFutureを得るそうです(Futures and responses)。
確認してみます。
print(KLIFS_CLIENT.Information.get_kinase_families()) # <bravado.http_future.HttpFuture object at 0x117469ef0>
確かに戻り値はHttpFutureオブジェクトとなっています。ここからレスポンスを得るにはHttpFuture.responce()とすればよく、この戻り値としてBravadoResponseインスタンスが得られます。Swaggerの結果を得るためにはこのインスタンスに対してBravadoResponse.resultとすれば良いそうです。
順番にやってみます。
print("レスポンス:", KLIFS_CLIENT.Information.get_kinase_families().response()) # レスポンス: <bravado.response.BravadoResponse object at 0x117da7048>
print("type:", type(KLIFS_CLIENT.Information.get_kinase_families().response().result)) print("result", KLIFS_CLIENT.Information.get_kinase_families().response().result) # type: <class 'list'> # result ['A6', 'ABC1', 'AKT', 'ALK', 'AUR', 'Abl', 'Ack', 'Akt', 'Alk', 'Alpha', 'Aur', 'Axl', 'BCR', 'BRD', 'BUB', 'Bud32', 'CAMK-Unique', 'CAMK1', 'CAMK2', 'CAMKK', 'CAMKL', 'CASK', 'CCK4', 'CDC7', 'CDK', 'CDKL', 'CK1', 'CK2', 'CLK', 'Csk', 'DAPK', 'DCAMKL', 'DDR', 'DMPK', 'DYRK', 'EGFR', 'Eph', 'FAK', 'FAST', 'FGFR', 'Fer', 'G11', 'GRK', 'GSK', 'H11', 'Haspin', 'IKK', 'IRAK', 'IRE', 'InsR', 'JakA', 'JakB', 'KIS', 'LISK', 'LRRK', 'Lmr', 'MAPK', 'MAPKAPK', 'MAST', 'MLCK', 'MLK', 'MOS', 'Met', 'Musk', 'NAK', 'NDR', 'NEK', 'NKF1', 'NKF2', 'NKF3', 'NKF4', 'NKF5', 'NRBP', 'Other-Unique', 'PAN3', 'PDGFR', 'PDHK', 'PDK1', 'PEK', 'PHK', 'PIK', 'PIKK', 'PIM', 'PIP', 'PIPK', 'PKA', 'PKC', 'PKD', 'PKG', 'PKN', 'PLK', 'PSK', 'RAD53', 'RAF', 'RCK', 'RGC', 'RIO', 'RIPK', 'RSK', 'RSKL', 'RSKR', 'RSKb', 'Ret', 'Ror', 'Ryk', 'SCY1', 'SGK', 'SRPK', 'STE-Unique', 'STE11', 'STE20', 'STE7', 'STKR', 'Sev', 'SgK071', 'SgK493', 'SgK495', 'SgK496', 'Slob', 'Src', 'Syk', 'TAF1', 'TBCK', 'TIF1', 'TK-Unique', 'TKL-Unique', 'TLK', 'TOPK', 'TSSK', 'TTBK', 'TTK', 'Tec', 'Tie', 'Trbl', 'Trio', 'Trk', 'ULK', 'VEGFR', 'VPS15', 'VRK', 'WEE', 'WNK', 'Wnk', 'YANK']
無事BravadoResponseオブジェクトの生成と、結果として全キナーゼファミリーを要素としてもつリストを得ることができました。
Bravadoの動作の流れをまとめると・・・
- SwaggerClient作成 > リソース選択 > get_URIで情報取得 >
HttpFuture> response()でレスポンス取得 >BravadoResponse> resultで結果取得
・・・長い。
以上が定義した_all_kinase_families()関数になります。
関数_kinases_from_family(family, species="HUMAN")の中身
こちらは指定したGETメソッドの引数にキナーゼファミリーの名前と種(デフォルトの引数はHUMANに設定)を与えてキナーゼの名前を取得する関数です。
操作自体は先の例と同様です。
def _kinases_from_family(family, species="HUMAN"): return KLIFS_CLIENT.Information.get_kinase_names(kinase_family=family, species=species).response().result
関数_protein_and_ligand_structure()の中身
こちらはキナーゼのIDを引数として与え、以下の3つを返す関数です。、
- キナーゼIDに基づく複合体構造(PDB形式)
- タンパク質のみの構造(mol2形式)
- キナーゼIDに基づく共結晶構造中のリガンドのリスト(SMILESのみを抜き出して出力)
1、2はStructuresリソースから、3はLigandsリソースから得ています。
def _protein_and_ligand_structure(*kinase_ids): structures = KLIFS_CLIENT.Structures.get_structures_list(kinase_ID=kinase_ids).response().result molcomplex = KLIFS_CLIENT.Structures.get_structure_get_pdb_complex(structure_ID=structures[0].structure_ID).response().result protein = KLIFS_CLIENT.Structures.get_structure_get_protein(structure_ID=structures[0].structure_ID).response().result ligands = KLIFS_CLIENT.Ligands.get_ligands_list(kinase_ID=kinase_ids).response().result print(f"Chosen KLIFS entry with PDB ID {structures[0].pdb} with chain {structures[0].chain} and alternate model {structures[0].alt}") return molcomplex, protein, [ligand.SMILES for ligand in ligands]
EGFRを例として順番に見ていきます。準備としてまず例とするEGFRのkinase_IDを取得します。
EGFR_id_result = KLIFS_CLIENT.Information.get_kinase_ID(kinase_name="EGFR").response().result print(EGFR_id_result) print("number of result:", len(EGFR_id_result)) # [KinaseInformation(HGNC='EGFR', family='EGFR', full_name='epidermal growth factor receptor', group='TK', iuphar=1797, kinase_ID=406, kinase_class='', name='EGFR', pocket='KVLGSGAFGTVYKVAIKELEILDEAYVMASVDPHVCRLLGIQLITQLMPFGCLLDYVREYLEDRRLVHRDLAARNVLVITDFGLA', species='Human', uniprot='P00533'), KinaseInformation(HGNC='Egfr', family='EGFR', full_name='epidermal growth factor receptor', group='TK', iuphar=0, kinase_ID=663, kinase_class='', name='EGFR', pocket='KVLGSGAFGTVYKVAIKELEILDEAYVMASVDPHVCRLLGIQLITQLMPYGCLLDYVREYLEDRRLVHRDLAARNVLVITDFGLA', species='Mouse', uniprot='Q01279')] # number of result: 2
2つ結果が得られました。今回はhumanの情報をとりたいので一つ目のIDを取り出します。
EGFR_ID = EGFR_id_result[0].kinase_ID print(EGFR_ID) # 406
では_protein_and_ligand_structure()関数の1行目、複合体の構造のリストの取得です。ここではkinase_IDをリストで与えてやる必要があるので[406]として渡します。
EGFR_structures = KLIFS_CLIENT.Structures.get_structures_list(kinase_ID=[406]).response().result print(len(EGFR_structures)) # 324
全部で324個の構造情報が得られました。次の工程で複合体のPDBファイルを得るには、構造情報のうちstructure_idが必要となります。一つ目の構造情報で確認します。
EGFR_structure_id = EGFR_structures[0].structure_ID print(EGFR_structure_id) # 782
このstructure_idで複合体を取得します。今回は引数はintのままで大丈夫です。
EGFR_molcomplex = KLIFS_CLIENT.Structures.get_structure_get_pdb_complex(structure_ID=782).response().result print(type(EGFR_molcomplex)) # str
冗長なので省略しますがprintするとPDB形式になっていることが確認できます。ちなみに中身を見るとPDB_ID: 3w33でKLIFSのWebページで眺めていたものと同じでした。
同様にして同じstructure_idでタンパク質単体も取り出します。
EGFR_protein = KLIFS_CLIENT.Structures.get_structure_get_protein(structure_ID=782).response().result print(type(EGFR_protein)) # str
こちらも省略しますがprintするとmol2形式になっていることが確認できます。
最後にリガンドのリストを取得します。EGFRのkinase_IDを使いますが、ここでもリスト[406]にして渡します。
EGFR_ligands = KLIFS_CLIENT.Ligands.get_ligands_list(kinase_ID=[406]).response().result print(len(EGFR_ligands)) # 94
94個のリガンド情報を取得できました。ここからSMILESを取り出しますが、参考までにどのような情報があるか見てみます。
print(type(EGFR_ligands[0])) print(dir(EGFR_ligands[0])) # <class 'abc.ligandDetails'> # ['InChIKey', 'Name', 'PDB-code', 'SMILES', 'ligand_ID']
SMILES以外にもInChIKeyやNameなどの情報があるようです。
EGFR_SMILES = [] for lig in EGFR_ligands: smi = lig.SMILES EGFR_SMILES.append(smi) print("number of ligands: ", len(EGFR_SMILES)) print("Example SMILES: ", EGFR_SMILES[0]) # number of ligands: 94 # Example SMILES: P(=O)(OP(=O)(O)O)(OC[C@H]1O[C@@H](N2c3ncnc(N)c3N=C2)[C@H](O)[C@@H]1O)O
94個全ての構造のSMILESを取り出すことができました!これで_protein_and_ligand_structure()関数の全行程の確認ができました。
T11パートAのKLIFSに関する部分は、ここまで見てきたコードを踏まえて見返すとなんとなくやりたいことがわかりました。
最後に取り出したリガンドの構造を描画して見ましょう。
from rdkit import Chem from rdkit.Chem import Draw m = Chem.MolFromSmiles(EGFR_SMILES[0]) Draw.MolToImage(m)

ADPでした。
T11パートAの結果を振り返って
以上、KLIFSのWebページ版とAPIとを比較してきました。T11パートAの結果と見比べることで、取り出されたリガンド構造に関して気づいた点があります。
T11パートAの最後、ケーススタディー: EGFRでEGFRのリガンドを取り出した時、同時に取り出したPDB共結晶構造中のリガンドが選ばれたと思っていました。実行時に得られたPDB_ID3w33とリガンドADPでした。ですが、上で見たようにKLIFSのWebページで同じPDB_ID3w33を確認するとorthosteric ligndはADPではない化合物でした。
この差が不思議だったのですが、APIを順番に見ていくことでT11 パートAの最後はkinase_IDで指定したリガンドを全て取り出し、その最初の一つを表示させているだけ、ということがわかりました。
もし同時にPDBのリガンドを指定して取得したいのなら、同じstructure_IDを使って以下のようにすれば良さそうです。
# mol2形式でリガンドを取得 ligand_782 = KLIFS_CLIENT.Structures.get_structure_get_ligand(structure_ID=782).response().result # Mol2の中身は3次元で見づらいのでSMILES経由で2次元に戻してから描画 m782 = Chem.MolFromMol2Block(ligand_782) smi782 = Chem.MolToSmiles(m782) m782_2d = Chem.MolFromSmiles(smi782) Draw.MolToImage(m782_2d)

先に確認したように、「structure_id: 782」のPDB_IDは3W33で、KLIFSのWebページで見ていた複合体構造と同じでした。確かにここでは同じリガンドが取り出されているようです。
まとめ
以上、今回はT11 パートAで扱われていたデータベースKLIFSで遊んで見ました。素晴らしいWebページ版が提供されているので、まずはこちらで遊んでどのような情報が得られるのか感触をつかんでからの方がプログラムの内容を理解しやすのではないかな?と思いました。一目で結果を理解しやすいGUIベースのページは、プログラムが意図した通りの動きをしているかを検証する目的でも良さそうです。
プログラムの内容についてREST、SWAGGERは相変わらず定義はよく分かりませんが、ぼんやりと何がしたいかはわかったような気がしないでもないような?仕様を定義ってのは、文法みたいなイメージで良いのでしょうか???
素人が適当にしらべたことをだらだら書いているので色々と間違いが多いと思います。ご指摘いただければ幸いです。
*1: キナーゼを標的とした構造生物学および創薬の現状 日本結晶学会誌 59,174-181(2017)
TeachOpenCADD トピック11(パートA)〜オンラインAPI/サービスを使った構造に基づくCADD〜
こちらはTeachOpenCADDの試訳です。TeachOpenCADD GitHubレポジトリのトークトリアル11をもととしておりCC BY 4.0のライセンスに従っています。
トークトリアル11は論文発表時のTeachOpenCADDには含まれておらず新しく追加されたものです。
4つのノートブックに分かれており、ここではその2つ目を訳出します。
トークトリアル11で取り上げられているWebサーバー、サービスには既に停止しているものもあり、そのまま使えるものではなくなってしまっていますが、基本的な考え方や手法を知る上では役にたつかもしれません。
以下、訳。
トークトリアル 11 (パート A)
オンラインAPI/サーバーを使った構造に基づくCADD
Developed at AG Volkamer, Charité
Dr. Jaime Rodríguez-Guerra, Dominique Sydow
このトークトリアルの目的
これは「オンラインWebサービス」についてのトークトリアルのパートAです。:
- 11a. キナーゼ阻害剤の候補をKLIFとPubChemで検索
- 11b. 11aで取得した候補化合物をターゲットタンパク質に対してドッキング
- 11c. 結果を評価し既知のデータと比較
KLIFデータベースにクエリを投げ、キナーゼの構造と阻害剤を一つ取得します。それから、PubChemで類似の化合物を検索します。
学習の目標
理論
- パイプラインと含まれているWebサービスの解説
実践
- パイプラインを構成する関数を書く
- ケーススタディー:EGFR阻害剤
レファレンス
- KLIFS:キナーゼー阻害剤相互作用データベース
- データベースそのものの解説 (J. Med. Chem. (2014), 57, 2, 249-277)
- オンラインサービスの解説 (Nucleic Acids Res. (2016), 44, 6, D365–D371)
- PubChem:低分子化合物のデータベース(Nucleic Acids Res. (2019), 47, D1102-1109)
- NGLView:Notebookのためのインタラクティブな可視化ソフト(Bioinformatics (2018), 34, 1241–124)
理論
ここまでで、さまざまなPythonライブラリをつかってオンラインWebサービスにクエリを投げる方法をみてきました。オンラインWebサービスからパイプラインを構築してみましょう!
パイプラインと含まれているWebサービスの解説
- キナーゼーリガンド相互作用フィンガープリントと構造のデータベース(KLIFS: Kinase-Ligand Interaction Fingerprints and Structures database)はアムステルダムのVU大学医薬品化学分野で開発されたデータベースで、触媒キナーゼドメインのタンパク質構造に関する情報(PDBから収集)と、リガンドとの相互作用に関する情報とを提供しています。このデータベースから精選されたタンパク質構造を取得し、そのリガンドの情報をつかってPubChemやChEMBLといった他のデータベースから類似したリガンドを取得することができます。
- KLIFによるリガンド情報を使って、PubChemで類似化合物を探します。
- タンパク質構造と候補となるリガンドをいくつか手に入れたら、OPALWebサービスで提供されている組み込みのVinaを使ってオンラインでドッキングを実施します。また、proteins.plus' DoGSiteScorerで化合物をドッキングする結合サイトの候補を探します。(パート B)
- 結果を
nglviewで可視化し、相互作用を PLIPを使ってレポートします。(パート C)
このトークトリアル パート(A)では、KLIFSデータベースから入力として用いるキナーゼー阻害剤構造を取得し、候補阻害剤として評価することが可能な類似化合物をPubChemで検索します。関連する出力はディスクのdata/に書き込み、あとで使います。
KLIFS
- 役割: キナーゼーリガンド相互作用プロファイルデータベース
- Webサイト: http://klifs.vu-compmedchem.nl/
- API: RESTベース、Swaggerで利用可能なものがあります。正式なクライアントはありません。
bravadoを使ってください。 - ドキュメンテーション: http://klifs.vu-compmedchem.nl/swagger/
- 文献:
- データベースそのものの解説 (J. Med. Chem. (2014), 57, 2, 249-277)
- オンラインサービスの解説 (Nucleic Acids Res. (2016), 44, 6, D365–D371)
キナーゼーリガンド相互作用フィンガープリントと構造のデータベース(KLIFS: Kinase-Ligand Interaction Fingerprints and Structures database)はアムステルダムのVU大学医薬品化学分野で開発されたデータベースで、触媒キナーゼドメインのタンパク質構造と、キナーゼ阻害剤の阻害様式に焦点をあてています。一貫したシステマティックなプロトコルに基づき、(現在、ヒトとマウスの)全てのキナーゼ構造とキナーゼリガンドの結合様式を直接互いに比較することができます。さらに、結合サイト全体を取り囲む85残基の分類がなされているので、例えば、キナーゼー阻害剤選択性を決める重要な相互作用を見つけるといった目的のため、キナーゼー阻害剤の相互作用パターンを互いに比較することができます。
PubChem
- 役割: 低分子化合物データベース
- Webサイト: https://pubchem.ncbi.nlm.nih.gov/
- API: RESTベースのものが利用可能です。正式なクライアントはありません。
bravadoを使ってください。 - ドキュメンテーション: https://pubchemdocs.ncbi.nlm.nih.gov/pug-rest
- 文献:
PubChemはアメリカ国立衛生研究所(NIH、National Institutes of Health)のオープン化学データベースです。「オープン」とはつまり、自分の科学的なデータをPubChemに入れることができ、他の利用者がそのデータを使用するかしれないということを意味します。2004年の立ち上げ以来、PubChemは科学者、学生そして一般の人々のための化学に関する重要な情報源となってきました。毎月、我々のWebサイトとプログラムによるサービスは世界中の数百万の利用者にデータを提供しています。
実践
パイプラインの構築
KLIFSから情報を取得する
KLIFSデータベースから、ランダムなキナーゼファミリーのランダムなキナーゼ(mol2形式)と、対応するリガンド(SMILES)を選びます。また、パートBで結合ポケットを計算するために、タンパク質ーリガンド複合体のPDB構造を取得します。
from bravado.client import SwaggerClient KLIFS_API_DEFINITIONS = "http://klifs.vu-compmedchem.nl/swagger/swagger.json" KLIFS_CLIENT = SwaggerClient.from_url(KLIFS_API_DEFINITIONS, config={'validate_responses': False})
def _all_kinase_families(): return KLIFS_CLIENT.Information.get_kinase_families().response().result def _kinases_from_family(family, species="HUMAN"): return KLIFS_CLIENT.Information.get_kinase_names(kinase_family=family, species=species).response().result def _protein_and_ligand_structure(*kinase_ids): structures = KLIFS_CLIENT.Structures.get_structures_list(kinase_ID=kinase_ids).response().result molcomplex = KLIFS_CLIENT.Structures.get_structure_get_pdb_complex(structure_ID=structures[0].structure_ID).response().result protein = KLIFS_CLIENT.Structures.get_structure_get_protein(structure_ID=structures[0].structure_ID).response().result ligands = KLIFS_CLIENT.Ligands.get_ligands_list(kinase_ID=kinase_ids).response().result print(f"Chosen KLIFS entry with PDB ID {structures[0].pdb} with chain {structures[0].chain} and alternate model {structures[0].alt}") return molcomplex, protein, [ligand.SMILES for ligand in ligands]
import random import time def random_kinase_structure(): """ ランダムなキナーゼファミリーのランダムなキナーゼを取得する """ attempts = 20 families = _all_kinase_families() while attempts: # キナーゼIDの中には利用可能な構造がないものもあります family = random.choice(families) kinase = random.choice(_kinases_from_family(family)) try: molcomplex, protein, ligands = _protein_and_ligand_structure(kinase.kinase_ID) except: attempts -= 1 time.sleep(1) else: print("Chosen", kinase.name, "kinase with ID", kinase.kinase_ID, "from family", family) return molcomplex, protein, ligands print("Could not find a valid kinase. Try again!") return None, None, None def kinase_structure_from_family(family): """ キナーゼファミリーの名前(`_all_kinase_families()`を確認してください)が与えられると、ランダムな構造を取得します。 """ attempts = 20 while attempts: # キナーゼIDの中には構造のリストで見つけ流ことができないものもあります・・・。 kinase = random.choice(_kinases_from_family(family)) try: molcomplex, protein, ligands = _protein_and_ligand_structure(kinase.kinase_ID) except: attempts -= 1 time.sleep(1) else: print("Chosen", kinase.name, "kinase with ID", kinase.kinase_ID, "from family", family) return molcomplex, protein, ligands print("Could not find a valid kinase. Try again!") return None, None, None
うまくいくか確認してみましょう!例えば、ランダムに選んだキナーゼファミリーの中からランダムにキナーゼが欲しいとします。その場合、次のようにrandom_kinase_structure()を使うことができます。
molcomplex, protein, ligands = random_kinase_structure() # Chosen KLIFS entry with PDB ID 3hng with chain A and alternate model A # Chosen FLT1 kinase with ID 483 from family VEGFR
ligands
# ['Clc1ccc(NC(=O)c2c(NCc3ccncc3)cccc2)cc1']
訳注(2020/05)
上記は実行の度に結果が変わります。オリジナルのノートブックでは以下を得ています。
- PDB ID 6cot、PKGファミリー(ID 42)、PRKG1キナーゼ
- リガンド: ['P(=O)(OP(=O)(O)NP(=O)(O)O)(OC[C@H]1OC@@Hc3N=C2)C@H[C@@H]1O)O', 'CCCOc1ccc(c(c1C(=O)c2ccc(cc2)C(=O)N[C@@H]3CNC[C@H]3NC(=O)c4ccc5c(c4)cn[nH]5)F)OC']
以降の本文はこの出力を踏まえたものであることをお知りおきください。
訳注ここまで
試しにタンパク質をnglviewで、リガンドをrdkitで見てみましょう。まだnglviewをインストールしていなければ以下のセルを実行してください。
!pip install nglview !nglview install !nglview enable
import nglview as nv from tempfile import NamedTemporaryFile from rdkit import Chem from rdkit.Chem.Draw import IPythonConsole, MolsToGridImage # 分子を描画するのに必要です。 def preview_molecule_contents(contents, ext="mol2"): # これは一時的なファイルで、自動的に消去されます。 v = nv.NGLWidget() v.add_component(contents, ext=ext) return v def preview_smiles(smiles): print(smiles) return Chem.MolFromSmiles(smiles) def multi_preview_smiles(*smiles): legends = [f"{s[:30]}..." for s in smiles] # テキストが重なるのを避けるためにSMILES文字列を短くします。 molecules = [Chem.MolFromSmiles(s) for s in smiles] return MolsToGridImage(molecules, molsPerRow=3, subImgSize=(300, 300), maxMols=len(molecules), legends=legends, useSVG=True)
v = preview_molecule_contents(protein)
v
# NGLWidget()
v.render_image(),
# (Image(value=b'', width='99%'),)
v._display_image()

preview_smiles(ligands[0]) # Clc1ccc(NC(=O)c2c(NCc3ccncc3)cccc2)cc1

PubChemを検索
上で取得したSMILES文字列を使って、PubChemで類似の化合物を検索しましょう。
ヒント:ChEMBLデータベースを使って似たような操作を行う方法はTalktorial T1 を参照してください。
import requests def similar_compounds_pubchem(smiles, threshold=75, n_records=10): # 類似化合物をPubChemで検索する url = f"https://pubchem.ncbi.nlm.nih.gov/rest/pug/compound/similarity/smiles/{smiles}/JSON?Threshold={threshold}&MaxRecords={n_records}" r = requests.get(url) r.raise_for_status() key = r.json()['Waiting']['ListKey'] # 応答は非同期なので、処理が終わったかどうか25秒間毎秒問い合わせます。 attempts = 25 while attempts: r = requests.get(f"https://pubchem.ncbi.nlm.nih.gov/rest/pug/compound/listkey/{key}/cids/JSON") r.raise_for_status() response = r.json() if 'IdentifierList' in response: cids = response['IdentifierList']['CID'] break attempts -= 1 time.sleep(1) else: raise IOError("Could not find matches for " + smiles) # 化合物IDを戻り値として得られますが、SMILESが必要です。 url = f"https://pubchem.ncbi.nlm.nih.gov/rest/pug/compound/cid/{','.join(map(str, cids))}/property/CanonicalSMILES/JSON" r = requests.get(url) r.raise_for_status() return [item['CanonicalSMILES'] for item in r.json()['PropertyTable']['Properties']]
def query_pubchem_for_similar_compounds(ligands): # 現在のキナーゼの最初のリガンドを取得します。 smiles = ligands[0] # PubChem上の最も類似した10個の化合物を探します。 return similar_compounds_pubchem(smiles, n_records=10)
取得した化合物をRDKitで描画します。
similar_smiles = query_pubchem_for_similar_compounds(ligands) multi_preview_smiles(*similar_smiles)

ケーススタディー: EGFR
対応するWikipediaの記事から改変して引用します。
上皮成長因子受容体(Epidermal Growth Factor Receptor; EGFR)はErbBファミリー受容体のメンバーで、密接に関係する4つの受容体チロシンキナーゼ、EGFR (ErbB-1)、HER2/neu (ErbB-2)、Her3 (ErbB-3)そしてHer4 (ErbB-4)のサブファミリーです。多くのがん種で、EGFRの発現あるいは活性に影響を与える変異が、発がんに繋がります。 ヒトにおいてEGFRと他の受容体チロシンキナーゼのシグナルの欠損はアルツハイマー病といった疾患と関連づけられており、一方で過剰発現はさまざまな種類の腫瘍の発達と関連づけられています。受容体細胞外ドメインのEGFR結合サイトの阻害、あるいは細胞内のチロシンキナーゼ活性の阻害によるEGFRシグナルの遮断により、EGFRを発現する腫瘍の成長を阻害したり患者の症状を改善することができます。
つまり、私たちはEGFRファミリーのメンバーをターゲットとすることができる阻害剤の候補を探すことに興味があります。さあ、上記と同じステップを繰り返しましょう!但し今回はこの特定のキナーゼファミリーをターッゲトとします。
egfr_molcomplex, egfr_protein, egfr_ligands = kinase_structure_from_family('EGFR') # Chosen KLIFS entry with PDB ID 3w33 with chain A and alternate model A # Chosen EGFR kinase with ID 406 from family EGFR
訳注(2020/05)
今回はたまたまオリジナルのノートブックと同じ結果が得られましたが、EGFRについては複数の構造が登録されているため、実行の度に取得されるPDB IDは異なります。
訳注ここまで
タンパク質を試しに見てみましょう。
v = preview_molecule_contents(egfr_protein)
v
# NGLWidget()
v.render_image(),
# (Image(value=b'', width='99%'),)
v._display_image()

EGFRのリガンド(ATP)を試しに見てみましょう。
preview_smiles(egfr_ligands[0]) # P(=O)(OP(=O)(O)O)(OC[C@H]1O[C@@H](N2c3ncnc(N)c3N=C2)[C@H](O)[C@@H]1O)O

(訳注 ADPの誤記だと思います。)
類似したリガンドを試しに見てみましょう。
similar_smiles_egfr = query_pubchem_for_similar_compounds(egfr_ligands) multi_preview_smiles(*similar_smiles_egfr)

トークトリアルの次のパートのためにディスクに結果を書き出しておきましょう!
import os os.makedirs('data', exist_ok=True) with open('data/similar_smiles.txt', 'w') as f: f.write('\n'.join(similar_smiles_egfr)) with open('data/protein.mol2', 'w') as f: f.write(egfr_protein) with open('data/complex.pdb', 'w') as f: f.write(egfr_molcomplex)
ディスカッション
このノートブックでは利用可能な技術に応じて異なるWebサービスにアクセスし使用する方法を学びました。きちんとした説明のあるAPIから、実際のWebブラウザーを真似しようとする手作りのスクレイパーまで取り上げました。
ここまで、KLIFSデータベースでEGFRファミリーのメンバーを検索し、キナーゼの構造と(当然のことながら)その内在性リガンドであるATPを取得しました。次に、PubChemから75%以上の類似度をもつ化合物を取得しました。次のパートではこれらの中から一つを選んで構造モデリングを実施します。
クイズ
- "EGFR"ファミリーについてKLIFは幾つのキナーゼを提供しているでしょうか?
- 類似性検索をより厳密にすることはできますか?
- 提案された候補のなかにすでに承認された阻害剤はあるでしょうか?(ヒント:PKIDBをスクレイプして、SMILESのリストを確認することができます)
訳以上
追記
今回も訳出、用語の選択に誤りがあるかもしれません。ご指摘いただければ幸いです。
今回でてきたPubChemにはPythonからアクセスするためのライブラリPubChemPyもあります。ドキュメントをざっとみる限り類似性検索の機能はなさそうでした。
PubChemPyの使い方と注意点は「化学の新しいカタチ」さんが以下の記事でわかりやすく解説してくださっていますのでご参照いただければと思います。
