RDKitで3次元構造に挑戦! part 3 〜HALAVENの共結晶構造との比較〜
前回までにRDKitを用いた3次元構造の生成、ならびに2次元構造の類似性比較を行いました。
今回はPDBに登録された共結晶構造との3次元構造の比較を行いたいと思います。
1. HALAVEN のおさらい
まずは今更ですが、そもそもHALAVEN®️( eribulin mesylate)ってどういう医薬品なの??という話から。
以下インタビューフォーム(IF)より抜粋
①ハラヴェンの有効成分エリブリンメシル酸塩はHalichondrin Bの合成誘導体 。(IF p.1)
②再発乳癌等を適応とする抗悪性腫瘍剤。(IF p.11)
③作用機序としては微小管のプラス端(伸長端)に結合することによる、微小管ダイナミクスの阻害。
微小管による正常な紡錘体形成を妨げることで、細胞分裂を停止させアポトーシスによる細胞死を誘導し、がん細胞の増殖を抑制します。(IF p.23)
④薬効評価の in vitro 試験(無細胞系)として、微小管ダイナミクスに対する作用が評価されており、微小管を形成するタンパク質であるチューブリンの重合阻害試験が用いられています。(IF p.25)
ちょうど良いことにチューブリン(Tublin) とHALAVEN®︎(Eribulin)の共結晶構造[PDB ID: 5JH7]が報告されていますので、早速眺めてみましょう。
(チューブリン命名したの日本人研究者の方だそうです。「チューブラリン」と迷ったってホンマかいな・・・チューブリン - Wikipedia)
2. 共結晶構造を眺める
まずはどのような構造となっているかPDB viewerで確認してみましょう。

図のようにαβ-tublinの複合体の間にEribulinが挟まった構造となっています。
Eribulinはちょうど「お椀」のような形となっており、β-tublin をキャップするかのように結合し、重合の伸長を阻害している様がわかります。
念のためcootでEribulin結合部位の電子密度を確認して見ましたが、リガンドと重なるよう電子密度を確認できました。
また、こちらの共結晶構造にはGTP/GDPも含まれており、チューブリンの伸長がGTPの結合・加水分解により駆動されている様もわかる興味深い構造となっています。
3. RDKitでリガンド構造をながめる
それではPDBに登録されているリガンドの構造をRDKitで眺めて見ましょう。
RSCB PDB のページより[Ligand ID : 6K9]のsdfファイルをダウンロードしました。
「Ligands_noHydrogens_withMissing_2_Instances.sdf」 という不穏な名前で保存されました・・・
モヤモヤしますがとりあえず「6K9_noH.sdf」とファイル名を変更して進めていきます。
まずはSDFファイルの読み込みから・・・(参考記事①)
#読み込み方法-1 (SDMolSupplier) from rdkit import Chem suppl = Chem.SDMolSupplier('./6K9_noH.sdf') mols = [x for x in suppl if x is not None] #分子が読み込めているかを確認 len(mols) #2と出力。2つ入っているらしい #読み込み方法-2 (Pandasデータフレームへの読み込み) from rdkit.Chem import PandasTools df = PandasTools.LoadSDF('./6K9_noH.sdf') #MolオブジェクトはROMolというカラムに格納されているらしい eri_PDB_noH = df.loc[0, 'ROMol'] #2次元描画 from rdkit.Chem import rdDepictor from rdkit.Chem.Draw import IPythonConsole rdDepictor.SetPreferCoordGen(True) eri_PDB_noH #3次元描画 IPythonConsole.drawMol3D(eri_PDB_noH)
以下のような構造が出力されました。

以前、PDBフォーマットの見方で確認したように、PDBの構造には水素原子が含まれていません。
Pandasデータフレームをみると、リガンドのsdfファイルには不斉や結合次数の情報は含まれているようですがやはり水素原子は含まれていないようです。
水素原子を付加してみましょう。
#水素原子を付加 from rdkit.Chem import AllChem eri_PDB_addH = AllChem.AddHs(eri_PDB_noH) IPythonConsole.drawMol3D(eri_PDB_addH)

・・・疾走感 !!!
どうやら3次元構造全体に対して水素原子1つを付け足しただけのようです。
AddHsの使い方を間違えたらしい。
いろいろ探しているといままで気づいていなかったページがPDBにありました。
こちらのリガンド詳細情報のページには
・Structure Data File (Ideal SDF)
・Structure Data File (Model SDF)
という、2つのSDF形式の情報があり、共に水素原子を含むものでした。
それぞれを
・6K9_SDF_ideal.sdf
・6K9_SDF_model.sdf
という名前で保存して、立体構造をながめてみましょう。
また、折角なのでRDKitでsmilesから生成させた3次元構造もならべて見ましょう。
#水素原子が大事なので 「removeHs=False」として読み込む SDF_ideal = Chem.SDMolSupplier('./6K9_SDF_ideal.sdf', removeHs=False) SDF_model = Chem.SDMolSupplier('./6K9_SDF_model.sdf', removeHs=False) #それぞれMolBlock形式に変換 SDF_ideal_mb = Chem.MolToMolBlock(SDF_ideal[0]) SDF_model_mb = Chem.MolToMolBlock(SDF_model[0]) #smilesからの3次元構造生成は省略 ( eri_molH_mb という名前でMolBlockを生成した) #py3Dmolで3つ並べて表示 import py3Dmol v = py3Dmol.view(width=1000, height=400, linked=True, viewergrid=(1,3)) v.addModel(eri_molH_mb, viewer=(0, 0)) v.addModel(SDF_ideal_mb, viewer=(0, 1)) v.addModel(SDF_model_mb, viewer=(0, 2)) v.setBackgroundColor('0xeeeeee') v.setStyle({'stick':{}}) v.zoomTo() v.show()
以下のようになりました。
左から①RDKitで生成した構造、②PDBのIdeal SDF、③ PDBのModel SDF となります。
Fig. 5 より①と②が類似しており、③は水素原子のないPDB構造(Fig. 2)に似ていることがわかります。
おそらく、Fig. 5 ②は2次元構造式から計算により生成された3次元構造で、
Fig. 5 ③ が Fig. 2の実験由来のモデル構造に水素原子を付加した形となっているのではないでしょうか?(すみません。憶測です。)
折角なので類似の①と②を重ね合わせて確認して見ましょう。
#水素原子を含まないPDBのSDFファイルをMolBlock形式に変換 eri_PDB_noH_mb = Chem.MolToMolBlock(eri_PDB_noH) #Model SDFの構造に上記を重ね合わせる AllChem.AlignMol(eri_PDB_noH, SDF_model[0]) #一つのviewオブジェクトに表示 p = py3Dmol.view(width=400,height=400) p.addModel(eri_PDB_noH_mb) p.addModel(SDF_model_mb) p.setStyle({'stick':{}}) p.setBackgroundColor('0xeeeeee') p.zoomTo() p.show()

・・・完全に一致!
Model SDFが実験データに水素原子の情報を付け足したものと考えて良さそうです。
ところでRDKit で生成した3次元構造(Fig.5 ①) は PDBのリガンド構造(Fig. 2)よりも平面的となっており、
計算結果と実験結果との差異が見られて興味深い結果となりました。
Fig. 1 から、EribulinはTublin をキャップするように重合阻害している様子でしたので、計算で出した構造ではあまりそのような構造の特徴が再現できていないように見えます。
4. 立体配座生成のアルゴリズムを比較
念のため、他のアルゴリズムでも3次元構造を生成して比較して見ましょう。(参考記事②)
#isomeric SMILES取得過程は省略(eri_isoという変数に格納) #それぞれ3次元構造を生成しMolBlock形式に変換 #①ディスタンスジオメトリー(DG)法 + Universal Force Field(UFF) eribulin_DG = AllChem.AddHs(Chem.MolFromSmiles(eri_iso)) AllChem.EmbedMolecule(eribulin_DG) AllChem.UFFHasAllMoleculeParams(eribulin_DG) #True AllChem.UFFOptimizeMolecule(eribulin_DG_UFF) eribulin_DG_UFF_mb = Chem.MolToMolBlock(eribulin_DG_UFF) #②ディスタンスジオメトリー(DG)法 + Merck Molecular Force Field (MMFF) 94 eribulin_DG_MMFF = AllChem.AddHs(Chem.MolFromSmiles(eri_iso)) AllChem.EmbedMolecule(eribulin_DG_MMFF) AllChem.MMFFOptimizeMolecule(eribulin_DG_MMFF) eribulin_DG_MMFF_mb = Chem.MolToMolBlock(eribulin_DG_MMFF) #③KDG (DG + basic Knowledge) eribulin_KDG = AllChem.AddHs(Chem.MolFromSmiles(eri_iso)) AllChem.EmbedMolecule(eribulin_KDG, AllChem.KDG()) eribulin_KDG_mb = Chem.MolToMolBlock(eribulin_KDG) #④ETDG (DG + Experimental Torsion angle preferences) eribulin_ETDG = AllChem.AddHs(Chem.MolFromSmiles(eri_iso)) AllChem.EmbedMolecule(eribulin_ETDG, AllChem.ETDG()) eribulin_ETDG_mb = Chem.MolToMolBlock(eribulin_ETDG) #⑤ETKDG (DG + K + ET) eribulin_ETKDG = AllChem.AddHs(Chem.MolFromSmiles(eri_iso)) AllChem.EmbedMolecule(eribulin_ETKDG, AllChem.ETKDG()) eribulin_ETKDG_mb = Chem.MolToMolBlock(eribulin_ETKDG) #py3Dmolで描画(省略) #PDBから取得した構造(Model SDF)とのRMSDを計算 mol_list = [eribulin_DG_UFF, eribulin_DG_MMFF, eribulin_KDG, eribulin_ETDG, eribulin_ETKDG] RMSD = [] for i in mol_list: a = AllChem.GetBestRMS(SDF_model[0], i) RMSD.append(a) #水素原子がない場合(Heavt Atom RMSD)も計算 Heavy_Atom_RMSD = [] for i in mol_list: a = AllChem.GetBestRMS(eri_PDB_noH, Chem.RemoveHs(i)) Heavy_Atom_RMSD.append(a)

今回の結果では⑤ETKDG が最もRMSDが小さくなりました。
DG +MMFF(②) > KDG(③) > DG + UFF(①) > ETDG(④) > ETKDG(⑤) となっています。
概ね改良された順番にPDBの共結晶構造に近い3次元構造を生成しているようです。
最もRMSDが大きい②と、最も小さい⑤についてそれぞれPDBの構造をと重ね合わせて見ましょう。
#Model SDFの構造に②DG+MMFF と ⑤ETKDGをそれぞれ重ね合わせる AllChem.AlignMol(eribulin_DG_MMFF, SDF_model[0]) AllChem.AlignMol(eribulin_ETKDG, SDF_model[0]) #MolBlock形式に変換 eribulin_DG_MMFF_mb = Chem.MolToMolBlock(eribulin_DG_MMFF) eribulin_ETKDG_mb = Chem.MolToMolBlock(eribulin_ETKDG) SDF_model_mb = Chem.MolToMolBlock(SDF_model[0]) #描画 v = py3Dmol.view(width=1000, height=400, linked=True, viewergrid=(1,2)) v.addModel(eribulin_DG_MMFF_mb, viewer=(0, 0)) v.addModel(SDF_model_mb, viewer=(0, 0)) v.addModel(eribulin_ETKDG_mb, viewer=(0, 1)) v.addModel(SDF_model_mb, viewer=(0, 1)) v.setBackgroundColor('0xeeeeee') v.setStyle({'stick':{}}) v.zoomTo() v.show()
こんな感じ
左右を比較してみるとRMSDの小さい右の方が重なりが良いというのがはっきりとわかります。
「化学の新しいカタチ」さんの記事(参考記事②)でDr Tomさんが 述べられているように、まずは⑤ETKDGを試しておけば良い結果が得られそうです。
5.EribulinとPaclitaxelの比較
これまでEribulin や元になった天然物 Halichondrin Bとの比較を行ってきました。
最後に「化学の新しいカタチ」さんでも何度か取り上げられている、複雑な天然物の代名詞 Paclitaxel とEribulin を比較して見たいと思います。
興味深いことにPaclitaxel(Taxol)も微小管を構成するチューブリンのβ-サブユニットに結合し、細胞分裂を阻害することで抗腫瘍活性を発揮します。
一方で、Eribulinは微小管の重合を阻害するのに対し、Paclitaxelは微小管を安定化させ脱重合を阻害するという、メカニズム上の違いもあります。
PDBを検索しましたが、残念ながらX線結晶構造は見当たりませんでしたので、Cryo-EMのうち比較的新しい[PDB id : 6EW0]を眺めて見ます。

Eliburinはβ-Tublinに対して伸長軸をキャップして妨げるように結合していましたが、Paclitaxelは伸長軸に沿って側面から結合しています。
2次元および3次元構造を比較しましょう。
3次元構造はいずれもPDBから水素原子の付加されたmodelのsdfファイルを取得して描画しています。

いずれも全合成が成し遂げられているのが不思議でたまらないほど複雑な構造をとっています。
この両者の類似性をFingerprintで評価したらどのような結果になるのでしょうか??
Morgan Fingerprint (radius =2, ビット長 =2048) で類似性を評価してみます。

案の定、Tanimoto係数、Dice係数ともに 0.1 ~ 0.2 と非常に低い値となっています。
・・・まあ標的は同じタンパク質でも結合している場所全然違いますし
原子ごとの寄与率の描画では複雑な縮環部位の寄与が大きくみえるのが面白いですね。
まとめ
以上、3回にわたってRDKitをもちいた構造の描画、立体構造の取り扱いを勉強してみました。
PDB viewerでは取り扱えない、低分子リガンド構造を様々な視点から検証でき、まだまだいろいろ遊べそうです。
・・・結局コピペしかしてないので自分ではコードの書き方がわからない
jupyter notebookに打ち込みつつ記載しましたが、使い方や解釈の仕方でいろいろと間違っている部分があるかと思います。ご指摘いただければ幸いです。
先ほど向山先生の訃報を目にしました。ご冥福をお祈りします。
直接の面識はもちろんありませんが、私のような有機化学の末端員でも数々の伝説を耳にしました。
向山先生によるTaxolの全合成は英語版のWikipediaに詳しいです。
Mukaiyama Taxol total synthesis - Wikipedia
~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~
(1) ハラヴェンに関する情報
医薬品インタビューフォーム (pmdaのページ)
ハラヴェン静注1mg
KEGG
医療用医薬品 : ハラヴェン
(2) 共結晶構造
Tubulin-Eribulin complex [PDB ID: 5JH7]
Eribulin [Ligand ID : 6K9]
RCSB PDB - 5JH7: Tubulin-Eribulin complex
元の文献(open access)
Termination of Protfilament Elongation by Eribulin INduces Lattice Defects that Promote Microtubule Catastrophes.
Doodhi, H., et. al., (2016) Curr.Biol. 26: 1713-1721
https://www.cell.com/current-biology/fulltext/S0960-9822(16)30407-9?_returnURL=https%3A%2F%2Flinkinghub.elsevier.com%2Fretrieve%2Fpii%2FS0960982216304079%3Fshowall%3Dtrue
(3)「化学の新しいカタチ」さんの以下の記事を参考にさせていただきました。
参考記事① : RDKitでケモインフォマティクスに入門
参考記事② : RDKitによる3次元構造の生成
(4) RDKit ブログ / Documentation
RDKit: 3D structure of a natural product
Getting Started with the RDKit in Python — The RDKit 2017.09.1.dev1 documentation
(5) RCSB PDB
PDB ID: 5JH7
Termination of Protofilament Elongation by Eribulin Induces Lattice Defects that Promote Microtubule Catastrophes.
Doodhi, H., et. al., (2016) Curr.Biol. 26: 1713-1721
PDB ID:6EW0
Cryo-EM structure of GDP-microtubule co-polymerised with doublecortin and supplemented with Taxol
Manka, S.W., Moores, C.A. (2018) Nat. Struct. Mol. Biol. 25: 607-615
NGL Viewer
AS Rose et al. (2018) NGL viewer: web-based molecular graphics for large complexes.
Bioinformatics dio:10.1093/bioinformatics/bty419
~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~
RDKitで3次元構造に挑戦! part 2 〜HALAVEN と Halichondrin Bの構造類似性比較〜
前回の記事でHALAVEN®️(eribulin mesylate)の元になった天然物 Halichondrin B の3次元構造を見ることができました。
今回は両者の比較を行って見たいと思います。
- 1. 3次元構造の比較
- 2. 2次元構造で共通部分構造を探索(MCS)
- 3. 2次元構造で類似性を比較(FingerPrint)
- 4. フィンガープリントへの各原子の寄与
- 5. フィンガープリントのビット配列を可視化
- まとめ
1. 3次元構造の比較
まずは前回の復習を兼ねてEribulinの3次元構造を生成します。
PubChemPyを用いた検索からEribulinは "CID: 11354606"ということが分かっていますので、こちらを利用したいと思います。
import pubchempy as pcp import pandas as pd from rdkit import Chem from rdkit.Chem import AllChem, Draw from rdkit.Chem.Draw import IPythonConsole import py3Dmol #Eribulin の3次元構造の生成 (参考記事①、②) #PubChem からEribulin (CID:11354606) のisomeric smilesを取得 df_eri = pcp.get_properties('IsomericSMILES', 11354606, 'cid', as_dataframe=True) eri_iso = df_eri.loc[11354606, 'IsomericSMILES'] eri_mol = Chem.MolFromSmiles(eri_iso) eri_molH = AllChem.AddHs(eri_mol) AllChem.EmbedMolecule(eri_molH, AllChem.ETKDG()) #Halicondrin B (CID : 548895) についても同様の処理(省略) #並べて表示 v = py3Dmol.view(width=1000, height=400, linked=False, viewergrid=(1,2)) eri_molH_mb = Chem.MolToMolBlock(eri_molH) v.addModel(eri_molH_mb, viewer=(0, 0)) hali_molH_mb = Chem.MolToMolBlock(hali_molH) v.addModel(hali_molH_mb, viewer=(0, 1)) v.setBackgroundColor('0xeeeeee') v.setStyle({'stick':{}}) v.zoomTo() v.show()

Fig. 1 よりEribulin はHalichondrin B の大員環状ケトン部位を基にしていることがわかります。
ただし完全に部分構造として抜き出されたものではなく、アナログ(構造類縁体)となっています。
ではどの部分構造がが両者で一致しているのでしょうか?
2. 2次元構造で共通部分構造を探索(MCS)
またしても「化学の新しいカタチ」さんの記事を参考にさせていただき、部分構造検索を実施したいと思います。(参考記事③)
使用するのは最大共通部分構造(MCS: Maximum Common Substructure)の探索[FindMCS]です。
では、早速・・・
#Moduleのimport from rdkit.Chem import rdFMCS #時間がかかるので水素原子を付加していない構造を使用し、探索終了の時間(timeout)も設定した mcs_01 = rdFMCS.FindMCS([eri_mol, hali_mol], timeout=60) #一致した部分構造の原子数と結合の数を出力 print('Num of Atoms:{}, Num of Bonds:{}'.format(mcs_01.numAtoms, mcs_01.numBonds))
探索時間を短くするため水素原子を付加しない状態で計算しました。(・・・我慢できない大人なので)
一致した原子の数と結合の数は下記のように出力されました。
「Num of Atoms:48, Num of Bonds:54」
構造としては下記のようになります。
#MCS探索で取得した部分構造のSmartsからMolオブジェクトを作成 mol_mcs_01 = Chem.MolFromSmarts(mcs_01.smartsString) #Eribulin、Halichondrin B それぞれから共通部分構造を取得 eri_match = eri_mol.GetSubstructMatch(mol_mcs_01) hali_match = hali_mol.GetSubstructMatch(mol_mcs_01) #それぞれで一致した原子番号の一覧を表示 print('Eribulin Matched Atoms:{}\n Halichondrin B Matched Atoms:{}'.format(eri_match, hali_match)) #マッチした部分構造をハイライトして描画 Draw.MolsToGridImage([eri_mol, hali_mol], molsPerRow=2, subImgSize=(400,400), highlightAtomLists=[eri_match, hali_match])

Fig 2. 構造式の赤くハイライトされた部分がMCSで共通部分構造として認識された構造となります。
大員環ケトン部位が確かに認識されていることがわかります。
また、大員環から伸びた部分についても、5員環と6員環の違いなどには関わらず、
原子としては一致として認識されていることがわかります。
もう少し格好良く描画したい・・・ということでSVG形式の描画というのに挑戦したいと思います。(参考記事④)
「svgファイル(Scalable Vector Graphics)はXMLをベースとした2次元ベクトル型の画像ファイルフォーマット」で「拡大・縮小しても画質が損なわれることが」ない、とのことです。(上記参考記事④より)
#コンテナとなるオブジェクトの作成 view = rdMolDraw2D.MolDraw2DSVG(300,300) #描画する分子の前処理 processed_eri_mol = rdMolDraw2D.PrepareMolForDrawing(eri_mol) #コンテナの描画設定 option = view.drawOptions() option.circleAtoms=False option.continuousHighlights=False #MCSで一致した部分構造をハイライトに設定 view.DrawMolecule(processed_eri_mol, highlightAtoms=eri_match) #コンテナをファイナライズ view.FinishDrawing() #コンテナに書き込んだデータを取り出す svg = view.GetDrawingText() #データを描画 SVG(svg.replace('svg:,',''))

・・・あまり先ほどと変わらない?
どうやらFig 2. では MolsToGridImage を用いましたが、こちらはChem.rdMolDraw2Dモジュールを使っているらしいので裏側で動いている仕組みが同じであるようです。
比較のためMolToImageで描画すると以下のようになります。
#比較用にMolToImageとMolsToGridImageでそれぞれ描画 Draw.MolToImage(eri_mol, highlightAtoms=eri_match) Draw.MolsToGridImage([eri_mol], molsPerRow=1, subImgSize=(400,400), highlightAtomLists=[eri_match])

確かにMolToImageよりもMolsToGrideImageの方が綺麗です。
ついでなのでもう少し、凝った描画に挑戦したいと思います。
#matplotlibによるRGBカラーコードの生成と色の指定 from matplotlib.colors import ColorConverter color_1 = {} color_2 = {} radius = {} for i, j in zip(eri_match, hali_match): color_1[i] = ColorConverter().to_rgb('skyblue') radius[i] = 0.5 color_2[j] = ColorConverter().to_rgb('khaki') radius[j] = 0.5 processed_eri_mol = rdMolDraw2D.PrepareMolForDrawing(eri_mol) processed_hali_mol = rdMolDraw2D.PrepareMolForDrawing(hali_mol) view2 = rdMolDraw2D.MolDraw2DSVG(1000, 300, 500, 300) #フォントサイズの設定 view2.SetFontSize(2*view2.FontSize()) option = view2.drawOptions() #多重結合の感覚を指定 option.multipleBondOffset = 0.2 #描画領域のマージンを設定 option.padding=0.13 #レジェンドのフォントサイズを設定 option.legendFontSize=18 view2.DrawMolecules([processed_eri_mol, processed_hali_mol], highlightAtoms=[eri_match, hali_match], highlightAtomColors=[color_1, color_2], highlightAtomRadii=[radius, radius], highlightBonds=[[],[]], legends=['Eribulin', 'Halichondrin B']) view2.FinishDrawing() svg = view2.GetDrawingText() SVG(svg.replace('svg:', ''))

原子ラベルのサイズなどを変更することで、かなり分かりやすい図になったように思います。
3. 2次元構造で類似性を比較(FingerPrint)
共通部分構造にもとづく比較を行ったので、次は新たな手法、
フィンガープリントを用いた類似性の検証に挑戦したいと思います。(参考記事⑤)
違いがわからないのでとりあえず全部計算! ・・・アホ丸出し
#比較する二つをまとめる mols = [eri_mol, hali_mol] #①MACCS Keys from rdkit import DataStructs maccs_fps = [AllChem.GetMACCSKeysFingerprint(mol) for mol in mols] maccs_Tanimoto = DataStructs.TanimotoSimilarity(maccs_fps[0], maccs_fps[1]) maccs_Dice = DataStructs.DiceSimilarity(maccs_fps[0], maccs_fps[1]) print('MACCS Keys\nTanimoto Similarity:{}, Dice Similarity:{}'.format(maccs_Tanimoto, maccs_Dice)) #②Topological フィンガープリント(RDKitフィンガープリント) from rdkit.Chem.Fingerprints import FingerprintMols rdkit_fps = [Chem.Fingerprints.FingerprintMols.FingerprintMol(mol) for mol in mols] rdkit_Tanimoto = DataStructs.TanimotoSimilarity(rdkit_fps[0], rdkit_fps[1]) rdkit_Dice = DataStructs.DiceSimilarity(rdkit_fps[0], rdkit_fps[1]) print('Topological Fingerprint\nTanimoto Similarity:{}, Dice Similarity:{}'.format(rdkit_Tanimoto, rdkit_Dice)) #③Morganフィンガープリント-1 ECFP類似 morgan_ECFP_fps = [AllChem.GetMorganFingerprint(mol, 2) for mol in mols] morgan_ECFP_Tanimoto = DataStructs.TanimotoSimilarity(morgan_ECFP_fps[0], morgan_ECFP_fps[1]) morgan_ECFP_Dice = DataStructs.DiceSimilarity(morgan_ECFP_fps[0], morgan_ECFP_fps[1]) print('Morgan Fingerprint(ECFP-like)\nTanimoto Similarity:{}, Dice Similarity:{}'.format(morgan_ECFP_Tanimoto, morgan_ECFP_Dice)) #③'Morganフィンガープリント-1 ECFP類似 (bit vector) morgan_ECFP_bit_fps = [AllChem.GetMorganFingerprintAsBitVect(mol, 2, 2048) for mol in mols] morgan_ECFP_bit_Tanimoto = DataStructs.TanimotoSimilarity(morgan_ECFP_bit_fps[0], morgan_ECFP_bit_fps[1]) morgan_ECFP_bit_Dice = DataStructs.DiceSimilarity(morgan_ECFP_bit_fps[0], morgan_ECFP_bit_fps[1]) print('Morgan Fingerprint(ECFP-like/bit vector)\nTanimoto Similarity:{}, Dice Similarity:{}'.format(morgan_ECFP_bit_Tanimoto, morgan_ECFP_bit_Dice)) #④Morganフィンガープリント-2 FCFP類似 morgan_FCFP_fps = [AllChem.GetMorganFingerprint(mol, 2, useFeatures=True) for mol in mols] morgan_FCFP_Tanimoto = DataStructs.TanimotoSimilarity(morgan_FCFP_fps[0], morgan_FCFP_fps[1]) morgan_FCFP_Dice = DataStructs.DiceSimilarity(morgan_FCFP_fps[0], morgan_FCFP_fps[1]) print('Morgan Fingerprint(FCFP-like)\nTanimoto Similarity:{}, Dice Similarity:{}'.format(morgan_FCFP_Tanimoto, morgan_FCFP_Dice)) #⑤Avalon フィンガープリント from rdkit.Avalon import pyAvalonTools avalon_fps = [pyAvalonTools.GetAvalonFP(mol) for mol in mols] avalon_Tanimoto = DataStructs.TanimotoSimilarity(avalon_fps[0], avalon_fps[1]) avalon_Dice = DataStructs.DiceSimilarity(avalon_fps[0], avalon_fps[1]) print('Avalon Fingerprint\nTanimoto Similarity:{}, Dice Similarity:{}'.format(avalon_Tanimoto, avalon_Dice)) #⑥AtomPair フィンガープリント #公式のDocumentationによるとatom-pairにはDice similarityを使うのが通常らしい from rdkit.Chem.AtomPairs import Pairs pair_fps = [Pairs.GetAtomPairFingerprint(mol) for mol in mols] pair_Tanimoto = DataStructs.TanimotoSimilarity(pair_fps[0], pair_fps[1]) pair_Dice = DataStructs.DiceSimilarity(pair_fps[0], pair_fps[1]) print('AtomPair Fingerprint\nTanimoto Similarity:{}, Dice Similarity:{}'.format(pair_Tanimoto, pair_Dice)) #⑥' AtomPair フィンガープリント(bit vector fingerprint) pair_bit_fps =[Pairs.GetAtomPairFingerprintAsBitVect(mol) for mol in mols] pair_bit_Tanimoto = DataStructs.TanimotoSimilarity(pair_bit_fps[0], pair_bit_fps[1]) pair_bit_Dice = DataStructs.DiceSimilarity(pair_bit_fps[0], pair_bit_fps[1]) print('AtomPair Fingerprint (bit vector)\nTanimoto Similarity:{}, Dice Similarity:{}'.format(pair_bit_Tanimoto, pair_bit_Dice)) #⑦Torsion フィンガープリント #bitが多すぎるのでこっちはAsBitVectは存在しないらしい from rdkit.Chem.AtomPairs import Torsions tts_fps = [Torsions.GetTopologicalTorsionFingerprintAsIntVect(mol) for mol in mols] tts_Tanimoto = DataStructs.TanimotoSimilarity(tts_fps[0], tts_fps[1]) tts_Dice = DataStructs.DiceSimilarity(tts_fps[0], tts_fps[1]) print('Torsion Fingerprint\nTanimoto Similarity:{}, Dice Similarity:{}'.format(tts_Tanimoto, tts_Dice)) #結果をPandasのデータフレームにまとめる import pandas as pd data = {"MACCS keys":{"Tanimoto": maccs_Tanimoto, "Dice":maccs_Dice}, "Topological FP":{"Tanimoto": rdkit_Tanimoto, "Dice":rdkit_Dice}, "Morgan FP(ECFP-like)":{"Tanimoto": morgan_ECFP_Tanimoto, "Dice":morgan_ECFP_Dice}, "Morgan FP(ECFP-like/bit vector)":{"Tanimoto": morgan_ECFP_bit_Tanimoto, "Dice":morgan_ECFP_bit_Dice}, "Morgan FP(FCFP-like)":{"Tanimoto": morgan_FCFP_Tanimoto, "Dice":morgan_FCFP_Dice}, "Avalon FP":{"Tanimoto": avalon_Tanimoto, "Dice":avalon_Dice}, "AtomPair FP":{"Tanimoto": pair_Tanimoto, "Dice":pair_Dice}, "AtomPair FP(bit vector)":{"Tanimoto": pair_bit_Tanimoto, "Dice":pair_bit_Dice}, "Torsion FP":{"Tanimoto": tts_Tanimoto, "Dice":tts_Dice}, } FP_df = pd.DataFrame(data).T
計算結果は以下のような感じ・・・・

Tanimoto係数とJaccard係数は(ほぼ)同じ・・・?
今回計算した7つのフィンガープリント全てで 「Dice 係数 > Tanimoto係数」となっています。
定義上、(Simpson ≧) Dice ≧ Tanimoto(Jaccard) となるようです。
EribulinとHalichondrin B の Fingerprintをそれぞれ a, b としてみると、
・Eribulinのほぼ全体が共通部分に含まれるので 「a ≒ c」 より 「Tanimoto係数 ≒ c / b」
・EribulinはHalichondrin B の(ほぼ)部分構造なので 「a < b」として 「Dice係数 > {2c / (b + b) } = c/b 」
と考えてみても、まあ、なるほどそうなるかなあ・・・という感じです。
ところで構造式のみをみると、共通部分構造(c)は8割くらいの構造を占めているので、
係数は全て0.8程度にはなるのではないかと思いましたが、意外と0.5前後の値もあります。
特にAtomPairやMorgan フィンガープリントはそこまで大きな値ではないのが気になります。
これらのフィンガープリントでは構造のどのあたりを考慮して計算されているのでしょうか?
各原子の寄与を可視化する機能がある、とのことなので試して見たいと思います。
4. フィンガープリントへの各原子の寄与
では早速AtomPairから・・・参照分子をEribulin、描画する分子をHalicondrin B として可視化します。
from rdkit.Chem.Draw import SimilarityMaps #AtomPair(規格化) fig1, maxweight = SimilarityMaps.GetSimilarityMapForFingerprint(eri_mol, hali_mol, SimilarityMaps.GetAPFingerprint) #AtomPair(ウェイトの絶対値を利用) weight = SimilarityMaps.GetAtomicWeightsForFingerprint(eri_mol, hali_mol, SimilarityMaps.GetAPFingerprint, metric=DataStructs.DiceSimilarity) fig2 = SimilarityMaps.GetSimilarityMapFromWeights(hali_mol, weight, size=(400, 400))

規格化も絶対値もほぼ同一のようにみえるので、sizeの設定可能な絶対値を利用して行きたいと思います。
次にTorsionとMorgan Fingerprint、Topological Fingerprint(RDKit Fingerprint)を見て見たいと思います。
#Torsionフィンガープリント weight = SimilarityMaps.GetAtomicWeightsForFingerprint(eri_mol, hali_mol, SimilarityMaps.GetTTFingerprint, metric=DataStructs.DiceSimilarity) fig3 = SimilarityMaps.GetSimilarityMapFromWeights(hali_mol, weight, size=(400, 400)) #Morganフィンガープリント(デフォルトは[radius=2, fpType='bv', useFeatures=False]なので ECFP-like/bit vector に対応) weight = SimilarityMaps.GetAtomicWeightsForFingerprint(eri_mol, hali_mol, SimilarityMaps.GetMorganFingerprint, metric=DataStructs.DiceSimilarity) fig4 = SimilarityMaps.GetSimilarityMapFromWeights(hali_mol, weight, size=(400, 400)) #Topological フィンガープリント (RDKitFingerprint) weight = SimilarityMaps.GetAtomicWeightsForFingerprint(eri_mol, hali_mol, SimilarityMaps.GetRDKFingerprint, metric=DataStructs.DiceSimilarity) fig5 = SimilarityMaps.GetSimilarityMapFromWeights(hali_mol, weight, size=(400, 400))

下に行くほど分子全体が緑色となっています。
Dice係数はMorgan Fingerprint よりもTorsion Fingerprintのほうが大きいですが、前者の方が全体的に緑色となっています。
また、RDKit フィンガープリントではHalichondrin B のうち Eribulinと重ならない大員環ケトン外部も緑色となっています。
これらは単純に構造に落とし込めるわけではなさそうです。
そこで最後にビット配列の可視化を行っておきたいと思います。
5. フィンガープリントのビット配列を可視化
それではMorganフィンガープリントおよび RDKit フィンガープリントのビット配列を眺めて見たいと思います。(参考記事⑥)
#Morgan フィンガープリント (Eribulin) bitI_morgan_eri = {} fp_morgan_eri = AllChem.GetMorganFingerprintAsBitVect(eri_mol, 2, bitInfo=bitI_morgan_eri) print(len(bitI_morgan_eri)) ###80 morgan_tuples_eri = ((eri_mol, bit, bitI_morgan_eri) for bit in list(bitI_morgan_eri.keys())[:12]) Draw.DrawMorganBits(morgan_tuples_eri, molsPerRow=6, legends=['bit: '+str(x) for x in list(bitI_morgan_eri.keys())[:12]]) #Morgan フィンガープリント (Halicondrin B) bitI_morgan_hali = {} fp_morgan_hali = AllChem.GetMorganFingerprintAsBitVect(hali_mol, 2, bitInfo=bitI_morgan_hali) print(len(bitI_morgan_hali)) ###93 morgan_tuples_hali = ((hali_mol, bit, bitI_morgan_hali) for bit in list(bitI_morgan_hali.keys())[:12]) Draw.DrawMorganBits(morgan_tuples_hali, molsPerRow=6, legends=['bit: '+str(x) for x in list(bitI_morgan_hali.keys())[:12]])
Eribulin でが全部で80個、Halichondrin B では全部で93個のMorgan フィンガープリントがONとなっています。
最初の12個の0でないビットについて両者を比較すると下図のようになります。
それぞれ枠で囲んだ部分が差異となります。

同様にRDKitフィンガープリントでも実施します。
#RDKit フィンガープリント (Eribulin) bitI_RDK_eri = {} fp_RDK_eri = Chem.RDKFingerprint(eri_mol, bitInfo=bitI_RDK_eri) print(len(bitI_RDK_eri)) ###779 RDK_tuples_eri = ((eri_mol, bit, bitI_RDK_eri) for bit in list(bitI_RDK_eri.keys())[:12]) Draw.DrawRDKitBits(RDK_tuples_eri, molsPerRow=6, legends=['bit: '+str(x) for x in list(bitI_RDK_eri.keys())[:12]]) #RDKit フィンガープリント (Halichondrin B) bitI_RDK_hali = {} fp_RDK_hali = Chem.RDKFingerprint(hali_mol, bitInfo=bitI_RDK_hali) print(len(bitI_RDK_hali)) ###753 RDK_tuples_hali = ((hali_mol, bit, bitI_RDK_hali) for bit in list(bitI_RDK_hali.keys())[:12]) Draw.DrawRDKitBits(RDK_tuples_hali, molsPerRow=6, legends=['bit: '+str(x) for x in list(bitI_RDK_hali.keys())[:12]])

Eribulin でが全部で779個、Halichondrin B では全部で753個のMorgan フィンガープリントがONとなっています。
興味深いことにRDKit フィンガープリントではHalichondrin Bの方がONになっているフィンガープリントの数が少なくなっています。
最初の12個の0でないビットについて両者を比較すると下図のようになります。
エステル部位が差異となります。
まとめ
以上、今回はEribulinとHalichondrin B について、その構造の比較、類似性の検証をRDKitの様々な指標計算、描画をもちいて行いました。
単純に構造式を目でみて判断するのとは異なった視点で検証ができ非常に興味深い結果でした。
コピペで手一杯で、裏側でどのような計算が行われているかについては全く踏み込めませんでしたが、これはこれで良しとしていきたい!!
いつか勉強する・・・多分・・・
追記
Twitter で より立体を反映した2次元構造の描写方法(rdDepictor.SetPreferCoordGen )を教えていただきました。
Nice work :) 話の本筋から逸れますが rdDepictor.SetPreferCoordGen
— yamasaKit_ (@yamasaKit_) 2018年11月25日
を使うともう少し綺麗な描写になりますよ。それでも少し変ですがw pic.twitter.com/ZJh4418gSU
@yamasaKit_ 先生ありがとうございました!
忘れてしまいそうなのでコードを転記させていただきます。
from rdkit import Chem, rdBase from rdkit.Chem import rdDepictor from rdkit.Chem.Draw import IPythonConsole # True or False で設定変更 rdDepictor.SetPreferCoordGen(True) eri_mol # isomeric smilesから生成したオブジェクトを使用

元々の描画(右)では、大きな丸となっており環員数もわかりづらくなっていましたが、
「rdDepictor.SetPreferCoordGen」(左)を用いると、エッジがはっきりしてよりマニュアルの描画に近い印象になりました。
こちらも使っていこう
rdkit.Chem.rdDepictor module — The RDKit 2018.09.1 documentation
~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~
上記記事の作成にあたって「化学の新しいカタチ」の以下の記事を参考にさせていただきました。
また、本記事の中に誤りがある場合、当ブログ作成者の責任です。
参考記事① : 化合物データベースPubChemをpythonで使いこなす
参考記事②: RDKitによる3次元構造の生成
参考記事③: RDKitを用いた部分構造検索とMCSアルゴリズム
参考記事④: RDKitでの構造式描画を詳しく解説
参考記事⑤: RDKitでフィンガープリントを使った分子類似性の判定
参考記事⑥: RDKitでフィンガープリントの可視化
RDKit documentation① 構造の描画
rdkit.Chem.Draw package — The RDKit 2018.09.1 documentation
RDKit documentation② MACCS Keys
http://www.rdkit.org/docs/GettingStartedInPython.html#maccs-keys
RDKit documentation③ Topological Fingerprints
http://www.rdkit.org/docs/GettingStartedInPython.html#topological-fingerprints
RDKit documentation④ Morgan Fingerprints
http://www.rdkit.org/docs/GettingStartedInPython.html#morgan-fingerprints-circular-fingerprints
RDKit documentation⑤Avalon フィンガープリント
http://rdkit.org/docs/source/rdkit.Avalon.pyAvalonTools.html
RDKit documentation⑥Similarity Maps using Fingerprints
https://www.rdkit.org/docs/GettingStartedInPython.html#generating-similarity-maps-using-fingerprints
集合類似度(係数)に関して参考にしたページ
https://www.conectron-networks.com/conectron/52
https://mieruca-ai.com/ai/jaccard_dice_simpson/
~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~
RDKitで3次元構造に挑戦!part 1 〜HALAVEN (Eribulin mesylate)を題材に〜
以前の記事で少し取り上げたHalaven (Eliburin)・・・
私は構造式をみても全く3次元的な構造がイメージできません。
ところで、ケモインフォマティクス について調べていると度々目にする"RDKit"。
どうやらタダで3Dの描画もできる様です。
さらには日本語で使い方を解説してくださっている素晴らしいブログ(↓)もあります。
future-chem.com
これは入門するしかない!
ということでRDKit に入門(コピペ)することにしました。。
備忘録を兼ねてコードを貼りますが主に上記ページの引用です。
Pythonも初心者なので怪しい記述がたくさん出てきます。ご容赦ください。
最終的には
1. Eribulin と 元になった天然物 Halichondrin B の比較
2. RDKitで出した構造と結晶構造の比較
をしてみたいのですが、とりあえずはHalichondrin B の描画に挑戦しました。
とりあえず、分かったことは
1. SMILES にもCanonical やら Isomeric やら色々ある
2. 立体構造の描画にプロトンを無視してはいけない
です。
(1) PubChemからの情報取得 (参考記事①)
まず、PubChemから化合物の構造に関する情報を取得します。
Halichondrin B と Eribulin の情報をそれぞれpandasのdataframeとして取得しました。
#PubChemPyをpcpとしてimport import pubchempy as pcp #列が多すぎるので全列表示するようにpandasを設定する import pandas as pd pd.set_option('display.max_columns', None) #Halichondrin B とEribulinを名前検索で情報取得 df_hali = pcp.get_compounds('Halichondrin B', 'name', as_dataframe=True) df_eri = pcp.get_compounds('Eribulin', 'name', as_dataframe=True)
取得される情報は右に記載されていました。API documentation — PubChemPy 1.0.4 documentation
| cid | PubChem Compound Identifier | elements | 含まれる元素記号のリスト |
|---|---|---|---|
| atoms | 原子のリスト | bonds | 結合のリスト |
| synonyms | 別名 | sids | PubChem Substance Identifier |
| aids | PubChem Assay Identifier | charge | 形式電荷 |
| molecular_formula | 分子式 | molecular_weight | 分子量 |
| canoical_smiles | Canoniacal SMILES | isomeric_smiles | Isomeric SMILES |
| inchi | InChI string | inchikey | InChIKey |
| iupac_name | IUPAC name | xlogp | XLogP |
| exact_mass | Exact mass | monoisotopic_mass | Monoisotopic mass |
| tpsa | Topological Polar Surface Area | complexity | Complexity |
| h_bond_donor_count | 水素結合ドナー数 | h_bond_acceptor_count | 水素結合アクセプター数 |
| rotatable_bond_count | Rotatable bond count | fingerprint | hex-encoded fingerprint |
| cactvs_fingerprint | PubChem CACTVS fingerprint | heavy_atom_count | Heavy atom count |
| isotope_atom_count | isotope atom count | atom_stereo_count | Atom stereocenter count |
| defined_atom_stereo_count | Defined atom stereocenter count | undefined_atom_stereo_count | Undefined atom stereocenter count |
| bond_stereo_count | Bond stereocenter count | defined_bond_stereo_count | Defined bond stereocenter count |
| undefined_bond_stereo_count | Undefined bond stereocenter count | covalent_unit_count | Covalently-bonded unit count |
"英語のまま = 意味がわからないからコピペした"
Halichondrin B はエントリーが7つでてきますが、どれも同じようなのでWikipediaにものっている”CID(Compound ID) : 5488895" を使用することにします。
#どうせWikipediaのCIDつかうならこうすりゃよかった。 pcp.get_compounds(5488895, 'cid', as_dataframe=True)
Eribulin は" CID: 11354606" のみでした。
Wikipedia にはメシル酸塩 "CID: 17755248" が記載されていますが、こちらは出てきませんでした。
名前検索は部分一致ではなく、全体一致なのでしょうか?
(2) Canonical SMILES からの2次元描画 ( 参考記事② )
次にHalichondrin B の Canonical SMILESから2次元構造を描画してみます。
#必要なものをimport from rdkit import Chem from rdkit.Chem import AllChem, Draw #取得したDataFrameからCanonical SMILESの情報を取り出す。 hali_smi_cano = df_hali.loc[5488895, "canonical_smiles"] #smilesからMolオブジェクトを作成 hali_mol_cano = Chem.MolFromSmiles(hali_smi_cano) #2次元座標の計算 AllChem.Compute2DCoords(hali_mol_cano) #2次元描画 Chem.Draw.MolToImage(hali_mol_cano)
できました!!

なるほど・・・そうきましたか。
流石にこういう感じ(↓)を自動生成は無理ですよね。
 ハリコンドリンB - Wikipedia
ハリコンドリンB - Wikipedia
上記は水素原子が省かれているのでつけてみます。
#プロトンをつけて描画
hali_mol_canoH = AllChem.AddHs(hali_mol_cano)
AllChem.Compute2DCoords(hali_mol_canoH)
Chem.Draw.MolToImage(hali_mol_canoH)

ぞわぞわする・・・サクサク行こう。
(3) 3次元構造を生成・描画 (参考記事③)
では、本題の3次元構造を作ってみましょう。
まずは見やすそうなので、そのまま"水素原子なし"でやって見ます。
#必要なものをimport from rdkit.Chem.Draw import IPythonConsole import py3Dmol #3次元構造を生成 AllChem.EmbedMolecule(hali_mol_cano, AllChem.ETKDG()) #Jupyter notebook上で描画 IPythonConsole.drawMol3D(hali_mol_cano)

おおおお!格好良い!これだ、これが見たかった!
Jupyter notebookでは回転とズームもできます!(上は画像なので無理です)
"水素原子あり"も作って、二つ並べて見ましょう。
#プロトン付きでも3次元構造を生成 AllChem.EmbedMolecule(hali_mol_canoH, AllChem.ETKDG()) #並べて表示するためにpy3Dmolを使用 #まずはviewオブジェクトを生成 v = py3Dmol.view(width=1000, height=400, linked=False, viewergrid=(1,2)) # "プロトンあり"と"なし"をそれぞれMolBlock形式としてグリッドに渡す。 hali_mol_cano_mb = Chem.MolToMolBlock(hali_mol_cano) v.addModel(hali_mol_cano_mb, viewer=(0, 0)) hali_mol_canoH_mb = Chem.MolToMolBlock(hali_mol_canoH) v.addModel(hali_mol_canoH_mb, viewer=(0, 1)) #格好良くする設定 v.setBackgroundColor('0xeeeeee') v.setStyle({'stick':{}}) v.zoomTo() v.show()

" linked=False " と設定しているので左と右それぞれの別々に動かせます。
うひょーっ・・・ん? なんか変。

なんと、水素あり、水素なしで立体が変わってしまっています。
水素付加の仕方に問題があるのでしょうか??
いろいろ調べてみると気になる記述が・・・SMILESにも色々種類があるそうです。
・generic SMILES: 原子と結合のみを記述
・isomeric SMILES: 同位体や不斉中心についての記述を含む
・canonical SMILES:generic SMILESをある定義に従って一義的に作成したもの言う
ESI友の会 - 2. SMILESとメタボロミクス
Dr Tomさんも別の記事でSMILES記法について解説してくださっていました。 (参考記事④)
今回、立体構造を生成するにあたって使用したのはcanonical SMILESですが、こちらには不斉中心の情報が含まれていないとのこと。
立体の情報をもたない線形表記法から図を作成したことに問題がありそうです。
ならば、isomeric SMILESでやって見ましょう。
(4) isomeric SMILES からの描画
PubChemから取得した情報にisomeric SMILESもあったので、Halichondrin B の構造をisomeric SMILESからおこしたいと思います。
まずは2次元。
#取得したDataFrameからisomeric SMILESの情報を取り出す。 hali_smi_iso = df_hali.loc[5488895, "isomeric_smiles"] #smilesからMolオブジェクトを作成し、座標の計算と描画 hali_mol_iso = Chem.MolFromSmiles(hali_smi_iso) AllChem.Compute2DCoords(hali_mol_iso) Chem.Draw.MolToImage(hali_mol_iso)

おお!立体を認識してそうです。
ちなみにそれぞれ線形表記は以下のようになります。@で不斉点を表している模様・・・
| canonical SMILES | CC1CC2CCC3C(=C)CC(O3)CCC45CC6C(O4)C7C(O6)C(O5)C8C(O7)CCC(O8)CC(=O)OC9C(C3C(CC4C(O3)CC3(O4)CC4C(O3)C(CC3(O4)CC(C4C(O3)CC(O4)C(CC(CO)O)O)C)C)OC9CC(C1=C)O2)C |
|---|---|
| isomeric SMILES | C[C@@H]1C[C@@H]2CC[C@H]3C(=C)C[C@@H](O3)CC[C@]45C[C@@H]6[C@H](O4)[C@H]7[C@@H](O6)[C@@H](O5)[C@@H]8[C@@H](O7)CC[C@@H](O8)CC(=O)O[C@@H]9[C@H]([C@H]3[C@H](C[C@@H]4[C@H](O3)C[C@@]3(O4)C[C@H]4[C@@H](O3)[C@H](C[C@]3(O4)C[C@@H]([C@H]4[C@@H](O3)C[C@H](O4)[C@H](C[C@H](CO)O)O)C)C)O[C@H]9C[C@H](C1=C)O2)C |
それでは、3次元構造をつくりましょう。
今回も"水素なし"と"水素あり"でやってみます。
#水素原子なしの3次元構造生成 AllChem.EmbedMolecule(hali_mol_iso, AllChem.ETKDG()) #水素原子を付加し3次元構造を生成 hali_mol_isoH = AllChem.AddHs(hali_mol_iso) AllChem.EmbedMolecule(hali_mol_isoH, AllChem.ETKDG()) #並べて表示 v = py3Dmol.view(width=1000, height=400, linked=False, viewergrid=(1,2)) hali_mol_iso_mb = Chem.MolToMolBlock(hali_mol_iso) v.addModel(hali_mol_iso_mb, viewer=(0, 0)) hali_mol_isoH_mb = Chem.MolToMolBlock(hali_mol_isoH) v.addModel(hali_mol_isoH_mb, viewer=(0, 1)) v.setBackgroundColor('0xeeeeee') v.setStyle({'stick':{}}) v.zoomTo() v.show()

水素を付加していない構造(左)は平面になってしまいました。
無駄に回転可能です・・・これはこれで嫌いじゃない!
水素付加したもの(右)は望み通り3次元構造となりました。
以上をまとめると、不斉中心が重要な医薬品に関しては
・立体情報を含むisomeric SMILESを使用
・3次元構造は水素を付加してから生成
と、するのが良さそうです。
次回からは関数っぽくして使おう・・・
import pubchempy as pcp import pandas as pd from rdkit import Chem from rdkit.Chem import AllChem, Draw from rdkit.Chem.Draw import IPythonConsole import py3Dmol def rittai(PubChemCID): #PubChemのCIDを別途用意して代入 #CIDからDataFrameとして化合物の情報を得る CID_df = pcp.get_compounds(PubChemCID, 'cid', as_dataframe=True) #DataFrameからisomeric smilesを抜き出す iso_smi = CID_df.loc[PubChemCID, 'isomeric_smiles'] #Molオブジェクトに変換 iso_mol = Chem.MolFromSmiles(iso_smi) #水素原子を付加 iso_molH = AllChem.AddHs(iso_mol) #3次元構造を生成 AllChem.EmbedMolecule(iso_molH, AllChem.ETKDG()) #Jupyter notebookで描画 IPythonConsole.drawMol3D(iso_molH)
それにしても初心者でもコピペで使えるとはDr Tom先生の記事クオリティ高すぎです。

*********************************************************************
上記記事の作成にあたって「化学の新しいカタチ」の以下の記事を参考にさせていただきました。
また、本記事の中に誤りがある場合、当ブログ作成者の責任です。
参考記事① : 化合物データベースPubChemをpythonで使いこなす
参考記事② : RDKitの分子Molオブジェクトを扱う
参考記事③: RDKitによる3次元構造の生成
参考記事④: SMILES記法は化学構造の線形表記法
********************************************************************
cootで電子密度を眺める

前回、結晶の質を眺めるにあたってPDB viewer を使用しましたが、cootというソフトウェアをご紹介いただきました。
cootで電子密度見ましょう!リガンドの化学構造が誤っている部分は、電子密度もけっこうプアでした。
— torusengoku (@torusengoku) 2018年11月13日
Sengoku先生、ありがとうございました!
早速導入(無料!)
以下のページからダウンロードしてきました。
Stand-Alone Coot - OS X Scientific Computing
オフィシャルHPはこちら
→ https://www2.mrc-lmb.cam.ac.uk/personal/pemsley/coot/
ターミナルで 「coot」と打ち込むと無事立ち上がりました。
ソフトを問題なくインストールできたのは初めてかも・・・
インターネット上でみつけたtutorialを参考に、課題の構造 [PDB id: 5NIX ]を眺めてみました。
tutorial 1 → Coot Tutorial X-ray Methods, CSHL 2016 Dec. 18, 2017 (pdf)
tutorial 2 → X線結晶構造解析における構造バイオインフォマティクス (pdf)
- 1. 設定の変更
- 2. ファイルの準備
- 3.ファイルの読み込み
- 4. 構造のおかしな場所を簡単に探す
- 5. アミノ酸側鎖の補正
- 6. リガンド部位の確認
- 7. validationのグラフ
- 8. 別の構造でも確認(PDB id: 5NIU)
1. 設定の変更
tutorial 1 のオススメに従い設定を変更。
① 分子の動かし方 "virtual trackball" (2.2章)
・"Sperical Surface" → "Flat" modeへ変更
② 動きを加速するための設定 (2.4章)
・ Preferences から "Active Map on Draggeing" を "No" へ変更
・"Smooth Recentering" の "Number of Steps" を 40 から 20 へ変更
③ 注目している原子の周辺の原子との距離を表示 (2.4章)
"Measures" の "Environment Distances" で
・"Show Residue Environment?"
・ "Label Atom?"
を表示させるように変更
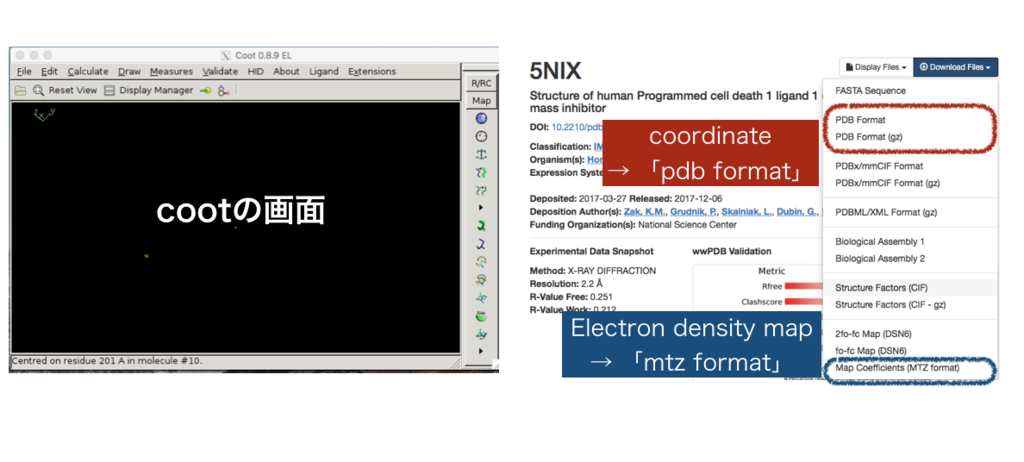
2. ファイルの準備
・原子座標ファイル(coordinate)・・・「PDB format」
・X線回折データ (電子密度)・・・「Map Coeficients (MTZ format)」
を取得しました。
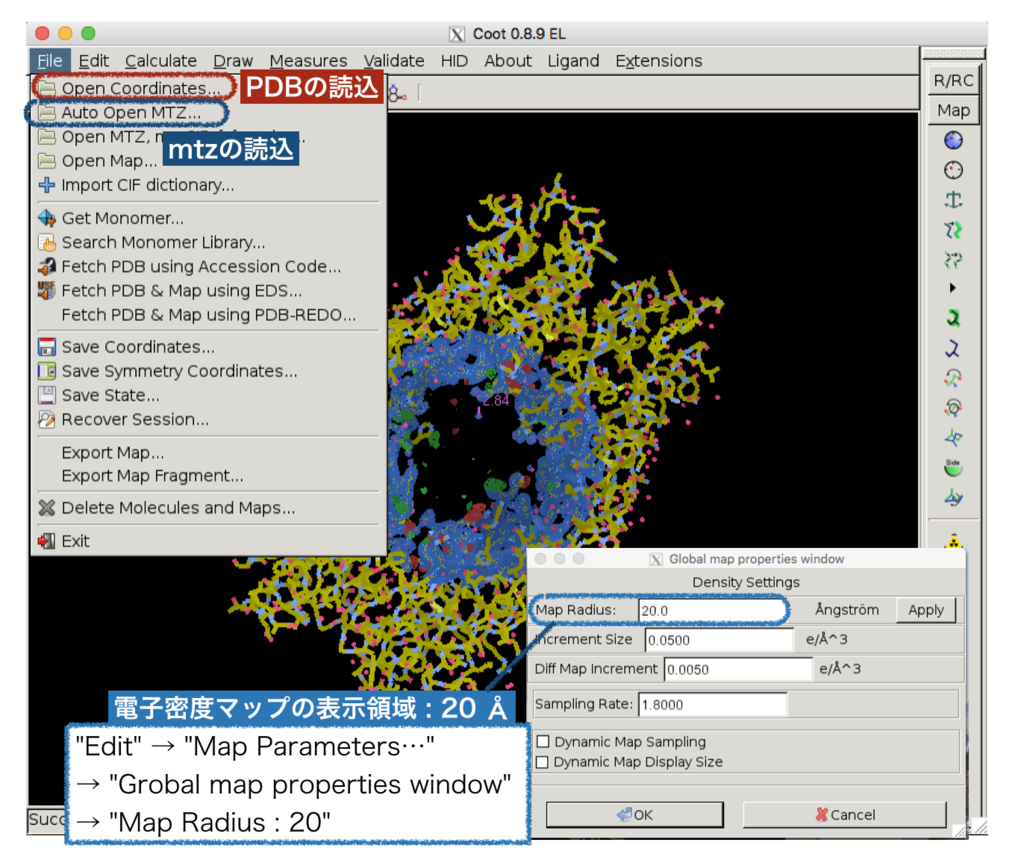
3.ファイルの読み込み
File の [Open Coordinates…] から PDB format を、[Auto Open MTZ…]から mtz format を読み込みます。
電子密度マップの表示領域を半径 20Åとすると以下のような図が得られました。(Fig. 2)
電子密度の色はそれぞれ、
・青 : 2Fo - Fc マップ → 電子の存在位置
・赤 : Fo - Fc マップ(正)→ 本来電子密度がないはずなのに構造が置かれている
・緑 : Fo - Fc マップ(負)→ 本来電子密度があるはずなのに構造が置かれていない
を表すそうで、つまり赤と緑がモデルのまずい場所だそうです。
4. 構造のおかしな場所を簡単に探す
電子密度の色の違いはわかりましたが、アミノ酸残基一つ一つを確認するのは面倒です。
Coot の 「Density Fit Analysis」をもちいると、残基ごとのモデルの良し悪しを簡便に確認可能でした。
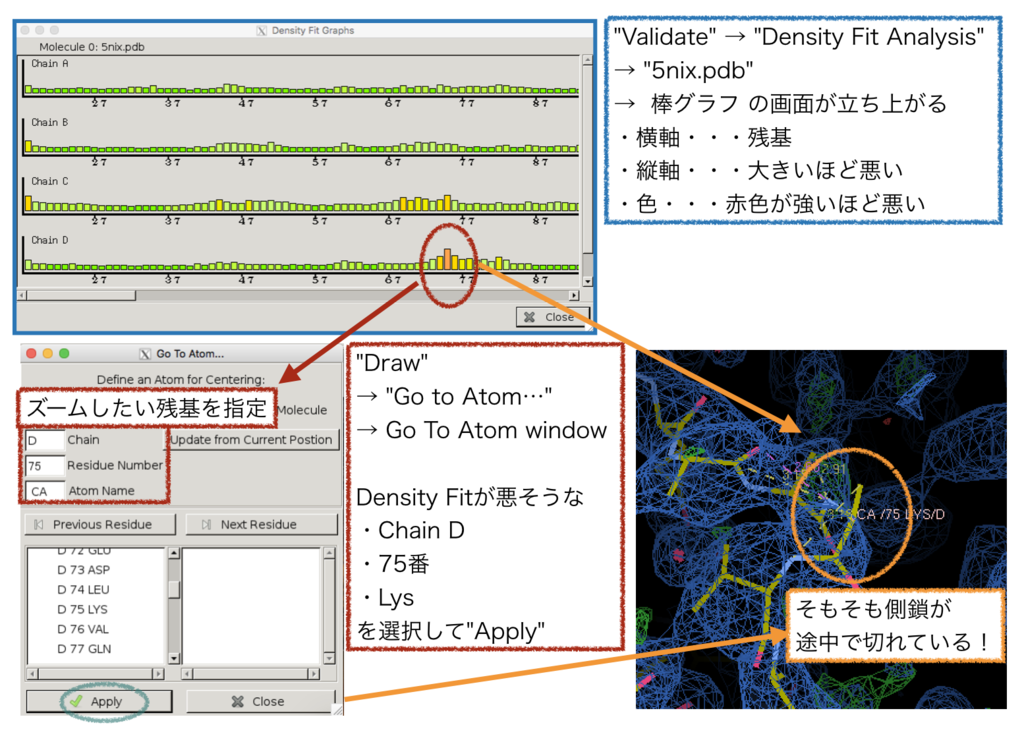
chain D の 残基番号 75 Lys が「Density Fit Analysis」でもっともFit が悪そうです。
確認したい残基の棒グラフをクリックすると、自動的に目的のアミノ酸残基がズームされます。また、「Go To Atom…」機能をもちいても、残基(原子)を指定したズームが可能です。
どうやらLys 側鎖に電子密度が見えておらず、そもそも側鎖もモデル化されていないようです。
途中で切れてる・・・
他にもChain A で Fit の悪い 82 ARG などでも側鎖が途中で切れており、モデル化されていませんでした。
長く、自由度の高いアミノ酸残基が特に溶媒露出面に位置している場合、電子密度がうまく観測されずモデル化も難しい、という感じでしょうか??
5. アミノ酸側鎖の補正
Chain A : 82 : ARG は側鎖の延長線上に緑色の電子密度があります。緑色は「構造が置かれていない電子密度」なので、試しに側鎖を伸ばしてみたいと思います。
「Mutate & Auto Fit…」機能を利用することで簡単に側鎖を伸ばすことができました。

「Rotamers」機能を使うと側鎖の取りうるRotamerの候補が出てきますが、今回は「Mutate & Auto Fit …」で導入したconformerが電子密度と最も重なりが良さそうに見えます。
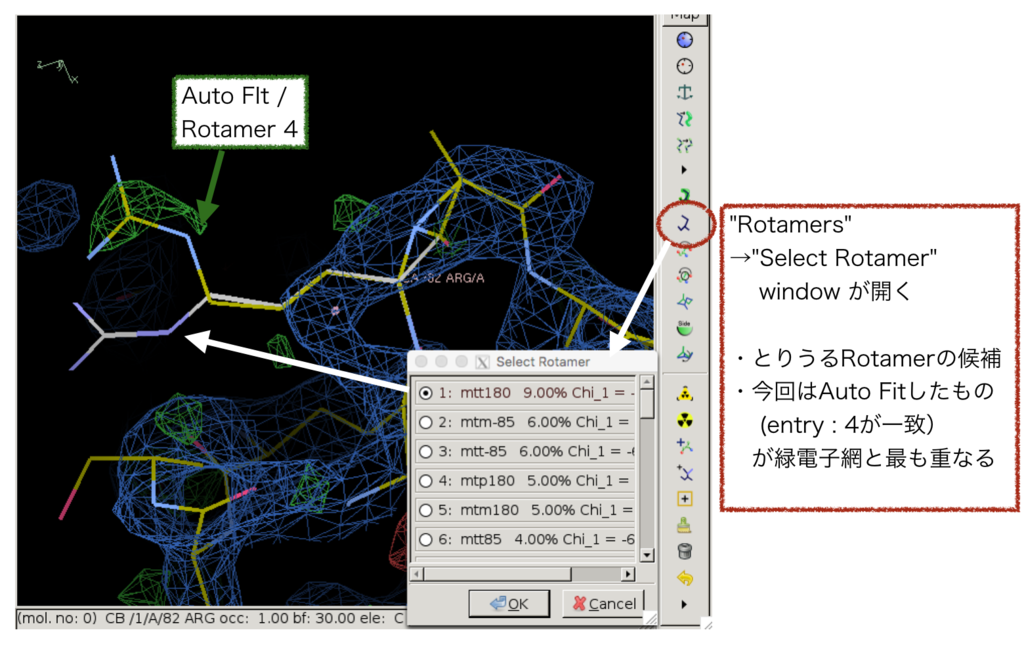
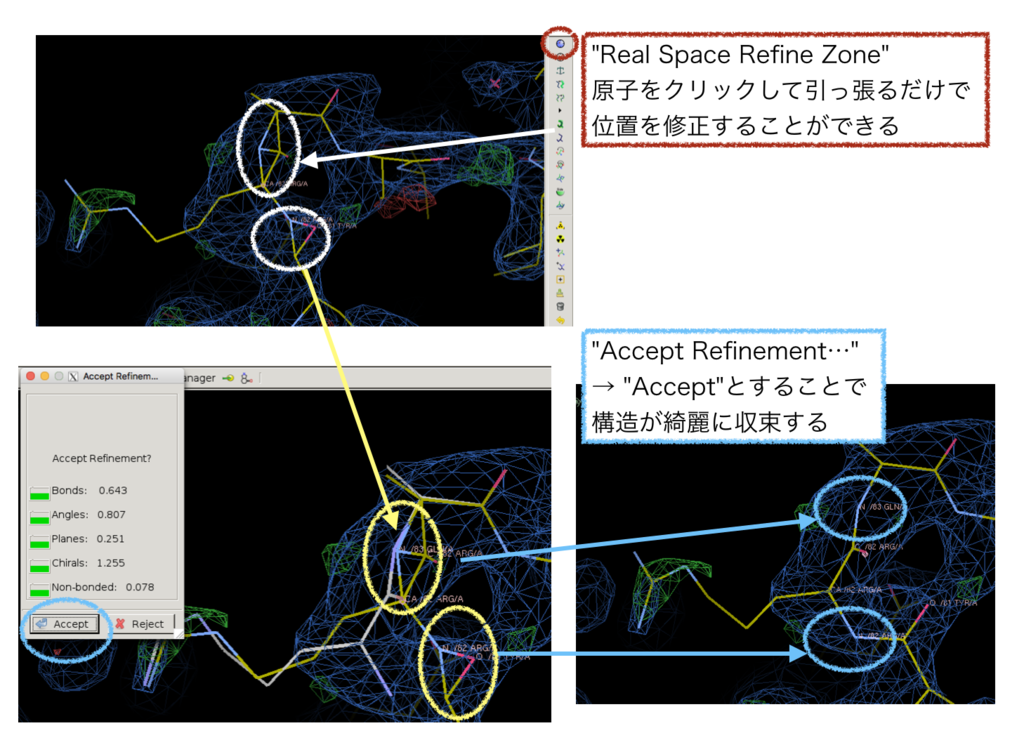
ARG 側鎖については問題ないように見えますが「Auto Fit」した主鎖はおかしな様子です。「Real Space Refine Zone」という機能をもちいると、該当の残基をクッリックしてドラッグすることで簡単に構造の修正を行うことができました。以下のような感じです。
6. リガンド部位の確認
問題のリガンド部位ですが、残基番号 201 として登録されています。
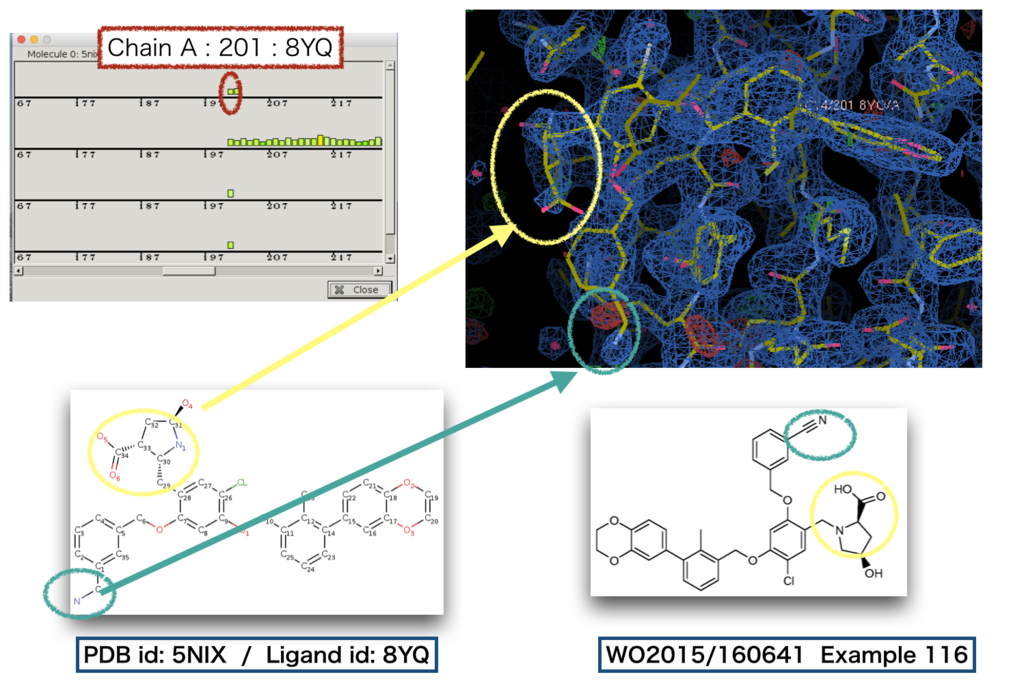
PDBに登録されている構造で元の文献と異なっている部位のうち
・ピロリジン部位(黄丸)は電子密度が薄い(poor)
・シアノ基(緑丸)は 赤い電子密度 とぶつかっています。(電子密度がないのに構造がモデル化されている)
と、なっています。どうやら、Fitさせた構造に無理がありそうです。
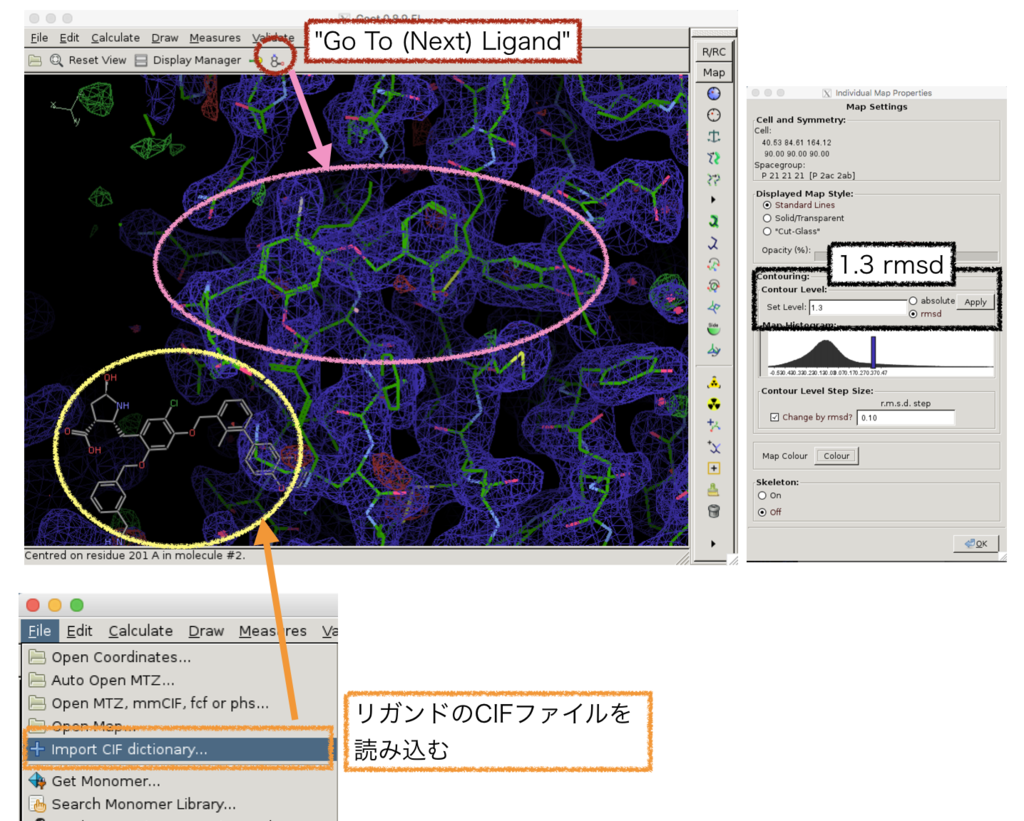
上記では残基番号からリガンドを辿りましたが、「Go To (Next) Ligand」機能を使用した方が、リガンドのズームが容易でした。
また、「import CIF dictionary…」からリガンドのCIFファイルを読み込むことで画面左下に2次元構造が表示されるようになりました。
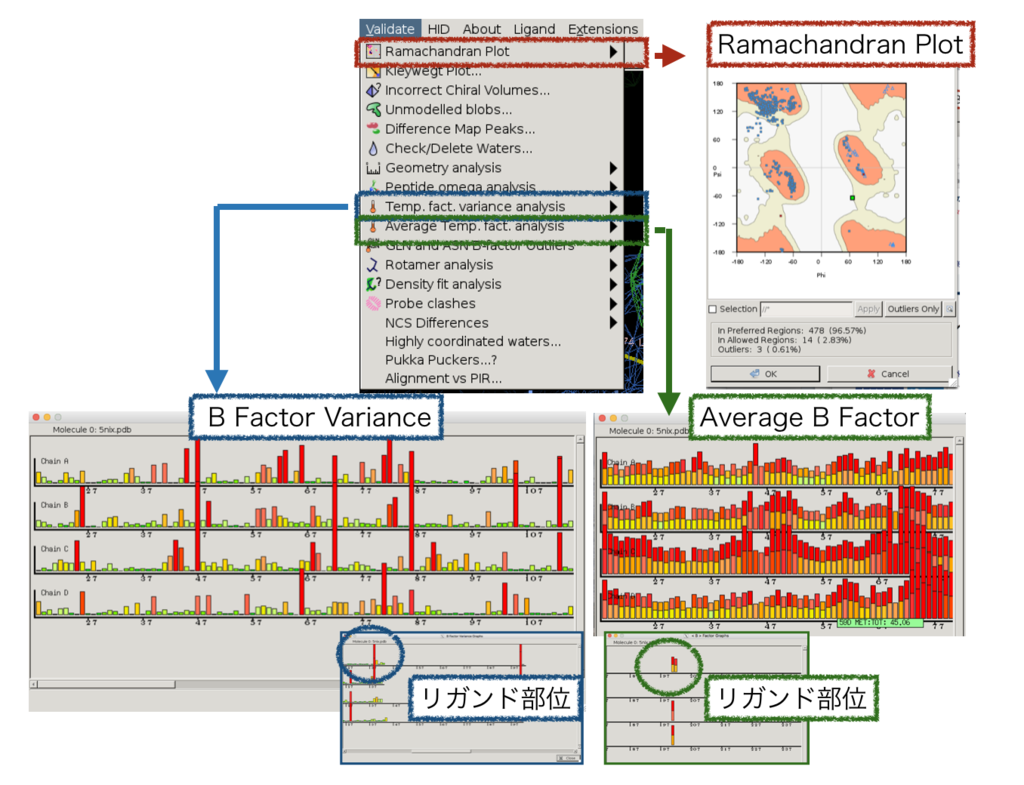
7. validationのグラフ
ちなみにその他のvalidation に関係しそうな指標は以下のように確認できます。
8. 別の構造でも確認(PDB id: 5NIU)
以上、構造式の誤っているPDB id : 5NIX の確認を行ってきましたが、同じ文献で報告されており別の構造 PDB id : 5NIUについても確認しておきたいと思います。
まずは2次元構造から・・・
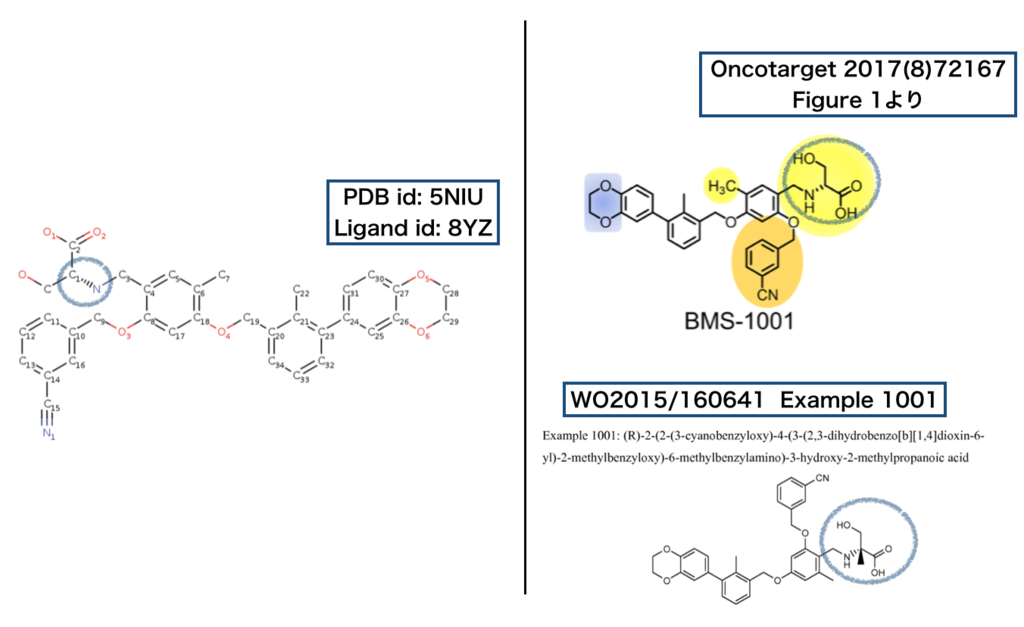
・・・今度は文献とPDBに登録された構造はあっていますが、さらに元となるBMSの特許とは構造が異なっており、メチル基が欠けています。
なんだか色々怪しくなってきました・・・
coot でリガンド部位を見てみましょう。
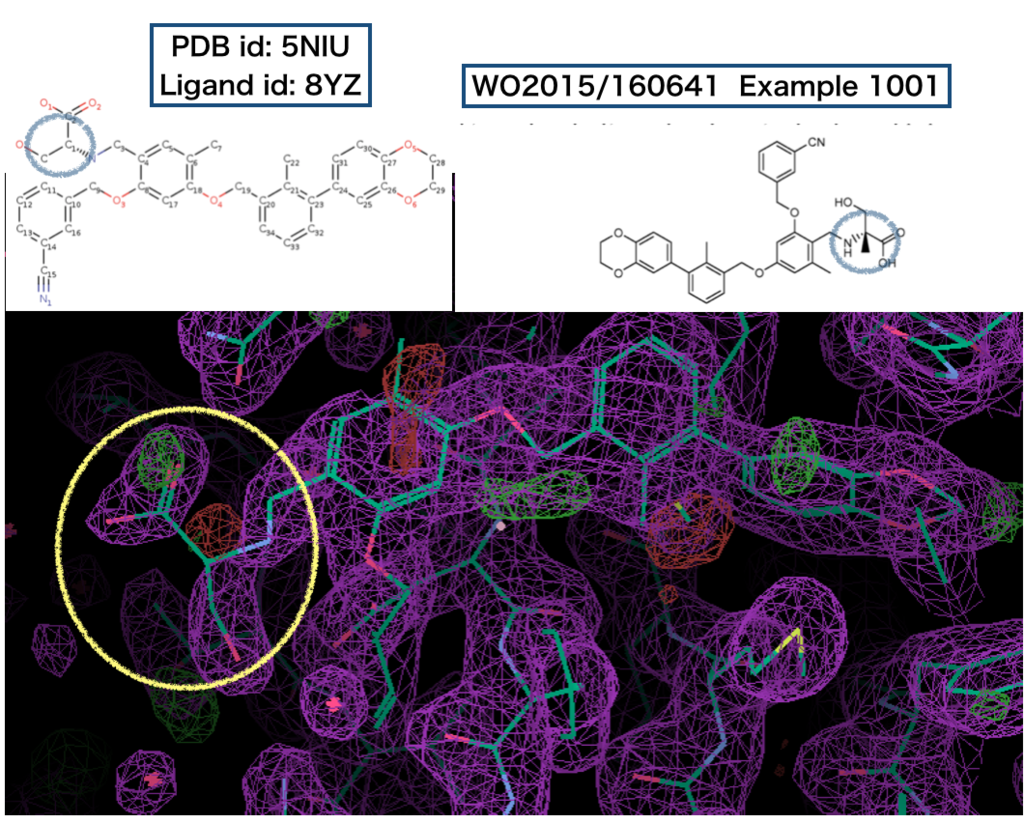
こちらもタンパク質の奥深くまで結合している部位は電子密度がはっきりしているように見えますが、溶媒に露出している部分は不明瞭になっているように見えます。
また、特許と構造の異なる部分(メチル基有無が異なる炭素)については特に電子密度がはっきりしていません。
先の構造で誤って登録されていた、シアノ基についてはどのようになっているでしょうか?
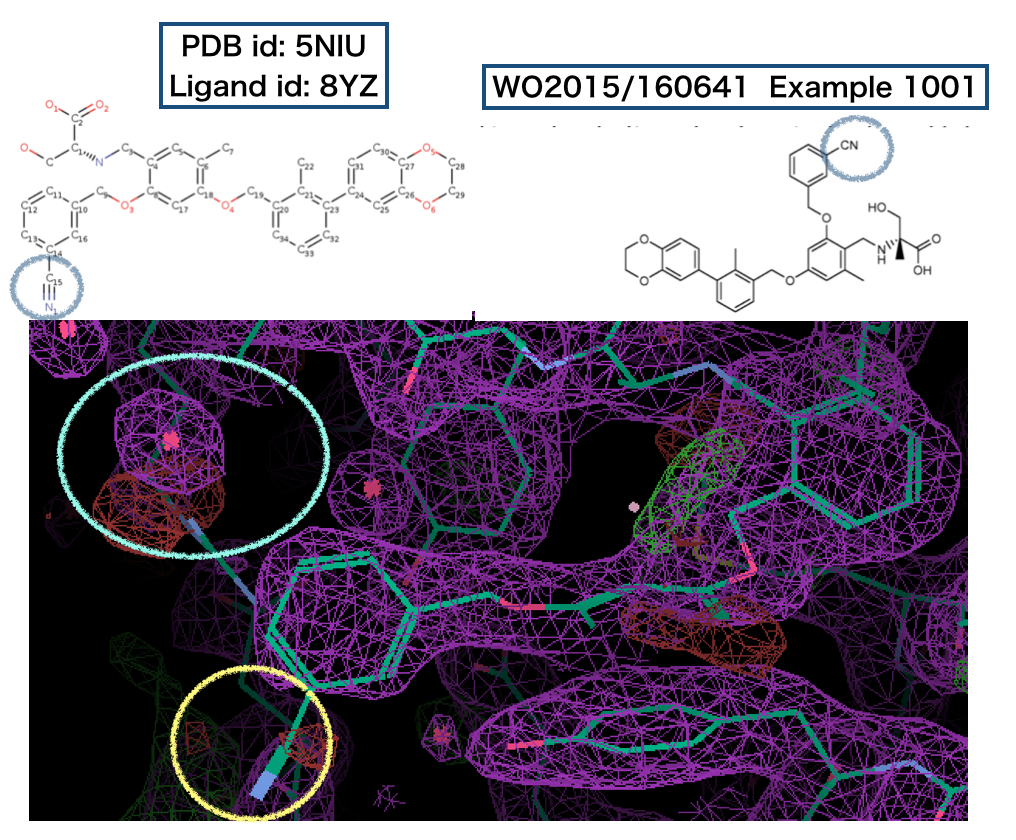
こちらは、シアノ基として登録されていますが少し曲がっており、赤い電子密度とも重なっているのが気になります。シアノ基の置かれた電子密度と対称の位置に、水(302 / HOH)が置かれた部位がありますが、こちら側にシアノ基が伸びていても良いようにみえます。
まとめ
以上、cootを利用して共結晶構造を眺めて見ました。今回取り上げた構造はどちらもそもそもの構造式に疑惑があり、適切な例ではなかったかもしれませんが、電子密度と比較してみることで構造の問題がよりクリアになったように思います。
結晶構造の解析等を実際に行ったことがないため、そもそも眺めている電子密度がどれほど質のものなのかもわかっておりません。誤り等あればご指摘いただければ幸いです。
あと、このページすごい・・・
*********************************************************************************************************
*1)
coot の tutorial
https://www2.mrc-lmb.cam.ac.uk/personal/pemsley/coot/web/tutorial/tutorial.pdf
"Features and Development of Coot" P Emsley, B Lohkamp, W Scott, and K Cowtan Acta Cryst. (2010). D66, 486-501 Acta Crystallographica Section D-Biological Crystallography 66: 486-501
*2)
使用にあたって参考にさせていただいたページ、ファイル
Refmac/cootでrefinement (finish編)
Introduction to ligand chemistry in protein X-ray structures
http://www.tsurumi.yokohama-cu.ac.jp/xtal-mls/coot/COOT.pdf
*3) MTZファイルについて
RCSB PDB: X-ray Electron Density Maps
*4) 共結晶構造の報告されている文献
Oncotarget. 2017; 8:72167-72181.
PDB viewer でバリデーション
PDBフォーマットの見方や注意点などについて調べてきました。折角ですので前記事を踏まえて再度、発端となった構造 [PDB id : 5NIX]の質について見てみましょう。
今まであまりにも知識がないため見て見ぬ振りをしていたのですが、X線結晶構造の実験データとバリデーションの情報についてもRCSB PDBページで提供されていました。
PDBのビューワーにもいろいろな機能がありそうなので遊んで見たいと思います。
1. 指標の確認
先ずは結晶の質の指標となる分解能とR因子の確認から・・・

前回のおさらいですが、原子レベルでの議論をするための結晶構造解析の質の目安として
・分解能 < 2Å
・Rfree < 0.20
が、挙げられていました。
Fig. 1 より、PDB :id 5NIXではいずれも目安より大きな値となっており、原子レベルの議論には向かない結晶構造かもしれません。
R因子の値のレンジですが、ランダムな構造を生成させて計算すると約0.6となるため、取りうる値の範囲 [ 0 ~ 約0.6 ] くらいの指標だそうです。( PDB-101:R-value and R-free )
数値のみを見ても、それがどの程度の質なのかはイメージできない・・・・
と思っていたら、wwPDB Validation と題した素敵なグラフもありました。

PDBの他のデータと比較した際に、該当のエントリーの構造の質がどの程度のものなのか、スライドバー形式で表示されています。
黒色がPDBの全エントリー(2017/12/27時点)と比較した場合、白抜きが類似の分解能の構造群と比較した場合にどのようなランク(%)に位置するかを示します。
Rfree は中央よりも左に寄っているため、他のデータと比較した場合、抜群に質の良い構造とは言えなさそうです。
Fig. 4 の Rfree の値が 0.264 でFig. 3の値 0.251と異なりますが、これはFig.3 が構造投稿者によって求められた値であるのに対し、Fig 4. はDCCというプログラムを用いて再計算した値が用いられているためです。( wwPDB: X-ray validation report user guide )
2. 3D viewer で全体の質をチェック
もっと格好良く!ということで3Dでも見てみたいと思います。
PDB の3D viewer で描画方法を変更してみました。

Fig. 3 左はこれまで用いてきた[Rainbow] 形式のカラーで、Fig. 3 右は[By Density FIt] としたものです。
こちらはモデルと実験データ(電子密度)がうまく一致しているか、その度合いをBetter(青)- Poor (赤) のグラデーションで色付けしています。
色付けの基準は以下のような指標に基づいているそうです(*1)。

RSRはモデルの1残基ごとに実空間のデータとの重なり度を評価する指標で、RSRZは、このRSRを分解能とアミノ酸残基のタイプに応じて正規化したものだそうです(*2)。
Fig. 3 をみると、リガンドは真っ白なので「まあまあの精度」といったところでしょうか?タンパク質(PD-L1)側は概ね青色で良い適合を示していますが、ところどころ白くなっています。周辺のループ構造のようにみえますので、動きがある部分なのかもしれません。
動きといえば・・・ということで、次の描画方法、温度因子(B-Factor) を見てみましょう。

B-factorは構造の中でもっとも高いB-factor値を持つ部位を赤、最も低い部位をタンとなるように色付けされています。
Density Fit(左)で白色となっている部分と、B-factor (右) で茶色を帯びている部分のいくつかは重なりがあるように見えます(Fig. 5 赤丸)。タンパク質の動きがある(柔らかい)部位でモデルと電子密度の差が生じているというのは、あながち間違いではないかもしれません。
雑な考察に無理やり辻褄を合わせていくスタイル・・・すみません。
数値的にはどうなっているでしょうか?

おそらくFig. 6 の Mean Isotropic B という値が等方性温度因子の平均値だと思いますが、43.81 となっています。
前回ご紹介した目安では
・占有率 < 1.0 かつ B-factor > 30 Å2
の場合、アミノ酸残基の位置が疑わしいということでした。
占有率 1.0 の場合B-factorが30を超えていても許容、ということなのでしょうか???
では、最後の描画方法 [By Geometry Quality] を見てみましょう。

こちらの描画方法では、構造に含まれる幾何学的な問題点の数に従って色付けされています。(0 : 青、1 : 黄、2 : オレンジ、3 以上 : 赤)
また、オプションで「Clashes」にチェックを入れると、ぶつかっている原子間にピンク色の円盤が表示されるようになります。円盤の大きさがvan der Waals半径の重なりの程度を表します。
Fig. 7をみるとリガンドは黄、オレンジ色の原子が多く、さらに周囲に"Clash"が複数みられます。
リガンド2次元構造の間違いから出発し、[PDB id : 5NIX] について調べてきましたが、どうやらリガンドの3次元構造のモデリングに課題がありそうです。
もう少し詳しく見てみたいと思います。
3. リガンドの構造を再調査
まずは2次元構造の問題点を再確認。

リガンドに関する指標もついでに確認。
wwPDB validation の[Full Report] の中に [Ligand Geometry] という項目がありました。
こちらは低分子の結晶構造データベースであるCambridge Structural Database (CSD)から導いた「適した幾何構造(preferred molecular geometries)」と、リガンドのモデル構造との比較結果が記載されているそうです。

Bond lengths、Bond anglesともにCounts の3つの数値のうち一番左、analyzed の数が他の2つよりも少なくなっていますが、CSDの情報の中に比較相手となるフラグメント構造が存在し無い場合に少なくなることがあるそうです。
リガンド分子全体について結合長、結合角、各々のRMSZ(root-mean-square value of the Z-scores)が求められていますが、こちらは[ 0 - 1 ] の間に収まることが期待れる値で、1よりも大きい場合over-fittingである可能性が示唆されるそうです。
また「#|Z| > 2」はZ-scoreの絶対値が2よりも大きく外れ値となっている bond / angle の数、および分子中に占める割合(%)だそうです。
Validation Report の中ですでに黄色にハイライトされているように「RMSZ」、「#|Z| > 2」ともにかなり怪しい値を示しています。
リガンド部位のモデルの指標としては以下のような値・・・

前回ご紹介した目安では
・占有率 < 0.5 or 温度因子 > 50 Å2 の場合、結合様式の信頼度低い
・原子レベルで比較対照する場合は、 占有率 1.0 かつ 温度因子 < 30 Å2 が好ましい
とのことでした。
占有率については問題ありませんが、温度因子としてはモデルを鵜呑みにしてはいけなさそうです。
それでは、Fig. 8 で指摘した4点の部位についてそれぞれ構造を見てみたいと思います。
①単結合を2重結合として認識している部位

赤丸で囲んだ部分、C19は 2 Geometry Problems、Clash ありと、原子の位置に問題がありそうです。
PDBでは3次元の配置から自動でアノテーションしているため、結合次数の誤りが生じることがあるということでした。
C19の位置が誤っており、O2-C19-C20-O3 が平面となるような配置となってしまったため、C19-C20 が2重結合として帰属されていると考えると理解できそうです。
② N の位置の違い、③不斉点の誤り

2次元構造レベルで複数の問題点を含むピロリジン骨格ですが、Fig. 11に抜き出したように3次元構造でみてみるとピロリジン環とフェニル基とを繋ぐ結合を中心としてGeometry Problemsを含む原子が並んでいます。
PDBファイルから該当の原子の情報を抜き出してみると、いずれも温度因子 が 50 Å2 以上 となっています。
「リガンドについては温度因子が 50 Å2 以上の場合、結合様式の信頼度が低い」とする目安と見事に一致しているように見えます。
格好いいので電子密度の図も貼っておきます。

・・・よくわかりませんがピロリジン環にぶら下がっているヒドロキシル基やカルボキシル基はあまり電子密度が内容にも見えます。
④シアノ基がアミノメチル基となっている

フェニルから直線的にのびるであろうシアノ基が曲がっています。確かにこの3次元座標からシアノ基として認識するのは無理です。
4.まとめ
以上、PDBのページで公開されている情報、および3D viewerをもちいて、「構造の質」という観点から、これまで眺めてみてきた構造を再度眺めてみました。
描画方法を変えてみるだけで、思った以上に問題点がわかりやすくなったように思います。
とりあえず、こちらの結晶構造をベースとしてそのまま議論するのはやめた方が良さそうです。
PDBのUser Guide等をざっと眺めただけで記載しているため、上記内容に誤りがあるかもしれません。ご指摘いただければ幸いです。
*************************************************************************************************************
*1) RCSB PDB: 3D View User Guide
*2)
7-1) PDBでは、RSRZをどのように計算していますか? - validation FAQ - - Help - 日本蛋白質構造データバンク
・EDSに関して
The Uppsala Electron-Density Server
Kleywegt, G.J., et al., Acta Cryst. (2004). D60, 2240-2249
http://scripts.iucr.org/cgi-bin/paper?S0907444904013253
・RSRZに関して
Statistical quality indicators for electron-density maps
Tickle, I.J., Acta Cryst., (2012). D68, 45-467
http://scripts.iucr.org/cgi-bin/paper?S0907444911035918
・RSCCに関して
Validation of lignads in macromolecular structures
Smart, O.S., et al., Acta Cryst. (2018). D74, 228-236
http://scripts.iucr.org/cgi-bin/paper?S2059798318002541
*3) 本記事の作成に以下を使用しました。
・RCSB PDB
・PDB ID : 5NIX
・Skalniak, L., et al., (2017) Oncotarget 8: 72167-72181
・AS Rose et al. (2018) NGL viewer: web-based molecular graphics for large complexes. Bioinformaticsdio:10.1093/bioinformatics/bty419
PDBの落とし穴にはまった話 〜非結晶学者が注意すべきこと編〜

PDBの中身もなんとなくわかったところで、当初の問題、PDBに登録された低分子の構造がなぜ間違っていたのかということを考えていきたいと思います。
その前に今回の内容の目次・・・
1. PDBフォーマットの問題点
では問題の構造を再掲。左がPDBで、右が特許(および元文献)中の構造です。

・・・ここまで違うともはや清々しい。
「PDBの低分子構造は自動でアノテーションされているため、間違いが生じる可能性がある」とのことでした。そこで、再度PDBファイルのリガンドの構造に関する部分を見てみます。

Fig. 2 に記載したように、PDBにはリガンドの構造にとって重要な下記の情報が欠けています。
①水素原子の情報がない(→ 水素の数や位置で不飽和度や立体配置を決められない)
②原子間に結合があることはわかるが、結合次数や立体配置が記載されていない。
PDBファイル本体にはこれらの情報が含まれておらず、原子の3次元座標のみから、結合角や位置関係をもとに、自動で結合次数や立体配置を割り当てているそうです。したがって、データの分解能が悪いと自動で生成した構造に誤りが生じ、間違ったまま登録されていることがあるようです。
Fig .1 で指摘した誤りのうち①、③、④については上記で説明がつきますが、構造式の誤り(②)もあるところを見ると、そもそも著者らのチェックにも問題がある気がします・・・。
PDBのフォーマットは古いため、記載方法や情報に課題があることは問題となっているようで、現在新しいフォーマット PDBx/mmCIF への移行が進められているそうです。
上記の説明は、ページ末尾の資料・文献を参考に作成しました(*1)が、私の理解不足により誤りがあるかもしれません。ご指摘いただければ幸いです。
2. PDBを利用する上で非結晶学者が注意すべきこと
ところで、私はタンパク質の結晶は言うまでもなく、低分子有機化合物の結晶化すらできなかったダメ学生だったのですが、今回参考とした資料の中には結晶学を専門としない私のような門外漢にとって、非常に勉強になる内容が多く記載されていました。
再度似たような間違いに陥らないよう、内容を紹介したいと思います。
(1) 共結晶構造中の構造の誤りについて
まずは文献③ Reynolds, C.H., ACS Med. Chem. Lett. 2014(5)727 より。
こちらの文献ではタンパク質-リガンド共結晶構造におけるリガンド構造の問題について下記5点が指摘されています。
タンパク質-リガンド共結晶構造におけるリガンド構造の問題
1. 構造式の誤り
・あるべき原子がない(missing atoms)
・結合次数が間違っている(incorrect bond orders)
・結合関係の間違い(connectivity issues)
2. 分子構造の問題
幾何的特徴が化学的に理想な値の範囲外にある場合(problems with geometric constraints and ideal values)
・結合長(bond distances)
・結合角(angles)
・二面角(dihedral angles)
3. タンパク質-リガンドの立体的衝突
タンパク質とリガンドがぶつかってしまっている(bad steric clashes)
4.コンフォメーションの間違い
化学的にみておかしな配座となっている(internal strain)。
・アミド結合の配座がシスとなっている(cis-amides)
・アミド結合の平面性が崩れ、ねじれている(twisted amides)
・環構造のねじれ( distorted rings (e.g. boat型やtwist型の配座))
・芳香族の平面性が崩れている(nonplanar aromatic groups)
・非平面のはずの構造が平面となっている(e.g., sufones and sulfoxides)
5. 結合モードの間違い
・結合サイト内でのリガンドの向きがおかしい(incorrect orientation)
→実験データから見てあきらかにとるはずのポーズをとっていない。
・ 水素付加(protonation states)、電荷(charges)の問題
上記の誤りが生じるテクニカルな要因
これらの問題が生じる要因として、3点のテクニカルな問題があげられています。
1.分解能不足
・超高分解能(< 1.0Å)でない限り電子密度分布のみからの分子構造の予測は困難である。
・したがって、X線結晶構造解析から導かれた構造は電子密度分布に合うようにつくられたモデル(fitted model)である。
2.力場の問題
・構造予測(モデルのフィット)に、力場が用いられるが、タンパク質にあわせてつくられたものであり低分子に適していない
・よく使われる力場(e.g. Engh-Huber)の問題点としては下記が挙げられている。
・united-atomモデルである(水素原子を炭素原子に含めて扱う
・静電力(electrostatics)が軽視(無視?)されている
・低分子医薬品向けにパラメーターが最適化されていない
3.リガンド精度の重みの低さ
・リガンドはタンパク質と比べて非常に小さいため、リガンドのフィッティングの精度は、結晶構造全体のフィッティング精度に占める割合が非常に小さい。
・したがってリガンド単体でみれば大きな間違いであっても、全体での精度の指標(一般的にはRfree)に与える影響が小さく、誤りが見過ごされてしまう。
(2) X線結晶解析結果を活用するための注意点
次に、文献② 東海大学 平山先生によるPerspectiveから。
こちらはX線結晶解析を専門としない研究者・技術者がX線結晶解析で得られた分子構造を参照する上で注意すべき点について詳しく説明されています。
低分子X線解析結果の注意点
1. X線解析単独では原子種(C、N、O)の決定が困難
解析の基本は・・・
・X線解析で求められるのは結晶内の電子密度の分布
・原子の位置 ・・・「電子密度の局所的極大点 = 原子核の場所」
・原子の種類 ・・・「原子の位置の電子密度の高さから決定」
電子数の差が小さい原子同士(CとNなど)では、電子状態によっては原子種の判別が困難で帰属を誤る可能性がある。
2. H原子の位置決定が困難
X線解析で得られるH原子の位置は実際の原子核の位置と有意に異なる。
理由は、
・H原子の電子密度は結合相手の原子に引き付けられている場合がほとんど
・電子密度の局所的極大点で原子核の位置を代表する。
・・・ H原子の位置が実際よりも、結合相手寄りの位置として解釈される
3. 温度因子(B factor)について
・結晶の単位胞内での原子の振動を考慮するために各原子に与えられるパラメータ
・・・温度因子が大きいほど結晶内での動きが大きい原子
・球状に近似した等方性温度因子(Isotropic B factor)と回転楕円体近似した異方性温度因子(Anisotropic B factor)がある
・・・低分子では主に後者が用いられ、運動の方向性がある程度分かる
*低分子X線解析の図でよく見るORTEP図(Oak Ridge Thermal Elliposid Plot)は異方性温度因子を考慮した表示方法だそうです。
4. 占有率
・全ての単位胞が等価とは限らない
例)水和物と無水物、複数の安定立体配座、糖のアノマーといった複数の構造の混在
・占有率は混在しているもののうち、ある構造を1単位胞内の存在量で表した値
・占有率と温度因子には相関がある
・・・占有率1.0でも温度因子が他の原子より異様に大きい原子は疑った方が良い
*占有率が1.0より小さい場合でも使用しているソフトウェアによっては一つの構造しか表示されないことがあるそうです。
5. R因子について
R = Σ(|Fo| - |Fc|) / Σ|Fo| ・・・(F: X線の振幅、Fo: 観測値、Fc: 計算値)
・「モデル構造がどれだけ観測データを説明するできるか?」の程度を表す指標であり良質な構造を選択する上で参考となる
・判断の目安
・・R 因子 5%以下・・・ほとんど問題なし
・・R 因子 7-10% ・・・原子の並びはよくても、原子種の判定が微妙なことがある
・重原子を含む構造や、自由度の高い分子の場合はR因子が高くなる傾向があることに注意
タンパク質X線解析結果の注意点
1. タンパク質結晶解析の3つの大きな特徴
①「結晶中にたくさんの水分子」かつ、「大部分は明確なX線回折に寄与しない」
② バルクの水に接する分子表面を中心に分子の運動性が高い
③ X線回折の条件が低分子結晶(緊密にパッキング)と比較して格段に悪い
2. タンパク質結晶解析の質を計る2つの指標
①分解能 (resolution)
・識別可能な最小の2点間の距離
↔︎ 観測できたデータの上限で、上限のデータの質を決定するものではない
→ 分解能の数字に相当する構造が求められていないことがありうる
・原子レベルの解析を目指す場合の目安
・・・分解能 < 2Å (低分子の分解能は通常1.0Å程度)
②R因子
・Rfree が使われる
・・・全てのデータを含めて計算されるRよりも信頼性が高い指標
・原子レベルの解析を目指す場合の目安
・・・Rfree < 0.20
3. 化学構造の曖昧さ
①タンパク質結晶では観測データだけでは、非水素原子の座標を決定するのに不十分
・・・構造についての予備知識なしに、電子密度のみから解釈することはできない
②各原子座標の原子パラメータの標準偏差を見積もることができない
・・・原子レベルでの分子構造や分子間相互作用について定量的な議論をするには精度が低い
4. 問題点
タンパク質側の問題
①原子種の帰属が不明瞭
・ヘテロ原子の帰属が困難
・・・AsnやGln側鎖の O / N原子の区別、His側鎖のN原子の位置の確定ができない
・水素原子の位置も推定できない
②温度因子と占有率
・アミノ酸残基・・・占有率 < 1.0 かつ 温度因子 > 30 Å2 の場合、位置が疑わしい
リガンド側の問題
①化学構造
・PDBデータの書誌的データの誤り
・電子密度のみから化学構造(結合の不飽和度、水素結合の有無)の定量的評価は困難
②解離状態
・タンパク質X線解析から直接的証拠を出せない
③温度因子と占有率
・リガンド・・・占有率 < 0.5 or 温度因子 > 50 Å2 の場合、結合様式の信頼度低い
・原子レベルで比較対照する場合は、 占有率 1.0 かつ 温度因子 < 30 Å2 が好ましい
平山先生のPerspectiveの紹介は以上です。2006年の文献であり、現在では精度等状況が変わっているかもしれませんが、個人的には知らなかったことばかりで大変勉強になりました。PDBのビューワーで綺麗な図をみているだけでは絶対にわからなかったことばかりだと思います。
3. まとめ
以上、PDBフォーマットの見方から、PDB利用の際の注意点まで見てきました。
「N/Oの区別ができない」や「水素結合の有無が不明瞭」など、ビューワーで眺めただけで「結合がありそうだから大事な部分構造だ」とか「活性に必須な官能基だ」と結論づけてしまうのは要注意ということですね。複数のデータを組み合わせて構造を見る必要がありそうです。
最後に文献⑤から、X線結晶構造を創薬にもちいる上で、 「知らず知らず前提としてしまっているけど正しいとは限らないよ」という3つの指摘を引用します。
(1) The protein structure is correct.
(2) The structure of the ligand and its interactions with the protein are correct
(3) The protein-ligand structure is relevant for drug design.
Davis, A.M.; St-Gallay, S.A.; Kleywegt, G.J. Drug Discov. Today 2009, 13, 81
・・・それは前提とさせて欲しかった!!
*************************************************************************************************************
*1) 参考文献
文献① 平成27年度日本結晶学会年会 大阪府立大学 PDBjランチョンセミナー資料(PDF)
文献②
構造活性相関部会・ニュースレター SAR News No.11 (Oct. 2006)
「結晶解析の結果を上手に活用するために」平山令明 教授
(日本薬学会構造活性相関部会 HPより閲覧可能)
文献③
Protein-Ligand Cocrystal Structures: We can Do Better ACS Med.Chem.Lett.,2014(5)727
(無料で読める)
文献④ (文献③で引用されている。なぜかネットにPDFが落ちている・・・)
Application and Limitations of X-ray Crystallographical Data in Structure-Based Ligand and Drug Design
Angew. Chem. Int. Ed. 2003(42)2718
文献⑤ (「Limitation X-ray」でググったら出てきた。これもなぜかPDFが・・・)
Limitations and lessons in the use of the X-ray structural information in drug design
Drug Discovery Today 2008(13)81
PDBの落とし穴にはまった話 〜PDBフォーマットの見方編〜

PDBで色々と遊んできましたが、肝心のPDBファイルの中身を理解していませんでした。PDBフォーマットについて調べたので忘れないようにメモ。
きっかけはTwitterでのやりとり。 PDBでは構造を自動で認識しているため間違っている可能性があると教えていただきました。
普通は、論文の方が正しくて(著者にまともな人がいたら見過ごさないと思う)、PDBでは自動システム(bond angleなどで検知)で低分子のアノテーションをするのでPDBが間違ってる可能性の方が少し高いですね。
— Shinya Fushinobu (@sugargroove) 2018年10月22日
Fushinobu先生ありがとうございました!(*1)
問題の構造はこちら

Fig. 1 左に示した2次元構造は、RCSB PDBで確認できる、BMS社のリガンドとPD-L1の共結晶[PDB id: 5NIX] のリガンドの構造ですが、末端部位が1,4-Benzodioxineとなっています。「こんな不安定そうな構造をBMSがつくるだろうか? そもそもどうやって合成するの?」ということで、PDBの元文献とBMSの特許を確認しました(Fig. 1 右)。
どちらも1,4-Benzodioxaneとなっており、どうやら先生のご指摘通りPDBの構造が間違っていそうです。
他にもPDB上の構造は、シアノ基の3重結合がなかったり、ピロリジン環のNの位置が違うなど、つっこみどころの多い構造となっています(Fig. 1 左、青枠で示した部分)。
これはPDBの中身についても知っておく必要がありそうです。
「中身って言ったってどうせ謎の文字列でしょ。マトリックスに出てくるスーパーハッカーみたいな人達が読むやつでしょー。」と思ってたのですが、意外に(?)普通のテキストファイルでした。
早速 PDB id : 5NIX をダウンロードし、テキストエディタで開いてみます。こんな感じ。

PDBファイルは1行80列の固定長からなっており、1行が1つのレコードに対応しています。左端の6列が各レコードの識別に割り当てられており、ここを見るとなんとなく何が書いてあるか内容がわかります。
固定長のため不自然に改行されていたりしますが、左端を頼りすれば読めそうです。
Fig. 2は PDBファイルの冒頭ですが、まずはタンパク質の情報や由来、文献、著者などのメタ・データが記載されています。
| レコード名 | FIELD | 定義 | 5NIXの場合 |
|---|---|---|---|
| HEADER | 11 - 50 列 : classification | 分子の分類 | 免疫系 (IMMUNE SYSTEM) |
| 51 - 59 列 : depDate | PDBがデータを受け取った日 (Deposition date) | 2017年5月27日 (27-MAR-17) | |
| 63 - 66列 : idCode | 割り当てられたPDB ID | 5NIX | |
| TITLE | 9 - 10 列 : continuation | 前の行からの続きかどうか | 改行されているので2行目に2の記載 |
| 11-80 列 : title | 実験のタイトル | PD-L1と低分子の複合体みたいな内容 | |
| COMPND | 8 - 10 列 : continuation | 5行目まで続くので「空白、2、3、4、5」となる | |
| 11-80 列 : compound | エントリー中の高分子(macromolecule)の説明 | ①MOL_ID:エントリー内の分子のID ②MOLECULE:分子の名前 ③CHAIN:含まれるChain IDのリスト ④SYNONYM:シノニムのリスト ⑤ENGINEERED:リコンビナントや化学合成したものか否か |
|
| SOURCE | 8 - 10 列 : continuation |
7行目まで続くので「空白、 2、3、4、5、6、7」となる |
|
| 11 - 79 列 : srcName | 高分子のソースの説明 | UniProtに書いてあるような情報や 発現に大腸菌を使った(EXPRESSION_SYSTEM) などなどが書いてある |
Table 1. 各レコード(Fig. 2)の説明
途中までですが、Fig. 2 各行は大体Table 2 のような感じです。
力尽きたので残りは こちら (→wwPDB Format version 3.3: Title Section) を参照してください。
残りは [KEYEDS : キーワード]、[EXPDTA : 構造決定の実験手法 ]、[AUTHOR : 著者]、[REVDAT : 更新履歴]、[JRNL : 文献]、[REMARK:その他詳細等の説明]・・・という感じで続いていきます。大体6文字でなんの略語か想像できますね。
いくつかレコードを挟んでタンパク質の3次元構造の情報が記載されています。

Fig. 3 はネットで見つけた右の講義資料(PDF: 構造バイオインフォマティクス 基礎 立体構造データベースとその利用)をもとに作成しました。
無理に色分け追加したせいでわかりにくくなったとか言わないで・・・
立体の情報は各原子の座標という形で記載されているようです。
原子名の記載方法(Fig. 3 ③)など、化学で扱うMOLファイルにはみられないような表記の仕方で、重視する情報の違いなどが見えて面白いですね。(*2)
温度因子や占有率はよく分かりません。すみません。
タンパク質以外のリガンドはどのように表記されているか?というと、タンパク質の次に、[HETATM]というレコード名で記載されていました。

[HETATM] の下には [CONECT] というレコードが続きます。こちらは原子間の結合(connectivity) についての情報で、結合に関与する原子の原子通し番号(Fig. 3 ②)を並べるという形で記載されています。

CONECT レコードは 水分子以外の HET グループについては記載が義務付けられており、以下のようなものが含まれます。( Connectivity Section )
(i) 水分子以外の non-standard (HET) residue における分子内の結合関係
(ii) HET グループから他の standard group (水 含む)、あるいは他のHET groupへの分子間の結合関係
(iii) SSBOND レコードに記載されているジスルフィド結合
話が前後してしまいましたが HETレコードは以下のような感じです。

PDBの低分子の表記に「~」などが含まれており、転記ミスかと思っていましたが、上付き文字を表すための特殊文字だそうです。
「~{R}」 は 「R」 の意味だろうか?この表記では PDB以外のフォーマットとの互換性が悪そう。
水の原子数が248になっていますが、エントリー数としては水は76個で3倍しても228個です。数が合わない?なぜだ???
SSBONDは以下のような感じ。

対称操作や点群は本当にわからないので間違っていたらご指摘いただければ幸いです。(*3)
REMARKの中にも興味深い項目がありました。
REMARK の 8-10列目の数字は、[ remarkNum : REAMRK number ]でどんな内容がかいてあることを表します( REMARK )。
例えばREMARK number 800 は結合サイトについての情報です。
・・・・こういう情報も書いてあるのか。

他にも2次構造情報のレコード [HELIX] や [SHEET] などの情報もあり、こういう情報があるからviewer でいろいろな形式で描画したり、結合サイトを抜き出した表示したりできるのか(・・・たぶん?)。楽屋裏を覗いている感じで面白いですね。
残りのレコードは
・[DBREF] :
複数のデータベースのクロスリファレンス (DataBase REFerence?)
データベースとしては 「GenBank : GB」、「Protein Data Bank : PDB」、「UNIPROT : UNP」、「Norine : NORINE」があります。
・[SRQRES]:
タンパク質のアミノ酸配列
・[SEQADV] :
PDBエントリーのアミノ酸配列(SEQRES)と他のデータベースの情報との差異
そして最後、チェック用の「MASTER」レコードと「END」レコードで終わります。

ああ、やっと見終わった。
・・・・・・
・・・・・・
・・・・・・
・・・・・・本題のPDBの低分子の構造が間違っていた問題解決していない!!
次回につづく!
P.S.
学生時代にX線結晶構造解析の授業に本当についていけなかったので間違いがあるかもしれません。ご指摘いただければ幸いです。
結晶学がわからないため飛ばしたレコード [CRTST1]も念のため・・・

・・・[ORIGXn]と[SCALEn] は本当にわからなかった
***********************************************************************************************************
*1
研究室のHP 東大・農・酵素学研究室
インタビューも掲載されていました。微生物の糖質分解酵素に着目し、酵素の産業応用につなげる〜伏信進矢・東京大学大学院教授 | Top Researchers
*2
PDBフォーマットの読み方については下記を参考に致しました。
PDBj 各書式の説明: Data Format - Help - Protein Data Bank Japan
より詳しい各項目の説明 (英語): Atomic Coordinate Entry Format Version 3.3
積ん読本: かつて勉強しようとして挫折した形跡が・・・
![タンパク質計算科学 ―基礎と創薬への応用― [CD-ROM付] タンパク質計算科学 ―基礎と創薬への応用― [CD-ROM付]](https://images-fe.ssl-images-amazon.com/images/I/51%2BUXm-2TuL._SL160_.jpg)
タンパク質計算科学 ―基礎と創薬への応用― [CD-ROM付]
- 作者: 神谷成敏,肥後順一,福西快文,中村春木
- 出版社/メーカー: 共立出版
- 発売日: 2009/08/25
- メディア: 単行本
- クリック: 36回
- この商品を含むブログ (4件) を見る
*2 MOLファイルの中身は「化学の新しいカタチ」さんの以下の記事がわかりやすかったです。
「有機合成化学者のための計算化学・ケモインフォマティクス 入門」と題された、ハイクオリティーすぎる記事満載のサイトです。
*3対称性について記載されたページ。難しい・・・
PDBの生物学的構造単位について — 生物学的集合体と対称性 1.0 ドキュメント
