Fingerprinting and Molecular Similarity part3 〜RDKit 直訳 Day14〜
(12/30追記)試訳をまとめたテストサイトを作成しました。よろしければご参照ください。
こちらはRDKit直訳 Advent Calendar 2018 - Adventar 14日目の記事です。基本的な進め方は1日目の記事をご覧ください。
本日の訳出に困った用語
picker: ピッカー
similarity map: 類似度マップ
similarity metric: 類似度の指標
Dice silmilarity: Dice類似度
convenience function: 簡易関数
normalization: 正規化
以下、訳
フィンガープリントを使って多種多様な分子を選択する(Picking Diverse Molecules Using Fingerprints)
[Link] https://www.rdkit.org/docs/GettingStartedInPython.html#picking-diverse-molecules-using-fingerprints
多数の分子を含む集合から、多種多様な分子を選択してより小さな部分集合をつくることはよく行われます。RDkitではこの作業のため、rdkit.SimDivFiltersモジュールに多数の方法を用意しています。最も効率的な方法はMaxMinアルゴリズムを使うことです[脚注6]。試しにやってみましょう。
[link] rdkit.SimDivFilters
[脚注6] Ashton, M. et al. “Identification of Diverse Database Subsets using Property-Based and Fragment-Based Molecular Descriptions.” Quantitative Structure-Activity Relationships 21:598-604 (2002).
まずは分子を1セット読み込んでMorganフィンガープリントを生成します:
>>> from rdkit import Chem >>> from rdkit.Chem.rdMolDescriptors import GetMorganFingerprint >>> from rdkit import DataStructs >>> from rdkit.SimDivFilters.rdSimDivPickers import MaxMinPicker >>> ms = [x for x in Chem.SDMolSupplier('data/actives_5ht3.sdf')] >>> while ms.count(None): ms.remove(None) >>> fps = [GetMorganFingerprint(x,3) for x in ms] >>> nfps = len(fps)
MaxMinアルゴリズムにはオブジェクト間の距離を計算する関数が必要です。ここではDiceSimilarityを使いましょう。
>>> def distij(i,j,fps=fps): ... return 1-DataStructs.DiceSimilarity(fps[i],fps[j])
それではピッカーを生成し、10個の多様な分子のセットを取りだしましょう:
>>> picker = MaxMinPicker() >>> pickIndices = picker.LazyPick(distij,nfps,10,seed=23) >>> list(pickIndices) [93, 109, 154, 6, 95, 135, 151, 61, 137, 139]
ピッカーはフィンガープリントのインデックスを返すだけ、ということに注意してください。分子そのものを取得するには次のようにします:
>>> picks = [ms[x] for x in pickIndices]
フィンガープリントを使って類似度マップを生成する(Generating Similarity Maps Using Fingerprints)
[Link] https://www.rdkit.org/docs/GettingStartedInPython.html#generating-similarity-maps-using-fingerprints
類似度マップはある分子と参照とする分子の間の類似度に対して、各原子の寄与を可視化する方法です。参考文献17 [脚注17] に方法が記載されています。rdkit.Chem.Draw.SimilarityMaps モジュールを使用してください:
[脚注17] Riniker, S.; Landrum, G. A. “Similarity Maps - A Visualization Strategy for Molecular Fingerprints and Machine-Learning Methods” J. Cheminf. 5:43 (2013).
[link] rdkit.Chem.Draw.SimilarityMaps
まずは分子を2つ作るところから始めます:
>>> from rdkit import Chem >>> mol = Chem.MolFromSmiles('COc1cccc2cc(C(=O)NCCCCN3CCN(c4cccc5nccnc54)CC3)oc21') >>> refmol = Chem.MolFromSmiles('CCCN(CCCCN1CCN(c2ccccc2OC)CC1)Cc1ccc2ccccc2c1')
SimilarityMapsモジュールはatom pairs、topological torsions、Morganフィンガープリントの3種類のフィンガープリントをサポートしています:
>>> from rdkit.Chem import Draw >>> from rdkit.Chem.Draw import SimilarityMaps >>> fp = SimilarityMaps.GetAPFingerprint(mol, fpType='normal') >>> fp = SimilarityMaps.GetTTFingerprint(mol, fpType='normal') >>> fp = SimilarityMaps.GetMorganFingerprint(mol, fpType='bv')
atom pairsとtopological torsionsにはデフォルトのノーマル(normal)とハッシュ(hashed)、そしてビットベクトル(bit vector, bv)の3つのタイプがあります。Morganフィンガープリントにはデフォルトのビットベクトル(bit vector, bv)と、カウントベクトル(count vector, count)の2つのタイプがあります。
2つのフィンガープリント間の類似度マップを生成する関数には、フィンガープリントの生成に用いた関数を特定する必要があり、またオプションとして類似度の指標を指定できます。デフォルトの指標はDice類似度です。Morganフィンガープリント関数をすべてデフォルト引数で使用した場合、類似度マップは次のように生成できます:
>>> fig, maxweight = SimilarityMaps.GetSimilarityMapForFingerprint(refmol, mol, SimilarityMaps.GetMorganFingerprint)
このような図が生成されます:
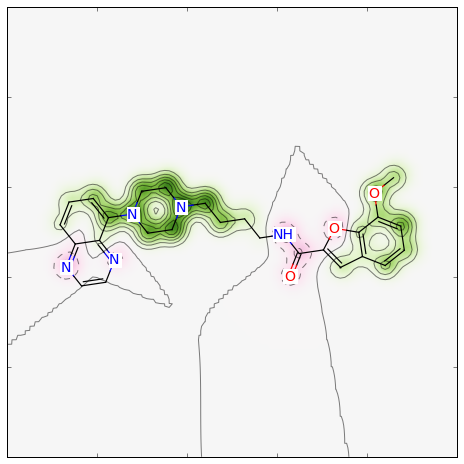
similarity_map_fp1.png
Morganフィンガープリントのタイプをデフォルトのビットベクトルからカウント(count)タイプに変更し、半径を2から1へ、類似度指標をDiceからTanimoto類似度へと変更した場合、以下のようになります:
>>> from rdkit import DataStructs >>> fig, maxweight = SimilarityMaps.GetSimilarityMapForFingerprint(refmol, mol, lambda m,idx: SimilarityMaps.GetMorganFingerprint(m, atomId=idx, radius=1, fpType='count'), metric=DataStructs.TanimotoSimilarity)
このような図が生成されます:
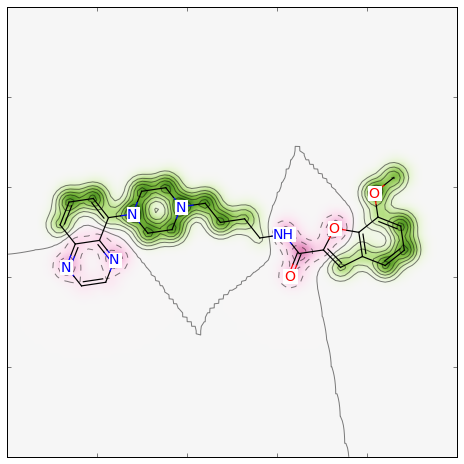
similarity_map_fp2.png
簡易関数のGetSimilarityMapForFingerprintには原子の重みの正規化が含まれており、重みの絶対値の最大が1となるよう正規化されます。したがって、この関数はマップを作成するときにみつかった、最大の重みを出力します。
>>> print(maxweight) 0.05747...
正規化の処理を行って欲しくない時は、次のようにしてマップを作ることができます:
>>> weights = SimilarityMaps.GetAtomicWeightsForFingerprint(refmol, mol, SimilarityMaps.GetMorganFingerprint) >>> print(["%.2f " % w for w in weights]) ['0.05 ', ... >>> fig = SimilarityMaps.GetSimilarityMapFromWeights(mol, weights)
このような図が生成されます:
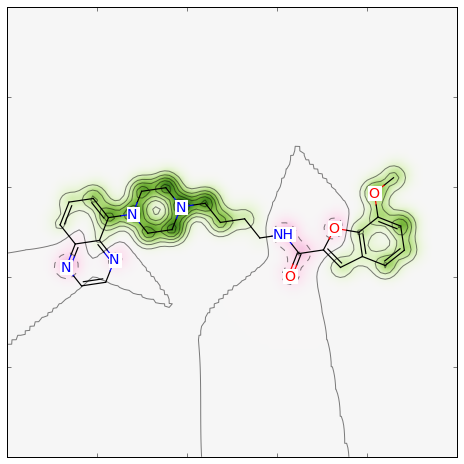
similarity_map_fp3.png
12/14/2018
このブログ記事のライセンス
以上はRDKit Documentationの試訳です。
ライセンスはCC BY-SA 4.0で、Greg Landrum氏の功績の元に作成しています。
Creative Commons — Attribution-ShareAlike 4.0 International — CC BY-SA 4.0