Non-Chemical Functionality 〜RDKit 直訳 Day21〜
(12/30追記)試訳をまとめたテストサイトを作成しました。よろしければご参照ください。
こちらはRDKit直訳 Advent Calendar 2018 - Adventar 21日目の記事です。基本的な進め方は1日目の記事をご覧ください。
本日の訳出に困った用語
binary value: 2進値、バイナリ値
sparsely: まばらな、低密度の
negate the vector: ベクトルの符号を反転する
completion: 補完
tooltip: ツールチップ
以下、訳
化学以外の機能(Non-Chemical Functionality)
[Link] https://www.rdkit.org/docs/GettingStartedInPython.html#non-chemical-functionality
ビットベクトル(Bit vetors)
[Link] https://www.rdkit.org/docs/GettingStartedInPython.html#bit-vectors
ビットベクトルは多数のバイナリ値(例えばフィンガープリントを表すバイナリ値)を効率的に格納するための入れ物です。RDKitには2つのタイプのフィンガープリントがあり、値を内部に格納する方法に違いがあります。2つのタイプは簡単に互いに変換することができますが、それぞれ最適な使用目的は異なっています:
- SparseBitVectsはベクトルに含まれるビットセットのリストだけを保存します。ファーマコフォアフィンガープリントのように非常に大きく、低密度のベクトルを保存するのにとても適しています。立っているビットのリストを取得するといった操作は非常に速いです。一方でベクトルの符号を反転するといった操作はとても、とても遅いです。
- ExplicitBitVectsは立っているビットも立っていないビットも両方の情報を記録します。一般にSparseBitVectsよりも速いですが、保存するのに必要なメモリーが大きくなります。
DIscrete value vectors
[Link] https://www.rdkit.org/docs/GettingStartedInPython.html#discrete-value-vectors
3D grids
[Link] https://www.rdkit.org/docs/GettingStartedInPython.html#d-grids
Points
[Link] https://www.rdkit.org/docs/GettingStartedInPython.html#points
ヘルプの使い方(Getting Help)
[Link] https://www.rdkit.org/docs/GettingStartedInPython.html#getting-help
RDKitのdocistringにはたくさんのドキュメントが含まれており、利用することができます。Pythonのヘルプコマンドを使って参照することができます。
>>> m = Chem.MolFromSmiles('Cc1ccccc1') >>> m.GetNumAtoms() 7 >>> help(m.GetNumAtoms) Help on method GetNumAtoms: GetNumAtoms(...) method of rdkit.Chem.rdchem.Mol instance GetNumAtoms( (Mol)arg1 [, (int)onlyHeavy=-1 [, (bool)onlyExplicit=True]]) -> int : Returns the number of atoms in the molecule. ARGUMENTS: - onlyExplicit: (optional) include only explicit atoms (atoms in the molecular graph) defaults to 1. NOTE: the onlyHeavy argument is deprecated C++ signature : int GetNumAtoms(RDKit::ROMol [,int=-1 [,bool=True]]) >>> m.GetNumAtoms(onlyExplicit=False) 15
コマンドの補完やツールチップを使うことができる環境で作業する場合、利用可能なメソッドをとても簡単に確認することができます。Jupyternotebookをつかった使用例のスクリーンショットはこのようになります:
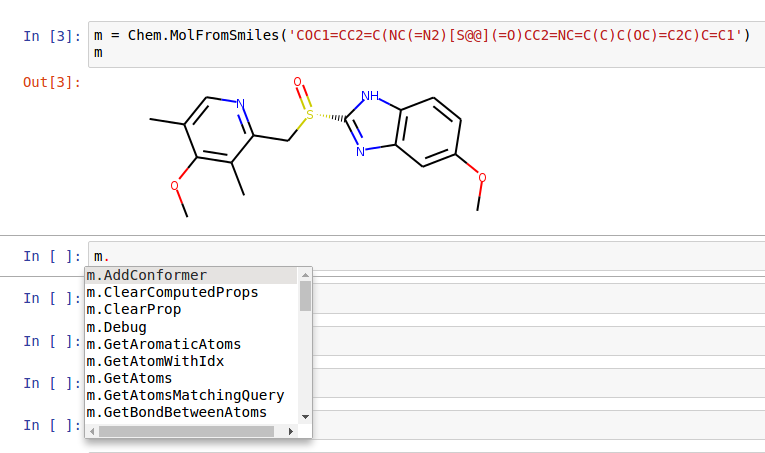
picutre_6.png
発展的なトピック/注意点(Advanced Topics/Warnings)
[Link] https://www.rdkit.org/docs/GettingStartedInPython.html#advanced-topics-warnings
分子の編集(Editing Molecules)
[Link] https://www.rdkit.org/docs/GettingStartedInPython.html#editing-molecules
RDKitで提供されているいくつかの機能では、"その場で"分子を編集することができます。
>>> m = Chem.MolFromSmiles('c1ccccc1') >>> m.GetAtomWithIdx(0).SetAtomicNum(7) >>> Chem.SanitizeMol(m) rdkit.Chem.rdmolops.SanitizeFlags.SANITIZE_NONE >>> Chem.MolToSmiles(m) 'c1ccncc1'
サニタイゼーションを行うのを忘れないようにしてください。忘れると、(深く考えない限り)一見うまくいったかのような結果を返しますが、
>>> m = Chem.MolFromSmiles('c1ccccc1') >>> m.GetAtomWithIdx(0).SetAtomicNum(8) >>> Chem.MolToSmiles(m) 'c1ccocc1'
この結果はもちろん完全にナンセンスで、サニタイゼーションをしようとすれば分かります:
>>> Chem.SanitizeMol(m) Traceback (most recent call last): File "/usr/lib/python2.6/doctest.py", line 1253, in __run compileflags, 1) in test.globs File "<doctest default[0]>", line 1, in <module> Chem.SanitizeMol(m) ValueError: Sanitization error: Can't kekulize mol
より複雑な変換はrdkit.Chem.rdchem.RWMolクラスを使うことで実行できます:
[link] rdkit.Chem.rdchem.RWMol
>>> m = Chem.MolFromSmiles('CC(=O)C=CC=C') >>> mw = Chem.RWMol(m) >>> mw.ReplaceAtom(4,Chem.Atom(7)) >>> mw.AddAtom(Chem.Atom(6)) 7 >>> mw.AddAtom(Chem.Atom(6)) 8 >>> mw.AddBond(6,7,Chem.BondType.SINGLE) 7 >>> mw.AddBond(7,8,Chem.BondType.DOUBLE) 8 >>> mw.AddBond(8,3,Chem.BondType.SINGLE) 9 >>> mw.RemoveAtom(0) >>> mw.GetNumAtoms() 8
RWMolはROMolと同じように使うことができます:
>>> Chem.MolToSmiles(mw) 'O=CC1=NC=CC=C1' >>> Chem.SanitizeMol(mw) rdkit.Chem.rdmolops.SanitizeFlags.SANITIZE_NONE >>> Chem.MolToSmiles(mw) 'O=Cc1ccccn1'
標準的な分子を扱うよりも、RWMolを使った方がナンセンスな結果を出しやすいです。化学的に妥当な結果が必要ならば、必ず結果にサニタイゼーションを行うようにしてください。
12/21/2018
このブログ記事のライセンス
以上はRDKit Documentationの試訳です。
ライセンスはCC BY-SA 4.0で、Greg Landrum氏の功績の元に作成しています。
Creative Commons — Attribution-ShareAlike 4.0 International — CC BY-SA 4.0
Molecular Fragments 〜RDKit 直訳 Day20〜
(12/30追記)試訳をまとめたテストサイトを作成しました。よろしければご参照ください。
こちらはRDKit直訳 Advent Calendar 2018 - Adventar 20日目の記事です。基本的な進め方は1日目の記事をご覧ください。
本日の訳出に困った用語
bit ranking functionality: ビットランキング機能
artificial example: モデルケース
以下、訳
分子フラグメント(Molecular Fragments)
[Link] https://www.rdkit.org/docs/GettingStartedInPython.html#molecular-fragments
RDKitには分子のフラグメント化を行うツールと、生成したフラグメントを扱うためのツールが揃っています。フラグメントは互いに結合した原子団から成るものと定義されていて、原子団が付随する官能基を持つ場合もあります。説明するよりもやってみせたほうがわかりやすいでしょう:
>>> fName=os.path.join(RDConfig.RDDataDir,'FunctionalGroups.txt') >>> from rdkit.Chem import FragmentCatalog >>> fparams = FragmentCatalog.FragCatParams(1,6,fName) >>> fparams.GetNumFuncGroups() 39 >>> fcat=FragmentCatalog.FragCatalog(fparams) >>> fcgen=FragmentCatalog.FragCatGenerator() >>> m = Chem.MolFromSmiles('OCC=CC(=O)O') >>> fcgen.AddFragsFromMol(m,fcat) 3 >>> fcat.GetEntryDescription(0) 'C<-O>C' >>> fcat.GetEntryDescription(1) 'C=C<-C(=O)O>' >>> fcat.GetEntryDescription(2) 'C<-C(=O)O>=CC<-O>'
フラグメントはrdkit.Chem.rdfragcatalog.FragCatalogにエントリーとして格納されています。エントリーの記述が山括弧で囲まれた部分(例えば'<'と'>'の間)を含むことに注意してください。この部分はフラグメントに結合する官能基を表します。例えば、上記の例の場合、カタログエントリー0はアルコールが一方の炭素に結合したエチル基のフラグメントに相当し、エントリー1はカルボン酸が一方の炭素に結合したエチレンに相当します。官能基についての詳細な情報は、官能基のIDをフラグメントに問い合わせ、そのIDをrdkit.Chem.rdfragcatalog.FragCatParamsオブジェクトの中から探し出すことで得られます。
[link] rdkit.Chem.rdfragcatalog.FragCatalog
[link] rdkit.Chem.rdfragcatalog.FragCatParams
>>> list(fcat.GetEntryFuncGroupIds(2)) [34, 1] >>> fparams.GetFuncGroup(1) <rdkit.Chem.rdchem.Mol object at 0x...> >>> Chem.MolToSmarts(fparams.GetFuncGroup(1)) '*-C(=O)[O&D1]' >>> Chem.MolToSmarts(fparams.GetFuncGroup(34)) '*-[O&D1]' >>> fparams.GetFuncGroup(1).GetProp('_Name') '-C(=O)O' >>> fparams.GetFuncGroup(34).GetProp('_Name') '-O'
カタログは階層構造になっており、小さなフラグメントが合わさってより大きなフラグメントを構成します。rdkit.Chem.rdfragcatalog.FragCatalog.GetEntryDownIds()メソッドを使って、小さなフラグメントから、そのフラグメントが寄与するより大きなフラグメントを見つけることができます。
[link] rdkit.Chem.rdfragcatalog.FragCatalog.GetEntryDownIds()
>>> fcat=FragmentCatalog.FragCatalog(fparams) >>> m = Chem.MolFromSmiles('OCC(NC1CC1)CCC') >>> fcgen.AddFragsFromMol(m,fcat) 15 >>> fcat.GetEntryDescription(0) 'C<-O>C' >>> fcat.GetEntryDescription(1) 'CN<-cPropyl>' >>> list(fcat.GetEntryDownIds(0)) [3, 4] >>> fcat.GetEntryDescription(3) 'C<-O>CC' >>> fcat.GetEntryDescription(4) 'C<-O>CN<-cPropyl>'
多数の分子からつくったフラグメントをカタログに加えることもできます。
>>> suppl = Chem.SmilesMolSupplier('data/bzr.smi') >>> ms = [x for x in suppl] >>> fcat=FragmentCatalog.FragCatalog(fparams) >>> for m in ms: nAdded=fcgen.AddFragsFromMol(m,fcat) >>> fcat.GetNumEntries() 1169 >>> fcat.GetEntryDescription(0) 'Cc' >>> fcat.GetEntryDescription(100) 'cc-nc(C)n
カタログ内のフラグメントは一意に定められたユニークなもので、分子をもう一度追加しても新しいエントリーは増えません:
>>> fcgen.AddFragsFromMol(ms[0],fcat) 0 >>> fcat.GetNumEntries() 1169
一旦rdkit.Chem.rdfragcatalog.FragCatalogを生成すれば、それを用いて分子のフィンガープリント化を行うこともできます。
[link] rdkit.Chem.rdfragcatalog.FragCatalog
>>> fpgen = FragmentCatalog.FragFPGenerator() >>> fp = fpgen.GetFPForMol(ms[8],fcat) >>> fp <rdkit.DataStructs.cDataStructs.ExplicitBitVect object at 0x...> >>> fp.GetNumOnBits() 189
これで、フィンガープリントに関連づけられた残りの機能をフラグメントフィンガープリントに適用することができるようになりました。例えば、二つの分子について、それぞれのフィンガープリントの積集合をとることで、共通して存在するフラグメントを簡単に見つけることができます:
>>> fp2 = fpgen.GetFPForMol(ms[7],fcat) >>> andfp = fp&fp2 >>> obl = list(andfp.GetOnBits()) >>> fcat.GetEntryDescription(obl[-1]) 'ccc(cc)NC<=O>' >>> fcat.GetEntryDescription(obl[-5]) 'c<-X>ccc(N)cc'
また、ある分子を別の分子と区別するフラグメントを見つけることもできます:
>>> combinedFp=fp&(fp^fp2) # can be more efficent than fp&(!fp2) >>> obl = list(combinedFp.GetOnBits()) >>> fcat.GetEntryDescription(obl[-1]) 'cccc(N)cc'
rdkit.ML.InfoTheory.rdInfoTheory.InfoBitRankerクラスのビットランキング機能を使って活性化合物を不活性化合物と区別するフラグメントを見つけ出すこともできます。
[link] rdkit.ML.InfoTheory.rdInfoTheory.InfoBitRanker
>>> suppl = Chem.SDMolSupplier('data/bzr.sdf') >>> sdms = [x for x in suppl] >>> fps = [fpgen.GetFPForMol(x,fcat) for x in sdms] >>> from rdkit.ML.InfoTheory import InfoBitRanker >>> ranker = InfoBitRanker(len(fps[0]),2) >>> acts = [float(x.GetProp('ACTIVITY')) for x in sdms] >>> for i,fp in enumerate(fps): ... act = int(acts[i]>7) ... ranker.AccumulateVotes(fp,act) ... >>> top5 = ranker.GetTopN(5) >>> for id,gain,n0,n1 in top5: ... print(int(id),'%.3f'%gain,int(n0),int(n1)) ... 702 0.081 20 17 328 0.073 23 25 341 0.073 30 43 173 0.073 30 43 1034 0.069 5 53
上記の例で、各列はbitId、infoGain、nInactive、nActiveを表します。上記モデルケースではこの手法がさほどうまくいっていないことに注意してください。
12/20/2018
このブログ記事のライセンス
以上はRDKit Documentationの試訳です。
ライセンスはCC BY-SA 4.0で、Greg Landrum氏の功績の元に作成しています。
Creative Commons — Attribution-ShareAlike 4.0 International — CC BY-SA 4.0
Chemical Features and Pharmacophores 〜RDKit 直訳 Day19〜
(12/30追記)試訳をまとめたテストサイトを作成しました。よろしければご参照ください。
こちらはRDKit直訳 Advent Calendar 2018 - Adventar 19日目の記事です。基本的な進め方は1日目の記事をご覧ください。
本日の訳出に困った用語
Chemical Features: 化学的特徴
Pharmacophore: ファーマコフォア
feature: フィーチャー
feature definition language: フィーチャー定義言語
feature factory: 特徴工場、フィーチャーファクトリ
bin: ビン分割
topological distance: 幾何学的距離
unique integer: 一意の整数値
encoding: 符号化、エンコーディング、コード化
feature-typing mechanism: フィーチャータイプ化機能
signature (fingerprint) factory: シグネチャー(フィンガープリント)ファクトリ
以下、訳
化学的特徴とファーマコフォア(Chemical Features and Pharmacophores)
[Link] https://www.rdkit.org/docs/GettingStartedInPython.html#chemical-features-and-pharmacophores
化学的特徴(Chemical Features)
[Link] https://www.rdkit.org/docs/GettingStartedInPython.html#chemical-features
RDKitにおいて化学的特徴(フィーチャー)は、SMARTSに基づくフィーチャー定義言語を使って定義されています(言語の詳細はthe RDKit bookに記載されています)。分子の化学的特徴を見つけ出すためには、まず初めにフィーチャーファクトリ[feature factory]を作らなければなりません:
>>> from rdkit import Chem >>> from rdkit.Chem import ChemicalFeatures >>> from rdkit import RDConfig >>> import os >>> fdefName = os.path.join(RDConfig.RDDataDir,'BaseFeatures.fdef') >>> factory = ChemicalFeatures.BuildFeatureFactory(fdefName)
次に、作成したファクトリーをつかってフィーチャーを検索します:
>>> m = Chem.MolFromSmiles('OCc1ccccc1CN') >>> feats = factory.GetFeaturesForMol(m) >>> len(feats) 8
個々のフィーチャーには、それが属するファミリー(たとえばドナーやアクセプターなど)やタイプ(より詳細な説明)、フィーチャーと結びつけられた原子(原子団)についての情報が紐付けられています:
>>> feats[0].GetFamily() 'Donor' >>> feats[0].GetType() 'SingleAtomDonor' >>> feats[0].GetAtomIds() (0,) >>> feats[4].GetFamily() 'Aromatic' >>> feats[4].GetAtomIds() (2, 3, 4, 5, 6, 7)
もし分子に座標の情報が含まれているなら、座標の中でフィーチャーが占める合理的な位置についての情報も紐付けられます:
>>> from rdkit.Chem import AllChem >>> AllChem.Compute2DCoords(m) 0 >>> feats[0].GetPos() <rdkit.Geometry.rdGeometry.Point3D object at 0x...> >>> list(feats[0].GetPos()) [2.07..., -2.335..., 0.0]
2Dファーマコフォアフィンガープリント(2D Pharmacophore Fingerprints)
[Link] https://www.rdkit.org/docs/GettingStartedInPython.html#d-pharmacophore-fingerprints
複数の化学的特徴と、フィーチャー間の2Dの(幾何学的、トポロジカル)距離とを合わせることで2Dファーマコフォアを作成することができます。距離がビン分割されると、それぞれのファーマコフォアに一意の整数値のIDを割り当てることができ、フィンガープリントにその情報を格納することができます。エンコーディングの詳細はThe RDKit Bookを参照してください。
[link] The RDKit book
ファーマコフォアフィンガープリントを生成するには、通常のRDKitのフィーチャータイプ化機能で作成した化学的特徴が必要です:
>>> from rdkit import Chem >>> from rdkit.Chem import ChemicalFeatures >>> fdefName = 'data/MinimalFeatures.fdef' >>> featFactory = ChemicalFeatures.BuildFeatureFactory(fdefName)
フィンガープリント自体は、ファーマコフォアを生成する際に必要だったすべてのパラメーターを保持しているシグネチャー(フィンガープリント)ファクトリを使って計算されます。
>>> from rdkit.Chem.Pharm2D.SigFactory import SigFactory >>> sigFactory = SigFactory(featFactory,minPointCount=2,maxPointCount=3) >>> sigFactory.SetBins([(0,2),(2,5),(5,8)]) >>> sigFactory.Init() >>> sigFactory.GetSigSize() 885
これでフィンガープリントを生成するシグネチャーファクトリーの準備が整いました。rdkit.Chem.Pharm2D.Generateモジュールを使って行います。
[link] rdkit.Chem.Pharm2D.Generate
>>> from rdkit.Chem.Pharm2D import Generate >>> mol = Chem.MolFromSmiles('OCC(=O)CCCN') >>> fp = Generate.Gen2DFingerprint(mol,sigFactory) >>> fp <rdkit.DataStructs.cDataStructs.SparseBitVect object at 0x...> >>> len(fp) 885 >>> fp.GetNumOnBits() 57
含まれるフィーチャーやビン分割されたフィーチャー間の距離行列といった情報も含めて、ビット自体の詳細についてはシグネチャーファクトリから得ることができます
>>> list(fp.GetOnBits())[:5] [1, 2, 6, 7, 8] >>> sigFactory.GetBitDescription(1) 'Acceptor Acceptor |0 1|1 0|' >>> sigFactory.GetBitDescription(2) 'Acceptor Acceptor |0 2|2 0|' >>> sigFactory.GetBitDescription(8) 'Acceptor Donor |0 2|2 0|' >>> list(fp.GetOnBits())[-5:] [704, 706, 707, 708, 714] >>> sigFactory.GetBitDescription(707) 'Donor Donor PosIonizable |0 1 2|1 0 1|2 1 0|' >>> sigFactory.GetBitDescription(714) 'Donor Donor PosIonizable |0 2 2|2 0 0|2 0 0|'
作業を簡便にするため(毎回fdefファイルを編集する必要をなくすため)に、SigFactoryの特定のフィーチャータイプを無効化することもできます。
>>> sigFactory.skipFeats=['PosIonizable'] >>> sigFactory.Init() >>> sigFactory.GetSigSize() 510 >>> fp2 = Generate.Gen2DFingerprint(mol,sigFactory) >>> fp2.GetNumOnBits() 36
RDKitで使用可能な、2Dファーマコフォアフィンガープリントのためのフィーチャー定義のセットにはもう一つあり、GobbiとPoppingerによって提案されたものです[脚注10]。rdkit.Chem.Pharm2D.Gobbi_Pharm2Dモジュールにこれらのフィンガープリントタイプのために予め設定されたシグネチャーファクトリがあります。使い方の例をお示しします:
[脚注10] Gobbi, A. & Poppinger, D. “Genetic optimization of combinatorial libraries.” Biotechnology and Bioengineering 61:47-54 (1998).
[link] rdkit.Chem.Pharm2D.Gobbi_Pharm2D
>>> from rdkit import Chem >>> from rdkit.Chem.Pharm2D import Gobbi_Pharm2D,Generate >>> m = Chem.MolFromSmiles('OCC=CC(=O)O') >>> fp = Generate.Gen2DFingerprint(m,Gobbi_Pharm2D.factory) >>> fp <rdkit.DataStructs.cDataStructs.SparseBitVect object at 0x...> >>> fp.GetNumOnBits() 8 >>> list(fp.GetOnBits()) [23, 30, 150, 154, 157, 185, 28878, 30184] >>> Gobbi_Pharm2D.factory.GetBitDescription(157) 'HA HD |0 3|3 0|' >>> Gobbi_Pharm2D.factory.GetBitDescription(30184) 'HA HD HD |0 3 0|3 0 3|0 3 0|'
12/19/2018
このブログ記事のライセンス
以上はRDKit Documentationの試訳です。
ライセンスはCC BY-SA 4.0で、Greg Landrum氏の功績の元に作成しています。
Creative Commons — Attribution-ShareAlike 4.0 International — CC BY-SA 4.0
Chemical Reactions part2 〜RDKit 直訳 Day18〜
(12/30追記)試訳をまとめたテストサイトを作成しました。よろしければご参照ください。
こちらはRDKit直訳 Advent Calendar 2018 - Adventar 18日目の記事です。基本的な進め方は1日目の記事をご覧ください。
本日の訳出に困った用語
dummy atom: ダミーアトム
generator object: ジェネレーターオブジェクト
valence: 原子価
generic function: ジェネリック関数
attachment point: 接着点
unsigned integer: 符号なし整数
以下、訳
BRICSの実装(BRICS Implemetation)
[Link] https://www.rdkit.org/docs/GettingStartedInPython.html#brics-implementation
RDKitはBRICSアルゴリズムの実装も提供しています[脚注9]。BRICSは合成的に利用可能な結合に沿って分子をフラグメント化するもう一つの方法です:
[脚注9] Degen, J.; Wegscheid-Gerlach, C.; Zaliani, A; Rarey, M. “On the Art of Compiling and Using ‘Drug-Like’ Chemical Fragment Spaces.” ChemMedChem 3:1503–7 (2008).
>>> from rdkit.Chem import BRICS >>> cdk2mols = Chem.SDMolSupplier('data/cdk2.sdf') >>> m1 = cdk2mols[0] >>> sorted(BRICS.BRICSDecompose(m1)) ['[14*]c1nc(N)nc2[nH]cnc12', '[3*]O[3*]', '[4*]CC(=O)C(C)C'] >>> m2 = cdk2mols[20] >>> sorted(BRICS.BRICSDecompose(m2)) ['[1*]C(=O)NN(C)C', '[14*]c1[nH]nc2c1C(=O)c1c([16*])cccc1-2', '[16*]c1ccc([16*])cc1', '[3*]OC', '[5*]N[5*]']
RDKitのBRICSの実装では、分子から作られたそれぞれのユニークなフラグメントを返します。また、ダミーアトムに、どのタイプの反応が適用されたかを示すタグがつけられたうえで返されます。
分子の集合に対して全てのフラグメントのリストを生成することがとても簡単にできます:
>>> allfrags=set() >>> for m in cdk2mols: ... pieces = BRICS.BRICSDecompose(m) ... allfrags.update(pieces) >>> len(allfrags) 90 >>> sorted(allfrags)[:5] ['NS(=O)(=O)c1ccc(N/N=C2\\C(=O)Nc3ccc(Br)cc32)cc1', '[1*]C(=O)C(C)C', '[1*]C(=O)NN(C)C', '[1*]C(=O)NN1CC[NH+](C)CC1', '[1*]C(C)=O']
BRICSモジュールは、フラグメントの集合にBRICSのルールを適用して新しい分子を生成するオプションも提供しています:
>>> import random >>> random.seed(127) >>> fragms = [Chem.MolFromSmiles(x) for x in sorted(allfrags)] >>> ms = BRICS.BRICSBuild(fragms)
結果はジェネレーターオブジェクトです:
>>> ms
<generator object BRICSBuild at 0x...>
リクエストに応じて分子を返します:
>>> prods = [next(ms) for x in range(10)] >>> prods[0] <rdkit.Chem.rdchem.Mol object at 0x...>
返された分子はサニタイズされていないので、処理を続ける前に少なくとも原子価を更新した方が良いでしょう:
>>> for prod in prods: ... prod.UpdatePropertyCache(strict=False) ... >>> Chem.MolToSmiles(prods[0],True) 'COCCO' >>> Chem.MolToSmiles(prods[1],True) 'O=C1Nc2ccc3ncsc3c2/C1=C/NCCO' >>> Chem.MolToSmiles(prods[2],True) 'O=C1Nc2ccccc2/C1=C/NCCO'
他のフラグメント化手法(Other fragmentation approaches)
[Link] https://www.rdkit.org/docs/GettingStartedInPython.html#other-fragmentation-approaches
上述の方法に加えて、RDKitはユーザーが指定した結合に沿って分子をフラグメント化する非常に柔軟なジェネリック関数を提供しています。
ここでは、簡単なデモンストレーションとして、環構造の中に含まれる原子と含まれない原子との間の結合を全て切断するという場合を試してみましょう。まずはペアとなる原子の組み合わせをすべて見つけることから始めます。
>>> m = Chem.MolFromSmiles('CC1CC(O)C1CCC1CC1') >>> bis = m.GetSubstructMatches(Chem.MolFromSmarts('[!R][R]')) >>> bis ((0, 1), (4, 3), (6, 5), (7, 8))
次に、原子のペアに一致する結合のインデックスを取得します:
>>> bs = [m.GetBondBetweenAtoms(x,y).GetIdx() for x,y in bis] >>> bs [0, 3, 5, 7]
そして、これらの結合のインデックスをフラグメント化関数のインプットとして使います:
>>> nm = Chem.FragmentOnBonds(m,bs)
アウトプットは、結合が切断された場所を示すダミーアトムを有する分子です:
>>> Chem.MolToSmiles(nm,True) '*C1CC([4*])C1[6*].[1*]C.[3*]O.[5*]CC[8*].[7*]C1CC1'
デフォルトでは、接着点は取り除かれた原子のインデックスとともに、(同位体[isotope]を使って)ラベルされています。独自の原子のラベルのセットを、符号なし整数のペアという形で設定することもできます。それぞれのペアの最初の値は、結合の出発点となる原子を置き換えるダミーのためのラベルとして使われ、ペアの2番目の値は、結合の終点となる原子を置き換えるダミーのためのラベルとして使われます。以下の例を見てみましょう。上述の解析を再度行い、接着点に関して、環構造に含まれない原子の場所は10とラベルをつけ、環構造の中の原子には1とラベルをつけています:
>>> bis = m.GetSubstructMatches(Chem.MolFromSmarts('[!R][R]')) >>> bs = [] >>> labels=[] >>> for bi in bis: ... b = m.GetBondBetweenAtoms(bi[0],bi[1]) ... if b.GetBeginAtomIdx()==bi[0]: ... labels.append((10,1)) ... else: ... labels.append((1,10)) ... bs.append(b.GetIdx()) >>> nm = Chem.FragmentOnBonds(m,bs,dummyLabels=labels) >>> Chem.MolToSmiles(nm,True) '[1*]C.[1*]CC[1*].[1*]O.[10*]C1CC([10*])C1[10*].[10*]C1CC1'
12/18/2018
このブログ記事のライセンス
以上はRDKit Documentationの試訳です。
ライセンスはCC BY-SA 4.0で、Greg Landrum氏の功績の元に作成しています。
Creative Commons — Attribution-ShareAlike 4.0 International — CC BY-SA 4.0
Chemical Reactions part1 〜RDKit 直訳 Day17〜
(12/30追記)試訳をまとめたテストサイトを作成しました。よろしければご参照ください。
こちらはRDKit直訳 Advent Calendar 2018 - Adventar 17日目の記事です。基本的な進め方は1日目の記事をご覧ください。
本日の訳出に困った用語
SMARTS-based language: SMARTSベースの記法
MDL rxn files: MDL rxnファイル
unique products: 単一の生成物
sanitize: サニタイズ
the hierarchy of transformations: 変換の階層構造
dummy atom: ダミーアトム
以下、訳
化学反応(Chemical Reactions)
[Link] https://www.rdkit.org/docs/GettingStartedInPython.html#chemical-reactions
RDKitは分子の集合に対して化学反応を適用することもサポートしています。化学反応を構築する一つの方法は、Daylight社のReaction SMILES [脚注11] に似たSMARTSベースの記法を使うことです。:
[脚注11] A more detailed description of reaction smarts, as defined by the rdkit, is in the The RDKit Book.
[link] The RDKit Book
>>> rxn = AllChem.ReactionFromSmarts('[C:1](=[O:2])-[OD1].[N!H0:3]>>[C:1](=[O:2])[N:3]') >>> rxn <rdkit.Chem.rdChemReactions.ChemicalReaction object at 0x...> >>> rxn.GetNumProductTemplates() 1 >>> ps = rxn.RunReactants((Chem.MolFromSmiles('CC(=O)O'),Chem.MolFromSmiles('NC'))) >>> len(ps) # one entry for each possible set of products 1 >>> len(ps[0]) # each entry contains one molecule for each product 1 >>> Chem.MolToSmiles(ps[0][0]) 'CNC(C)=O' >>> ps = rxn.RunReactants((Chem.MolFromSmiles('C(COC(=O)O)C(=O)O'),Chem.MolFromSmiles('NC'))) >>> len(ps) 2 >>> Chem.MolToSmiles(ps[0][0]) 'CNC(=O)OCCC(=O)O' >>> Chem.MolToSmiles(ps[1][0]) 'CNC(=O)CCOC(=O)O
MDL rxnファイルから反応を構築することもできます:
>>> rxn = AllChem.ReactionFromRxnFile('data/AmideBond.rxn') >>> rxn.GetNumReactantTemplates() 2 >>> rxn.GetNumProductTemplates() 1 >>> ps = rxn.RunReactants((Chem.MolFromSmiles('CC(=O)O'), Chem.MolFromSmiles('NC'))) >>> len(ps) 1 >>> Chem.MolToSmiles(ps[0][0]) 'CNC(C)=O'
もちろんアミド結合の形成よりももっと複雑な反応を行うこともできます:
>>> rxn = AllChem.ReactionFromSmarts('[C:1]=[C:2].[C:3]=[*:4][*:5]=[C:6]>>[C:1]1[C:2][C:3][*:4]=[*:5][C:6]1') >>> ps = rxn.RunReactants((Chem.MolFromSmiles('OC=C'), Chem.MolFromSmiles('C=CC(N)=C'))) >>> Chem.MolToSmiles(ps[0][0]) 'NC1=CCCC(O)C1'
この場合、テンプレートへの反応物質のマッピングが1通りではないので、得られるものが複数の生成物を含むセットであることに注意してください:
>>> len(ps) 4
カノニカルSMILESとpythonの辞書型を使うことで、それぞれのユニークな生成物を得ることができます。
>>> uniqps = {}
>>> for p in ps:
... smi = Chem.MolToSmiles(p[0])
... uniqps[smi] = p[0]
...
>>> sorted(uniqps.keys())
['NC1=CCC(O)CC1', 'NC1=CCCC(O)C1']
化学反応を処理するコードから生成される分子はサニタイズされていないことに注意して下さい。あえて不自然な反応例をつかって示します:
>>> rxn = AllChem.ReactionFromSmarts('[C:1]=[C:2][C:3]=[C:4].[C:5]=[C:6]>>[C:1]1=[C:2][C:3]=[C:4][C:5]=[C:6]1') >>> ps = rxn.RunReactants((Chem.MolFromSmiles('C=CC=C'), Chem.MolFromSmiles('C=C'))) >>> Chem.MolToSmiles(ps[0][0]) 'C1=CC=CC=C1' >>> p0 = ps[0][0] >>> Chem.SanitizeMol(p0) rdkit.Chem.rdmolops.SanitizeFlags.SANITIZE_NONE >>> Chem.MolToSmiles(p0) 'c1ccccc1'
より高度な反応機能(Advanced Reaction Functionality)
[Link] https://www.rdkit.org/docs/GettingStartedInPython.html#advanced-reaction-functionality
原子の保護(Protecting Atoms)
[Link] https://www.rdkit.org/docs/GettingStartedInPython.html#protecting-atoms
ありえない生成物を生み出してしまわないように十分に正確に反応を表現することは、時折難しく、特にrxnファイルを扱っているときにこのような問題が生じます。RDKitでは原子を"保護する"方法を提供しており、その原子が反応に参加しないようにすることができます。
上のセクジョンで用いたアミド結合形成の反応を再度用いてデモンストレーションしてみましょう。アミンのクエリが十分に明確ではなく、少なくとも一つの水素原子の結合した窒素原子はどれでもマッチしてしまいます。したがって、この反応をすでにアミド結合を有する分子に適用した場合、アミドのNも反応点として扱われてしまいます:
>>> rxn = AllChem.ReactionFromRxnFile('data/AmideBond.rxn') >>> acid = Chem.MolFromSmiles('CC(=O)O') >>> base = Chem.MolFromSmiles('CC(=O)NCCN') >>> ps = rxn.RunReactants((acid,base)) >>> len(ps) 2 >>> Chem.MolToSmiles(ps[0][0]) 'CC(=O)N(CCN)C(C)=O' >>> Chem.MolToSmiles(ps[1][0]) 'CC(=O)NCCNC(C)=O'
最初の生成物がアミドのNで反応が起きたものに相当します。
全てのアミドのNを保護することで、このような事態が生じるのを防ぐことができます。ここでは、アミドとチオアミドにマッチする部分構造クエリをつかって保護を行い、マッチする原子に"_protected"プロパティをセットしましょう:
>>> amidep = Chem.MolFromSmarts('[N;$(NC=[O,S])]') >>> for match in base.GetSubstructMatches(amidep): ... base.GetAtomWithIdx(match[0]).SetProp('_protected','1')
これで、たった一つだけ生成物を生じるようになります:
>>> ps = rxn.RunReactants((acid,base)) >>> len(ps) 1 >>> Chem.MolToSmiles(ps[0][0]) 'CC(=O)NCCNC(C)=O'
Recapの実装(Recap Implementation)
[Link] https://www.rdkit.org/docs/GettingStartedInPython.html#recap-implementation
化学反応機能と関連して、Recapアルゴリズムの実装を取り上げます。[脚注8] Recapは、実験室でよく使われる反応を模倣した化学変換のセットを使って、分子を一連の合理的なフラグメントに分解します。
[脚注8] Lewell, X.Q.; Judd, D.B.; Watson, S.P.; Hann, M.M. “RECAP-Retrosynthetic Combinatorial Analysis Procedure: A Powerful New Technique for Identifying Privileged Molecular Fragments with Useful Applications in Combinatorial Chemistry” J. Chem. Inf. Comp. Sci. 38:511-22 (1998).
RDKitのrdkit.Chem.Recapの実装は、適用される変換の階層構造の流れを保存します。
[link] rdkit.Chem.Recap
>>> from rdkit import Chem >>> from rdkit.Chem import Recap >>> m = Chem.MolFromSmiles('c1ccccc1OCCOC(=O)CC') >>> hierarch = Recap.RecapDecompose(m) >>> type(hierarch) <class 'rdkit.Chem.Recap.RecapHierarchyNode'>
階層構造の起点は元々の分子です:
>>> hierarch.smiles
'CCC(=O)OCCOc1ccccc1'
それぞれのノードはSMILESをキーとする辞書型を使って、子ノードの情報を保持します。
>>> ks=hierarch.children.keys() >>> sorted(ks) ['*C(=O)CC', '*CCOC(=O)CC', '*CCOc1ccccc1', '*OCCOc1ccccc1', '*c1ccccc1']
階層構造の末端のノード(葉ノード)は簡単にアクセスすることができ、これもSMILESをキーとする辞書型となっています。
>>> ks=hierarch.GetLeaves().keys() >>> ks=sorted(ks) >>> ks ['*C(=O)CC', '*CCO*', '*CCOc1ccccc1', '*c1ccccc1']
分子がフラグメント化された場所では、ダミーアトムをつかって印がつけられていることに注意して下さい。
ノード自体には関連づけられた分子が入っています:
>>> leaf = hierarch.GetLeaves()[ks[0]] >>> Chem.MolToSmiles(leaf.mol) '*C(=O)CC'
12/17/2018
このブログ記事のライセンス
以上はRDKit Documentationの試訳です。
ライセンスはCC BY-SA 4.0で、Greg Landrum氏の功績の元に作成しています。
Creative Commons — Attribution-ShareAlike 4.0 International — CC BY-SA 4.0
RDKit Cookbook 〜RDKit 直訳 Day16〜
(12/30追記)試訳をまとめたテストサイトを作成しました。よろしければご参照ください。
こちらはRDKit直訳 Advent Calendar 2018 - Adventar 16日目の記事です。基本的な進め方は1日目の記事をご覧ください。
本日の訳出に困った用語
align: アラインメントを取る
shape comparison: 形状比較
Shape protrude distance: 形状はみ出し距離
Shape Tanimoto distance: 形状タニモト距離
volume: 体積
3D distance matrix: 3D距離行列
2D-pharmacophore machinery: 2Dファーマコフォア機能
Tanimoto similarity: タニモト類似度
approach: 手法
rotatable bond: 回転可能な結合
ring system: 環系
torsional angle: 二面角
deviation: 偏差
normalization: 規格化
Butina clustering: Butinaクラスタリング
reordering: 並べ替え
helper function: ヘルパー関数
bogus: 偽の、インチキの
shebang: シバン(UNIXのスクリプトの#!から始まる1行目のこと)
TL;DR : too long didn't read(長すぎたから読まなかった)の略語、転じて要約の意。
以下、訳
RDKitクックブック(RDKit Cookbook)
[Link] https://www.rdkit.org/docs/Cookbook.html#rdkit-cookbook
このページについて(What is this?)
[Link] https://www.rdkit.org/docs/Cookbook.html#what-is-this
このページはPythonからRDKitの機能を使って特定のタスクをどうやって実行すれば良いか例を示します。このコンテンツはRDKitコミュニティの協力で作られています。
間違いを見つけたり、より良い方法を提案する場合は、ソースドキュメント(.rstファイル)をご自身で修正していただくか、メーリングリスト: rdkit-discuss@lists.sourceforge.net に送ってください。(メーリングリストを利用する場合はまず加入する必要があります。)
雑多な話題(Miscellaneous Topics)
[Link] https://www.rdkit.org/docs/Cookbook.html#miscellaneous-topics
異なる芳香族性モデルを使う方法(Using a differnt aromaticity model)
[Link] https://www.rdkit.org/docs/Cookbook.html#using-a-different-aromaticity-model
デフォルトでは、RDKitは分子を読み込むときにRDKit自体の芳香族性モデル(RDKit Theory Bookで説明されています)を適用します。ですが、このモデルを上書きし、自分の芳香族性モデルを使うのはとても簡単です。
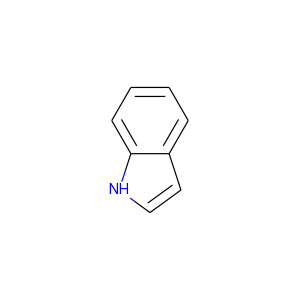
最も簡単な方法は、保持してほしい芳香族性のセットと合わせてSMILESで分子を提供することです。例えばインドールの場合を考えましょう:
indole1.png
デフォルトではRDKitはどちらの環構造も芳香族であると認識します:
>>> from rdkit import Chem >>> m = Chem.MolFromSmiles('N1C=Cc2ccccc12') >>> m.GetSubstructMatches(Chem.MolFromSmarts('c')) ((1,), (2,), (3,), (4,), (5,), (6,), (7,), (8,))
5員環を脂肪族として扱いたいなら、これは入力のSMILESがどのように書かれているかという問題ですが、ケクレ化[kekulization]と芳香族性を認識する過程をスキップする、部分的なサニタイゼーションを行う必要があるだけです:
>>> m2 = Chem.MolFromSmiles('N1C=Cc2ccccc12',sanitize=False) >>> Chem.SanitizeMol(m2,sanitizeOps=Chem.SanitizeFlags.SANITIZE_ALL^Chem.SanitizeFlags.SANITIZE_KEKULIZE^Chem.SanitizeFlags.SANITIZE_SETAROMATICITY) rdkit.Chem.rdmolops.SanitizeFlags.SANITIZE_NONE >>> m2.GetSubstructMatches(Chem.MolFromSmarts('c')) ((3,), (4,), (5,), (6,), (7,), (8,))
もちろん自分の芳香族性認識関数を書くことも可能ですが、このドキュメントで扱う範囲を超えています。
分子の操作(Manipulating Molecules)
[Link] https://www.rdkit.org/docs/Cookbook.html#manipulating-molecules
ヘテロ環のクリーンアップ(Cleaning up heterocycles)
[Link] https://www.rdkit.org/docs/Cookbook.html#cleaning-up-heterocycles
メーリングリストの議論; * http://www.mail-archive.com/rdkit-discuss@lists.sourceforge.net/msg01185.html * http://www.mail-archive.com/rdkit-discuss@lists.sourceforge.net/msg01162.html * http://www.mail-archive.com/rdkit-discuss@lists.sourceforge.net/msg01900.html * http://www.mail-archive.com/rdkit-discuss@lists.sourceforge.net/msg01901.html
コード:
使用例:
結果:
並列化したコンフォメーション生成(Parallel conformation generation)
[Link] https://www.rdkit.org/docs/Cookbook.html#parallel-conformation-generation
メーリングリストでのディスカッション: http://www.mail-archive.com/rdkit-discuss@lists.sourceforge.net/msg02648.html
コード:
""" オリジナルはAndrew Dalke氏によるものです """ import sys from rdkit import Chem from rdkit.Chem import AllChem # これは右のリンクからダウンロードできます http://pypi.python.org/pypi/futures from concurrent import futures # これは右のリンクからダウンロードできます http://pypi.python.org/pypi/progressbar import progressbar ## 私のマシンではwoker1つで39秒、4つで10秒かかりました。 ## 29.055u 0.102s 0:28.68 101.6% 0+0k 0+3io 0pf+0w #max_workers=1 ## スレッド4つでは11秒かかりました。 ## 34.933u 0.188s 0:10.89 322.4% 0+0k 125+1io 0pf+0w max_workers=4 # (ユーザータイム(u)は子プロセスに使われた時間も含みます # 実時間はそれぞれ28.68秒と10.89でした。) # この関数はサブプロセスで呼び出されます。 # パラメータ(分子とコンフォマーの数)はPythonを介して渡されます。 def generateconformations(m, n): m = Chem.AddHs(m) ids=AllChem.EmbedMultipleConfs(m, numConfs=n, params=AllChem.ETKDG()) # EmbedMultipleConfsはBoost-wrappedタイプを返しますが、これはピッケル化できません。 # Pythonのリストに変換することはできます。 return m, list(ids) smi_input_file, sdf_output_file = sys.argv[1:3] n = int(sys.argv[3]) writer = Chem.SDWriter(sdf_output_file) suppl = Chem.SmilesMolSupplier(smi_input_file, titleLine=False) with futures.ProcessPoolExecutor(max_workers=max_workers) as executor: # 非同期性のジョブのセットを投げます。 jobs = [] for mol in suppl: if mol: job = executor.submit(generateconformations, mol, n) jobs.append(job) widgets = ["Generating conformations; ", progressbar.Percentage(), " ", progressbar.ETA(), " ", progressbar.Bar()] pbar = progressbar.ProgressBar(widgets=widgets, maxval=len(jobs)) for job in pbar(futures.as_completed(jobs)): mol,ids=job.result() for id in ids: writer.write(mol, confId=id) writer.close()
電荷を帯びた分子の中和(Neutralizing Charged Molecules)
[Link] https://www.rdkit.org/docs/Cookbook.html#neutralizing-charged-molecules
メーリングリストでのディスカッション:http://www.mail-archive.com/rdkit-discuss@lists.sourceforge.net/msg02669.html
コード:
""" Hans de Winter氏によるものです """ from rdkit import Chem from rdkit.Chem import AllChem def _InitialiseNeutralisationReactions(): patts= ( # Imidazoles ('[n+;H]','n'), # Amines ('[N+;!H0]','N'), # Carboxylic acids and alcohols ('[$([O-]);!$([O-][#7])]','O'), # Thiols ('[S-;X1]','S'), # Sulfonamides ('[$([N-;X2]S(=O)=O)]','N'), # Enamines ('[$([N-;X2][C,N]=C)]','N'), # Tetrazoles ('[n-]','[nH]'), # Sulfoxides ('[$([S-]=O)]','S'), # Amides ('[$([N-]C=O)]','N'), ) return [(Chem.MolFromSmarts(x),Chem.MolFromSmiles(y,False)) for x,y in patts] _reactions=None def NeutraliseCharges(smiles, reactions=None): global _reactions if reactions is None: if _reactions is None: _reactions=_InitialiseNeutralisationReactions() reactions=_reactions mol = Chem.MolFromSmiles(smiles) replaced = False for i,(reactant, product) in enumerate(reactions): while mol.HasSubstructMatch(reactant): replaced = True rms = AllChem.ReplaceSubstructs(mol, reactant, product) mol = rms[0] if replaced: return (Chem.MolToSmiles(mol,True), True) else: return (smiles, False)
使用例:
smis=("c1cccc[nH+]1", "C[N+](C)(C)C","c1ccccc1[NH3+]", "CC(=O)[O-]","c1ccccc1[O-]", "CCS", "C[N-]S(=O)(=O)C", "C[N-]C=C","C[N-]N=C", "c1ccc[n-]1", "CC[N-]C(=O)CC") for smi in smis: (molSmiles, neutralised) = NeutraliseCharges(smi) print(smi + "->" + molSmiles)
結果:
c1cccc[nH+]1 -> c1ccncc1 C[N+](C)(C)C -> C[N+](C)(C)C c1ccccc1[NH3+] -> Nc1ccccc1 CC(=O)[O-] -> CC(=O)O c1ccccc1[O-] -> Oc1ccccc1 CCS -> CCS C[N-]S(=O)(=O)C -> CNS(C)(=O)=O C[N-]C=C -> C=CNC C[N-]N=C -> C=NNC c1ccc[n-]1 -> c1cc[nH]c1 CC[N-]C(=O)CC -> CCNC(=O)CC
RDKitの3D機能(3D functionality in the RDKit)
[Link] https://www.rdkit.org/docs/Cookbook.html#d-functionality-in-the-rdkit
RDKitには3Dを取り扱う広範囲な機能があります。たとえば:
- Shape alignment
- RMS Calculation
- Shape Tanimoto Distance
- Shape Protrude Distance
- 3D pharmacophore fingerprint
- Torsion fingerprint (deviation)
現在、RDKitでは利用可能なアラインメントのメソッドが2つあります。例として、PDBの同じ分子から2つの結晶構造を使います。
コード:
from rdkit import Chem, RDConfig from rdkit.Chem import AllChem, rdMolAlign # 参照となる分子(reference molecule) ref = Chem.MolFromSmiles('NC(=[NH2+])c1ccc(C[C@@H](NC(=O)CNS(=O)(=O)c2ccc3ccccc3c2)C(=O)N2CCCCC2)cc1') # PDBのコンフォメーション mol1 = Chem.MolFromPDBFile(RDConfig.RDBaseDir+'/rdkit/Chem/test_data/1DWD_ligand.pdb') mol1 = AllChem.AssignBondOrdersFromTemplate(ref, mol1) mol2 = Chem.MolFromPDBFile(RDConfig.RDBaseDir+'/rdkit/Chem/test_data/1PPC_ligand.pdb') mol2 = AllChem.AssignBondOrdersFromTemplate(ref, mol2) # アラインメントを取ります rms = rdMolAlign.AlignMol(mol1, mol2) print(rms) # OPEN3DAlignを使ってアラインメントを取ります pyO3A = rdMolAlign.GetO3A(mol1, mol2) score = pyO3A.Align() print(score)
結果:
1.55001955728 0.376459885045
分子に一つ以上のコンフォマーが含まれている場合、最初のコンフォマーに関してアラインメントを取ります。リストがオプションのRMSlistに渡された場合、アライメントによるRMS値が格納されます。一つの分子の二つのコンフォマーのRMS値は別々に計算することもでき、(prealignedフラグを使うことで)アライメント有り、無しのどちらでも計算可能です。
使用例:
from rdkit import Chem from rdkit.Chem import AllChem mol = Chem.MolFromSmiles('NC(=[NH2+])c1ccc(C[C@@H](NC(=O)CNS(=O)(=O)c2ccc3ccccc3c2)C(=O)N2CCCCC2)cc1') cids = AllChem.EmbedMultipleConfs(mol, numConfs=50, maxAttempts=1000, pruneRmsThresh=0.1) print(len(cids)) # コンフォマーのアラインメントを取ります rmslist = [] AllChem.AlignMolConformers(mol, RMSlist=rmslist) print(len(rmslist)) # コンフォマー1から9のRMSを別々に計算します rms = AllChem.GetConformerRMS(mol, 1, 9, prealigned=True)
結果:
50 49
形状比較のため、RDKitはあらかじめアラインメントされた分子あるいはコンフォマーに対して、形状に基づく距離[Shape-based distances]を2つ用意しています。形状はみ出し距離[Shape protrude distance]は体積のミスマッチに焦点をあてていて、形状タニモト距離[Shape Tanimoto distance]は重ね合わせた体積全体を考慮に入れます。
使用例:
from rdkit import Chem, RDConfig from rdkit.Chem import AllChem, rdMolAlign, rdShapeHelpers ref = Chem.MolFromSmiles('NC(=[NH2+])c1ccc(C[C@@H](NC(=O)CNS(=O)(=O)c2ccc3ccccc3c2)C(=O)N2CCCCC2)cc1') mol1 = Chem.MolFromPDBFile(RDConfig.RDBaseDir+'/rdkit/Chem/test_data/1DWD_ligand.pdb') mol1 = AllChem.AssignBondOrdersFromTemplate(ref, mol1) mol2 = Chem.MolFromPDBFile(RDConfig.RDBaseDir+'/rdkit/Chem/test_data/1PPC_ligand.pdb') mol2 = AllChem.AssignBondOrdersFromTemplate(ref, mol2) rms = rdMolAlign.AlignMol(mol1, mol2) tani = rdShapeHelpers.ShapeTanimotoDist(mol1, mol2) prtr = rdShapeHelpers.ShapeProtrudeDist(mol1, mol2) print(rms, tani, prtr)
結果:
1.55001955728 0.18069102331 0.0962800875274
3Dファーマコフォアフィンガープリントは、3D距離行列を2Dファーマコフォア機能に与えることで、RDKitを使って計算することができます。
使用例:
from rdkit import Chem, DataStructs, RDConfig from rdkit.Chem import AllChem from rdkit.Chem.Pharm2D import Gobbi_Pharm2D, Generate ref = Chem.MolFromSmiles('NC(=[NH2+])c1ccc(C[C@@H](NC(=O)CNS(=O)(=O)c2ccc3ccccc3c2)C(=O)N2CCCCC2)cc1') mol1 = Chem.MolFromPDBFile(RDConfig.RDBaseDir+'/rdkit/Chem/test_data/1DWD_ligand.pdb') mol1 = AllChem.AssignBondOrdersFromTemplate(ref, mol1) mol2 = Chem.MolFromPDBFile(RDConfig.RDBaseDir+'/rdkit/Chem/test_data/1PPC_ligand.pdb') mol2 = AllChem.AssignBondOrdersFromTemplate(ref, mol2) # ファーマコフォアフィンガープリント factory = Gobbi_Pharm2D.factory fp1 = Generate.Gen2DFingerprint(mol1, factory, dMat=Chem.Get3DDistanceMatrix(mol1)) fp2 = Generate.Gen2DFingerprint(mol2, factory, dMat=Chem.Get3DDistanceMatrix(mol2)) # Tanimoto類似度 tani = DataStructs.TanimotoSimilarity(fp1, fp2) print(tani)
結果:
0.451665312754
RDKitはSchulz-Gaschら(J. Chem. Inf. Model, 52, 1499, 2012)により開発された手法、トーションフィンガープリントデビエーション(torsion fingerprint deviation:TFD)も実装しています。一つの分子のコンフォマーのペアについて、回転可能な結合と環系の二面角をトーションフィンガープリント(torsion fingerprint: TF)に記録し、そしてTF間の偏差を計算、規格化、そして合計を取ります。各二面角について。a-b-c-dの4つの原子のセットが選ばれます。
RDKitの実装では、次に示すようにトーションフィンガープリントをカスタマイズすることが可能です。
- 元々の手法では、二面角は分子の中心に対する距離に基づいて重みづけられていました。デフォルトでは、この重みづけが実行されますが、useWeights=Falseフラグを使うことで、実行しないようにできます。
- 対称的な原子aとd(あるいは、aかd)が存在しているとき、全てのとりうる二面角が計算されます。2つの原子が対称的かどうか決定するため、与えられた半径(デフォルトは半径=2)のMorganアルゴリズムに基づくハッシュ・コードが使われます。
- 元々の方法では規格化に使われる最大の偏差は、全ての二面角に対して180.0度です(デフォルト)。maxDev='spec'とすることで、最大偏差に依存する二面角のタイプが規格化に使われます。
- 元々の方法では、三重結合とアレンに隣接する単結合は無視されます(デフォルト)。ignoreColinearBonds='False'とすることで、"連結された(combined)"二面角が使われます。
以上に加えて、Schulz-Gaschらによる方法と異なる点が2、3あります:
- 水素原子は決して考慮されません。
- 元々の方法では、原子bとc(あるいはbかc)に複数の非対称な隣接原子がある場合、原子aとd(あるいはaかd)はランダムに選ばれていました。RDKitの実装では最小のMorgan不変量をもつ原子を選びます。この方法では分子の原子の順番に選択する原子が依存しません。
- 原子aとd(あるいはaかd)が対称的な場合、元々の手法では最も小さい二面角のみを保存しますが、RDKitの実装では全ての取りうる二面角をTFに保存します。続いて、全ての取りうる偏差が決定され、そして最も小さいものがTFDの計算に使われます。この手順により最も小さい偏差がTFDに入ることを保証します。
使用例:
from rdkit import Chem, RDConfig from rdkit.Chem import AllChem, TorsionFingerprints ref = Chem.MolFromSmiles('NC(=[NH2+])c1ccc(C[C@@H](NC(=O)CNS(=O)(=O)c2ccc3ccccc3c2)C(=O)N2CCCCC2)cc1') mol1 = Chem.MolFromPDBFile(RDConfig.RDBaseDir+'/rdkit/Chem/test_data/1DWD_ligand.pdb') mol1 = AllChem.AssignBondOrdersFromTemplate(ref, mol1) mol2 = Chem.MolFromPDBFile(RDConfig.RDBaseDir+'/rdkit/Chem/test_data/1PPC_ligand.pdb') mol2 = AllChem.AssignBondOrdersFromTemplate(ref, mol2) tfd1 = TorsionFingerprints.GetTFDBetweenMolecules(mol1, mol2) tfd2 = TorsionFingerprints.GetTFDBetweenMolecules(mol1, mol2, useWeights=False) tfd3 = TorsionFingerprints.GetTFDBetweenMolecules(mol1, mol2, maxDev='spec') print(tfd1, tfd2, tfd3)
結果:
0.0691236990428 0.111475253992 0.0716255058804
同じ分子のコンフォマー間のTFDを計算する場合、性能上の理由でGetTFDBetweenConformers()関数を使うべきです。
使用例:
from rdkit import Chem, RDConfig from rdkit.Chem import AllChem, TorsionFingerprints ref = Chem.MolFromSmiles('NC(=[NH2+])c1ccc(C[C@@H](NC(=O)CNS(=O)(=O)c2ccc3ccccc3c2)C(=O)N2CCCCC2)cc1') mol1 = Chem.MolFromPDBFile(RDConfig.RDBaseDir+'/rdkit/Chem/test_data/1DWD_ligand.pdb') mol1 = AllChem.AssignBondOrdersFromTemplate(ref, mol1) mol2 = Chem.MolFromPDBFile(RDConfig.RDBaseDir+'/rdkit/Chem/test_data/1PPC_ligand.pdb') mol1.AddConformer(mol2.GetConformer(), assignId=True) tfd = TorsionFingerprints.GetTFDBetweenConformers(mol1, confIds1=[0], confIds2=[1]) print(tfd)
結果:
[0.0691...]
コンフォマーRMSとTFDの値のため、RDKitでは、Butinaクラスタリングといったクラスタリングアルゴリズムに与えることができる対称行列を直接計算する簡易関数を用意しています。フラグの並べ替えにより、クラスターが作られるたびに毎回、クラスタリングされていない分子について近接するものの数が更新されることを保証します。
使用例:
from rdkit import Chem from rdkit.Chem import AllChem, TorsionFingerprints from rdkit.ML.Cluster import Butina mol = Chem.MolFromSmiles('NC(=[NH2+])c1ccc(C[C@@H](NC(=O)CNS(=O)(=O)c2ccc3ccccc3c2)C(=O)N2CCCCC2)cc1') cids = AllChem.EmbedMultipleConfs(mol, numConfs=50, maxAttempts=1000, pruneRmsThresh=0.1) # RMS行列(RMS matrix) rmsmat = AllChem.GetConformerRMSMatrix(mol, prealigned=False) # TFD行列(TFD matrix) tfdmat = TorsionFingerprints.GetTFDMatrix(mol) # クラスタリング num = mol.GetNumConformers() rms_clusters = Butina.ClusterData(rmsmat, num, 2.0, isDistData=True, reordering=True) tfd_clusters = Butina.ClusterData(tfdmat, num, 0.3, isDistData=True, reordering=True)
RDKitと一緒にscikit-learnを使う方法(Using scikit-learn with RDKit)
[Link] https://www.rdkit.org/docs/Cookbook.html#using-scikit-learn-with-rdkit
scikit-learnはPythoのための機械学習のライブラリで、様々な教師あり、教師無しの手法を含みます。siki-learnに関するドキュメンテーションはこちらにあります:http://scikit-learn.org/stable/user_guide.html 。
RDKitのフィンガープリントはscikit-learnの機械学習モデルを訓練するのに使うことができます。これはランダムフォレストの例です。
コード:
from rdkit import Chem, DataStructs from rdkit.Chem import AllChem from sklearn.ensemble import RandomForestClassifier import numpy # 4つの分子を生成 m1 = Chem.MolFromSmiles('c1ccccc1') m2 = Chem.MolFromSmiles('c1ccccc1CC') m3 = Chem.MolFromSmiles('c1ccncc1') m4 = Chem.MolFromSmiles('c1ccncc1CC') mols = [m1, m2, m3, m4] # フィンガープリントを生成: 半径2のMorganフィンガープリント fps = [AllChem.GetMorganFingerprintAsBitVect(m, 2) for m in mols] # RDKitの明示的なベクトルをNumpyのアレイに変換 np_fps = [] for fp in fps: arr = numpy.zeros((1,)) DataStructs.ConvertToNumpyArray(fp, arr) np_fps.append(arr) # 100個のツリーをもつランダムフォレスト分類器を手に入れます rf = RandomForestClassifier(n_estimators=100, random_state=1123) # 最初の2つの分子を活性あり(active, class 1)、 # 残りの2つを活性無し(inactive, class 0)として、 # ランダムフォレストを訓練します ys_fit = [1, 1, 0, 0] rf.fit(np_fps, ys_fit) # 新しい分子を予測するためにランダムフォレストを使います m5 = Chem.MolFromSmiles('c1ccccc1O') fp = numpy.zeros((1,)) DataStructs.ConvertToNumpyArray(AllChem.GetMorganFingerprintAsBitVect(m5, 2), fp) print(rf.predict((fp,))) print(rf.predict_proba((fp,)))
scikit-learn バージョン0.13での出力結果は:
[1] [[ 0.14 0.86]]
このモデルに対して類似度マップを作成しましょう。
コードは:
from rdkit.Chem.Draw import SimilarityMaps # ヘルパー関数 def getProba(fp, predictionFunction): return predictionFunction((fp,))[0][1] m5 = Chem.MolFromSmiles('c1ccccc1O') fig, maxweight = SimilarityMaps.GetSimilarityMapForModel(m5, SimilarityMaps.GetMorganFingerprint, lambda x: getProba(x, rf.predict_proba))
これで、次の結果が返ってきます:
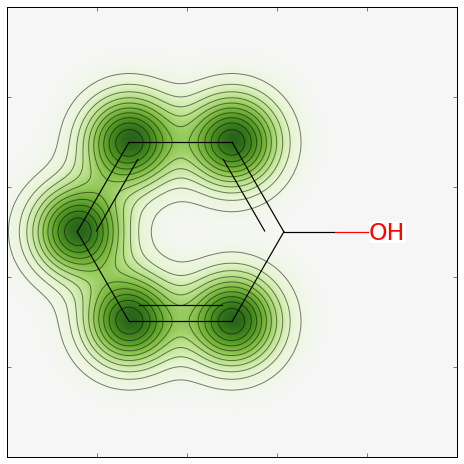
similarity_map_rf.png
カスタムMCSアトムタイプを使う方法(Using custom MCS atom types)
[Link] https://www.rdkit.org/docs/Cookbook.html#using-custom-mcs-atom-types
メーリングリストの議論: http://www.mail-archive.com/rdkit-discuss@lists.sourceforge.net/msg03676.html
IPython notebook:
http://nbviewer.ipython.org/gist/greglandrum/8351725
https://gist.github.com/greglandrum/8351725
MCSについては可読性のあるSMILESを手に入れることができますが、このセクションの目標はMCSコードでカスタムアトムタイプを使えるようになることです。マッチングに同位体の情報を使うために、MCSコードのオプションを使います。そして、我々の同位体の情報を含む偽の同位体の値をセットします。
コード:
from rdkit import Chem from rdkit.Chem import rdFMCS # テストに用いる分子: smis=["COc1ccc(C(Nc2nc3c(ncn3COCC=O)c(=O)[nH]2)(c2ccccc2)c2ccccc2)cc1", "COc1ccc(C(Nc2nc3c(ncn3COC(CO)(CO)CO)c(=O)[nH]2)(c2ccccc2)c2ccccc2)cc1"] ms = [Chem.MolFromSmiles(x) for x in smis] def label(a): " a simple hash combining atom number and hybridization " return 100*int(a.GetHybridization())+a.GetAtomicNum() # あとで分子を変えるので、コピーしておきます: nms = [Chem.Mol(x) for x in ms] for nm in nms: for at in nm.GetAtoms(): at.SetIsotope(label(at)) mcs=rdFMCS.FindMCS(nms,atomCompare=rdFMCS.AtomCompare.CompareIsotopes) print(mcs.smartsString)
これで次の出力が得られます:
[406*]-[308*]-[306*]1:[306*]:[306*]:[306*](:[306*]:[306*]:1)-[406*](-[307*]-[306*]1:[307*]:[306*]2:[306*](:[306*](:[307*]:1)=[308*]):[307*]:[306*]:[307*]:2-[406*]-[408*]-[406*])(-[306*]1:[306*]:[306*]:[306*]:[306*]:[306*]:1)-[306*]1:[306*]:[306*]:[306*]:[306*]:[306*]:1
これが我々が求めたものではありますが、必ずしも可読性の高いものではありません。2ステップのプロセスで、より読みやすい形式とすることができます:
- MCSの部分構造マッチングをコピーした分子に対して行う
- コピーとマッチした原子だけを使って、元々の分子のSMILESを生成する
我々はコピーと元々の分子のアトムインデックスが同じであると知っているので、これで上手くいきます。
def getMCSSmiles(mol,labelledMol,mcs): mcsp = Chem.MolFromSmarts(mcs.smartsString) match = labelledMol.GetSubstructMatch(mcsp) return Chem.MolFragmentToSmiles(mol,atomsToUse=match, isomericSmiles=True, canonical=False) print(getMCSSmiles(ms[0],nms[0],mcs)) COc1ccc(C(Nc2nc3c(ncn3COC)c(=O)[nH]2)(c2ccccc2)c2ccccc2)cc1
これが我々が探し求めていたものです。
分子のクラスタリング(Clustering molecules)
[Link] https://www.rdkit.org/docs/Cookbook.html#clustering-molecules
大きな分子のセット(1000から2000以上)には、Butinaクラスタリングアルゴリズムを使うのが最も効率が良いです。
これが、フィンガープリントのセットに対してクラスタリングを行うためのコードです:
def ClusterFps(fps,cutoff=0.2): from rdkit import DataStructs from rdkit.ML.Cluster import Butina # まず距離行列を生成します: dists = [] nfps = len(fps) for i in range(1,nfps): sims = DataStructs.BulkTanimotoSimilarity(fps[i],fps[:i]) dists.extend([1-x for x in sims]) # データをクラスタリングします: cs = Butina.ClusterData(dists,nfps,cutoff,isDistData=True) return cs
戻り値はクラスターのタプルで、各クラスターはidのタプルとなっています。
使用例:
from rdkit import Chem from rdkit.Chem import AllChem import gzip ms = [x for x in Chem.ForwardSDMolSupplier(gzip.open('zdd.sdf.gz')) if x is not None] fps = [AllChem.GetMorganFingerprintAsBitVect(x,2,1024) for x in ms] clusters=ClusterFps(fps,cutoff=0.4)
変数のクラスターが結果を含んでいます:
>>> print(clusters[200]) (6164, 1400, 1403, 1537, 1543, 6575, 6759)
このクラスタには7点含まれており、クラスターの重心は6164です。
N個の分子の間でRMSDを計算(RMSD Calculation between N molecules)
[Link] https://www.rdkit.org/docs/Cookbook.html#rmsd-calculation-between-n-molecules
イントロダクション(Introduction)
[Link] https://www.rdkit.org/docs/Cookbook.html#introduction
時々、2つ(あるいはそれ以上の)分子間のRMSD距離を計算する必要が生じます。これは2つのコンフォマーのがどれだけ近いかを計算するのに使われます。ほとんどのRMSD計算は類似の化合物、あるいは、少なくとも共通の部分をもつ異なる化合物についてのみ、意味をなします。
詳細(Details)
[Link] https://www.rdkit.org/docs/Cookbook.html#details
(Python 2.7で書かれた)次のプログラムはSDFファイルを入力とし、ファイルに含まれる分子間の全てのRMSD距離を生成します。これらの距離は(ユーザーが定義した)出力ファイルに書き込まれます。
5つのコンフォマーを持つSDFにたいしては、10個のRMSDスコアが得られます。典型的な、n個から反復無しにk個を選び出す問題です。すなわち、5!/2!(5-2)!。
コード:
#!/usr/bin/python ''' ファイルの中の全ての構造間のRMSDの差を計算 @author: JP <jp@javaclass.co.uk> ''' import os import getopt import sys # rdkitをインポート from rdkit import Chem from rdkit.Chem import AllChem ''' 文字列のコンテンツをファイルに書き込む ''' def write_contents(filename, contents): # 基本的なチェックをいくつか行います。厳密に言えばassertを使うことができます。 assert filename is not None, "filename cannot be None" assert contents is not None, "contents cannot be None" f = open(filename, "w") f.write(contents) f.close() # ファイルをクローズします ''' リストをファイルに書き込みます ''' def write_list_to_file(filename, list, line_sep = os.linesep): # 基本的なチェックをいくつか行います。厳密に言えばassertを使うことができます。 assert list is not None and len(list) > 0, "list cannot be None or empty" write_contents(filename, line_sep.join(list)) ''' RMSDのスプレッドを計算 ''' def calculate_spread(molecules_file): assert os.path.isfile(molecules_file), "File %s does not exist!" % molecules # イテレータの取得 mols = Chem.SDMolSupplier(molecules_file) spread_values = [] # いくつの分子がファイルの中に含まれているか? mol_count = len(mols) # それぞれの分子を他の全ての分子と比較します。 # 典型的なn個からk個を選択する話です(nから2個選択) # 組み合わせの数は(n!) / k!(n-k)! で与えら得れます(私の数学能力が錆ついていなければ) for i in range(mol_count - 1): for j in range(i+1, mol_count): # 何かの処理が行われていることを表示するようにしましょう・・・mol_countの値が大きい場合、ある程度の時間がかかるので print("Aligning molecule #%d with molecule #%d (%d molecules in all)" % (i, j, mol_count)) # RMSDを計算し、アレイに格納します # AlignMolと異なって、これは対称性の処理も行います spread_values.append(str(AllChem.GetBestRMS(mols[i], mols[j]))) # アレイを返します return spread_values def main(): try: # オプションは次の通りです: # f - 実際の構造のファイル opts, args = getopt.getopt(sys.argv[1:], "vf:o:") except getopt.GetoptError, err: # ヘルプの情報をprint関数で出力し、終了します: print(str(err)) # これは "option -a not recognized" といったようなものを出力します sys.exit(401) # デフォルト molecules_file = None output_file = None for opt, arg in opts: if opt == "-v": print("RMSD Spread 1.1") sys.exit() elif opt == "-f": molecules_file = arg elif opt == "-o": output_file = arg else: assert False, "Unhandled option: " + opt # 次をassertしてください - 最も綺麗な方法ではありませんが、上手くいきます assert molecules_file is not None, "file containing molecules must be specified, add -f to command line arguments" assert output_file is not None, "output file must be specified, add -o to command line arguments" # RMSDスプレッドの値を得ます spread_values = calculate_spread(molecules_file) # ファイルに書き込みます write_list_to_file(output_file, spread_values) if __name__ == "__main__": main()
このプログラムは次のような方法でコマンドラインから実行されるかもしれません(Pythonのインタプリタが/usr/bin/pythonにある場合です。その他の場合は最初の行(滑稽な名前がつけられたシバン)を編集してください:
calculate_spread.py -f my_conformers.sdf -o my_conformers.rmsd_spread.txt
要約: AllChem.GetBestRMS(mol1, mol2)の行はRMSDを不動小数点型として返し、このプログラムの要点となっています。GetBestRMS()はAlignMol()と異なり対称性を考慮に入れた処理を行います。
ライセンス(License)
[Link] https://www.rdkit.org/docs/Cookbook.html#license
https://www.rdkit.org/docs/Overview.html#license
この文書の著作権は copyright (C) 2013-2018 by Greg Landrum に所属しています。
この文書はCreative Commons Attribution-ShareAlike 4.0 Licenseのもとでライセンスされています。このライセンスを見るためにはhttp://creativecommons.org/licenses/by-sa/4.0/ にアクセスするか、Creative Commons, 543 Howard Street, 5th Floor, San Francisco, California, 94105, USA. に手紙を送ってください。
このライセンスの意図はRDKitそのものの意図と似ています。簡単に言えば "これを使ってなんでもやりたいことをやっていいけど、私たちの功績にも言及してね”
12/16/2018
このブログ記事のライセンス
以上はRDKit Documentationの試訳です。
ライセンスはCC BY-SA 4.0で、Greg Landrum氏の功績の元に作成しています。
Creative Commons — Attribution-ShareAlike 4.0 International — CC BY-SA 4.0
Descriptor Calculation 〜RDKit 直訳 Day15〜
(12/30追記)試訳をまとめたテストサイトを作成しました。よろしければご参照ください。
こちらはRDKit直訳 Advent Calendar 2018 - Adventar 15日目の記事です。基本的な進め方は1日目の記事をご覧ください。
本日の訳出に困った用語
Gasteiger partial charges: Gasteiger部分電荷
color sheme: カラースキーム、色彩設計
Crippen contributions: Crippen寄与
以下、訳
記述子計算(Descriptor Calculation)
[Link] https://www.rdkit.org/docs/GettingStartedInPython.html#descriptor-calculation
RDKitには様々な記述子が実装されています。記述子の完全なリストはList of Available Descriptorsを参照してください。
[list] List of Available Descriptors
記述子のほとんどはPythonから集中型のrdkit.Chem.Descriptorsモジュールを介して直接使うことができます:
[list] rdkit.Chem.Descriptors
>>> from rdkit.Chem import Descriptors >>> m = Chem.MolFromSmiles('c1ccccc1C(=O)O') >>> Descriptors.TPSA(m) 37.3 >>> Descriptors.MolLogP(m) 1.3848
部分電荷の扱い方は少し異なります:
>>> m = Chem.MolFromSmiles('c1ccccc1C(=O)O') >>> AllChem.ComputeGasteigerCharges(m) >>> float(m.GetAtomWithIdx(0).GetProp('_GasteigerCharge')) -0.047...
記述子の可視化(Visualization of Descriptors)
[Link] https://www.rdkit.org/docs/GettingStartedInPython.html#visualization-of-descriptors
類似度マップは原子ごとの寄与に分割された記述子の可視化に使うことができます。
Gasteiger部分電荷は(異なるカラースキームを使って)次のように可視化できます:
>>> from rdkit.Chem.Draw import SimilarityMaps >>> mol = Chem.MolFromSmiles('COc1cccc2cc(C(=O)NCCCCN3CCN(c4cccc5nccnc54)CC3)oc21') >>> AllChem.ComputeGasteigerCharges(mol) >>> contribs = [float(mol.GetAtomWithIdx(i).GetProp('_GasteigerCharge')) for i in range(mol.GetNumAtoms())] >>> fig = SimilarityMaps.GetSimilarityMapFromWeights(mol, contribs, colorMap='jet', contourLines=10)
このような図が生成されます:
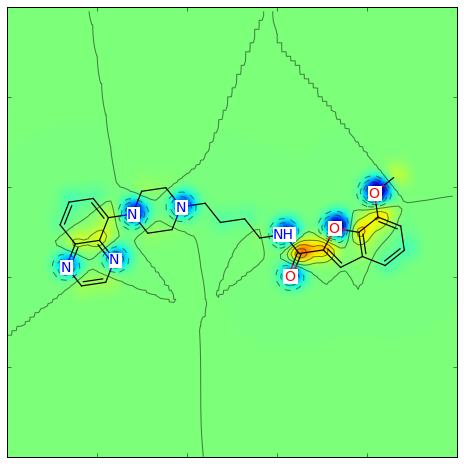 similarity_map_charges.png
similarity_map_charges.png
logPへのCrippen寄与を可視化するには次のようにします:
>>> from rdkit.Chem import rdMolDescriptors >>> contribs = rdMolDescriptors._CalcCrippenContribs(mol) >>> fig = SimilarityMaps.GetSimilarityMapFromWeights(mol,[x for x,y in contribs], colorMap='jet', contourLines=10)
このような図が生成されます:
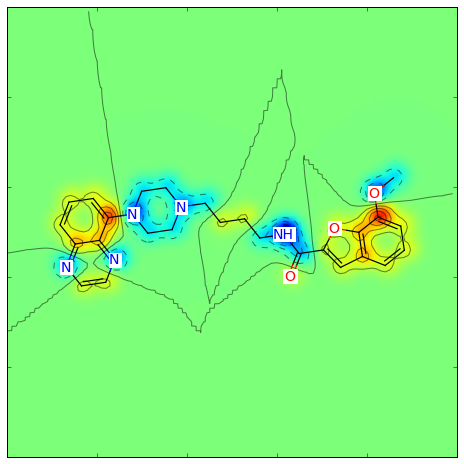 similarity_map_crippen.png
similarity_map_crippen.png
12/15/2018
このブログ記事のライセンス
以上はRDKit Documentationの試訳です。
ライセンスはCC BY-SA 4.0で、Greg Landrum氏の功績の元に作成しています。
Creative Commons — Attribution-ShareAlike 4.0 International — CC BY-SA 4.0