Fingerprinting and Molecular Similarity part3 〜RDKit 直訳 Day14〜
(12/30追記)試訳をまとめたテストサイトを作成しました。よろしければご参照ください。
こちらはRDKit直訳 Advent Calendar 2018 - Adventar 14日目の記事です。基本的な進め方は1日目の記事をご覧ください。
本日の訳出に困った用語
picker: ピッカー
similarity map: 類似度マップ
similarity metric: 類似度の指標
Dice silmilarity: Dice類似度
convenience function: 簡易関数
normalization: 正規化
以下、訳
フィンガープリントを使って多種多様な分子を選択する(Picking Diverse Molecules Using Fingerprints)
[Link] https://www.rdkit.org/docs/GettingStartedInPython.html#picking-diverse-molecules-using-fingerprints
多数の分子を含む集合から、多種多様な分子を選択してより小さな部分集合をつくることはよく行われます。RDkitではこの作業のため、rdkit.SimDivFiltersモジュールに多数の方法を用意しています。最も効率的な方法はMaxMinアルゴリズムを使うことです[脚注6]。試しにやってみましょう。
[link] rdkit.SimDivFilters
[脚注6] Ashton, M. et al. “Identification of Diverse Database Subsets using Property-Based and Fragment-Based Molecular Descriptions.” Quantitative Structure-Activity Relationships 21:598-604 (2002).
まずは分子を1セット読み込んでMorganフィンガープリントを生成します:
>>> from rdkit import Chem >>> from rdkit.Chem.rdMolDescriptors import GetMorganFingerprint >>> from rdkit import DataStructs >>> from rdkit.SimDivFilters.rdSimDivPickers import MaxMinPicker >>> ms = [x for x in Chem.SDMolSupplier('data/actives_5ht3.sdf')] >>> while ms.count(None): ms.remove(None) >>> fps = [GetMorganFingerprint(x,3) for x in ms] >>> nfps = len(fps)
MaxMinアルゴリズムにはオブジェクト間の距離を計算する関数が必要です。ここではDiceSimilarityを使いましょう。
>>> def distij(i,j,fps=fps): ... return 1-DataStructs.DiceSimilarity(fps[i],fps[j])
それではピッカーを生成し、10個の多様な分子のセットを取りだしましょう:
>>> picker = MaxMinPicker() >>> pickIndices = picker.LazyPick(distij,nfps,10,seed=23) >>> list(pickIndices) [93, 109, 154, 6, 95, 135, 151, 61, 137, 139]
ピッカーはフィンガープリントのインデックスを返すだけ、ということに注意してください。分子そのものを取得するには次のようにします:
>>> picks = [ms[x] for x in pickIndices]
フィンガープリントを使って類似度マップを生成する(Generating Similarity Maps Using Fingerprints)
[Link] https://www.rdkit.org/docs/GettingStartedInPython.html#generating-similarity-maps-using-fingerprints
類似度マップはある分子と参照とする分子の間の類似度に対して、各原子の寄与を可視化する方法です。参考文献17 [脚注17] に方法が記載されています。rdkit.Chem.Draw.SimilarityMaps モジュールを使用してください:
[脚注17] Riniker, S.; Landrum, G. A. “Similarity Maps - A Visualization Strategy for Molecular Fingerprints and Machine-Learning Methods” J. Cheminf. 5:43 (2013).
[link] rdkit.Chem.Draw.SimilarityMaps
まずは分子を2つ作るところから始めます:
>>> from rdkit import Chem >>> mol = Chem.MolFromSmiles('COc1cccc2cc(C(=O)NCCCCN3CCN(c4cccc5nccnc54)CC3)oc21') >>> refmol = Chem.MolFromSmiles('CCCN(CCCCN1CCN(c2ccccc2OC)CC1)Cc1ccc2ccccc2c1')
SimilarityMapsモジュールはatom pairs、topological torsions、Morganフィンガープリントの3種類のフィンガープリントをサポートしています:
>>> from rdkit.Chem import Draw >>> from rdkit.Chem.Draw import SimilarityMaps >>> fp = SimilarityMaps.GetAPFingerprint(mol, fpType='normal') >>> fp = SimilarityMaps.GetTTFingerprint(mol, fpType='normal') >>> fp = SimilarityMaps.GetMorganFingerprint(mol, fpType='bv')
atom pairsとtopological torsionsにはデフォルトのノーマル(normal)とハッシュ(hashed)、そしてビットベクトル(bit vector, bv)の3つのタイプがあります。Morganフィンガープリントにはデフォルトのビットベクトル(bit vector, bv)と、カウントベクトル(count vector, count)の2つのタイプがあります。
2つのフィンガープリント間の類似度マップを生成する関数には、フィンガープリントの生成に用いた関数を特定する必要があり、またオプションとして類似度の指標を指定できます。デフォルトの指標はDice類似度です。Morganフィンガープリント関数をすべてデフォルト引数で使用した場合、類似度マップは次のように生成できます:
>>> fig, maxweight = SimilarityMaps.GetSimilarityMapForFingerprint(refmol, mol, SimilarityMaps.GetMorganFingerprint)
このような図が生成されます:
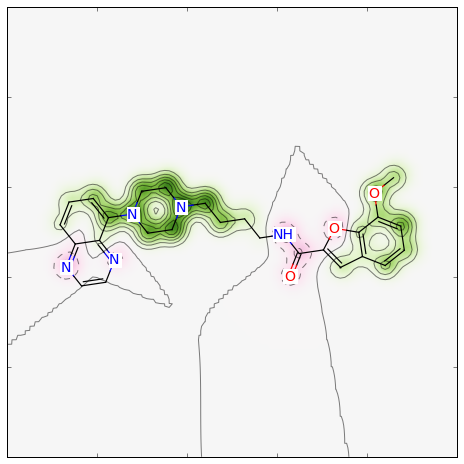
similarity_map_fp1.png
Morganフィンガープリントのタイプをデフォルトのビットベクトルからカウント(count)タイプに変更し、半径を2から1へ、類似度指標をDiceからTanimoto類似度へと変更した場合、以下のようになります:
>>> from rdkit import DataStructs >>> fig, maxweight = SimilarityMaps.GetSimilarityMapForFingerprint(refmol, mol, lambda m,idx: SimilarityMaps.GetMorganFingerprint(m, atomId=idx, radius=1, fpType='count'), metric=DataStructs.TanimotoSimilarity)
このような図が生成されます:
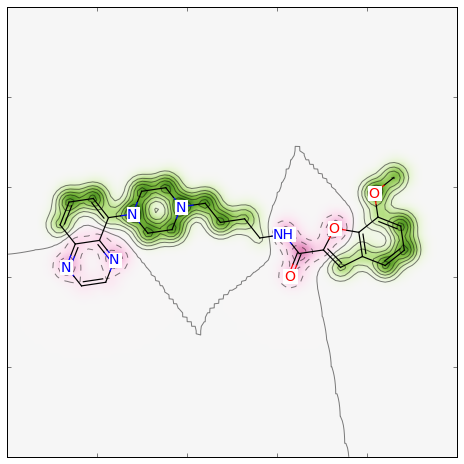
similarity_map_fp2.png
簡易関数のGetSimilarityMapForFingerprintには原子の重みの正規化が含まれており、重みの絶対値の最大が1となるよう正規化されます。したがって、この関数はマップを作成するときにみつかった、最大の重みを出力します。
>>> print(maxweight) 0.05747...
正規化の処理を行って欲しくない時は、次のようにしてマップを作ることができます:
>>> weights = SimilarityMaps.GetAtomicWeightsForFingerprint(refmol, mol, SimilarityMaps.GetMorganFingerprint) >>> print(["%.2f " % w for w in weights]) ['0.05 ', ... >>> fig = SimilarityMaps.GetSimilarityMapFromWeights(mol, weights)
このような図が生成されます:
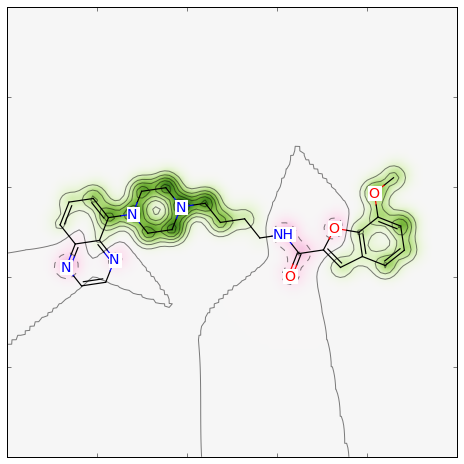
similarity_map_fp3.png
12/14/2018
このブログ記事のライセンス
以上はRDKit Documentationの試訳です。
ライセンスはCC BY-SA 4.0で、Greg Landrum氏の功績の元に作成しています。
Creative Commons — Attribution-ShareAlike 4.0 International — CC BY-SA 4.0
低分子でタンパク質の量を制御 〜アゴニストでもアンタゴニストでも阻害剤でもないMOA〜
創薬 Advent Calendar 2018 - Adventar 13日目が空いていたので埋めます。
創薬アドベントカレンダーを読むような方々には釈迦に説法という感があり恐縮なのですが、しばしお付き合いいただければと思います。
言うまでもありませんが、医薬品となる化合物の多くがタンパク質と結合し、その薬効を発揮してます(*1)。インシリコ創薬に関する話題でいえば、結合するか否かの予測(ドッキングやSBVS)や活性の予測ということになると思いますが、ではその活性ってどんな種類があるの?ということについて調べました。
活性の種類(作用機序: MOA)といえば、多くはアゴニストやアンタゴニスト・阻害剤として機能していると思います。タンパク質と結合して、本来のシグナルを流すスイッチをONにするか、流れているシグナルを止める、酵素活性を阻害するといった感じです。これらは基本的に結合の対象となる”タンパク質の機能"に対して影響を及ぼすことで、薬効を発揮しています。
ところで低分子医薬品の中には、"タンパク質の量"のコントロールを行うものもあるそうです。そこで今回、そのようなメカニズム、プロテインノックダウン法とケミカルシャペロンについてとりあげたいと思います。
①タンパク質の分解を誘導する (プロテインノックダウン法)
GPCRのようにシグナルに関与するわけでもなく、キナーゼのように酵素活性があるわけでもない、そのようなタンパク質を標的として創薬を行うにはどうすればよいか?
「そんなの壊してしまえばいいじゃない」
というラディカルな発想がこのメカニズムです。
ユビキチン-プロテアソーム系
タンパク質の分解を低分子によって誘導してやろう、というわけなのですが、このため生体内にもとからあるタンパク質分解機構「ユビキチン-プロテアソーム系」を利用します。こちらは2004年にノーベル化学賞を受賞した機構ですが、2016年のノーベル生理学・医学賞「オートファジー」が不要なゴミをざざっと分解するのに対して、もう少し精密(?)で特定のタンパク質を狙って分解するという仕組みです。
具体的には標的となるタンパク質をユビキチンで標識し、その標識を頼りにプロテアソームが分解を行います。ユビキチン標識にはユビキチン活性化酵素(E1)、ユビキチン結合酵素(E2)、ユビキチンリガーゼ(E3)と呼ばれる酵素群素が関与しますが、このうちE3リガーゼが分解の標的となるタンパク質の認識に重要となります。

プロテアソーム阻害剤
本題のプロテインノックダウン法とは異なりますが、すでにユビキチンープロテアソーム系を標的とした医薬品は上市されており、ボルテゾミブ、カルフィルゾミブというものがあります。両者ともにプロテアソームを標的とするプロテアソーム阻害剤で、ホウ素を含んでいたりエポキシケトンがあったりと、あまり医薬品では見かけないような(毒性が出ないか心配になる)構造をしています。
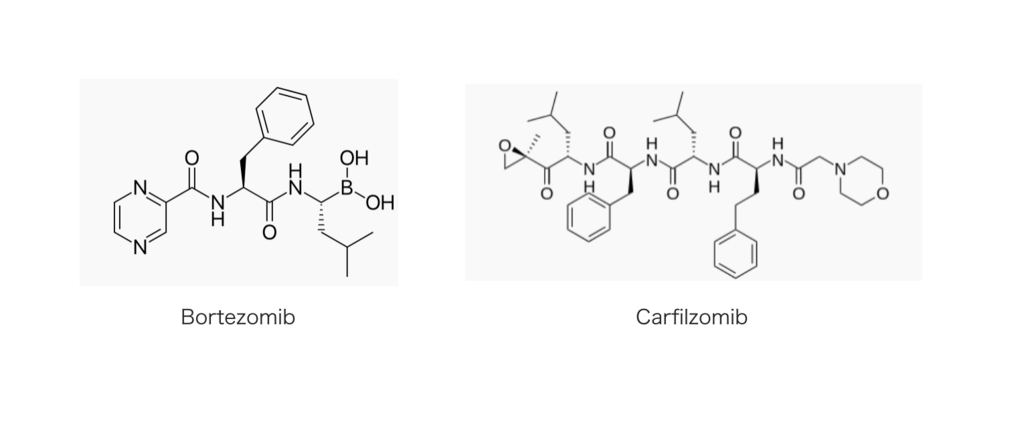
ボルテゾミブはプロテアソームの可逆的阻害剤、カルフィルゾミブは不可逆的阻害剤です。ざっと見る限り副作用も多いようですが、適応は多発性骨髄腫。生死に関わる疾患であるだけに、薬効とのバランスで、ある程度の副作用性は許容されるのでしょうか? ( 薬効と毒性の問題については創薬アドベントカレンダー 7日目のこちらの記事 でも取り上げられていましたので、未読の方は是非ご一読を!)
ところで、上記のカルフィルゾミブの発見に携わったのがYale大学のCraig M. Crews教授(Crews Laboratory)ですが、プロテインノックダウン法においてもキーパーソンです。
プロテインノックダウン法
プロテインノックダウン法では、プロテアソームではなくユビキチン標識に働く酵素群をターゲットとしています。今、ユビキチン化の基質認識に働くユビキチンE3リガーゼ(A)と結合する化合物(X)が手元にあるとします。これとは別に、壊してしまいたい標的のタンパク質(B)と結合する化合物(Y)を見つけてきます。この2つの化合物XとYをうまくリンカーでつないで、新しい化合物(Z)を作ります。ZはE3リガーゼ(A)とタンパク質(B)の両方に結合するため、Zによって両者が引き寄せられます。するとE3リガーゼがタンパク質Bを分解すべきタンパク質だ、と認識してユビキチン標識を行います。ユビキチン標識されたタンパク質Bはプロテアソームにより分解されるため、狙ったタンパク質を分解することができる、というのがこの手法のコンセプトです。

プロテインノックダウン法ですが、いくつか流派(?)があり、それぞれ利用するE3リガーゼや、E3リガーゼに結合する部分構造などに違いがあるようです。
上述のCrews教授らはPROTAC法(Proteolysis Targeting Chimeraの略)と名付け、von Hippel-Lindau (VHL)やcelebron(CRBN)をE3リガーゼとして用いています(Fig.3 参照)。また、Blander博士らはDegronimidと名付け、同じくCRBNを標的E3リガーゼとしていますが、E3リガーゼのリガンドとしてサリドマイド誘導体を用いています。日本では国立医薬品食品衛生え研究所の内藤先生・大岡先生らのグループでE3リガーゼとしてIAPを用いたSNIPER(Specific and Nongenetic Inhibitor of Apoptosis Protein(IAP)-dependent Portein Eraser)という技術の開発が行われています。
新しい創薬の手法としてプロテインノックダウン 法は注目を集めており、今年は第一回目となる国際会議Targeted Protein Degradation Summit 2018なども開かれたそうです。
長所・短所
では、プロテインノックダウン法には従来の創薬手法と比較してどのような長所があるのでしょうか?
長所①
まず標的とできるタンパク質の範囲が広がったということがあげられます。"Drugging Undruggable Targets"というキャッチーなフレーズが表すように、従来の低分子医薬品では標的とすることが難しかったタンパク質を標的とすることができる、と言われています。繰り返しになりますが、酵素として働くわけでも、シグナル伝達に関わるわけでもない、それ自体の機能が明確ではないタンパク質であったとしても、結合する部分構造さえ見つけてしまえば、分解というアプローチで標的とすることができます。
(妥当かどうかは置いておいて)わかりやすい例ではアルツハイマー病との関連が示唆されているアミロイドβや、パーキンソン病と関連するα-シヌクレインがあげられるでしょうか? 両者ともに細胞内に蓄積することで神経細胞死を起こす、といわれているため、分解を誘導し蓄積を解消できれば治療効果が期待できるかもしれません(*4)。
長所②
また、結合の位置がタンパク質の機能と関わりのある場所である必要がないため、高い選択性をもつ化合物が得られる可能性があります。例えばキナーゼを標的とする場合、多くの低分子阻害剤は酵素活性と密接に関係するATP結合サイトを結合サイトとして利用しています。ところが、機能と密接に関係する部位のため、異なるキナーゼ間でもタンパク質の構造の類似性が高く、狙ったキナーゼのみを阻害することが難しくなります。
一方で、プロテインノックダウン 法であれば、目的は酵素機能の阻害ではなく、とにかく結合してユビキチンE3リガーゼへと誘導できればよいわけですから、化合物の結合部位の選択肢が広がります。したがって、狙ったタンパク質に特有の部分構造(あまり進化で保存されて部位など?)を結合部位として利用できる可能性があり、その意味で高い選択性を発揮するポテンシャルがあります。
もちろん長所ばかりではありません。
短所①
Fig. 3のPROTACの分子構造を見ていただければ一目瞭然ですが、二つの構造を繋ぐという手法のため、分子が巨大化しやすいという問題点があります(単純化すれば従来の低分子 x 2以上の分子量)。昨年の創薬アドベントカレンダーの記事(Druglikenessについてのよもやま話 – y__sama – Medium )でもとりあげられていましたが、一般に、分子量の増大は低分子医薬品にとってはできれば避けたいといわれています(Rule of 5)。理由としては細胞膜透過性の低下による経口吸収性の低下や、薬物動態の悪化、などでしょうか?、、、とにかくあんまり良く無いっぽい(適当)
短所②
また、プロテインノックダウン 法の化合物は2種類のタンパク質に同時に結合しなければなりません。したがって、単純に「化合物 対 タンパク質 = 1 対 1」とくらべて結合のために出会う確率が下がります。そのため、細胞系の実験では高濃度にしなければ望みの効果が発揮されないといった問題点も指摘されていました。この点については、より洗練された化合物の開発により低濃度でも薬効を示すものが見出されているそうです。
実用化に向けて
肯定派・否定派さまざまな意見があるようですが、低分子で標的とできるターゲットタンパク質はあらかた出尽くした、とか、ターゲットが枯渇している、などと言われてている医薬品業界で、このように新しい創薬の手法・標的タンパク質の可能性を広げるプロテインノックダウン 法は期待されており、様々なベンチャー企業が開発に乗り出しているようです。例えば、Crews教授らは現在Arvinas 社を設立し、経口投与で薬効を示すPROTAC化合物の臨床試験を準備しているとのことです。また、Blander博士はC4 Therapeutics社を設立しています。他にもkymera 社や、 日本ではFIMECS, Inc. 社が設立されたりと、これからの動向が大変興味深い分野です。(*5)
さて、タンパク質の量を減らす試みを見てきましたが、ついでタンパク質を増やす方向を見てみたいと思います。
②ケミカルシャペロン(ファーマコロジカルシャペロン)
上述のプロテインノックダウン法では生体内の不要なタンパク質を分解する仕組み、ユビキチンープロテアソーム系を利用していました。この不要な"ゴミ"タンパク質の中には、本来の機能を発揮するための3次元構造をとれず、機能の失われたミスフォールドタンパク質も含まれます。
機能しないタンパク質とその治療という話題では、近年、脊髄性筋萎縮症を対象とするSpinrazaや デュシェンヌ型筋ジストロフィーを対象とするExondys 51といった核酸医薬品が注目を集めています。これよりすこし遡りますが2012年、嚢胞性線維症(Cystic Fibrosis)を対象としてKalydeco(一般名; ivacaftor、Vertex社)という低分子医薬品が、ついで2015年Orkambi(ivacaftorとlumacaftorの合剤)がFDAにより承認されました。ivacaftor、lumacafrorは機能するタンパク質を増やすことにより治療効果を発揮する医薬品です。
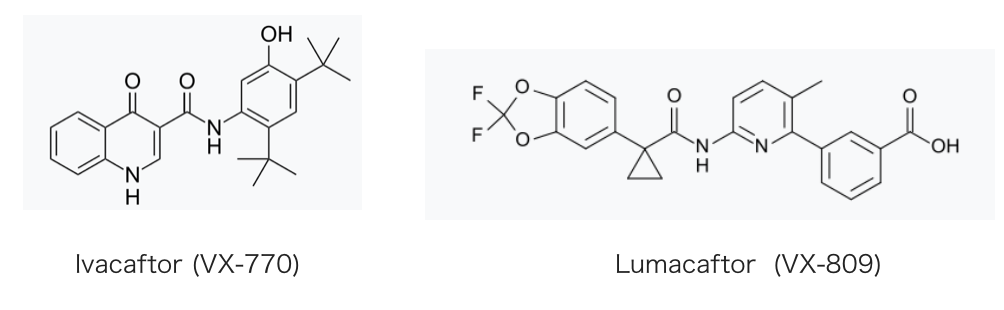
嚢胞性線維症?
嚢胞性線維症(Cystic fibrosis: 以下CF)は、常染色体劣性遺伝疾患で、日本では非常に稀な疾患ですが、米国では約3万人のCF患者がいるとされています。原因は、塩素イオン・水の輸送を調節する塩素イオンチャネルをコードするCFTR(cystic fibrosis transmembrane conductance regulator)遺伝子の変異で、このイオンチャネルがうまく機能しないことで全身の外分泌機能に異常をきたします。平均生存期間が約20年ともいわれることから、深刻な疾患であることがわかると思います。(*6)
CFにおける変異の分類とタンパク質の機能
CFと一口に言っても様々な遺伝子の変異の種類があり、各変異遺伝子の産物であるイオンチャネルの機能の欠損の度合いには差があります。文献 (*7)を頼りにその分類を見て見ましょう。
CFTRの変異は大きく2つ、
分類①: そもそもイオンチャネルが本来機能する場所である細胞膜上に達することができない(Class I、Class II)
分類②: 細胞膜上には移行するもののイオンチャネルとしての開閉機能に問題がある(Class III、Class IV、Class V)
とに分けることができます。
より詳細には、Class I の変異では核で産生されるタンパク質が短く欠失、Class II はミスフォールドによりゴルジ体にとどまる。Class III、Class IVは全長のCFTRが細胞膜上に発現できるものの、イオンチャネルのゲート開閉機能がうまく働かない(Class III)、イオン透過能(conductivity)が低い(Class IV)といった問題があります。また、Class Vは正常のCFTRですが数が少なくなるという変異です。
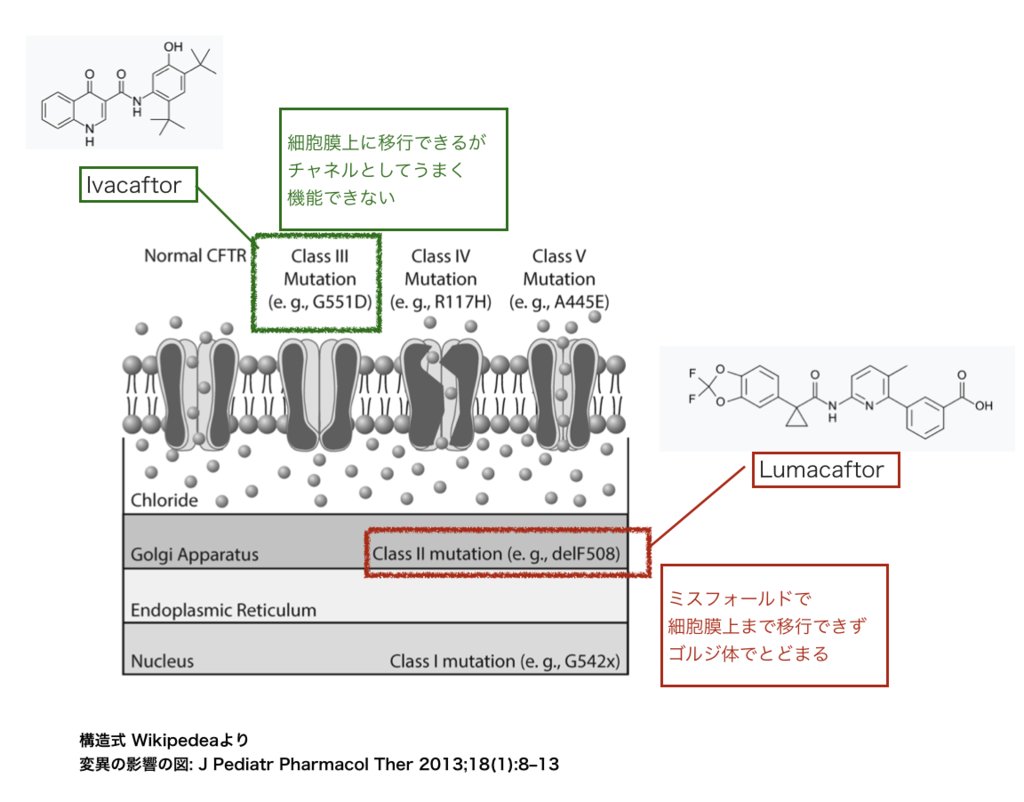
各変異をレスキューするための作用機序(MOA: Mode of Action )
では、各変異に対しどのような治療戦略が考えられるでしょうか?
変異の分類①の時は、まずは細胞膜上のCFTRを増やすことが治療の第一歩となります。つまりCFTRの合成を改善(対Class I)、細胞膜への輸送異常を改善(対Class II)となります。一方、分類②では、細胞膜上でCFTRがイオンチャンネルとしてうまく機能できるように手助けしてやる(対Class III)ことがあげられます。
上述の薬剤では、LumacaftorはClass II の変異(delF508)に対して膜移行を助けるケミカルシャペロンとして、Ivacaftor はClass III の変異(G551)に対してイオン透過能を上昇させるポテンシエーターとして働くとされています。
ケミカルシャペロンって?
前置き無しにケミカルシャペロンという言葉を導入しましたが、そもそもシャペロンとは何でしょうか?
シャペロン(英: chaperone)とは、他のタンパク質分子が正しい折りたたみ(フォールディング)をして機能を獲得するのを助けるタンパク質の総称である。分子シャペロン(英: molecular chaperone)、タンパク質シャペロンともいう。 (シャペロン - Wikipedia)(*8)
ケミカルシャペロン(ファーマコロジカルシャペロン)は低分子化合物でありながら、タンパク質のミスフォールドを修正し、適切な高次構造形成の補助・安定化に働くもののことを言います。
Lumacaftorのシャペロン効果の検証
ケミカルシャペロン、コンセプトとしてはわかりますが、その効果はどのように検証すれば良いのでしょうか?2011年Vertex社の論文 (*9)を参照して見ましょう。
まず、ミスフォールドする変異CFTRは、翻訳後修飾(グリコシル化など)がうまく行われず、修飾が正しく行われた正常なCFTR(mature : 成熟)と糖鎖修飾のパターンが異なります(immature : 未成熟)。糖鎖のパターンは細胞内でのタンパク質輸送を左右するため、これが、未成熟な変異CFTRが小胞体-ゴルジ体から細胞膜上へと移行できない理由となります (*10)。
下図は、上記文献Fig. 3Aの引用ですが、左上の図から正常のCFTRと変異CFTRでゲル上の位置が変わってくることがわかります。さらに図中左下から、Lumacaftor処理により濃度依存的に成熟型CFTRの比率が増えている様がわかります。
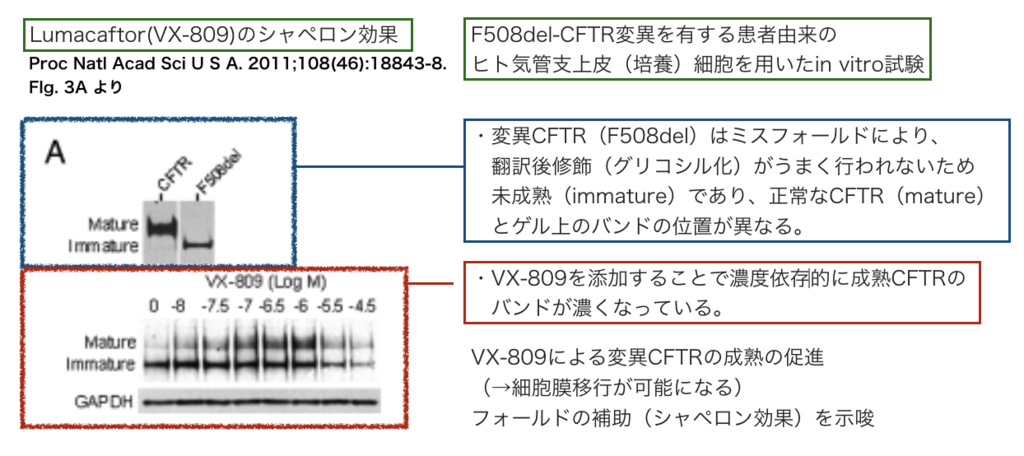
文献中にはCFTRの成熟に関する他の実験、および細胞膜上でのイオンチャネル機能に対する効果の検証もおこなわれており、複数の実験の組合せから、Lumacaftorが変異CFTRのフォールディング補助と、それに続く翻訳語修飾(プロセッシング)を促進する効果が検証されています。オープンアクセスとなっていますのでご興味があるかたは是非元の文献を参照していただければと思います。 私の読解は間違っている可能性が高いので・・・ (*11)
Ivacaftor、Lumacaftorという画期的な医薬品によりCFは完全に治療できるようになったか?というと残念ながらそうではありません。見てきたようにCFには様々な変異の種類があり、これら医薬品で治療可能な範囲には限界があります。Vertex社は継続して様々な変異への適応拡大を目指した臨床試験・新規医薬品の研究開発を進めているようなので、治療環境が改善していくと期待されます。
ケミカルシャペロンの利点?
上記のように遺伝子変異に由来する疾患に非常に有用なことが示唆されるケミカルシャペロンというMOAですが、このような疾患に対する治療戦略としては他にも核酸医薬、ウイルスべクターによる遺伝子治療や酵素補充療法など、様々なアプローチがありうると思います。これらと比較した場合の利点は何でしょうか?よく言われるように低分子はコストが安い、ということでしょうか?
この点については明確にそうだとは私は言い切れません。確かに製造コストは安くなるかもしれませんが、希少疾患である以上、莫大な研究開発費を補うため、一人当たりのCFの治療費は非常に高額なものとなっています。
では低分子ならではのメリットとはなんでしょうか?
長所①:
低分子の他のアプローチに対する利点はまずは薬物動態上のメリットです。核酸医薬では血中の安定性が悪かったり、特定の組織に集積しやすく全身暴露がかかりづらい、といった問題や、酵素補充療法では血液脳関門(BBB)を透過できないため中枢の症状には効果がでない、といった問題があります。低分子では全身暴露・中枢移行性ともに解決可能な可能性が高く、もちろん経口投与も大きなメリットです。
長所②:
また、酵素補充療法(リコンビナントタンパク質補充療法)では、抗タンパク質抗体の出現により、効果がなくなったり、過敏反応、アナフィラキシーショック、半減期の短縮といった問題が生じる可能性があります。この点でもおそらく低分子が優位でしょう。(*12)
もったいぶったわりに普通の結論になってしまった!!
他の事例と今後の期待
これまでVertex社のアプローチをみてきましたが、ここしかないの?と言われるとそんなことはありません。
「小さな命が呼ぶとき」というアメリカ映画のモデルともなったAmicus Therapeuticsという希少疾患をメインターゲットととする製薬会社があります。Amicusの開発したファブリ病(α-ガラクトシダーゼの変異による遺伝性疾患)治療薬、Migalastat(Wikipedia) もその作用機序はケミカルシャペロンで、日本ではガラフォルド®️として今年(2018年3月)製造販売承認が取得されています(*13)。
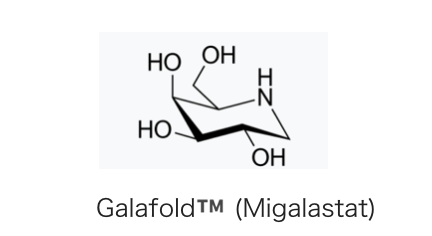
Vertex、Amicusともに遺伝性疾患を対象として、変異により機能障害のおきたタンパク質を低分子により機能回復させています。何故、上述のケミカルシャペロンや、ポテンシエーター(アロステリックモジュレーターに近い?)といった作用機序が魅力があるか? というと、ゲノム解析の進展との関係で興味深いと思うからです。遺伝子レベルの変異の同定と、タンパク質構造・機能の違いの解析が加速した際に、変異し機能が損なわれたタンパク質の機能回復、という選択肢はまだまだポテンシャルがあるのではないかなあ、と思います。・・・こいつ何にもわかってないなと聞き流してください
まとめ
以上、本記事では"タンパク質の量を低分子でコントロールするMOA"という内容で、分解させるプロテインノックダウン法と、増加させるケミカルシャペロンというアプローチについてざっと見てきました。あまり薬理学の教科書でメジャーに扱われる作用機序では無いと思うので、そんなのもあるのかーと思っていただけたら幸いです。
(私が知らないだけで薬学会では一般常識かも・・・)
私自身は専門家では無いため、記事のなかに誤りや不適切な表現が多数あると思います。ご指摘いただければ幸いです。
以上。お付き合いいただきありがとうございました。
~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~
*1
低分子化合物に限っています。DNAと相互作用するシスプラチンのようなものもありますが、話を簡単にするために省いています。
*2
Fig. 1 は以下を引用し、改変して作成
2-1) Wikipedia (https://ja.wikipedia.org/wiki/プロテアソーム )
By Rogerdodd, CC 表示-継承 3.0, https://commons.wikimedia.org/w/index.php?curid=1291009
2-2)
脱ユビキチン化酵素Ubp6はプロテアソームの分子集合を制御する(http://first.lifesciencedb.jp/archives/3121)
佐伯 泰・田中啓二 (東京都医学総合研究所 蛋白質代謝研究室)
ライフサイエンス新着論文レビュー 2011年7月11日 DOI: 107875/first.author.2011.105
http://first.lifesciencedb.jp/archives/3121
© 2011 佐伯 泰・田中啓二 Licensed under CC 表示 2.1 日本
*3
Fig. 3作成にあたって、以下を改変・引用
Proc Natl Acad Sci U S A. 2016 Jun 28; 113(26): 7124–7129.
*4
アミロイド仮説は臨床試験がことごとくうまくいっていないためそれ自体を疑う意見もあるようですが、わかりやすいのであくまで例としてあげました。
*5
その他、プロテインノックダウンに関係して参考にさせていただいたもの
5-1) SNIPERの解説・およびプロテインノックダウン法の日本語によるレビュー
https://www.jstage.jst.go.jp/article/yakushi/138/9/138_18-00113/_pdf/-char/ja
5-2) とてもわかりやすいブログ記事:
タンパク質分解誘導薬とは?これまでの薬と違う新しいタイプの薬 | Luck Is What Happens When Preparation Meets Opportunity
*6
嚢胞性線維症の概要に関しては以下を参考にしました。
https://www.shouman.jp/disease/details/03_06_008/
http://www.nanbyou.or.jp/entry/4532
6-2)
*7
7-1) Ivacaftor: A Novel Gene-Based Therapeutic Approach for Cystic Fibrosis
Condren ME, Bradshaw SM. J. Pediatr. Pharmacol. Ther. 2013;18(1): 8-13
上記文献はFig. 5作成にあたっても改変し引用
Sloane PA, Rowe SM.Curr Opin Pulm Med. 2010;16(6):591-7.
*8
シャペロンの代表的な例としては大腸菌のGroELがよく取り上げられています。
GroELの機能などのわかりやすい解説(日本語)
Journal of Japanese Biochemical Society 87(2): 194-204 (2015)
*9
Correction of the F508del-CFTR protein processing defect in vitro by the investigational drug VX-809
Proc Natl Acad Sci U S A. 2011;108(46):18843-8.
上記文献はFig. 6作成にあたっても改変し引用
*10
糖鎖修飾に関しては以下のページの説明がわかりやすかったです。
梶原研究室HP 研究の背景
*11
なお上記文献およびVertex社の他の文献中で、Lumacaftorはシャペロンとは呼ばれておらず、タンパク質のプロセシングを正常化する"corrector"と呼ばれていました。あえてなのか意図はわかりませんが、Fig. 6からもわかるように、ミスフォールドしたタンパク質を正常な高次構造に戻す様を直接明確に示す実験は困難で、糖鎖修飾やイオンの流出入量の調節といった間接的な機能改善の実験的証拠のみがあげられています。したがってケミカルシャペロンだ、と言い切るのは誤りかもしれませんが、簡単のためケミカルシャペロンの例として今回取り上げさせていただきました。
*12
もちろん低分子でも代謝産物がハプテン化し、過剰な免疫応答を起こすといったリスクはあります。
*13
トランプ大統領が映画のモデルとなったAmicusのCEOと面会したのが承認申請の強い後押しになった、なんて話も・・・
Fingerprinting and Molecular Similarity part2 〜RDKit 直訳 Day13〜
(12/30追記)試訳をまとめたテストサイトを作成しました。よろしければご参照ください。
こちらはRDKit直訳 Advent Calendar 2018 - Adventar 13日目の記事です。基本的な進め方は1日目の記事をご覧ください。
本日の訳出に困った用語
Morgan fingerprint: Morganフィンガープリント
Circular fingerprint: Circularフィンガープリント
family of fingerprints: 一連のフィンガープリント
Morgan algorithm: Morganアルゴリズム
user-supplied atom invariants: ユーザー定義のアトム不変量
cout: ビットのカウント数
bit vector: ビットベクトル
ECFP family of fingerprint: ECFPフィンガープリントファミリー
connectivity information: 結合関係の情報
feature-based invariant: 特徴量ベースの不変量
FCFP fingerprint: FCFPフィンガープリント
similarity score: 類似性スコア
the diameter of the atom environments considered: 考慮している原子の環境の直径
dictionary: 辞書型のデータ
submolecule: 擬似分子
以下、訳
Morganフィンガープリント(Circularフィンガープリント)(Morgan FIngerprints(Circular Fingerprints))
[Link] https://www.rdkit.org/docs/GettingStartedInPython.html#morgan-fingerprints-circular-fingerprints
circularフィンガープリント[脚注5]としてより知られているかもしれませんが、Morganフィンガープリントと呼ばれる一連のフィンガープリントは、ユーザー定義のアトム不変量のセットに対してMorganアルゴリズムを適用することで生成されます。Morganフィンガープリントを生成するときには、フィンガープリントの半径を定義しなければなりません:
[脚注5] Rogers, D.; Hahn, M. “Extended-Connectivity Fingerprints.” J. Chem. Inf. and Model. 50:742-54 (2010).
>>> from rdkit.Chem import AllChem >>> m1 = Chem.MolFromSmiles('Cc1ccccc1') >>> fp1 = AllChem.GetMorganFingerprint(m1,2) >>> fp1 <rdkit.DataStructs.cDataStructs.UIntSparseIntVect object at 0x...> >>> m2 = Chem.MolFromSmiles('Cc1ncccc1') >>> fp2 = AllChem.GetMorganFingerprint(m2,2) >>> DataStructs.DiceSimilarity(fp1,fp2) 0.55...
アトムペアやトポロジカルトーションのようにMorganフィンガープリントは、デフォルトではビットのカウント数を使いますが、ビットベクトルとして計算することもできます:
>>> fp1 = AllChem.GetMorganFingerprintAsBitVect(m1,2,nBits=1024) >>> fp1 <rdkit.DataStructs.cDataStructs.ExplicitBitVect object at 0x...> >>> fp2 = AllChem.GetMorganFingerprintAsBitVect(m2,2,nBits=1024) >>> DataStructs.DiceSimilarity(fp1,fp2) 0.51...
デフォルトのアトム不変量は、よく知られたECFPフィンガープリントファミリーで使われているものと類似した、結合関係の情報を使います。FCFPフィンガープリントで使われているものに類似した、特徴ベースの不変量を使うこともできます。特徴量の定義はFeature Definitions Used in the Morgan Fingerprintsセクションに記載されています。時折、とても異なる類似性スコアを出すことがあります。
[link] Feature Definitions Used in the Morgan Fingerprints
>>> m1 = Chem.MolFromSmiles('c1ccccn1') >>> m2 = Chem.MolFromSmiles('c1ccco1') >>> fp1 = AllChem.GetMorganFingerprint(m1,2) >>> fp2 = AllChem.GetMorganFingerprint(m2,2) >>> ffp1 = AllChem.GetMorganFingerprint(m1,2,useFeatures=True) >>> ffp2 = AllChem.GetMorganFingerprint(m2,2,useFeatures=True) >>> DataStructs.DiceSimilarity(fp1,fp2) 0.36... >>> DataStructs.DiceSimilarity(ffp1,ffp2) 0.90...
RDKitで生成したECFP/FCFPフィンガープリントとMorganフィンガープリントを比較する際には、Morganフィンガープリントのパラメーターが半径であるのに対して、ECFP4の4は考慮している原子の環境の直径に一致していることを思い出してください。したがって、上記の例では半径=2とすることで、ECFP4とFCFP4とおおよそ等価となります。
rdkit.Chem.rdMolDescriptors.GetMorganFingerprint()のオプションの不変量の引数を使うことで、ユーザー自身でアトム不変量を設定することもできます。不変量に定数を使った簡単な例が以下になります。得られたフィンカープリントで分子のトポロジーを比較しています。
[link] rdkit.Chem.rdMolDescriptors.GetMorganFingerprint()
>>> m1 = Chem.MolFromSmiles('Cc1ccccc1') >>> m2 = Chem.MolFromSmiles('Cc1ncncn1') >>> fp1 = AllChem.GetMorganFingerprint(m1,2,invariants=[1]*m1.GetNumAtoms()) >>> fp2 = AllChem.GetMorganFingerprint(m2,2,invariants=[1]*m2.GetNumAtoms()) >>> fp1==fp2 True
デフォルトでは結合次数が考慮されていることに注意してください:
>>> m3 = Chem.MolFromSmiles('CC1CCCCC1') >>> fp3 = AllChem.GetMorganFingerprint(m3,2,invariants=[1]*m3.GetNumAtoms()) >>> fp1==fp3 False
この設定を外すこともできます:
>>> fp1 = AllChem.GetMorganFingerprint(m1,2,invariants=[1]*m1.GetNumAtoms(), ... useBondTypes=False) >>> fp3 = AllChem.GetMorganFingerprint(m3,2,invariants=[1]*m3.GetNumAtoms(), ... useBondTypes=False) >>> fp1==fp3 True
モーガンフィンガープリントのビットの説明(Explaining bits from Morgan Fingerprints)
[Link] https://www.rdkit.org/docs/GettingStartedInPython.html#explaining-bits-from-morgan-fingerprints
bitinfo引数を介して、Morganフィンガープリントの特定のビットに寄与する原子についての情報を手に入れることができます。この辞書型のデータには、フィンガープリントのビットセットごとに一つのエントリーが取り込まれており、キー[key]はビットID、値[value]は(アトムインデックス、半径)のタプルのリストとなっています。
>>> m = Chem.MolFromSmiles('c1cccnc1C') >>> info={} >>> fp = AllChem.GetMorganFingerprint(m,2,bitInfo=info) >>> len(fp.GetNonzeroElements()) 16 >>> len(info) 16 >>> info[98513984] ((1, 1), (2, 1)) >>> info[4048591891] ((5, 2),)
上記を解釈すると、ビット98513984はatom 1とatom 2に一度ずつ、それぞれ半径1で計2回セットされています。また、ビット4048591891はatom 5に半径2でセットされています。
ビット4048591891にフォーカスを当ててみましょう。分子全体から、atom 5の半径2の中にある全ての原子からなる部分[擬似分子 submolecule]を抜き出すことができます:
>>> env = Chem.FindAtomEnvironmentOfRadiusN(m,2,5) >>> amap={} >>> submol=Chem.PathToSubmol(m,env,atomMap=amap) >>> submol.GetNumAtoms() 6 >>> amap {0: 3, 1: 5, 3: 4, 4: 0, 5: 1, 6: 2}
この擬似分子のSMILESを生成することでビットを"説明"しましょう:
>>> Chem.MolToSmiles(submol)
'ccc(C)nc'
中心の原子にSMILESの起点[root]を置くともっと役に立ちます:
>>> Chem.MolToSmiles(submol,rootedAtAtom=amap[5],canonical=False) 'c(nc)(C)cc'
上記の代わりとなる別の方法として、rdkit.Chem.MolFragmentToSmiles()関数をつかうと、同じことを(特に多数の分子を扱う際に、より高速に)行うことができます。
[link] rdkit.Chem.MolFragmentToSmiles()
>>> atoms=set() >>> for bidx in env: ... atoms.add(m.GetBondWithIdx(bidx).GetBeginAtomIdx()) ... atoms.add(m.GetBondWithIdx(bidx).GetEndAtomIdx()) ... >>> Chem.MolFragmentToSmiles(m,atomsToUse=list(atoms),bondsToUse=env,rootedAtAtom=5) 'c(C)(cc)nc'
フィンガープリントのビットのイメージの生成(Generating images of fingerprint bits)
[Link] https://www.rdkit.org/docs/GettingStartedInPython.html#generating-images-of-fingerprint-bits
MorganフィンガープリントとRDKitフィンガープリントに対しては、rdkit.Chem.Draw.DrawMorganBit()関数とrdkit.Chem.Draw.DrawRDKitBit()関数を使うことでビットを定義する原子の環境のイメージを生成することができます。
[link] rdkit.Chem.Draw.DrawMorganBit()
[link] rdkit.Chem.Draw.DrawRDKitBit()
>>> from rdkit.Chem import Draw >>> mol = Chem.MolFromSmiles('c1ccccc1CC1CC1') >>> bi = {} >>> fp = AllChem.GetMorganFingerprintAsBitVect(mol, radius=2, bitInfo=bi) >>> bi[872] ((6, 2),) >>> mfp2_svg = Draw.DrawMorganBit(mol, 872, bi) >>> rdkbi = {} >>> rdkfp = Chem.RDKFingerprint(mol, maxPath=5, bitInfo=rdkbi) >>> rdkbi[1553] [[0, 1, 9, 5, 4], [2, 3, 4, 9, 5]] >>> rdk_svg = Draw.DrawRDKitBit(mol, 1553, rdkbi)
このようなイメージができます:
svg未対応のためFigureは省略(mfp2_bit872.svg, rdk_bit1553.svg)
Morgan bit RDKit bit
Morganビットのデフォルトのハイライトの色は以下を示します:
* blue: 環境の中心となるアトム
* yellow: 芳香族のアトム
* gray: 脂肪族環構造のアトム
RDKitビットのデフォルトのハイライトの色は以下を示します:
* yellow: 芳香族のアトム
(上記のRDKitフィンガープリントのビット1553の例のように)分子の複数の原子に同じビットがセットされている場合には、描画機能は一番最初の例を表示することに気をつけてください。どの例を表示するか明示することで変更することができます:
>>> rdk_svg = Draw.DrawRDKitBit(mol, 1553, rdkbi, whichExample=1)
このようなイメージができます:
svg未対応のためFigureは省略(rdk_bit_1553_2.svg)
RDKit bit
12/13/2018
このブログ記事のライセンス
以上はRDKit Documentationの試訳です。
ライセンスはCC BY-SA 4.0で、Greg Landrum氏の功績の元に作成しています。
Creative Commons — Attribution-ShareAlike 4.0 International — CC BY-SA 4.0
Fingerprinting and Molecular Similarity part1 〜RDKit 直訳 Day12〜
(12/30追記)試訳をまとめたテストサイトを作成しました。よろしければご参照ください。
こちらはRDKit直訳 Advent Calendar 2018 - Adventar 12日目の記事です。基本的な進め方は1日目の記事をご覧ください。
本日の訳出に困った用語
Fingerprinting : フィンガープリントの生成
topologial path: トポロジカルパス
a particular density of set bits : 特定のビット密度
fingerprinter: フィンガープリント生成器
path size: パス長
target on-bit density: オンとなっているビット密度の目標値
unfolded fingerprint: 分散したフィンガープリント (畳み込まれていない(スパースな?)フィンガープリント)
similarity metric : 類似性指標
Tanimoto similarity : Tanimoto係数による類似性評価
public MACCS key: 公開されたMACCS キー
Atom-pair fingerprint : アトムペアフィンガープリント
space of bits: ビット空間
sparse manner : スパースな形式
Dice similarity:Dice類似度評価
encode: 符号化、コード化
bit-vector: ビットベクトル
Tlpological torsion descriptor:トポロジカルトーション記述子
以下、訳
フィンガープリントの生成と分子の類似性(Fingerprinting and Molecular Similarity)
[Link] https://www.rdkit.org/docs/GettingStartedInPython.html#fingerprinting-and-molecular-similarity
RDKitには分子の様々なフィンガープリントを生成する機能があらかじめ用意されており、これを使って分子の類似性を計算することができます。
トポロジカルフィンガープリント(Topological Fingerprints)
[link] https://www.rdkit.org/docs/GettingStartedInPython.html#topological-fingerprints
>>> from rdkit import DataStructs >>> from rdkit.Chem.Fingerprints import FingerprintMols >>> ms = [Chem.MolFromSmiles('CCOC'), Chem.MolFromSmiles('CCO'), ... Chem.MolFromSmiles('COC')] >>> fps = [FingerprintMols.FingerprintMol(x) for x in ms] >>> DataStructs.FingerprintSimilarity(fps[0],fps[1]) 0.6... >>> DataStructs.FingerprintSimilarity(fps[0],fps[2]) 0.4... >>> DataStructs.FingerprintSimilarity(fps[1],fps[2]) 0.25
このフィンガープリントのアルゴリズムはDaylight フィンガープリントの生成で使われているものと類似しています:分子のトポロジカルパス(たとえば結合)を同定しハッシュを生成します。それを利用してユーザー指定の長さのフィンガープリントにビットを立てます。全てのパスを同定した後で、フィンガープリントは一般的に、特定のビット密度になるまで畳み込まれます。
フィンガープリント生成器で使われているデフォルトのパラメーターセットは次の様になっています。- 最小パス長:1結合 - 最大パス長:7結合 - フィンガープリントのサイズ:2048 - ハッシュ毎のビット数:2 - 最小のフィンガープリントのサイズ:64ビット - オンとなっているビット密度の目標値:0.3
これらのパラメーターはrdkit.Chem.rdmolops.RDKFingerprint() を直接呼び出すことで制御できます:分散したフィンガープリント[unfolded fingerprint]を返すので、望みの密度に畳み込む[fold]ことができます。Pythonで書かれたrdkit.Chem.Fingerprints.FingerprintMols.FingerprintMol()関数を見ればどうやってこれを行うかがわかります。
[link] rdkit.Chem.rdmolops.RDKFingerprint(): https://www.rdkit.org/docs/source/rdkit.Chem.rdmolops.html#rdkit.Chem.rdmolops.RDKFingerprint [link] rdkit.Chem.Fingerprints.FingerprintMols.FingerprintMol(): https://www.rdkit.org/docs/source/rdkit.Chem.Fingerprints.FingerprintMols.html#rdkit.Chem.Fingerprints.FingerprintMols.FingerprintMol
rdkit.DataStructs.FingerprintSimilarity()で使われているデフォルトの類似性指標はTanimoto係数です。他の類似性指標を使うこともできます。
[link] rdkit.DataStructs.FingerprintSimilarity(): https://www.rdkit.org/docs/source/rdkit.DataStructs.html#rdkit.DataStructs.FingerprintSimilarity
>>> DataStructs.FingerprintSimilarity(fps[0],fps[1], metric=DataStructs.DiceSimilarity) 0.75
利用可能な類似性指標は、Tanimoto、Dice、Cosine、Sokal、Russel、Kulczynski、McConnaughey、そしてTverskyです。
MACCS キー(MACCS Keys)
[Link] https://www.rdkit.org/docs/GettingStartedInPython.html#maccs-keys
公開されている166種類のMACCSキーを使ったSMARTSベースの実装もあります。
>>> from rdkit.Chem import MACCSkeys >>> fps = [MACCSkeys.GenMACCSKeys(x) for x in ms] >>> DataStructs.FingerprintSimilarity(fps[0],fps[1]) 0.5 >>> DataStructs.FingerprintSimilarity(fps[0],fps[2]) 0.538... >>> DataStructs.FingerprintSimilarity(fps[1],fps[2]) 0.214...
Q3 2008で、MACCS keyは厳密に評価され他のMACCSの実装と比較されました。公開されたキーを完全に定義した場合は非常にうまく機能しました。
アトムペアとトポロジカルトーション(Atom Pairs and Topological Torsions)
[Link] https://www.rdkit.org/docs/GettingStartedInPython.html#atom-pairs-and-topological-torsions
アトムペア記述子[脚注3]はいくつかの異なる方法で利用可能です。標準的な形式ではフィンガープリントを0か1で表すのではなく、それぞれのビットの数をカウントしてフィンガープリントに含めます:
[脚注3]Carhart, R.E.; Smith, D.H.; Venkataraghavan R. “Atom Pairs as Molecular Features in Structure-Activity Studies: Definition and Applications” J. Chem. Inf. Comp. Sci. 25:64-73 (1985).
>>> from rdkit.Chem.AtomPairs import Pairs >>> ms = [Chem.MolFromSmiles('C1CCC1OCC'),Chem.MolFromSmiles('CC(C)OCC'),Chem.MolFromSmiles('CCOCC')] >>> pairFps = [Pairs.GetAtomPairFingerprint(x) for x in ms]
アトムペアフィンガープイントに含められるビット空間は非常に巨大なので、スパースな形式で保存されます。各フィンガープリントのビットのリストとそのカウント数を辞書型のデータとして得ることができます。
>>> d = pairFps[-1].GetNonzeroElements() >>> d[541732] 1 >>> d[1606690] 2
ビットの説明も手に入ります:
>>> Pairs.ExplainPairScore(558115) (('C', 1, 0), 3, ('C', 2, 0))
上記の意味は「隣接原子が1つあり、π電子が0個の炭素(C)で、隣接原子が2つあり、π電子が0個の炭素(C)から、結合3つ分離れている」です。
アトムペアフィンガープリント間の類似性評価に通常用いられる指標はDice類似度評価です。
>>> from rdkit import DataStructs >>> DataStructs.DiceSimilarity(pairFps[0],pairFps[1]) 0.333... >>> DataStructs.DiceSimilarity(pairFps[0],pairFps[2]) 0.258... >>> DataStructs.DiceSimilarity(pairFps[1],pairFps[2]) 0.56
(カウント数の情報を無視した)標準的なビットベクトルのフィンガープリントとしてコード化されたアトムペアを得ることもできます。
airFps = [Pairs.GetAtomPairFingerprintAsBitVect(x) for x in ms]
これらは標準的なビットベクトルなので、rdkit.DataStructsモジュールを使って類似性評価を行うこともできます。
[link] rdkit.DataStructs: https://www.rdkit.org/docs/source/rdkit.DataStructs.html#module-rdkit.DataStructs
>>> from rdkit import DataStructs >>> DataStructs.DiceSimilarity(pairFps[0],pairFps[1]) 0.48 >>> DataStructs.DiceSimilarity(pairFps[0],pairFps[2]) 0.380... >>> DataStructs.DiceSimilarity(pairFps[1],pairFps[2]) 0.625
トポロジカルトーション記述子も基本的には同じ方法で計算されます。[脚注4]
[脚注4] Nilakantan, R.; Bauman N.; Dixon J.S.; Venkataraghavan R. “Topological Torsion: A New Molecular Descriptor for SAR Applications. Comparison with Other Desciptors.” J. Chem.Inf. Comp. Sci. 27:82-5 (1987)
>>> from rdkit.Chem.AtomPairs import Torsions >>> tts = [ .GetTopologicalTorsionFingerprintAsIntVect(x) for x in ms] >>> DataStructs.DiceSimilarity(tts[0],tts[1]) 0.166...
このページを書いている段階では、トポロジカルトーションフィンガープリントには多すぎてBitVector機能を使ってコード化できないほどたくさんビットがあるので、GetTopologicalTorsionFingerprintAsBitVect 関数といった関数は存在しません。
12/12/2018
このブログ記事のライセンス
以上はRDKit Documentationの試訳です。
ライセンスはCC BY-SA 4.0で、Greg Landrum氏の功績の元に作成しています。
Creative Commons — Attribution-ShareAlike 4.0 International — CC BY-SA 4.0
Maximum Common Substructure 〜RDKit 直訳 Day11〜
(12/30追記)試訳をまとめたテストサイトを作成しました。よろしければご参照ください。
こちらはRDKit直訳 Advent Calendar 2018 - Adventar 11日目の記事です。基本的な進め方は1日目の記事をご覧ください。
本日の訳出に困った用語
SMARTS string: SMARTS 文字列
bond type : 結合タイプ
comparison function : 比較関数
Isotope label : 同位体ラベル
wall-clock senconds:実時間
CPU seconds:CPU時間
以下、訳
最大共通部分構造(Maximum Common Substructure)
[Link] https://www.rdkit.org/docs/GettingStartedInPython.html#maximum-common-substructure
FindMCS関数は2つ以上の分子から最大共通部分構造(MCS)を見つけ出します:
>>> from rdkit.Chem import rdFMCS >>> mol1 = Chem.MolFromSmiles("O=C(NCc1cc(OC)c(O)cc1)CCCC/C=C/C(C)C") >>> mol2 = Chem.MolFromSmiles("CC(C)CCCCCC(=O)NCC1=CC(=C(C=C1)O)OC") >>> mol3 = Chem.MolFromSmiles("c1(C=O)cc(OC)c(O)cc1") >>> mols = [mol1,mol2,mol3] >>> res=rdFMCS.FindMCS(mols) >>> res <rdkit.Chem.rdFMCS.MCSResult object at 0x...> >>> res.numAtoms 10 >>> res.numBonds 10 >>> res.smartsString '[#6]1(-[#6]):[#6]:[#6](-[#8]-[#6]):[#6](:[#6]:[#6]:1)-[#8]' >>> res.canceled False
FindMCS変数は、MCS中の原子と結合の番号の情報をもつMCSReultインスタンスと、見つかったMCSに一致するSMARTS文字列、そしてアルゴリズムがタイムアウトした場合に教えてくれるフラグを返します。MCSが見つからなかった場合は、原子と結合の番号は0、SMARTSは''となります。
デフォルトでは、2つの原子は同じ元素の時に、2つの結合は同じ結合のタイプの時に一致となります。異なる比較関数をつかうにはatomCompareやbondCompareを明示してください。例えば以下の様に:
>>> mols = (Chem.MolFromSmiles('NCC'),Chem.MolFromSmiles('OC=C')) >>> rdFMCS.FindMCS(mols).smartsString '' >>> rdFMCS.FindMCS(mols, atomCompare=rdFMCS.AtomCompare.CompareAny).smartsString '[#7,#8]-[#6]' >>> rdFMCS.FindMCS(mols, bondCompare=rdFMCS.BondCompare.CompareAny).smartsString '[#6]-,=[#6]'
atomCompare引数のオプションは、CompareAnyはいずれの原子も他のいかなる原子にもマッチする、CompareElementsは元素のタイプによって比較する、CompareIsotopesは同位体ラベルに基づいてマッチする、となっています。同位体ラベルはユーザー定義されたアトムタイプの実装にも使うことができます。CompareAnyのbondCompareはいずれの結合も他のいかなる結合にもマッチし、CompareOrderExactは同じ結合タイプを有する時のみ等価であるとします。そしてCompareOrderは単結合と芳香族結合が互いにマッチすることは許容しますが、それ以外については正確な結合次数の一致を要求します。
>>> mols = (Chem.MolFromSmiles('c1ccccc1'),Chem.MolFromSmiles('C1CCCC=C1')) >>> rdFMCS.FindMCS(mols,bondCompare=rdFMCS.BondCompare.CompareAny).smartsString '[#6]1:,-[#6]:,-[#6]:,-[#6]:,-[#6]:,=[#6]:,-1' >>> rdFMCS.FindMCS(mols,bondCompare=rdFMCS.BondCompare.CompareOrderExact).smartsString '' >>> rdFMCS.FindMCS(mols,bondCompare=rdFMCS.BondCompare.CompareOrder).smartsString '[#6](:,-[#6]:,-[#6]:,-[#6]):,-[#6]:,-[#6]'
部分構造は原子と結合の両方を保持します。デフォルトではアルゴリズムは見つかる結合の数を最大化しようとします。この設定はmaximizeBonds引数をFalseに設定することで変更できます。2つの小さな環構造は一つの大きな環構造よりも結合の数が少なくなるかもしれませんが、結合の数を最大化しようとする設定では環構造の数を最大化しようとする傾向があります。
価数3の窒素原子を価数5の窒素原子とマッチさせたくない場合があるかもしれません。デフォルトではmachValenceがFlaseとなっており、価数の情報を無視します。Trueとすると、atomCompareの設定が、2つの原子が同じ価数を有することも要求する様に変更されます。
>>> mols = (Chem.MolFromSmiles('NC1OC1'),Chem.MolFromSmiles('C1OC1[N+](=O)[O-]')) >>> rdFMCS.FindMCS(mols).numAtoms 4 >>> rdFMCS.FindMCS(mols, matchValences=True).numBonds 3
炭素原子からなる直鎖構造が環構造とマッチするのが奇妙に見えるかもしれませんが、ringMatchesRingOnlyがデフォルトではFalseとなっているからです。Trueに変更すると、環構造の結合は、環構造とだけマッチする様になります。
>>> mols = [Chem.MolFromSmiles("C1CCC1CCC"), Chem.MolFromSmiles("C1CCCCCC1")] >>> rdFMCS.FindMCS(mols).smartsString '[#6](-[#6]-[#6])-[#6]-[#6]-[#6]-[#6]' >>> rdFMCS.FindMCS(mols, ringMatchesRingOnly=True).smartsString '[#6](-[#6]-[#6])-[#6]'
さらに制限を加えることもでき、(この場合の様に)環構造のうち部分構造の場合は許容されないように設定することもできます。つまり、ある原子がMCSの一部で、分子全体の環構造の中にあるなら、その原子はMCSの環構造に含まれます。completeRingsOnlyをTrueとすることで、この必要条件のオン・オフを切り替えることができます。ringMatchesRingOnlyもTrueとして下さい。
>>> mols = [Chem.MolFromSmiles("CCC1CC2C1CN2"), Chem.MolFromSmiles("C1CC2C1CC2")] >>> rdFMCS.FindMCS(mols).smartsString '[#6]1-[#6]-[#6](-[#6]-1-[#6])-[#6]' >>> rdFMCS.FindMCS(mols, ringMatchesRingOnly=True).smartsString '[#6](-[#6]-[#6]-[#6]-[#6])-[#6]' >>> rdFMCS.FindMCS(mols, completeRingsOnly=True).smartsString '[#6]1-[#6]-[#6]-[#6]-1'
MCSアルゴリズムは最大共通部分構造を網羅的に探索します。大抵の場合あっという間に終わりますが、数分あるいはもっと時間がかかる場合があります。timeoutパラメータを使えば、設定した秒数(実時間でCPU時間ではありません)で検索を打ち切り、時間内で見つかった最も良い一致構造を返します。タイムアウトの時間に達するとMCSResultのcanceledプロパティがFalseではなくTrueとなります。
>>> mols = [Chem.MolFromSmiles("Nc1ccccc1"*10), Chem.MolFromSmiles("Nc1ccccccccc1"*10)] >>> rdFMCS.FindMCS(mols, timeout=1).canceled True
(50秒後のMCSは511の原子を含みました。)
12/11/2018
このブログ記事のライセンス
以上はRDKit Documentationの試訳です。
ライセンスはCC BY-SA 4.0で、Greg Landrum氏の功績の元に作成しています。
Creative Commons — Attribution-ShareAlike 4.0 International — CC BY-SA 4.0
Chemical Transformations 〜RDKit 直訳 Day10〜
(12/30追記)試訳をまとめたテストサイトを作成しました。よろしければご参照ください。
こちらはRDKit直訳 Advent Calendar 2018 - Adventar 10日目の記事です。基本的な進め方は1日目の記事をご覧ください。
本日の訳出に困った用語
core: 骨格
sidechain: 側鎖
scaffold : 骨格
Murcko Decomposition : Murcko型分解
Murcko-type Decomposition : Murcko型分解
framework: 骨格
以下、訳
化学変換(Chemical Transformations)
[Link] https://www.rdkit.org/docs/GettingStartedInPython.html#chemical-transformations
RDKitには分子に修正を加える多数の関数があります。これらの変換用の関数は、分子に単純な修正を加えるのに簡単な方法を提供することを目的として作られていることに注意してください。より複雑な変換を行うにはChemical Reactions機能を使用してください。
[link] Chemical Reactions
部分構造ベースの変換(Substructure-based transformations)
[Link] https://www.rdkit.org/docs/GettingStartedInPython.html#substructure-based-transformations
RDKitの部分構造一致検索機能には分子の変換を素早く行うための様々な関数があります。利用可能な変換の例を挙げていくと、部分構造の削除:
>>> m = Chem.MolFromSmiles('CC(=O)O') >>> patt = Chem.MolFromSmarts('C(=O)[OH]') >>> rm = AllChem.DeleteSubstructs(m,patt) >>> Chem.MolToSmiles(rm) 'C'
部分構造の置換:
>>> repl = Chem.MolFromSmiles('OC') >>> patt = Chem.MolFromSmarts('[$(NC(=O))]') >>> m = Chem.MolFromSmiles('CC(=O)N') >>> rms = AllChem.ReplaceSubstructs(m,patt,repl) >>> rms (<rdkit.Chem.rdchem.Mol object at 0x...>,) >>> Chem.MolToSmiles(rms[0]) 'COC(C)=O'
また、側鎖を取り除くといった簡単なSARテーブルの変換や:
>>> m1 = Chem.MolFromSmiles('BrCCc1cncnc1C(=O)O') >>> core = Chem.MolFromSmiles('c1cncnc1') >>> tmp = Chem.ReplaceSidechains(m1,core) >>> Chem.MolToSmiles(tmp) '[1*]c1cncnc1[2*]'
骨格の削除も可能です:
>>> tmp = Chem.ReplaceCore(m1,core)
>>> Chem.MolToSmiles(tmp)
'[1*]CCBr.[2*]C(=O)O'
デフォルトでは側鎖は見つかった順番にラベルがつけられます。側鎖が結合した、骨格の原子の番号に従ってラベルをつけることも可能です。
>>> m1 = Chem.MolFromSmiles('c1c(CCO)ncnc1C(=O)O') >>> tmp=Chem.ReplaceCore(m1,core,labelByIndex=True) >>> Chem.MolToSmiles(tmp) '[1*]CCO.[5*]C(=O)O'
rdkit.Chem.rdmolops.ReplaceCore() は一つの分子の側鎖を返します。これはrdkit.Chem.rdmolops.GetMolFrags() を使って別々の分子に分割することができます。
[link] rdkit.Chem.rdmolops.ReplaceCore() [link] rdkit.Chem.rdmolops.GetMolFrags()
>>> rs = Chem.GetMolFrags(tmp,asMols=True) >>> len(rs) 2 >>> Chem.MolToSmiles(rs[0]) '[1*]CCO' >>> Chem.MolToSmiles(rs[1]) '[5*]C(=O)O'
Murcko型分解(Murcko Decomposition)
[Link] https://www.rdkit.org/docs/GettingStartedInPython.html#murcko-decomposition
RDKitでは標準的なMrucko型分解[脚注7]により分子を骨格に分割できます:
[脚注7] Bemis, G. W.; Murcko, M. A. “The Properties of Known Drugs. 1. Molecular Frameworks.” J. Med. Chem. 39:2887-93 (1996).
>>> from rdkit.Chem.Scaffolds import MurckoScaffold >>> cdk2mols = Chem.SDMolSupplier('data/cdk2.sdf') >>> m1 = cdk2mols[0] >>> core = MurckoScaffold.GetScaffoldForMol(m1) >>> Chem.MolToSmiles(core) 'c1ncc2nc[nH]c2n1'
また、一般的な骨格にも分解できます:
>>> fw = MurckoScaffold.MakeScaffoldGeneric(core)
>>> Chem.MolToSmiles(fw)
'C1CCC2CCCC2C1'
12/10/2018
このブログ記事のライセンス
以上はRDKit Documentationの試訳です。
ライセンスはCC BY-SA 4.0で、Greg Landrum氏の功績の元に作成しています。
Creative Commons — Attribution-ShareAlike 4.0 International — CC BY-SA 4.0
Installation 〜RDKit 直訳 Day9〜
(12/30追記)試訳をまとめたテストサイトを作成しました。よろしければご参照ください。
こちらはRDKit直訳 Advent Calendar 2018 - Adventar 9日目の記事です。基本的な進め方は1日目の記事をご覧ください。
以下、訳
インストールの仕方(Installation)
[Link] https://www.rdkit.org/docs/Install.html#installation
以下は様々なインストール方法を多数示していますが、複雑さの度合いが異なります。
anaconda pythonを使ったクロスプラットフォームな方法(最速のインストール方法) (Cross-platform under anaconda python(fastest install))
[Link] https://www.rdkit.org/docs/Install.html#cross-platform-under-anaconda-python-fastest-install
anacondaの紹介(Introduction to anaconda)
[Link] https://www.rdkit.org/docs/Install.html#introduction-to-anaconda
condaはオープンソースで、クロスプラットフォームのソフトウェアパッケージマネージャーです。ソフトウェアコンポーネントのパッケージ化と配布をサポートしており、独立した実行環境内部でのインストールを管理します。pipやvirtaulenvといくつかの類似性がありますが、より"Pythonに依存しない"形になるようデザインされていて、バイナリパッケージの配布と依存関係により適しています。
condaを手にいれる方法(How to get conda)
[Link] https://www.rdkit.org/docs/Install.html#how-to-get-conda
condaを手に入れる最も簡単な方法はAnaconda Python distribution の一部分としてインストールすることです。その他の方法として(少し使用方法が複雑になりますが)より小さく、必要なものに絞られたMinicondaをインストールするという選択肢もあります。condaのソースコードレポジトリはgithub で手に入ります。追加のドキュメントもプロジェクトのwebsite上で提供されています.
[link] Anaconda Python distribution
[link] Miniconda
[link] github
[link] website
Condaを使ってRDKitをインストールする方法(How to install RDKit with Conda)
[Link] https://www.rdkit.org/docs/Install.html#how-to-install-rdkit-with-conda
RDKitがインストールされた新しいconda環境を作るには、次のようなコマンドを一つ打ち込むだけでOKです::
$ conda create -c rdkit -n my-rdkit-env rdkit
最後に、同じシェル上で相当するPythonインタープリターが利用可能になるように新しい環境をアクティベートする必要があります:
$ source activate my-rdkit-env
もし、何らかの理由でうまくいかなかった場合は次を試してください:
$ cd [anaconda folder]/bin $ source activate my-rdkit-env
Windowsユーザーは少し違うコマンドが必要です:
C:\> activate my-rdkit-env
conda-forge package
[Link] https://www.rdkit.org/docs/Install.html#conda-forge-package
conda-forge のRDKitパッケージも利用可能です。すでに他のconda-forgeパッケージを使っているユーザーにはもしかしたらより簡単なインストール方法かもしれません。このパッケージは次のようにインストールすることができます:
[link] conda-forge
$ conda install -c conda-forge rdkit
Condaを使ってソースからビルドする方法(How to bulid from source with Conda)
[Link] https://www.rdkit.org/docs/Install.html#how-to-build-from-source-with-conda
condaを使ってソースからビルドする方法の詳細は、conda-rdkit repositoryを見てください。
[link] conda-rdkit repository
macOS 10.12 (Sierra): Python 3 environment
[Link] https://www.rdkit.org/docs/Install.html#macos-10-12-sierra-python-3-environment
次のコマンドでmacOS Sierra と Python3のための開発環境を作ることができます。condaからMiniconda3-latest-MacOSX-x86_64.shをダウンロードし次のコマンドを実行してください:
[link] Conda
bash Miniconda3-latest-MacOSX-x86_64.sh conda install numpy matplotlib conda install cmake cairo pillow eigen pkg-config conda install boost-cpp boost py-boost
オプションとして、便利な開発用ツールとして、次のパッケージを付け足すこともできます:
pip install yapf==0.11.1 pip install coverage==3.7.1
そして、通常のビルドの方法を実施します。PYTHON_INCLUDE_DIRをcmakeコマンドにセットする必要があります。
PYROOT=<path to miniconda3> cmake -DPYTHON_INCLUDE_DIR=$PYROOT/include/python3.6m \ -DRDK_BUILD_AVALON_SUPPORT=ON \ -DRDK_BUILD_CAIRO_SUPPORT=ON \ -DRDK_BUILD_INCHI_SUPPORT=ON \ ..
"make"と"make install"が最後までうまくいったら、次のコマンドでテストを行ってください:
RDBASE=$RDBASE DYLD_FALLBACK_LIBRARY_PATH="$RDBASE/lib:$PYROOT/lib" PYTHONPATH=$RDBASE ctest
これは最近のmacOSバージョンに導入されたSystem Integrity Protection SIPのため、必要となります。
[link] System Integrity Protection SIP
Linux x86_64: Python 3 environment
[Link] https://www.rdkit.org/docs/Install.html#linux-x86-64-python-3-environment
次のコマンドでLinux x86_64 と Python3のための開発環境を作ることができます。
まずはAnacondaから最新のanacondaインストーラーをダウンロードしてください。そして、必要なパッケージをインストールしてください:
[link] Anaconda
bash Anaconda3-5.2.0-x86_64.sh conda install -y cmake cairo pillow eigen pkg-config conda install -y boost-cpp boost py-boost
Numpyとmatplotlibはanacondaの基本インストールの一部としてすでに含まれています。最新のboostライブラリが、現在、anacondaのデフォルトよりも新しいGLIBICバージョンを使ってビルドされているため、より新しいバージョンにアップデートする必要があります:
conda install -y gxx_linux-64
この時点で、ビルドしたい場所にRDKitレポジトリをクローンし、ビルドを開始できる必要があります。anacondaがNumpyパッケージの中に隠してしまうので、RDKitがnumpyのヘッダを見つけられるようにパスを示す必要があります:
git clone https://github.com/rdkit/rdkit.git
cd rdkit
mkdir build && cd build
cmake .. -DPy_ENABLE_SHARED=1 \
-DRDK_INSTALL_INTREE=ON \
-DRDK_INSTALL_STATIC_LIBS=OFF \
-DRDK_BUILD_CPP_TESTS=ON \
-DPYTHON_NUMPY_INCLUDE_PATH="$CONDA_PREFIX/lib/python3.6/site-packages/numpy/core/include"
最後に、make、make install、ctestを実行してください。
conda環境からPostgreSQLとRDKit PostgreSQLカートリッジを使ってインストールする方法(Installing and using PostgreSQL and the RDKit PostgreSQL cartridge from a conda environment )
conda pythonディストリビューションがシステムのPythonのバージョンと異なるため、PostgreSQLとPostgreSQL pythonクライアントはcondaを介してインストールするのが最も簡単です。
環境をアクティベートした状態で、次のコマンドを実行するだけです:
conda install -c rdkit rdkit-postgresql
condaパッケージのPostgreSQLバージョンは[conda folder]/envs/my-rdkit-env/binの中にあるinitdbコマンドを走らせて初期化する必要があります:
[conda folder]/envs/my-rdkit-env/bin/initdb -D /folder/where/data/should/be/stored
初期化後、ターミナルから次のコマンドでPostgreSQLを実行することができます:
[conda folder]/envs/my-rdkit-env/bin/postgres -D /folder/where/data/should/be/stored
多くの場合、代わりにdaemonとしてPostgreSQLを走らせる必要があると思います。これを行う一つの方法はsupervisorを使うことです。より詳細な情報とsupervisorのインストール方法は ここで手に入ります。必要なコンフィギュレーションファイルは以下ようなものを見てください:
[link] ここ
[program:postgresql] command=[conda folder]/envs/my-rdkit-env/bin/postgres -D /folder/where/data/should/be/stored user=[your username] autorestart=true
PostgreSQLが起動し、走り始めたら、conda環境をアクティベートすれば、通常のPostgreSQLコマンドは全て実行可能となります。したがってデータベースを作るには次を実行してください:
createdb my_rdkit_db psql my_rdkit_db # create extension rdkit;
もし異なる環境でPostgreSQLを複数インストールして使おうとしているなら、 PostgreSQL configファイルを編集 して、異なるpidファイルとunixソケット、およびポートをセットアップする必要があります。上記のコンフィギュレーションの場合、これらのファイルは /folder/where/data/should/be/stored で見つけることができます。
[link] PostgreSQL configファイルを編集
Linux and OS X
[Link] https://www.rdkit.org/docs/Install.html#linux-and-os-x
Installation from repositories
[Link] https://www.rdkit.org/docs/Install.html#installation-from-repositories
Ubuntu 12.04 and later
[Link] https://www.rdkit.org/docs/Install.html#ubuntu-12-04-and-later
Debichemチームの努力のおかげで、RDKitはUbuntuレポジトリを介して手に入れることができます。インストールするには次を実行してください:
sudo apt-get install python-rdkit librdkit1 rdkit-data
Fedora, CentOS, and RHEL
[Link] https://www.rdkit.org/docs/Install.html#fedora-centos-and-rhel
Gianluca Sfornaのおかげで、RDKitのためのバイナリRPMが公式のFedoraレポジトリの一部となっています: https://admin.fedoraproject.org/pkgdb/package/rpms/rdkit/
OS X
[Link] https://www.rdkit.org/docs/Install.html#os-x
Eddie CaoがRDKitを簡単にビルドするのに使うことができるhomebrewのフォーミュラを作りました https://github.com/rdkit/homebrew-rdkit
Building from Source
[Link] https://www.rdkit.org/docs/Install.html#building-from-source
2018_03 リリースから、RDKitのコアとなるC++コードはモダンなC++で書かれています;このリリースの場合C++11です。つまり、ビルドするのに使うコパイラとして古いものを使うことが完全にできなくなったということです。最低限のテスト済みのバージョンは次のものです: * g++ v4.8: SLNパーサーコードはv4.8でビルドすることができないことに注意してください。古いコンパイラが使われると自動的に無効にされます。 * clang v3.9: もっと古いバージョンも機能するかもしれませんが試していません。 * Visual Studio 2015: もっと古いバージョンも機能するかもしれませんが試していません。
Installing prerequisites from source
[Link] https://www.rdkit.org/docs/Install.html#installing-prerequisites-from-source
- 必要なパッケージ:
- cmake。version 3.1(以上)が必要です。Linuxディストリビューションが適したパーッケージを持っていなければhttp://www.cmake.org を参照してください。
- Pythonラッパーを使おうと思っているなら次が必要です
- Pythonヘッダ。おそらくLinuxディストリビューションにpython-devパッケージ(あるいはそれが必要とするものを何でも)をインストールする必要があることを意味します。
- sqlite3。共有ライブラリも必要です。sqlite3-devパッケージをインストールする必要が有るかもしれません。
- Numpy (http://www.scipy.org/NumPy)をインストールしている必要があります。 注意 OS XでXCode4を使ってビルドするには、XCode4に付随するnumpyのバージョンに問題があるようです。応急処置の方法は下のセクション(FAQセクション)を参照してください。
Installing Boost
[Link] https://www.rdkit.org/docs/Install.html#installing-boost
linxusディストリビューションに、Pythonとシリアライズ化ライブラリを含む、バージョン1.58以上のboost-develパッケージがあれば次のステップは必要ありません。
**注意**
プレインストールされたboostライブラリのバージョンがある状態で、自分のバージョンを使いたい場合はコードをビルドする際に気をつけてください。我々は少なくとも一つ、Fedoraシステム上で、ユーザーがインストールしたバージョンのboostを使ってcmakeでコンパイルし、システムのバージョンにリンクさせた、という事例を知っていますが、この時はセグメンテーションフォールトになりました。応急処置の方法は下のセクション(FAQセクション)を参照してください。
- boostのウェブサイト からboostのソースディストリビューションをダウンロード
- マシンからソースを取得する(例えば
/usr/local/src/boost_1_58_0) - 必要なboostライブラリをビルドする。boostのサイトに詳細な説明がありますが、全体の流れは次のようになります:
cd \$BOOST- Pythonラッパーを使いたい場合は:
./bootstrap.sh --with-libraries=python,serialization - Pythonラッパーを使わない場合は:
./bootstrap.sh --with-libraries=serialization ./b2 install
このステップで問題が生じた場合はboostのインストールマニュアルを参照してください。
[link] boost web site
[link] 詳細な説明
[link] インストールマニュアル
Building the RDKit
[Link] https://www.rdkit.org/docs/Install.html#building-the-rdkit
ソースを取得します。ここではtar.gzを使っていますがgitを使うこともできます。
wget https://github.com/rdkit/rdkit/archive/Release_XXXX_XX_X.tar.gz
- あらかじめ必要なものがインストールされているか確認しておいてください
- 環境変数:
- RDBASE: RDKitディストリビューションのルートディレクトリ(例 ~/RDKit)
- Linux: LD_LIBRARY_PATH: \$RDBASE/libとboost共有ライブラリがインストールされている場所が確実に含まれるようにしてください
- OS X: DYLD_LIBRARY_PATH: \$RDBASE/libとboost共有ライブラリがインストールされている場所が確実に含まれるようにしてください
- Pythonラッパーを使おうと思っているなら次が必要です:
- PYTHONPATH: \$RDBASEを含むことを確認してください
- ビルド:
- \$RDBASE に cd
mkdir buildcd buildcmake ..: デフォルトのバージョンではないPythonを指定する必要がある場合か、標準的ではない場所にboostがある場合は、ビルドを設定する際に下のセクションを参照してくださいmake: (デフォルトでは)これで全てのライブラリとリグレッションテスト、ラッパーをビルドされます。make install
FAQと解決方法のリストは下を見てください。
Testing the build (optional, but recomended)
[Link] https://www.rdkit.org/docs/Install.html#testing-the-build-optional-but-recommended
- \$RDBASE/build にcd、次に
ctestを実行 - これでおしまい
Advanced
[Link] https://www.rdkit.org/docs/Install.html#advanced
Specifying an alternate Boost installation
[Link] https://www.rdkit.org/docs/Install.html#specifying-an-alternate-boost-installation
cmakeにboostライブラリとヘッダファイルの場所を教える必要があります: boostを/opt/localにおいている場合、cmakeの呼び出しは次のようになります:
cmake -DBOOST_ROOT=/opt/local ..
システムがインストールしたboostがあるシステム上で、自分のboostを使っている場合は注意してください。通常、cmakeコマンドに引数-D Boost_NO_SYSTEM_PATHS=ONも含めた方が良いです。
Specifying an alternate Python installation
[Link] https://www.rdkit.org/docs/Install.html#specifying-an-alternate-python-installation
デフォルトでインストールされたPythonを使っていない場合は、リンクすべきPythonライブラリの場所とPythonヘッダファイルの場所をcmakeに教える必要があります。
これがサンプルコマンドラインです:
cmake -D PYTHON_LIBRARY=/usr/lib/python2.7/config/libpython2.7.a -D PYTHON_INCLUDE_DIR=/usr/include/python2.7/ -D PYTHON_EXECUTABLE=/usr/bin/python ..
正しいPythonが、あなたのPATHの最初のバージョンなら、PYTHON_EXECUTABLEの部分はオプションです。
Disabling the Python wrappers
[Link] https://www.rdkit.org/docs/Install.html#disabling-the-python-wrappers
Pythonラッパーのビルドを完全に無効化することができます:
cmake -DRDK_BUILD_PYTHON_WRAPPERS=OFF ..
Recommended extras
[Link] https://www.rdkit.org/docs/Install.html#recommended-extras
- cmakeコマンドラインに
-DRDK_BUILD_INCHI_SUPPORT=ON引数を加えることでInChI文字列とInChIキーを生成するためのサポートを有効にすることができます。 - cmakeコマンドラインに
-DRDK_BUILD_AVALON_SUPPORT=ON引数を加えることで Avalonツールキットのサポートを有効にすることができます。 - 構造描画のために高画質のPNGを生成できるようにしたいなら、cairoをシステムにインストールし、cairoのサポートを有効にしてRDKitをビルドする必要があります:
-DRDK_BUILD_CAIRO_SUPPORT=ON - 3D記述子を使えるようにしたいなら、eigen3のコピーがインストールされている状態にする必要があります。ほとんどのOSは適切なパッケージを持っています。
Building the Java wrappers
[Link] https://www.rdkit.org/docs/Install.html#building-the-java-wrappers
Additional Requirements
- SWIG >v2.0: http://www.swig.org
Building
* cmakeを呼び出すときに-D RDK_BUILD_SWIG_WRAPPERS=ONを引数に加えてください。例えば:cmake -D RDK_BUILD_SWIG_WRAPPERS=ON ..
* ビルドとインストールは通常makeを使います。$RDBASE/Code/JavaWrappers/gmwrapperディレクトリに3つの必要なファイルが入っています: libGraphMolWrap.so (libGraphMolWrap.jnilib on OS X)と org.RDKit.jar、そしてorg.RDKitDoc.jarです。
Using the wrappers
ラッパーを使うには3つのファイルが同じディレクトリに入っている必要があり、そしてそれがCLASSPATHとjava.library.pathに含まれている必要があります。jythonを使った例を示します:
% CLASSPATH=$CLASSPATH:$RDBASE/Code/JavaWrappers/gmwrapper/org.RDKit.jar; jython -Djava.library.path=$RDBASE/Code/JavaWrappers/gmwrapper
Jython 2.2.1 on java1.6.0_20
Type "copyright", "credits" or "license" for more information.
>>> from org.RDKit import *
>>> from java import lang
>>> lang.System.loadLibrary('GraphMolWrap')
>>> m = RWMol.MolFromSmiles('c1ccccc1')
>>> m.getNumAtoms()
6L
optional packages
[Link] https://www.rdkit.org/docs/Install.html#optional-packages
- RDKit InChIのサポートをインストールしたいなら
$RDBASE/External/INCHI-API/READMEの指示に従ってください。 - RDKit Avalonツールキットのサポートをインストールしたいなら
$RDBASE/External/AvalonTool/READMEの指示に従ってください。 - PostgreSQLカートリッジをビルドしインストールしたいなら
$RDBASE/Code/PgSQL/rdkit/READMEの指示に従ってください。
Frequently Encountered Problems
[Link] https://www.rdkit.org/docs/Install.html#frequently-encountered-problems
以下の各事例ではパスの特定の部分を...で置き換えています。
Problem:
Linking CXX shared library libSLNParse.so /usr/bin/ld: .../libboost_regex.a(cpp_regex_traits.o): relocation R_X86_64_32S against `std::basic_string<char, std::char_traits<char>, std::allocator<char> >::_Rep::_S_empty_rep_storage' can not be used when making a shared object; recompile with -fPIC .../libboost_regex.a: could not read symbols: Bad value collect2: ld returned 1 exit status make[2]: *** [Code/GraphMol/SLNParse/libSLNParse.so] Error 1 make[1]: *** [Code/GraphMol/SLNParse/CMakeFiles/SLNParse.dir/all] Error 2 make: *** [all] Error 2
Solution:
cmakeを呼び出すときに次を引数に加えてください:-DBoost_USE_STATIC_LIBS=OFF
追加の情報はこちら:http://www.mail-archive.com/rdkit-discuss@lists.sourceforge.net/msg01119.html
Problem:
.../Code/GraphMol/Wrap/EditableMol.cpp:114: instantiated from here .../boost/type_traits/detail/cv_traits_impl.hpp:37: internal compiler error: in make_rtl_for_nonlocal_decl, at cp/decl.c:5067 Please submit a full bug report, with preprocessed source if appropriate. See \<URL:<http://bugzilla.redhat.com/bugzilla>\> for instructions. Preprocessed source stored into /tmp/ccgSaXge.out file, please attach this to your bugreport. make[2]: **\* [Code/GraphMol/Wrap/CMakeFiles/rdchem.dir/EditableMol.cpp.o] Error 1 make[1]:**\* [Code/GraphMol/Wrap/CMakeFiles/rdchem.dir/all] Error 2 make: *\** [all] Error 2
Solution:
Code/GraphMol/Wrap/EditableMol.cppの一番上に#define BOOST_PYTHON_NO_PY_SIGNATURESを加えてください。
追加の情報はこちら: http://www.mail-archive.com/rdkit-discuss@lists.sourceforge.net/msg01178.html
Problem:
システムには/usr/libにインストールされたバージョンのboostがあるが、RDKitにはより最新のものを使わせたい。
Solution:
-D Boost_NO_SYSTEM_PATHS=ON引数を渡すことで解決できます。
Problem:
OS XでXCode4を使ってビルドする場合
XCode4とともに配布されているNumpyのバージョンによって問題が起きているように見えるので、新しいコピーをビルドする必要があります。
Solution:
Numpyのコピーを手に入れて、次のようにroot:をroot:としてビルドしてください:
export MACOSX_DEPLOYMENT_TARGET=10.6 export LDFLAGS="-Wall -undefined dynamic_lookup -bundle -arch x86_64" export CFLAGS="-arch x86_64" ln -s /usr/bin/gcc /usr/bin/gcc-4.2 ln -s /usr/bin/g++ /usr/bin/g++-4.2 python setup.py build python setup.py install
確実に新しいNumpyがビルドで使われるようにしてしてください:
PYTHON_NUMPY_INCLUDE_PATH /Library/Python/2.6/site-packages/numpy/core/include
また、PYTHONPATHの最初になるようにしてください:
export PYTHONPATH="/Library/Python/2.6/site-packages:$PYTHONPATH"
これで、安全にboostとRDKitをビルドすることができます。
Windows
[Link] https://www.rdkit.org/docs/Install.html#windows
Prerequisites
[Link] https://www.rdkit.org/docs/Install.html#prerequisites
- Python 2.7 あるいは 3.4+ (http://www.python.org/)
- numpy (http://numpy.scipy.org/ あるいは pip install numpy)。win64用のバイナリはここで手に入る: http://www.lfd.uci.edu/~gohlke/pythonlibs/#numpy
- Pillow:(https://python-pillow.github.io/> あるいはpip install Pillow)。
Recommeded extras
[Link] https://www.rdkit.org/docs/Install.html#recommended-extras
- aggdraw:Pythonで高画質の描画を行うためのライブラリ。ダウンロードの方法はこちら: http://effbot.org/zone/aggdraw-index.htm 新しい描画コード(2008年5月時点)はaggdraw v1.2a3 でテストした。alpha labelにも関わらず、コードは安定していてかつ機能する。
- matplotlib: Pythonから科学的なプロットを作成するためのライブラリ。 http://matplotlib.sourceforge.net/
- ipython : Python(とそれ以上に多くのこと)のためのとても役に立つインタラクティブなシェル http://ipython.scipy.org/dist/
- win32all: PythonのためのWindowsの拡張 http://sourceforge.net/projects/pywin32/
Installation of RDKit binaries
[Link] https://www.rdkit.org/docs/Install.html#installation-of-rdkit-binaries
- 適切なWindows用のバイナリビルドを https://github.com/rdkit/rdkit/releases から入手する
- zipファイルをどこかから取り出す(i.e.
C:\)。名前にスペースを入れないこと。 - 以下は
C:\RDKit_2015_09_2にインストールされていると仮定して進める - 次の環境変数を設定する:
- RDBASE:
C:\RDKit_2015_09_2 - PYTHONPATH:
%RDBASE%もしすでにPYTHONPATHがあるなら、末尾に;%RDBASE%を付け足す。。 - PATH:
;%RDBASE%\libを末尾に付け足す。
Win7システムではDLLが見つからないことでトラブルに見舞われるかもしれません。メーリングリストのスレッド(http://www.mail-archive.com/rdkit-discuss@lists.sourceforge.net/msg01632.html) を参照してください。必要なDLLはここからダウンロードできます: http://www.microsoft.com/en-us/download/details.aspx?id=5555
Installation from source
[Link] https://www.rdkit.org/docs/Install.html#installation-from-source
Extra software to install
[Link] https://www.rdkit.org/docs/Install.html#extra-software-to-install
- Microsoft Visual C++ : コミュニティバージョンに必要なものは全て揃っていて、無料でダウンロードすることができます(https://www.visualstudio.com/vs/community)。大きなインストールになるので時間がかかります。RDKitはVisual Studio 2015と2017を使ってビルドされています、より最新のバージョンの方が良いでしょう。
- cmake : (http://www.cmake.org/cmake/resources/software.html) インストールする必要があります。
- boost : boostライブラリのコンパイル済みのバージョンを http://sourceforge.net/projects/boost/files/boost-binaries/ からダウンロートして使うことを強くお勧めします。インストーラーを実行するときに必要なバイナリライブラリはPythonとserializationだけです。ソースからboostをインストールしたいなら、http://www.boost.org からコピーをダウンロードして、ドキュメントの"Getting Started"セクションの指示に従ってください。確実にライブラリとヘッダが C:\boost にインストールされるようにしてください。
- a git client : これはRDKitのdevelopment versionでビルドしようと思っている時だけ必要になります。 http://git-scm.com/downloads からダウンロードすることができます。gitはMicrosoft Visual Studio 2015のオプションのアドオンとしても含まれています。
Setup and Preparation
[Link] https://www.rdkit.org/docs/Install.html#setup-and-preparation
このセクションはPythonがC:\Python27に、boostライブラリがC:\boostにインストールされていて、RDKitをC:\RDKitという名前のディレクトリからビルドしようとしていると仮定して進めます。以上の条件のどれか一つでも違っているなら、対応するパスを変更してください。
- 名前にスペースを含むパスにインストールしようとしているなら、確実に環境変数の定義に適切に引用を使うようにしてください。
- RDKitのdevelopment versionを使おうとしているなら、gitを使っている現在のRDKitのソースのコピーを手に入れてください。コマンドラインクライアントを使っているなら、コマンドは
git clone https://github.com/rdkit/rdkit.git C:\RDKitです。 - リリースバージョンのRDKitを使おうとしているなら、最新のリリースのコピーを手に入れ、
C:\RDKitディレクトリに取り出してください。 - 必要な環境変数を設定します:
RDBASE = C:\RDKitC:\Python27がPATHに入っていることを確認してくださいC:\RDKit\libがPATHに入っていることを確認してくださいC:\boost\libがPATHに入っていることを確認してくださいC:\RDKitがPYTHONPATHに入っていることを確認してください
Building from the command line (recommended)
[Link] https://www.rdkit.org/docs/Install.html#building-from-the-command-line-recommended
C:\RDKit\buildディレクトリを作ってそこにcdしてください- cmakeを実行してください。InChIとAvalonツールキットのソースをそれぞれInChI TrustとSourceForgeレポジトリからダウンロードし、インストール済みのバージョンのPostgreSQLのためのPostgreSQL カートリッジをビルドする、64bit版Windowsのための基本的なコマンドラインの例は次のようになります。
cmake -DRDK_BUILD_PYTHON_WRAPPERS=ON -DBOOST_ROOT=C:/boost -DRDK_BUILD_INCHI_SUPPORT=ON -DRDK_BUILD_AVALON_SUPPORT=ON -DRDK_BUILD_PGSQL=ON -DPostgreSQL_ROOT="C:\Program Files\PostgreSQL\9.5" -G"Visual Studio 14 2015 Win64" .. - コードをビルドしてください。コマンドラインの例は次のようになります:
C:/Windows/Microsoft.NET/Framework64/v4.0.30319/MSBuild.exe /m:4 /p:Configuration=Release INSTALL.vcxproj - PostgreSQLサポートでビルドした場合、管理者権限でシェルを開き、PostgreSQLサービスをとめ、インストール用のスクリプト
pgsql_install.batを実行し、そしてPostgreSQLサービスを再起動する必要があります。 (より詳細には%RDBASE%\Code\PgSQL\rdkit\READMEを参照してください):- '"C:\Program Files\PostgreSQL\9.5\bin\pg_ctl.exe" -N "postgresql-9.5" -D "C:\Program Files\PostgreSQL\9.5\data" -w stop`
C:\RDKit\build\Code\PgSQL\rdkit\pgsql_install.bat"C:\Program Files\PostgreSQL\9.5\bin\pg_ctl.exe" -N "postgresql-9.5" -D "C:\Program Files\PostgreSQL\9.5\data" -w start- PostgreSQLサービスを再起動する前に、RDKitをビルドする際に使ったBoostライブラリがシステムのPATHにあることを確認してください。さもないとPostgreSQLは
rdkitの拡張を作るのに失敗し、次のような紛らわしいエラーメッセージを出します:ERROR: could not load library "C:/Program Files/PostgreSQL/9.5/lib/rdkit.dll": The specified module could not be found.
Testing the Build (optional, but recommended)
[Link] https://www.rdkit.org/docs/Install.html#testing-the-build-optional-but-recommended
C:\RDKit\buildにcdし、ctestを実行してください。PostgreSQLサポート内部でビルドした場合は、現在のログインユーザーが、データベースの作成とスーパーユーザーの権限を持ったPostgreSQLユーザーである必要があることに注意してください。さもないとPostgreSQLのテストは失敗します。認証のための便利なオプションは、ctestを実行しているシェルの現在のログインユーザーのPostgreSQLパスワードに、PGPASSWORD環境変数をセットすることです。- これでおしまい
License
[Link] https://www.rdkit.org/docs/Install.html#license
この文書の著作権は copyright (C) 2013-2018 by Greg Landrum に所属しています。
この文書はCreative Commons Attribution-ShareAlike 4.0 Licenseのもとでライセンスされています。このライセンスを見るためにはhttp://creativecommons.org/licenses/by-sa/4.0/ にアクセスするか、Creative Commons, 543 Howard Street, 5th Floor, San Francisco, California, 94105, USA. に手紙を送ってください。
このライセンスの意図はRDKitそのものの意図と似ています。簡単に言えば "これを使ってなんでもやりたいことをやっていいけど、私たちの功績にも言及してね”
12/09/2018
このブログ記事のライセンス
以上はRDKit Documentationの試訳です。
ライセンスはCC BY-SA 4.0で、Greg Landrum氏の功績の元に作成しています。
Creative Commons — Attribution-ShareAlike 4.0 International — CC BY-SA 4.0