Chemical Features and Pharmacophores 〜RDKit 直訳 Day19〜
(12/30追記)試訳をまとめたテストサイトを作成しました。よろしければご参照ください。
こちらはRDKit直訳 Advent Calendar 2018 - Adventar 19日目の記事です。基本的な進め方は1日目の記事をご覧ください。
本日の訳出に困った用語
Chemical Features: 化学的特徴
Pharmacophore: ファーマコフォア
feature: フィーチャー
feature definition language: フィーチャー定義言語
feature factory: 特徴工場、フィーチャーファクトリ
bin: ビン分割
topological distance: 幾何学的距離
unique integer: 一意の整数値
encoding: 符号化、エンコーディング、コード化
feature-typing mechanism: フィーチャータイプ化機能
signature (fingerprint) factory: シグネチャー(フィンガープリント)ファクトリ
以下、訳
化学的特徴とファーマコフォア(Chemical Features and Pharmacophores)
[Link] https://www.rdkit.org/docs/GettingStartedInPython.html#chemical-features-and-pharmacophores
化学的特徴(Chemical Features)
[Link] https://www.rdkit.org/docs/GettingStartedInPython.html#chemical-features
RDKitにおいて化学的特徴(フィーチャー)は、SMARTSに基づくフィーチャー定義言語を使って定義されています(言語の詳細はthe RDKit bookに記載されています)。分子の化学的特徴を見つけ出すためには、まず初めにフィーチャーファクトリ[feature factory]を作らなければなりません:
>>> from rdkit import Chem >>> from rdkit.Chem import ChemicalFeatures >>> from rdkit import RDConfig >>> import os >>> fdefName = os.path.join(RDConfig.RDDataDir,'BaseFeatures.fdef') >>> factory = ChemicalFeatures.BuildFeatureFactory(fdefName)
次に、作成したファクトリーをつかってフィーチャーを検索します:
>>> m = Chem.MolFromSmiles('OCc1ccccc1CN') >>> feats = factory.GetFeaturesForMol(m) >>> len(feats) 8
個々のフィーチャーには、それが属するファミリー(たとえばドナーやアクセプターなど)やタイプ(より詳細な説明)、フィーチャーと結びつけられた原子(原子団)についての情報が紐付けられています:
>>> feats[0].GetFamily() 'Donor' >>> feats[0].GetType() 'SingleAtomDonor' >>> feats[0].GetAtomIds() (0,) >>> feats[4].GetFamily() 'Aromatic' >>> feats[4].GetAtomIds() (2, 3, 4, 5, 6, 7)
もし分子に座標の情報が含まれているなら、座標の中でフィーチャーが占める合理的な位置についての情報も紐付けられます:
>>> from rdkit.Chem import AllChem >>> AllChem.Compute2DCoords(m) 0 >>> feats[0].GetPos() <rdkit.Geometry.rdGeometry.Point3D object at 0x...> >>> list(feats[0].GetPos()) [2.07..., -2.335..., 0.0]
2Dファーマコフォアフィンガープリント(2D Pharmacophore Fingerprints)
[Link] https://www.rdkit.org/docs/GettingStartedInPython.html#d-pharmacophore-fingerprints
複数の化学的特徴と、フィーチャー間の2Dの(幾何学的、トポロジカル)距離とを合わせることで2Dファーマコフォアを作成することができます。距離がビン分割されると、それぞれのファーマコフォアに一意の整数値のIDを割り当てることができ、フィンガープリントにその情報を格納することができます。エンコーディングの詳細はThe RDKit Bookを参照してください。
[link] The RDKit book
ファーマコフォアフィンガープリントを生成するには、通常のRDKitのフィーチャータイプ化機能で作成した化学的特徴が必要です:
>>> from rdkit import Chem >>> from rdkit.Chem import ChemicalFeatures >>> fdefName = 'data/MinimalFeatures.fdef' >>> featFactory = ChemicalFeatures.BuildFeatureFactory(fdefName)
フィンガープリント自体は、ファーマコフォアを生成する際に必要だったすべてのパラメーターを保持しているシグネチャー(フィンガープリント)ファクトリを使って計算されます。
>>> from rdkit.Chem.Pharm2D.SigFactory import SigFactory >>> sigFactory = SigFactory(featFactory,minPointCount=2,maxPointCount=3) >>> sigFactory.SetBins([(0,2),(2,5),(5,8)]) >>> sigFactory.Init() >>> sigFactory.GetSigSize() 885
これでフィンガープリントを生成するシグネチャーファクトリーの準備が整いました。rdkit.Chem.Pharm2D.Generateモジュールを使って行います。
[link] rdkit.Chem.Pharm2D.Generate
>>> from rdkit.Chem.Pharm2D import Generate >>> mol = Chem.MolFromSmiles('OCC(=O)CCCN') >>> fp = Generate.Gen2DFingerprint(mol,sigFactory) >>> fp <rdkit.DataStructs.cDataStructs.SparseBitVect object at 0x...> >>> len(fp) 885 >>> fp.GetNumOnBits() 57
含まれるフィーチャーやビン分割されたフィーチャー間の距離行列といった情報も含めて、ビット自体の詳細についてはシグネチャーファクトリから得ることができます
>>> list(fp.GetOnBits())[:5] [1, 2, 6, 7, 8] >>> sigFactory.GetBitDescription(1) 'Acceptor Acceptor |0 1|1 0|' >>> sigFactory.GetBitDescription(2) 'Acceptor Acceptor |0 2|2 0|' >>> sigFactory.GetBitDescription(8) 'Acceptor Donor |0 2|2 0|' >>> list(fp.GetOnBits())[-5:] [704, 706, 707, 708, 714] >>> sigFactory.GetBitDescription(707) 'Donor Donor PosIonizable |0 1 2|1 0 1|2 1 0|' >>> sigFactory.GetBitDescription(714) 'Donor Donor PosIonizable |0 2 2|2 0 0|2 0 0|'
作業を簡便にするため(毎回fdefファイルを編集する必要をなくすため)に、SigFactoryの特定のフィーチャータイプを無効化することもできます。
>>> sigFactory.skipFeats=['PosIonizable'] >>> sigFactory.Init() >>> sigFactory.GetSigSize() 510 >>> fp2 = Generate.Gen2DFingerprint(mol,sigFactory) >>> fp2.GetNumOnBits() 36
RDKitで使用可能な、2Dファーマコフォアフィンガープリントのためのフィーチャー定義のセットにはもう一つあり、GobbiとPoppingerによって提案されたものです[脚注10]。rdkit.Chem.Pharm2D.Gobbi_Pharm2Dモジュールにこれらのフィンガープリントタイプのために予め設定されたシグネチャーファクトリがあります。使い方の例をお示しします:
[脚注10] Gobbi, A. & Poppinger, D. “Genetic optimization of combinatorial libraries.” Biotechnology and Bioengineering 61:47-54 (1998).
[link] rdkit.Chem.Pharm2D.Gobbi_Pharm2D
>>> from rdkit import Chem >>> from rdkit.Chem.Pharm2D import Gobbi_Pharm2D,Generate >>> m = Chem.MolFromSmiles('OCC=CC(=O)O') >>> fp = Generate.Gen2DFingerprint(m,Gobbi_Pharm2D.factory) >>> fp <rdkit.DataStructs.cDataStructs.SparseBitVect object at 0x...> >>> fp.GetNumOnBits() 8 >>> list(fp.GetOnBits()) [23, 30, 150, 154, 157, 185, 28878, 30184] >>> Gobbi_Pharm2D.factory.GetBitDescription(157) 'HA HD |0 3|3 0|' >>> Gobbi_Pharm2D.factory.GetBitDescription(30184) 'HA HD HD |0 3 0|3 0 3|0 3 0|'
12/19/2018
このブログ記事のライセンス
以上はRDKit Documentationの試訳です。
ライセンスはCC BY-SA 4.0で、Greg Landrum氏の功績の元に作成しています。
Creative Commons — Attribution-ShareAlike 4.0 International — CC BY-SA 4.0
Chemical Reactions part2 〜RDKit 直訳 Day18〜
(12/30追記)試訳をまとめたテストサイトを作成しました。よろしければご参照ください。
こちらはRDKit直訳 Advent Calendar 2018 - Adventar 18日目の記事です。基本的な進め方は1日目の記事をご覧ください。
本日の訳出に困った用語
dummy atom: ダミーアトム
generator object: ジェネレーターオブジェクト
valence: 原子価
generic function: ジェネリック関数
attachment point: 接着点
unsigned integer: 符号なし整数
以下、訳
BRICSの実装(BRICS Implemetation)
[Link] https://www.rdkit.org/docs/GettingStartedInPython.html#brics-implementation
RDKitはBRICSアルゴリズムの実装も提供しています[脚注9]。BRICSは合成的に利用可能な結合に沿って分子をフラグメント化するもう一つの方法です:
[脚注9] Degen, J.; Wegscheid-Gerlach, C.; Zaliani, A; Rarey, M. “On the Art of Compiling and Using ‘Drug-Like’ Chemical Fragment Spaces.” ChemMedChem 3:1503–7 (2008).
>>> from rdkit.Chem import BRICS >>> cdk2mols = Chem.SDMolSupplier('data/cdk2.sdf') >>> m1 = cdk2mols[0] >>> sorted(BRICS.BRICSDecompose(m1)) ['[14*]c1nc(N)nc2[nH]cnc12', '[3*]O[3*]', '[4*]CC(=O)C(C)C'] >>> m2 = cdk2mols[20] >>> sorted(BRICS.BRICSDecompose(m2)) ['[1*]C(=O)NN(C)C', '[14*]c1[nH]nc2c1C(=O)c1c([16*])cccc1-2', '[16*]c1ccc([16*])cc1', '[3*]OC', '[5*]N[5*]']
RDKitのBRICSの実装では、分子から作られたそれぞれのユニークなフラグメントを返します。また、ダミーアトムに、どのタイプの反応が適用されたかを示すタグがつけられたうえで返されます。
分子の集合に対して全てのフラグメントのリストを生成することがとても簡単にできます:
>>> allfrags=set() >>> for m in cdk2mols: ... pieces = BRICS.BRICSDecompose(m) ... allfrags.update(pieces) >>> len(allfrags) 90 >>> sorted(allfrags)[:5] ['NS(=O)(=O)c1ccc(N/N=C2\\C(=O)Nc3ccc(Br)cc32)cc1', '[1*]C(=O)C(C)C', '[1*]C(=O)NN(C)C', '[1*]C(=O)NN1CC[NH+](C)CC1', '[1*]C(C)=O']
BRICSモジュールは、フラグメントの集合にBRICSのルールを適用して新しい分子を生成するオプションも提供しています:
>>> import random >>> random.seed(127) >>> fragms = [Chem.MolFromSmiles(x) for x in sorted(allfrags)] >>> ms = BRICS.BRICSBuild(fragms)
結果はジェネレーターオブジェクトです:
>>> ms
<generator object BRICSBuild at 0x...>
リクエストに応じて分子を返します:
>>> prods = [next(ms) for x in range(10)] >>> prods[0] <rdkit.Chem.rdchem.Mol object at 0x...>
返された分子はサニタイズされていないので、処理を続ける前に少なくとも原子価を更新した方が良いでしょう:
>>> for prod in prods: ... prod.UpdatePropertyCache(strict=False) ... >>> Chem.MolToSmiles(prods[0],True) 'COCCO' >>> Chem.MolToSmiles(prods[1],True) 'O=C1Nc2ccc3ncsc3c2/C1=C/NCCO' >>> Chem.MolToSmiles(prods[2],True) 'O=C1Nc2ccccc2/C1=C/NCCO'
他のフラグメント化手法(Other fragmentation approaches)
[Link] https://www.rdkit.org/docs/GettingStartedInPython.html#other-fragmentation-approaches
上述の方法に加えて、RDKitはユーザーが指定した結合に沿って分子をフラグメント化する非常に柔軟なジェネリック関数を提供しています。
ここでは、簡単なデモンストレーションとして、環構造の中に含まれる原子と含まれない原子との間の結合を全て切断するという場合を試してみましょう。まずはペアとなる原子の組み合わせをすべて見つけることから始めます。
>>> m = Chem.MolFromSmiles('CC1CC(O)C1CCC1CC1') >>> bis = m.GetSubstructMatches(Chem.MolFromSmarts('[!R][R]')) >>> bis ((0, 1), (4, 3), (6, 5), (7, 8))
次に、原子のペアに一致する結合のインデックスを取得します:
>>> bs = [m.GetBondBetweenAtoms(x,y).GetIdx() for x,y in bis] >>> bs [0, 3, 5, 7]
そして、これらの結合のインデックスをフラグメント化関数のインプットとして使います:
>>> nm = Chem.FragmentOnBonds(m,bs)
アウトプットは、結合が切断された場所を示すダミーアトムを有する分子です:
>>> Chem.MolToSmiles(nm,True) '*C1CC([4*])C1[6*].[1*]C.[3*]O.[5*]CC[8*].[7*]C1CC1'
デフォルトでは、接着点は取り除かれた原子のインデックスとともに、(同位体[isotope]を使って)ラベルされています。独自の原子のラベルのセットを、符号なし整数のペアという形で設定することもできます。それぞれのペアの最初の値は、結合の出発点となる原子を置き換えるダミーのためのラベルとして使われ、ペアの2番目の値は、結合の終点となる原子を置き換えるダミーのためのラベルとして使われます。以下の例を見てみましょう。上述の解析を再度行い、接着点に関して、環構造に含まれない原子の場所は10とラベルをつけ、環構造の中の原子には1とラベルをつけています:
>>> bis = m.GetSubstructMatches(Chem.MolFromSmarts('[!R][R]')) >>> bs = [] >>> labels=[] >>> for bi in bis: ... b = m.GetBondBetweenAtoms(bi[0],bi[1]) ... if b.GetBeginAtomIdx()==bi[0]: ... labels.append((10,1)) ... else: ... labels.append((1,10)) ... bs.append(b.GetIdx()) >>> nm = Chem.FragmentOnBonds(m,bs,dummyLabels=labels) >>> Chem.MolToSmiles(nm,True) '[1*]C.[1*]CC[1*].[1*]O.[10*]C1CC([10*])C1[10*].[10*]C1CC1'
12/18/2018
このブログ記事のライセンス
以上はRDKit Documentationの試訳です。
ライセンスはCC BY-SA 4.0で、Greg Landrum氏の功績の元に作成しています。
Creative Commons — Attribution-ShareAlike 4.0 International — CC BY-SA 4.0
Chemical Reactions part1 〜RDKit 直訳 Day17〜
(12/30追記)試訳をまとめたテストサイトを作成しました。よろしければご参照ください。
こちらはRDKit直訳 Advent Calendar 2018 - Adventar 17日目の記事です。基本的な進め方は1日目の記事をご覧ください。
本日の訳出に困った用語
SMARTS-based language: SMARTSベースの記法
MDL rxn files: MDL rxnファイル
unique products: 単一の生成物
sanitize: サニタイズ
the hierarchy of transformations: 変換の階層構造
dummy atom: ダミーアトム
以下、訳
化学反応(Chemical Reactions)
[Link] https://www.rdkit.org/docs/GettingStartedInPython.html#chemical-reactions
RDKitは分子の集合に対して化学反応を適用することもサポートしています。化学反応を構築する一つの方法は、Daylight社のReaction SMILES [脚注11] に似たSMARTSベースの記法を使うことです。:
[脚注11] A more detailed description of reaction smarts, as defined by the rdkit, is in the The RDKit Book.
[link] The RDKit Book
>>> rxn = AllChem.ReactionFromSmarts('[C:1](=[O:2])-[OD1].[N!H0:3]>>[C:1](=[O:2])[N:3]') >>> rxn <rdkit.Chem.rdChemReactions.ChemicalReaction object at 0x...> >>> rxn.GetNumProductTemplates() 1 >>> ps = rxn.RunReactants((Chem.MolFromSmiles('CC(=O)O'),Chem.MolFromSmiles('NC'))) >>> len(ps) # one entry for each possible set of products 1 >>> len(ps[0]) # each entry contains one molecule for each product 1 >>> Chem.MolToSmiles(ps[0][0]) 'CNC(C)=O' >>> ps = rxn.RunReactants((Chem.MolFromSmiles('C(COC(=O)O)C(=O)O'),Chem.MolFromSmiles('NC'))) >>> len(ps) 2 >>> Chem.MolToSmiles(ps[0][0]) 'CNC(=O)OCCC(=O)O' >>> Chem.MolToSmiles(ps[1][0]) 'CNC(=O)CCOC(=O)O
MDL rxnファイルから反応を構築することもできます:
>>> rxn = AllChem.ReactionFromRxnFile('data/AmideBond.rxn') >>> rxn.GetNumReactantTemplates() 2 >>> rxn.GetNumProductTemplates() 1 >>> ps = rxn.RunReactants((Chem.MolFromSmiles('CC(=O)O'), Chem.MolFromSmiles('NC'))) >>> len(ps) 1 >>> Chem.MolToSmiles(ps[0][0]) 'CNC(C)=O'
もちろんアミド結合の形成よりももっと複雑な反応を行うこともできます:
>>> rxn = AllChem.ReactionFromSmarts('[C:1]=[C:2].[C:3]=[*:4][*:5]=[C:6]>>[C:1]1[C:2][C:3][*:4]=[*:5][C:6]1') >>> ps = rxn.RunReactants((Chem.MolFromSmiles('OC=C'), Chem.MolFromSmiles('C=CC(N)=C'))) >>> Chem.MolToSmiles(ps[0][0]) 'NC1=CCCC(O)C1'
この場合、テンプレートへの反応物質のマッピングが1通りではないので、得られるものが複数の生成物を含むセットであることに注意してください:
>>> len(ps) 4
カノニカルSMILESとpythonの辞書型を使うことで、それぞれのユニークな生成物を得ることができます。
>>> uniqps = {}
>>> for p in ps:
... smi = Chem.MolToSmiles(p[0])
... uniqps[smi] = p[0]
...
>>> sorted(uniqps.keys())
['NC1=CCC(O)CC1', 'NC1=CCCC(O)C1']
化学反応を処理するコードから生成される分子はサニタイズされていないことに注意して下さい。あえて不自然な反応例をつかって示します:
>>> rxn = AllChem.ReactionFromSmarts('[C:1]=[C:2][C:3]=[C:4].[C:5]=[C:6]>>[C:1]1=[C:2][C:3]=[C:4][C:5]=[C:6]1') >>> ps = rxn.RunReactants((Chem.MolFromSmiles('C=CC=C'), Chem.MolFromSmiles('C=C'))) >>> Chem.MolToSmiles(ps[0][0]) 'C1=CC=CC=C1' >>> p0 = ps[0][0] >>> Chem.SanitizeMol(p0) rdkit.Chem.rdmolops.SanitizeFlags.SANITIZE_NONE >>> Chem.MolToSmiles(p0) 'c1ccccc1'
より高度な反応機能(Advanced Reaction Functionality)
[Link] https://www.rdkit.org/docs/GettingStartedInPython.html#advanced-reaction-functionality
原子の保護(Protecting Atoms)
[Link] https://www.rdkit.org/docs/GettingStartedInPython.html#protecting-atoms
ありえない生成物を生み出してしまわないように十分に正確に反応を表現することは、時折難しく、特にrxnファイルを扱っているときにこのような問題が生じます。RDKitでは原子を"保護する"方法を提供しており、その原子が反応に参加しないようにすることができます。
上のセクジョンで用いたアミド結合形成の反応を再度用いてデモンストレーションしてみましょう。アミンのクエリが十分に明確ではなく、少なくとも一つの水素原子の結合した窒素原子はどれでもマッチしてしまいます。したがって、この反応をすでにアミド結合を有する分子に適用した場合、アミドのNも反応点として扱われてしまいます:
>>> rxn = AllChem.ReactionFromRxnFile('data/AmideBond.rxn') >>> acid = Chem.MolFromSmiles('CC(=O)O') >>> base = Chem.MolFromSmiles('CC(=O)NCCN') >>> ps = rxn.RunReactants((acid,base)) >>> len(ps) 2 >>> Chem.MolToSmiles(ps[0][0]) 'CC(=O)N(CCN)C(C)=O' >>> Chem.MolToSmiles(ps[1][0]) 'CC(=O)NCCNC(C)=O'
最初の生成物がアミドのNで反応が起きたものに相当します。
全てのアミドのNを保護することで、このような事態が生じるのを防ぐことができます。ここでは、アミドとチオアミドにマッチする部分構造クエリをつかって保護を行い、マッチする原子に"_protected"プロパティをセットしましょう:
>>> amidep = Chem.MolFromSmarts('[N;$(NC=[O,S])]') >>> for match in base.GetSubstructMatches(amidep): ... base.GetAtomWithIdx(match[0]).SetProp('_protected','1')
これで、たった一つだけ生成物を生じるようになります:
>>> ps = rxn.RunReactants((acid,base)) >>> len(ps) 1 >>> Chem.MolToSmiles(ps[0][0]) 'CC(=O)NCCNC(C)=O'
Recapの実装(Recap Implementation)
[Link] https://www.rdkit.org/docs/GettingStartedInPython.html#recap-implementation
化学反応機能と関連して、Recapアルゴリズムの実装を取り上げます。[脚注8] Recapは、実験室でよく使われる反応を模倣した化学変換のセットを使って、分子を一連の合理的なフラグメントに分解します。
[脚注8] Lewell, X.Q.; Judd, D.B.; Watson, S.P.; Hann, M.M. “RECAP-Retrosynthetic Combinatorial Analysis Procedure: A Powerful New Technique for Identifying Privileged Molecular Fragments with Useful Applications in Combinatorial Chemistry” J. Chem. Inf. Comp. Sci. 38:511-22 (1998).
RDKitのrdkit.Chem.Recapの実装は、適用される変換の階層構造の流れを保存します。
[link] rdkit.Chem.Recap
>>> from rdkit import Chem >>> from rdkit.Chem import Recap >>> m = Chem.MolFromSmiles('c1ccccc1OCCOC(=O)CC') >>> hierarch = Recap.RecapDecompose(m) >>> type(hierarch) <class 'rdkit.Chem.Recap.RecapHierarchyNode'>
階層構造の起点は元々の分子です:
>>> hierarch.smiles
'CCC(=O)OCCOc1ccccc1'
それぞれのノードはSMILESをキーとする辞書型を使って、子ノードの情報を保持します。
>>> ks=hierarch.children.keys() >>> sorted(ks) ['*C(=O)CC', '*CCOC(=O)CC', '*CCOc1ccccc1', '*OCCOc1ccccc1', '*c1ccccc1']
階層構造の末端のノード(葉ノード)は簡単にアクセスすることができ、これもSMILESをキーとする辞書型となっています。
>>> ks=hierarch.GetLeaves().keys() >>> ks=sorted(ks) >>> ks ['*C(=O)CC', '*CCO*', '*CCOc1ccccc1', '*c1ccccc1']
分子がフラグメント化された場所では、ダミーアトムをつかって印がつけられていることに注意して下さい。
ノード自体には関連づけられた分子が入っています:
>>> leaf = hierarch.GetLeaves()[ks[0]] >>> Chem.MolToSmiles(leaf.mol) '*C(=O)CC'
12/17/2018
このブログ記事のライセンス
以上はRDKit Documentationの試訳です。
ライセンスはCC BY-SA 4.0で、Greg Landrum氏の功績の元に作成しています。
Creative Commons — Attribution-ShareAlike 4.0 International — CC BY-SA 4.0
RDKit Cookbook 〜RDKit 直訳 Day16〜
(12/30追記)試訳をまとめたテストサイトを作成しました。よろしければご参照ください。
こちらはRDKit直訳 Advent Calendar 2018 - Adventar 16日目の記事です。基本的な進め方は1日目の記事をご覧ください。
本日の訳出に困った用語
align: アラインメントを取る
shape comparison: 形状比較
Shape protrude distance: 形状はみ出し距離
Shape Tanimoto distance: 形状タニモト距離
volume: 体積
3D distance matrix: 3D距離行列
2D-pharmacophore machinery: 2Dファーマコフォア機能
Tanimoto similarity: タニモト類似度
approach: 手法
rotatable bond: 回転可能な結合
ring system: 環系
torsional angle: 二面角
deviation: 偏差
normalization: 規格化
Butina clustering: Butinaクラスタリング
reordering: 並べ替え
helper function: ヘルパー関数
bogus: 偽の、インチキの
shebang: シバン(UNIXのスクリプトの#!から始まる1行目のこと)
TL;DR : too long didn't read(長すぎたから読まなかった)の略語、転じて要約の意。
以下、訳
RDKitクックブック(RDKit Cookbook)
[Link] https://www.rdkit.org/docs/Cookbook.html#rdkit-cookbook
このページについて(What is this?)
[Link] https://www.rdkit.org/docs/Cookbook.html#what-is-this
このページはPythonからRDKitの機能を使って特定のタスクをどうやって実行すれば良いか例を示します。このコンテンツはRDKitコミュニティの協力で作られています。
間違いを見つけたり、より良い方法を提案する場合は、ソースドキュメント(.rstファイル)をご自身で修正していただくか、メーリングリスト: rdkit-discuss@lists.sourceforge.net に送ってください。(メーリングリストを利用する場合はまず加入する必要があります。)
雑多な話題(Miscellaneous Topics)
[Link] https://www.rdkit.org/docs/Cookbook.html#miscellaneous-topics
異なる芳香族性モデルを使う方法(Using a differnt aromaticity model)
[Link] https://www.rdkit.org/docs/Cookbook.html#using-a-different-aromaticity-model
デフォルトでは、RDKitは分子を読み込むときにRDKit自体の芳香族性モデル(RDKit Theory Bookで説明されています)を適用します。ですが、このモデルを上書きし、自分の芳香族性モデルを使うのはとても簡単です。
最も簡単な方法は、保持してほしい芳香族性のセットと合わせてSMILESで分子を提供することです。例えばインドールの場合を考えましょう:
indole1.png
デフォルトではRDKitはどちらの環構造も芳香族であると認識します:
>>> from rdkit import Chem >>> m = Chem.MolFromSmiles('N1C=Cc2ccccc12') >>> m.GetSubstructMatches(Chem.MolFromSmarts('c')) ((1,), (2,), (3,), (4,), (5,), (6,), (7,), (8,))
5員環を脂肪族として扱いたいなら、これは入力のSMILESがどのように書かれているかという問題ですが、ケクレ化[kekulization]と芳香族性を認識する過程をスキップする、部分的なサニタイゼーションを行う必要があるだけです:
>>> m2 = Chem.MolFromSmiles('N1C=Cc2ccccc12',sanitize=False) >>> Chem.SanitizeMol(m2,sanitizeOps=Chem.SanitizeFlags.SANITIZE_ALL^Chem.SanitizeFlags.SANITIZE_KEKULIZE^Chem.SanitizeFlags.SANITIZE_SETAROMATICITY) rdkit.Chem.rdmolops.SanitizeFlags.SANITIZE_NONE >>> m2.GetSubstructMatches(Chem.MolFromSmarts('c')) ((3,), (4,), (5,), (6,), (7,), (8,))
もちろん自分の芳香族性認識関数を書くことも可能ですが、このドキュメントで扱う範囲を超えています。
分子の操作(Manipulating Molecules)
[Link] https://www.rdkit.org/docs/Cookbook.html#manipulating-molecules
ヘテロ環のクリーンアップ(Cleaning up heterocycles)
[Link] https://www.rdkit.org/docs/Cookbook.html#cleaning-up-heterocycles
メーリングリストの議論; * http://www.mail-archive.com/rdkit-discuss@lists.sourceforge.net/msg01185.html * http://www.mail-archive.com/rdkit-discuss@lists.sourceforge.net/msg01162.html * http://www.mail-archive.com/rdkit-discuss@lists.sourceforge.net/msg01900.html * http://www.mail-archive.com/rdkit-discuss@lists.sourceforge.net/msg01901.html
コード:
使用例:
結果:
並列化したコンフォメーション生成(Parallel conformation generation)
[Link] https://www.rdkit.org/docs/Cookbook.html#parallel-conformation-generation
メーリングリストでのディスカッション: http://www.mail-archive.com/rdkit-discuss@lists.sourceforge.net/msg02648.html
コード:
""" オリジナルはAndrew Dalke氏によるものです """ import sys from rdkit import Chem from rdkit.Chem import AllChem # これは右のリンクからダウンロードできます http://pypi.python.org/pypi/futures from concurrent import futures # これは右のリンクからダウンロードできます http://pypi.python.org/pypi/progressbar import progressbar ## 私のマシンではwoker1つで39秒、4つで10秒かかりました。 ## 29.055u 0.102s 0:28.68 101.6% 0+0k 0+3io 0pf+0w #max_workers=1 ## スレッド4つでは11秒かかりました。 ## 34.933u 0.188s 0:10.89 322.4% 0+0k 125+1io 0pf+0w max_workers=4 # (ユーザータイム(u)は子プロセスに使われた時間も含みます # 実時間はそれぞれ28.68秒と10.89でした。) # この関数はサブプロセスで呼び出されます。 # パラメータ(分子とコンフォマーの数)はPythonを介して渡されます。 def generateconformations(m, n): m = Chem.AddHs(m) ids=AllChem.EmbedMultipleConfs(m, numConfs=n, params=AllChem.ETKDG()) # EmbedMultipleConfsはBoost-wrappedタイプを返しますが、これはピッケル化できません。 # Pythonのリストに変換することはできます。 return m, list(ids) smi_input_file, sdf_output_file = sys.argv[1:3] n = int(sys.argv[3]) writer = Chem.SDWriter(sdf_output_file) suppl = Chem.SmilesMolSupplier(smi_input_file, titleLine=False) with futures.ProcessPoolExecutor(max_workers=max_workers) as executor: # 非同期性のジョブのセットを投げます。 jobs = [] for mol in suppl: if mol: job = executor.submit(generateconformations, mol, n) jobs.append(job) widgets = ["Generating conformations; ", progressbar.Percentage(), " ", progressbar.ETA(), " ", progressbar.Bar()] pbar = progressbar.ProgressBar(widgets=widgets, maxval=len(jobs)) for job in pbar(futures.as_completed(jobs)): mol,ids=job.result() for id in ids: writer.write(mol, confId=id) writer.close()
電荷を帯びた分子の中和(Neutralizing Charged Molecules)
[Link] https://www.rdkit.org/docs/Cookbook.html#neutralizing-charged-molecules
メーリングリストでのディスカッション:http://www.mail-archive.com/rdkit-discuss@lists.sourceforge.net/msg02669.html
コード:
""" Hans de Winter氏によるものです """ from rdkit import Chem from rdkit.Chem import AllChem def _InitialiseNeutralisationReactions(): patts= ( # Imidazoles ('[n+;H]','n'), # Amines ('[N+;!H0]','N'), # Carboxylic acids and alcohols ('[$([O-]);!$([O-][#7])]','O'), # Thiols ('[S-;X1]','S'), # Sulfonamides ('[$([N-;X2]S(=O)=O)]','N'), # Enamines ('[$([N-;X2][C,N]=C)]','N'), # Tetrazoles ('[n-]','[nH]'), # Sulfoxides ('[$([S-]=O)]','S'), # Amides ('[$([N-]C=O)]','N'), ) return [(Chem.MolFromSmarts(x),Chem.MolFromSmiles(y,False)) for x,y in patts] _reactions=None def NeutraliseCharges(smiles, reactions=None): global _reactions if reactions is None: if _reactions is None: _reactions=_InitialiseNeutralisationReactions() reactions=_reactions mol = Chem.MolFromSmiles(smiles) replaced = False for i,(reactant, product) in enumerate(reactions): while mol.HasSubstructMatch(reactant): replaced = True rms = AllChem.ReplaceSubstructs(mol, reactant, product) mol = rms[0] if replaced: return (Chem.MolToSmiles(mol,True), True) else: return (smiles, False)
使用例:
smis=("c1cccc[nH+]1", "C[N+](C)(C)C","c1ccccc1[NH3+]", "CC(=O)[O-]","c1ccccc1[O-]", "CCS", "C[N-]S(=O)(=O)C", "C[N-]C=C","C[N-]N=C", "c1ccc[n-]1", "CC[N-]C(=O)CC") for smi in smis: (molSmiles, neutralised) = NeutraliseCharges(smi) print(smi + "->" + molSmiles)
結果:
c1cccc[nH+]1 -> c1ccncc1 C[N+](C)(C)C -> C[N+](C)(C)C c1ccccc1[NH3+] -> Nc1ccccc1 CC(=O)[O-] -> CC(=O)O c1ccccc1[O-] -> Oc1ccccc1 CCS -> CCS C[N-]S(=O)(=O)C -> CNS(C)(=O)=O C[N-]C=C -> C=CNC C[N-]N=C -> C=NNC c1ccc[n-]1 -> c1cc[nH]c1 CC[N-]C(=O)CC -> CCNC(=O)CC
RDKitの3D機能(3D functionality in the RDKit)
[Link] https://www.rdkit.org/docs/Cookbook.html#d-functionality-in-the-rdkit
RDKitには3Dを取り扱う広範囲な機能があります。たとえば:
- Shape alignment
- RMS Calculation
- Shape Tanimoto Distance
- Shape Protrude Distance
- 3D pharmacophore fingerprint
- Torsion fingerprint (deviation)
現在、RDKitでは利用可能なアラインメントのメソッドが2つあります。例として、PDBの同じ分子から2つの結晶構造を使います。
コード:
from rdkit import Chem, RDConfig from rdkit.Chem import AllChem, rdMolAlign # 参照となる分子(reference molecule) ref = Chem.MolFromSmiles('NC(=[NH2+])c1ccc(C[C@@H](NC(=O)CNS(=O)(=O)c2ccc3ccccc3c2)C(=O)N2CCCCC2)cc1') # PDBのコンフォメーション mol1 = Chem.MolFromPDBFile(RDConfig.RDBaseDir+'/rdkit/Chem/test_data/1DWD_ligand.pdb') mol1 = AllChem.AssignBondOrdersFromTemplate(ref, mol1) mol2 = Chem.MolFromPDBFile(RDConfig.RDBaseDir+'/rdkit/Chem/test_data/1PPC_ligand.pdb') mol2 = AllChem.AssignBondOrdersFromTemplate(ref, mol2) # アラインメントを取ります rms = rdMolAlign.AlignMol(mol1, mol2) print(rms) # OPEN3DAlignを使ってアラインメントを取ります pyO3A = rdMolAlign.GetO3A(mol1, mol2) score = pyO3A.Align() print(score)
結果:
1.55001955728 0.376459885045
分子に一つ以上のコンフォマーが含まれている場合、最初のコンフォマーに関してアラインメントを取ります。リストがオプションのRMSlistに渡された場合、アライメントによるRMS値が格納されます。一つの分子の二つのコンフォマーのRMS値は別々に計算することもでき、(prealignedフラグを使うことで)アライメント有り、無しのどちらでも計算可能です。
使用例:
from rdkit import Chem from rdkit.Chem import AllChem mol = Chem.MolFromSmiles('NC(=[NH2+])c1ccc(C[C@@H](NC(=O)CNS(=O)(=O)c2ccc3ccccc3c2)C(=O)N2CCCCC2)cc1') cids = AllChem.EmbedMultipleConfs(mol, numConfs=50, maxAttempts=1000, pruneRmsThresh=0.1) print(len(cids)) # コンフォマーのアラインメントを取ります rmslist = [] AllChem.AlignMolConformers(mol, RMSlist=rmslist) print(len(rmslist)) # コンフォマー1から9のRMSを別々に計算します rms = AllChem.GetConformerRMS(mol, 1, 9, prealigned=True)
結果:
50 49
形状比較のため、RDKitはあらかじめアラインメントされた分子あるいはコンフォマーに対して、形状に基づく距離[Shape-based distances]を2つ用意しています。形状はみ出し距離[Shape protrude distance]は体積のミスマッチに焦点をあてていて、形状タニモト距離[Shape Tanimoto distance]は重ね合わせた体積全体を考慮に入れます。
使用例:
from rdkit import Chem, RDConfig from rdkit.Chem import AllChem, rdMolAlign, rdShapeHelpers ref = Chem.MolFromSmiles('NC(=[NH2+])c1ccc(C[C@@H](NC(=O)CNS(=O)(=O)c2ccc3ccccc3c2)C(=O)N2CCCCC2)cc1') mol1 = Chem.MolFromPDBFile(RDConfig.RDBaseDir+'/rdkit/Chem/test_data/1DWD_ligand.pdb') mol1 = AllChem.AssignBondOrdersFromTemplate(ref, mol1) mol2 = Chem.MolFromPDBFile(RDConfig.RDBaseDir+'/rdkit/Chem/test_data/1PPC_ligand.pdb') mol2 = AllChem.AssignBondOrdersFromTemplate(ref, mol2) rms = rdMolAlign.AlignMol(mol1, mol2) tani = rdShapeHelpers.ShapeTanimotoDist(mol1, mol2) prtr = rdShapeHelpers.ShapeProtrudeDist(mol1, mol2) print(rms, tani, prtr)
結果:
1.55001955728 0.18069102331 0.0962800875274
3Dファーマコフォアフィンガープリントは、3D距離行列を2Dファーマコフォア機能に与えることで、RDKitを使って計算することができます。
使用例:
from rdkit import Chem, DataStructs, RDConfig from rdkit.Chem import AllChem from rdkit.Chem.Pharm2D import Gobbi_Pharm2D, Generate ref = Chem.MolFromSmiles('NC(=[NH2+])c1ccc(C[C@@H](NC(=O)CNS(=O)(=O)c2ccc3ccccc3c2)C(=O)N2CCCCC2)cc1') mol1 = Chem.MolFromPDBFile(RDConfig.RDBaseDir+'/rdkit/Chem/test_data/1DWD_ligand.pdb') mol1 = AllChem.AssignBondOrdersFromTemplate(ref, mol1) mol2 = Chem.MolFromPDBFile(RDConfig.RDBaseDir+'/rdkit/Chem/test_data/1PPC_ligand.pdb') mol2 = AllChem.AssignBondOrdersFromTemplate(ref, mol2) # ファーマコフォアフィンガープリント factory = Gobbi_Pharm2D.factory fp1 = Generate.Gen2DFingerprint(mol1, factory, dMat=Chem.Get3DDistanceMatrix(mol1)) fp2 = Generate.Gen2DFingerprint(mol2, factory, dMat=Chem.Get3DDistanceMatrix(mol2)) # Tanimoto類似度 tani = DataStructs.TanimotoSimilarity(fp1, fp2) print(tani)
結果:
0.451665312754
RDKitはSchulz-Gaschら(J. Chem. Inf. Model, 52, 1499, 2012)により開発された手法、トーションフィンガープリントデビエーション(torsion fingerprint deviation:TFD)も実装しています。一つの分子のコンフォマーのペアについて、回転可能な結合と環系の二面角をトーションフィンガープリント(torsion fingerprint: TF)に記録し、そしてTF間の偏差を計算、規格化、そして合計を取ります。各二面角について。a-b-c-dの4つの原子のセットが選ばれます。
RDKitの実装では、次に示すようにトーションフィンガープリントをカスタマイズすることが可能です。
- 元々の手法では、二面角は分子の中心に対する距離に基づいて重みづけられていました。デフォルトでは、この重みづけが実行されますが、useWeights=Falseフラグを使うことで、実行しないようにできます。
- 対称的な原子aとd(あるいは、aかd)が存在しているとき、全てのとりうる二面角が計算されます。2つの原子が対称的かどうか決定するため、与えられた半径(デフォルトは半径=2)のMorganアルゴリズムに基づくハッシュ・コードが使われます。
- 元々の方法では規格化に使われる最大の偏差は、全ての二面角に対して180.0度です(デフォルト)。maxDev='spec'とすることで、最大偏差に依存する二面角のタイプが規格化に使われます。
- 元々の方法では、三重結合とアレンに隣接する単結合は無視されます(デフォルト)。ignoreColinearBonds='False'とすることで、"連結された(combined)"二面角が使われます。
以上に加えて、Schulz-Gaschらによる方法と異なる点が2、3あります:
- 水素原子は決して考慮されません。
- 元々の方法では、原子bとc(あるいはbかc)に複数の非対称な隣接原子がある場合、原子aとd(あるいはaかd)はランダムに選ばれていました。RDKitの実装では最小のMorgan不変量をもつ原子を選びます。この方法では分子の原子の順番に選択する原子が依存しません。
- 原子aとd(あるいはaかd)が対称的な場合、元々の手法では最も小さい二面角のみを保存しますが、RDKitの実装では全ての取りうる二面角をTFに保存します。続いて、全ての取りうる偏差が決定され、そして最も小さいものがTFDの計算に使われます。この手順により最も小さい偏差がTFDに入ることを保証します。
使用例:
from rdkit import Chem, RDConfig from rdkit.Chem import AllChem, TorsionFingerprints ref = Chem.MolFromSmiles('NC(=[NH2+])c1ccc(C[C@@H](NC(=O)CNS(=O)(=O)c2ccc3ccccc3c2)C(=O)N2CCCCC2)cc1') mol1 = Chem.MolFromPDBFile(RDConfig.RDBaseDir+'/rdkit/Chem/test_data/1DWD_ligand.pdb') mol1 = AllChem.AssignBondOrdersFromTemplate(ref, mol1) mol2 = Chem.MolFromPDBFile(RDConfig.RDBaseDir+'/rdkit/Chem/test_data/1PPC_ligand.pdb') mol2 = AllChem.AssignBondOrdersFromTemplate(ref, mol2) tfd1 = TorsionFingerprints.GetTFDBetweenMolecules(mol1, mol2) tfd2 = TorsionFingerprints.GetTFDBetweenMolecules(mol1, mol2, useWeights=False) tfd3 = TorsionFingerprints.GetTFDBetweenMolecules(mol1, mol2, maxDev='spec') print(tfd1, tfd2, tfd3)
結果:
0.0691236990428 0.111475253992 0.0716255058804
同じ分子のコンフォマー間のTFDを計算する場合、性能上の理由でGetTFDBetweenConformers()関数を使うべきです。
使用例:
from rdkit import Chem, RDConfig from rdkit.Chem import AllChem, TorsionFingerprints ref = Chem.MolFromSmiles('NC(=[NH2+])c1ccc(C[C@@H](NC(=O)CNS(=O)(=O)c2ccc3ccccc3c2)C(=O)N2CCCCC2)cc1') mol1 = Chem.MolFromPDBFile(RDConfig.RDBaseDir+'/rdkit/Chem/test_data/1DWD_ligand.pdb') mol1 = AllChem.AssignBondOrdersFromTemplate(ref, mol1) mol2 = Chem.MolFromPDBFile(RDConfig.RDBaseDir+'/rdkit/Chem/test_data/1PPC_ligand.pdb') mol1.AddConformer(mol2.GetConformer(), assignId=True) tfd = TorsionFingerprints.GetTFDBetweenConformers(mol1, confIds1=[0], confIds2=[1]) print(tfd)
結果:
[0.0691...]
コンフォマーRMSとTFDの値のため、RDKitでは、Butinaクラスタリングといったクラスタリングアルゴリズムに与えることができる対称行列を直接計算する簡易関数を用意しています。フラグの並べ替えにより、クラスターが作られるたびに毎回、クラスタリングされていない分子について近接するものの数が更新されることを保証します。
使用例:
from rdkit import Chem from rdkit.Chem import AllChem, TorsionFingerprints from rdkit.ML.Cluster import Butina mol = Chem.MolFromSmiles('NC(=[NH2+])c1ccc(C[C@@H](NC(=O)CNS(=O)(=O)c2ccc3ccccc3c2)C(=O)N2CCCCC2)cc1') cids = AllChem.EmbedMultipleConfs(mol, numConfs=50, maxAttempts=1000, pruneRmsThresh=0.1) # RMS行列(RMS matrix) rmsmat = AllChem.GetConformerRMSMatrix(mol, prealigned=False) # TFD行列(TFD matrix) tfdmat = TorsionFingerprints.GetTFDMatrix(mol) # クラスタリング num = mol.GetNumConformers() rms_clusters = Butina.ClusterData(rmsmat, num, 2.0, isDistData=True, reordering=True) tfd_clusters = Butina.ClusterData(tfdmat, num, 0.3, isDistData=True, reordering=True)
RDKitと一緒にscikit-learnを使う方法(Using scikit-learn with RDKit)
[Link] https://www.rdkit.org/docs/Cookbook.html#using-scikit-learn-with-rdkit
scikit-learnはPythoのための機械学習のライブラリで、様々な教師あり、教師無しの手法を含みます。siki-learnに関するドキュメンテーションはこちらにあります:http://scikit-learn.org/stable/user_guide.html 。
RDKitのフィンガープリントはscikit-learnの機械学習モデルを訓練するのに使うことができます。これはランダムフォレストの例です。
コード:
from rdkit import Chem, DataStructs from rdkit.Chem import AllChem from sklearn.ensemble import RandomForestClassifier import numpy # 4つの分子を生成 m1 = Chem.MolFromSmiles('c1ccccc1') m2 = Chem.MolFromSmiles('c1ccccc1CC') m3 = Chem.MolFromSmiles('c1ccncc1') m4 = Chem.MolFromSmiles('c1ccncc1CC') mols = [m1, m2, m3, m4] # フィンガープリントを生成: 半径2のMorganフィンガープリント fps = [AllChem.GetMorganFingerprintAsBitVect(m, 2) for m in mols] # RDKitの明示的なベクトルをNumpyのアレイに変換 np_fps = [] for fp in fps: arr = numpy.zeros((1,)) DataStructs.ConvertToNumpyArray(fp, arr) np_fps.append(arr) # 100個のツリーをもつランダムフォレスト分類器を手に入れます rf = RandomForestClassifier(n_estimators=100, random_state=1123) # 最初の2つの分子を活性あり(active, class 1)、 # 残りの2つを活性無し(inactive, class 0)として、 # ランダムフォレストを訓練します ys_fit = [1, 1, 0, 0] rf.fit(np_fps, ys_fit) # 新しい分子を予測するためにランダムフォレストを使います m5 = Chem.MolFromSmiles('c1ccccc1O') fp = numpy.zeros((1,)) DataStructs.ConvertToNumpyArray(AllChem.GetMorganFingerprintAsBitVect(m5, 2), fp) print(rf.predict((fp,))) print(rf.predict_proba((fp,)))
scikit-learn バージョン0.13での出力結果は:
[1] [[ 0.14 0.86]]
このモデルに対して類似度マップを作成しましょう。
コードは:
from rdkit.Chem.Draw import SimilarityMaps # ヘルパー関数 def getProba(fp, predictionFunction): return predictionFunction((fp,))[0][1] m5 = Chem.MolFromSmiles('c1ccccc1O') fig, maxweight = SimilarityMaps.GetSimilarityMapForModel(m5, SimilarityMaps.GetMorganFingerprint, lambda x: getProba(x, rf.predict_proba))
これで、次の結果が返ってきます:
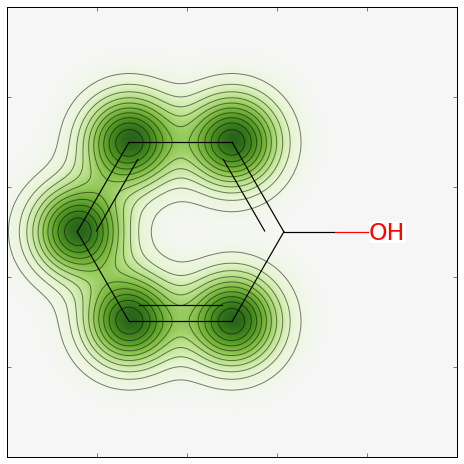
similarity_map_rf.png
カスタムMCSアトムタイプを使う方法(Using custom MCS atom types)
[Link] https://www.rdkit.org/docs/Cookbook.html#using-custom-mcs-atom-types
メーリングリストの議論: http://www.mail-archive.com/rdkit-discuss@lists.sourceforge.net/msg03676.html
IPython notebook:
http://nbviewer.ipython.org/gist/greglandrum/8351725
https://gist.github.com/greglandrum/8351725
MCSについては可読性のあるSMILESを手に入れることができますが、このセクションの目標はMCSコードでカスタムアトムタイプを使えるようになることです。マッチングに同位体の情報を使うために、MCSコードのオプションを使います。そして、我々の同位体の情報を含む偽の同位体の値をセットします。
コード:
from rdkit import Chem from rdkit.Chem import rdFMCS # テストに用いる分子: smis=["COc1ccc(C(Nc2nc3c(ncn3COCC=O)c(=O)[nH]2)(c2ccccc2)c2ccccc2)cc1", "COc1ccc(C(Nc2nc3c(ncn3COC(CO)(CO)CO)c(=O)[nH]2)(c2ccccc2)c2ccccc2)cc1"] ms = [Chem.MolFromSmiles(x) for x in smis] def label(a): " a simple hash combining atom number and hybridization " return 100*int(a.GetHybridization())+a.GetAtomicNum() # あとで分子を変えるので、コピーしておきます: nms = [Chem.Mol(x) for x in ms] for nm in nms: for at in nm.GetAtoms(): at.SetIsotope(label(at)) mcs=rdFMCS.FindMCS(nms,atomCompare=rdFMCS.AtomCompare.CompareIsotopes) print(mcs.smartsString)
これで次の出力が得られます:
[406*]-[308*]-[306*]1:[306*]:[306*]:[306*](:[306*]:[306*]:1)-[406*](-[307*]-[306*]1:[307*]:[306*]2:[306*](:[306*](:[307*]:1)=[308*]):[307*]:[306*]:[307*]:2-[406*]-[408*]-[406*])(-[306*]1:[306*]:[306*]:[306*]:[306*]:[306*]:1)-[306*]1:[306*]:[306*]:[306*]:[306*]:[306*]:1
これが我々が求めたものではありますが、必ずしも可読性の高いものではありません。2ステップのプロセスで、より読みやすい形式とすることができます:
- MCSの部分構造マッチングをコピーした分子に対して行う
- コピーとマッチした原子だけを使って、元々の分子のSMILESを生成する
我々はコピーと元々の分子のアトムインデックスが同じであると知っているので、これで上手くいきます。
def getMCSSmiles(mol,labelledMol,mcs): mcsp = Chem.MolFromSmarts(mcs.smartsString) match = labelledMol.GetSubstructMatch(mcsp) return Chem.MolFragmentToSmiles(mol,atomsToUse=match, isomericSmiles=True, canonical=False) print(getMCSSmiles(ms[0],nms[0],mcs)) COc1ccc(C(Nc2nc3c(ncn3COC)c(=O)[nH]2)(c2ccccc2)c2ccccc2)cc1
これが我々が探し求めていたものです。
分子のクラスタリング(Clustering molecules)
[Link] https://www.rdkit.org/docs/Cookbook.html#clustering-molecules
大きな分子のセット(1000から2000以上)には、Butinaクラスタリングアルゴリズムを使うのが最も効率が良いです。
これが、フィンガープリントのセットに対してクラスタリングを行うためのコードです:
def ClusterFps(fps,cutoff=0.2): from rdkit import DataStructs from rdkit.ML.Cluster import Butina # まず距離行列を生成します: dists = [] nfps = len(fps) for i in range(1,nfps): sims = DataStructs.BulkTanimotoSimilarity(fps[i],fps[:i]) dists.extend([1-x for x in sims]) # データをクラスタリングします: cs = Butina.ClusterData(dists,nfps,cutoff,isDistData=True) return cs
戻り値はクラスターのタプルで、各クラスターはidのタプルとなっています。
使用例:
from rdkit import Chem from rdkit.Chem import AllChem import gzip ms = [x for x in Chem.ForwardSDMolSupplier(gzip.open('zdd.sdf.gz')) if x is not None] fps = [AllChem.GetMorganFingerprintAsBitVect(x,2,1024) for x in ms] clusters=ClusterFps(fps,cutoff=0.4)
変数のクラスターが結果を含んでいます:
>>> print(clusters[200]) (6164, 1400, 1403, 1537, 1543, 6575, 6759)
このクラスタには7点含まれており、クラスターの重心は6164です。
N個の分子の間でRMSDを計算(RMSD Calculation between N molecules)
[Link] https://www.rdkit.org/docs/Cookbook.html#rmsd-calculation-between-n-molecules
イントロダクション(Introduction)
[Link] https://www.rdkit.org/docs/Cookbook.html#introduction
時々、2つ(あるいはそれ以上の)分子間のRMSD距離を計算する必要が生じます。これは2つのコンフォマーのがどれだけ近いかを計算するのに使われます。ほとんどのRMSD計算は類似の化合物、あるいは、少なくとも共通の部分をもつ異なる化合物についてのみ、意味をなします。
詳細(Details)
[Link] https://www.rdkit.org/docs/Cookbook.html#details
(Python 2.7で書かれた)次のプログラムはSDFファイルを入力とし、ファイルに含まれる分子間の全てのRMSD距離を生成します。これらの距離は(ユーザーが定義した)出力ファイルに書き込まれます。
5つのコンフォマーを持つSDFにたいしては、10個のRMSDスコアが得られます。典型的な、n個から反復無しにk個を選び出す問題です。すなわち、5!/2!(5-2)!。
コード:
#!/usr/bin/python ''' ファイルの中の全ての構造間のRMSDの差を計算 @author: JP <jp@javaclass.co.uk> ''' import os import getopt import sys # rdkitをインポート from rdkit import Chem from rdkit.Chem import AllChem ''' 文字列のコンテンツをファイルに書き込む ''' def write_contents(filename, contents): # 基本的なチェックをいくつか行います。厳密に言えばassertを使うことができます。 assert filename is not None, "filename cannot be None" assert contents is not None, "contents cannot be None" f = open(filename, "w") f.write(contents) f.close() # ファイルをクローズします ''' リストをファイルに書き込みます ''' def write_list_to_file(filename, list, line_sep = os.linesep): # 基本的なチェックをいくつか行います。厳密に言えばassertを使うことができます。 assert list is not None and len(list) > 0, "list cannot be None or empty" write_contents(filename, line_sep.join(list)) ''' RMSDのスプレッドを計算 ''' def calculate_spread(molecules_file): assert os.path.isfile(molecules_file), "File %s does not exist!" % molecules # イテレータの取得 mols = Chem.SDMolSupplier(molecules_file) spread_values = [] # いくつの分子がファイルの中に含まれているか? mol_count = len(mols) # それぞれの分子を他の全ての分子と比較します。 # 典型的なn個からk個を選択する話です(nから2個選択) # 組み合わせの数は(n!) / k!(n-k)! で与えら得れます(私の数学能力が錆ついていなければ) for i in range(mol_count - 1): for j in range(i+1, mol_count): # 何かの処理が行われていることを表示するようにしましょう・・・mol_countの値が大きい場合、ある程度の時間がかかるので print("Aligning molecule #%d with molecule #%d (%d molecules in all)" % (i, j, mol_count)) # RMSDを計算し、アレイに格納します # AlignMolと異なって、これは対称性の処理も行います spread_values.append(str(AllChem.GetBestRMS(mols[i], mols[j]))) # アレイを返します return spread_values def main(): try: # オプションは次の通りです: # f - 実際の構造のファイル opts, args = getopt.getopt(sys.argv[1:], "vf:o:") except getopt.GetoptError, err: # ヘルプの情報をprint関数で出力し、終了します: print(str(err)) # これは "option -a not recognized" といったようなものを出力します sys.exit(401) # デフォルト molecules_file = None output_file = None for opt, arg in opts: if opt == "-v": print("RMSD Spread 1.1") sys.exit() elif opt == "-f": molecules_file = arg elif opt == "-o": output_file = arg else: assert False, "Unhandled option: " + opt # 次をassertしてください - 最も綺麗な方法ではありませんが、上手くいきます assert molecules_file is not None, "file containing molecules must be specified, add -f to command line arguments" assert output_file is not None, "output file must be specified, add -o to command line arguments" # RMSDスプレッドの値を得ます spread_values = calculate_spread(molecules_file) # ファイルに書き込みます write_list_to_file(output_file, spread_values) if __name__ == "__main__": main()
このプログラムは次のような方法でコマンドラインから実行されるかもしれません(Pythonのインタプリタが/usr/bin/pythonにある場合です。その他の場合は最初の行(滑稽な名前がつけられたシバン)を編集してください:
calculate_spread.py -f my_conformers.sdf -o my_conformers.rmsd_spread.txt
要約: AllChem.GetBestRMS(mol1, mol2)の行はRMSDを不動小数点型として返し、このプログラムの要点となっています。GetBestRMS()はAlignMol()と異なり対称性を考慮に入れた処理を行います。
ライセンス(License)
[Link] https://www.rdkit.org/docs/Cookbook.html#license
https://www.rdkit.org/docs/Overview.html#license
この文書の著作権は copyright (C) 2013-2018 by Greg Landrum に所属しています。
この文書はCreative Commons Attribution-ShareAlike 4.0 Licenseのもとでライセンスされています。このライセンスを見るためにはhttp://creativecommons.org/licenses/by-sa/4.0/ にアクセスするか、Creative Commons, 543 Howard Street, 5th Floor, San Francisco, California, 94105, USA. に手紙を送ってください。
このライセンスの意図はRDKitそのものの意図と似ています。簡単に言えば "これを使ってなんでもやりたいことをやっていいけど、私たちの功績にも言及してね”
12/16/2018
このブログ記事のライセンス
以上はRDKit Documentationの試訳です。
ライセンスはCC BY-SA 4.0で、Greg Landrum氏の功績の元に作成しています。
Creative Commons — Attribution-ShareAlike 4.0 International — CC BY-SA 4.0
Descriptor Calculation 〜RDKit 直訳 Day15〜
(12/30追記)試訳をまとめたテストサイトを作成しました。よろしければご参照ください。
こちらはRDKit直訳 Advent Calendar 2018 - Adventar 15日目の記事です。基本的な進め方は1日目の記事をご覧ください。
本日の訳出に困った用語
Gasteiger partial charges: Gasteiger部分電荷
color sheme: カラースキーム、色彩設計
Crippen contributions: Crippen寄与
以下、訳
記述子計算(Descriptor Calculation)
[Link] https://www.rdkit.org/docs/GettingStartedInPython.html#descriptor-calculation
RDKitには様々な記述子が実装されています。記述子の完全なリストはList of Available Descriptorsを参照してください。
[list] List of Available Descriptors
記述子のほとんどはPythonから集中型のrdkit.Chem.Descriptorsモジュールを介して直接使うことができます:
[list] rdkit.Chem.Descriptors
>>> from rdkit.Chem import Descriptors >>> m = Chem.MolFromSmiles('c1ccccc1C(=O)O') >>> Descriptors.TPSA(m) 37.3 >>> Descriptors.MolLogP(m) 1.3848
部分電荷の扱い方は少し異なります:
>>> m = Chem.MolFromSmiles('c1ccccc1C(=O)O') >>> AllChem.ComputeGasteigerCharges(m) >>> float(m.GetAtomWithIdx(0).GetProp('_GasteigerCharge')) -0.047...
記述子の可視化(Visualization of Descriptors)
[Link] https://www.rdkit.org/docs/GettingStartedInPython.html#visualization-of-descriptors
類似度マップは原子ごとの寄与に分割された記述子の可視化に使うことができます。
Gasteiger部分電荷は(異なるカラースキームを使って)次のように可視化できます:
>>> from rdkit.Chem.Draw import SimilarityMaps >>> mol = Chem.MolFromSmiles('COc1cccc2cc(C(=O)NCCCCN3CCN(c4cccc5nccnc54)CC3)oc21') >>> AllChem.ComputeGasteigerCharges(mol) >>> contribs = [float(mol.GetAtomWithIdx(i).GetProp('_GasteigerCharge')) for i in range(mol.GetNumAtoms())] >>> fig = SimilarityMaps.GetSimilarityMapFromWeights(mol, contribs, colorMap='jet', contourLines=10)
このような図が生成されます:
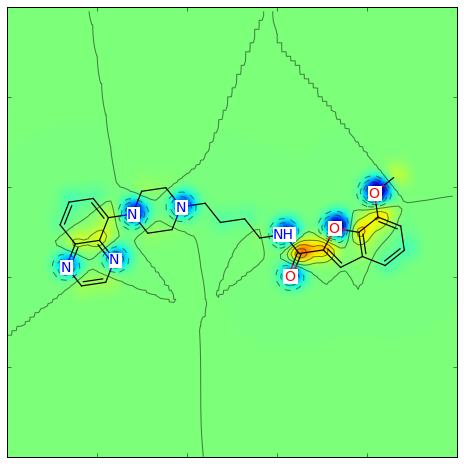 similarity_map_charges.png
similarity_map_charges.png
logPへのCrippen寄与を可視化するには次のようにします:
>>> from rdkit.Chem import rdMolDescriptors >>> contribs = rdMolDescriptors._CalcCrippenContribs(mol) >>> fig = SimilarityMaps.GetSimilarityMapFromWeights(mol,[x for x,y in contribs], colorMap='jet', contourLines=10)
このような図が生成されます:
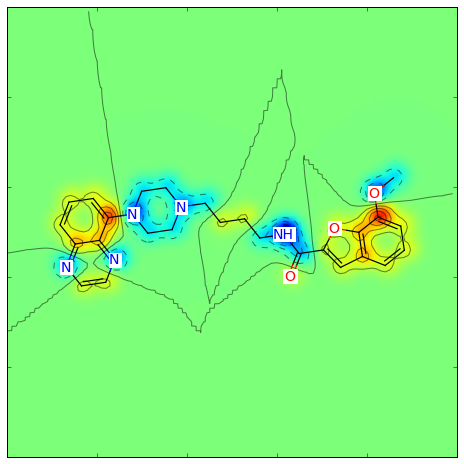 similarity_map_crippen.png
similarity_map_crippen.png
12/15/2018
このブログ記事のライセンス
以上はRDKit Documentationの試訳です。
ライセンスはCC BY-SA 4.0で、Greg Landrum氏の功績の元に作成しています。
Creative Commons — Attribution-ShareAlike 4.0 International — CC BY-SA 4.0
Fingerprinting and Molecular Similarity part3 〜RDKit 直訳 Day14〜
(12/30追記)試訳をまとめたテストサイトを作成しました。よろしければご参照ください。
こちらはRDKit直訳 Advent Calendar 2018 - Adventar 14日目の記事です。基本的な進め方は1日目の記事をご覧ください。
本日の訳出に困った用語
picker: ピッカー
similarity map: 類似度マップ
similarity metric: 類似度の指標
Dice silmilarity: Dice類似度
convenience function: 簡易関数
normalization: 正規化
以下、訳
フィンガープリントを使って多種多様な分子を選択する(Picking Diverse Molecules Using Fingerprints)
[Link] https://www.rdkit.org/docs/GettingStartedInPython.html#picking-diverse-molecules-using-fingerprints
多数の分子を含む集合から、多種多様な分子を選択してより小さな部分集合をつくることはよく行われます。RDkitではこの作業のため、rdkit.SimDivFiltersモジュールに多数の方法を用意しています。最も効率的な方法はMaxMinアルゴリズムを使うことです[脚注6]。試しにやってみましょう。
[link] rdkit.SimDivFilters
[脚注6] Ashton, M. et al. “Identification of Diverse Database Subsets using Property-Based and Fragment-Based Molecular Descriptions.” Quantitative Structure-Activity Relationships 21:598-604 (2002).
まずは分子を1セット読み込んでMorganフィンガープリントを生成します:
>>> from rdkit import Chem >>> from rdkit.Chem.rdMolDescriptors import GetMorganFingerprint >>> from rdkit import DataStructs >>> from rdkit.SimDivFilters.rdSimDivPickers import MaxMinPicker >>> ms = [x for x in Chem.SDMolSupplier('data/actives_5ht3.sdf')] >>> while ms.count(None): ms.remove(None) >>> fps = [GetMorganFingerprint(x,3) for x in ms] >>> nfps = len(fps)
MaxMinアルゴリズムにはオブジェクト間の距離を計算する関数が必要です。ここではDiceSimilarityを使いましょう。
>>> def distij(i,j,fps=fps): ... return 1-DataStructs.DiceSimilarity(fps[i],fps[j])
それではピッカーを生成し、10個の多様な分子のセットを取りだしましょう:
>>> picker = MaxMinPicker() >>> pickIndices = picker.LazyPick(distij,nfps,10,seed=23) >>> list(pickIndices) [93, 109, 154, 6, 95, 135, 151, 61, 137, 139]
ピッカーはフィンガープリントのインデックスを返すだけ、ということに注意してください。分子そのものを取得するには次のようにします:
>>> picks = [ms[x] for x in pickIndices]
フィンガープリントを使って類似度マップを生成する(Generating Similarity Maps Using Fingerprints)
[Link] https://www.rdkit.org/docs/GettingStartedInPython.html#generating-similarity-maps-using-fingerprints
類似度マップはある分子と参照とする分子の間の類似度に対して、各原子の寄与を可視化する方法です。参考文献17 [脚注17] に方法が記載されています。rdkit.Chem.Draw.SimilarityMaps モジュールを使用してください:
[脚注17] Riniker, S.; Landrum, G. A. “Similarity Maps - A Visualization Strategy for Molecular Fingerprints and Machine-Learning Methods” J. Cheminf. 5:43 (2013).
[link] rdkit.Chem.Draw.SimilarityMaps
まずは分子を2つ作るところから始めます:
>>> from rdkit import Chem >>> mol = Chem.MolFromSmiles('COc1cccc2cc(C(=O)NCCCCN3CCN(c4cccc5nccnc54)CC3)oc21') >>> refmol = Chem.MolFromSmiles('CCCN(CCCCN1CCN(c2ccccc2OC)CC1)Cc1ccc2ccccc2c1')
SimilarityMapsモジュールはatom pairs、topological torsions、Morganフィンガープリントの3種類のフィンガープリントをサポートしています:
>>> from rdkit.Chem import Draw >>> from rdkit.Chem.Draw import SimilarityMaps >>> fp = SimilarityMaps.GetAPFingerprint(mol, fpType='normal') >>> fp = SimilarityMaps.GetTTFingerprint(mol, fpType='normal') >>> fp = SimilarityMaps.GetMorganFingerprint(mol, fpType='bv')
atom pairsとtopological torsionsにはデフォルトのノーマル(normal)とハッシュ(hashed)、そしてビットベクトル(bit vector, bv)の3つのタイプがあります。Morganフィンガープリントにはデフォルトのビットベクトル(bit vector, bv)と、カウントベクトル(count vector, count)の2つのタイプがあります。
2つのフィンガープリント間の類似度マップを生成する関数には、フィンガープリントの生成に用いた関数を特定する必要があり、またオプションとして類似度の指標を指定できます。デフォルトの指標はDice類似度です。Morganフィンガープリント関数をすべてデフォルト引数で使用した場合、類似度マップは次のように生成できます:
>>> fig, maxweight = SimilarityMaps.GetSimilarityMapForFingerprint(refmol, mol, SimilarityMaps.GetMorganFingerprint)
このような図が生成されます:
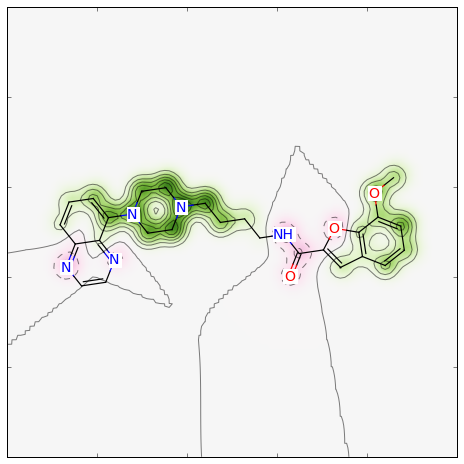
similarity_map_fp1.png
Morganフィンガープリントのタイプをデフォルトのビットベクトルからカウント(count)タイプに変更し、半径を2から1へ、類似度指標をDiceからTanimoto類似度へと変更した場合、以下のようになります:
>>> from rdkit import DataStructs >>> fig, maxweight = SimilarityMaps.GetSimilarityMapForFingerprint(refmol, mol, lambda m,idx: SimilarityMaps.GetMorganFingerprint(m, atomId=idx, radius=1, fpType='count'), metric=DataStructs.TanimotoSimilarity)
このような図が生成されます:
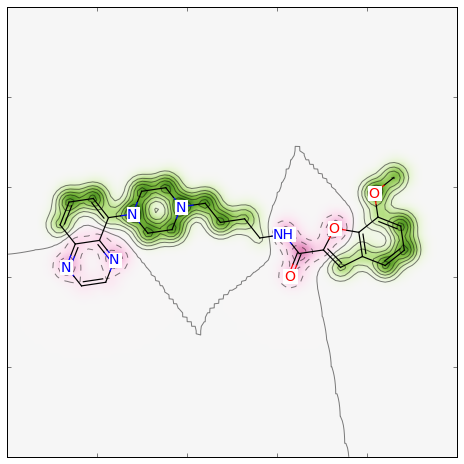
similarity_map_fp2.png
簡易関数のGetSimilarityMapForFingerprintには原子の重みの正規化が含まれており、重みの絶対値の最大が1となるよう正規化されます。したがって、この関数はマップを作成するときにみつかった、最大の重みを出力します。
>>> print(maxweight) 0.05747...
正規化の処理を行って欲しくない時は、次のようにしてマップを作ることができます:
>>> weights = SimilarityMaps.GetAtomicWeightsForFingerprint(refmol, mol, SimilarityMaps.GetMorganFingerprint) >>> print(["%.2f " % w for w in weights]) ['0.05 ', ... >>> fig = SimilarityMaps.GetSimilarityMapFromWeights(mol, weights)
このような図が生成されます:
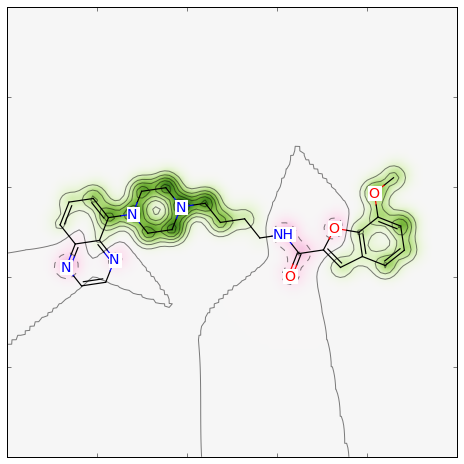
similarity_map_fp3.png
12/14/2018
このブログ記事のライセンス
以上はRDKit Documentationの試訳です。
ライセンスはCC BY-SA 4.0で、Greg Landrum氏の功績の元に作成しています。
Creative Commons — Attribution-ShareAlike 4.0 International — CC BY-SA 4.0
低分子でタンパク質の量を制御 〜アゴニストでもアンタゴニストでも阻害剤でもないMOA〜
創薬 Advent Calendar 2018 - Adventar 13日目が空いていたので埋めます。
創薬アドベントカレンダーを読むような方々には釈迦に説法という感があり恐縮なのですが、しばしお付き合いいただければと思います。
言うまでもありませんが、医薬品となる化合物の多くがタンパク質と結合し、その薬効を発揮してます(*1)。インシリコ創薬に関する話題でいえば、結合するか否かの予測(ドッキングやSBVS)や活性の予測ということになると思いますが、ではその活性ってどんな種類があるの?ということについて調べました。
活性の種類(作用機序: MOA)といえば、多くはアゴニストやアンタゴニスト・阻害剤として機能していると思います。タンパク質と結合して、本来のシグナルを流すスイッチをONにするか、流れているシグナルを止める、酵素活性を阻害するといった感じです。これらは基本的に結合の対象となる”タンパク質の機能"に対して影響を及ぼすことで、薬効を発揮しています。
ところで低分子医薬品の中には、"タンパク質の量"のコントロールを行うものもあるそうです。そこで今回、そのようなメカニズム、プロテインノックダウン法とケミカルシャペロンについてとりあげたいと思います。
①タンパク質の分解を誘導する (プロテインノックダウン法)
GPCRのようにシグナルに関与するわけでもなく、キナーゼのように酵素活性があるわけでもない、そのようなタンパク質を標的として創薬を行うにはどうすればよいか?
「そんなの壊してしまえばいいじゃない」
というラディカルな発想がこのメカニズムです。
ユビキチン-プロテアソーム系
タンパク質の分解を低分子によって誘導してやろう、というわけなのですが、このため生体内にもとからあるタンパク質分解機構「ユビキチン-プロテアソーム系」を利用します。こちらは2004年にノーベル化学賞を受賞した機構ですが、2016年のノーベル生理学・医学賞「オートファジー」が不要なゴミをざざっと分解するのに対して、もう少し精密(?)で特定のタンパク質を狙って分解するという仕組みです。
具体的には標的となるタンパク質をユビキチンで標識し、その標識を頼りにプロテアソームが分解を行います。ユビキチン標識にはユビキチン活性化酵素(E1)、ユビキチン結合酵素(E2)、ユビキチンリガーゼ(E3)と呼ばれる酵素群素が関与しますが、このうちE3リガーゼが分解の標的となるタンパク質の認識に重要となります。

プロテアソーム阻害剤
本題のプロテインノックダウン法とは異なりますが、すでにユビキチンープロテアソーム系を標的とした医薬品は上市されており、ボルテゾミブ、カルフィルゾミブというものがあります。両者ともにプロテアソームを標的とするプロテアソーム阻害剤で、ホウ素を含んでいたりエポキシケトンがあったりと、あまり医薬品では見かけないような(毒性が出ないか心配になる)構造をしています。
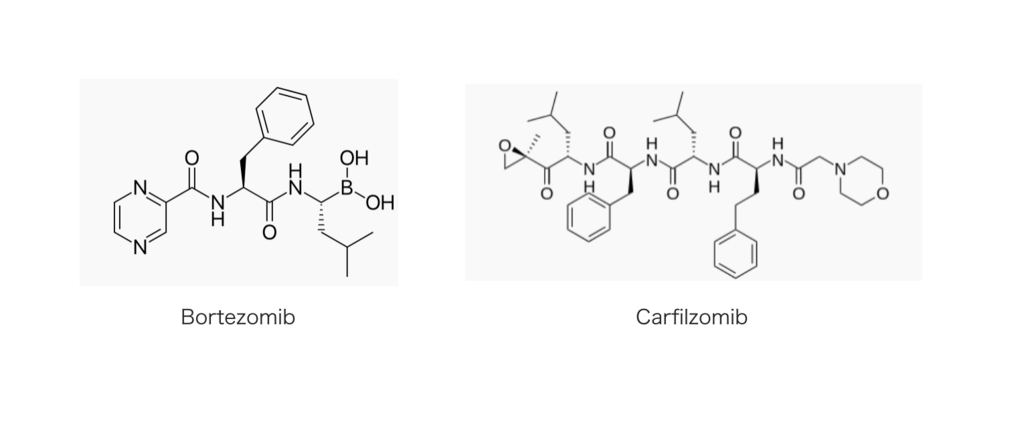
ボルテゾミブはプロテアソームの可逆的阻害剤、カルフィルゾミブは不可逆的阻害剤です。ざっと見る限り副作用も多いようですが、適応は多発性骨髄腫。生死に関わる疾患であるだけに、薬効とのバランスで、ある程度の副作用性は許容されるのでしょうか? ( 薬効と毒性の問題については創薬アドベントカレンダー 7日目のこちらの記事 でも取り上げられていましたので、未読の方は是非ご一読を!)
ところで、上記のカルフィルゾミブの発見に携わったのがYale大学のCraig M. Crews教授(Crews Laboratory)ですが、プロテインノックダウン法においてもキーパーソンです。
プロテインノックダウン法
プロテインノックダウン法では、プロテアソームではなくユビキチン標識に働く酵素群をターゲットとしています。今、ユビキチン化の基質認識に働くユビキチンE3リガーゼ(A)と結合する化合物(X)が手元にあるとします。これとは別に、壊してしまいたい標的のタンパク質(B)と結合する化合物(Y)を見つけてきます。この2つの化合物XとYをうまくリンカーでつないで、新しい化合物(Z)を作ります。ZはE3リガーゼ(A)とタンパク質(B)の両方に結合するため、Zによって両者が引き寄せられます。するとE3リガーゼがタンパク質Bを分解すべきタンパク質だ、と認識してユビキチン標識を行います。ユビキチン標識されたタンパク質Bはプロテアソームにより分解されるため、狙ったタンパク質を分解することができる、というのがこの手法のコンセプトです。

プロテインノックダウン法ですが、いくつか流派(?)があり、それぞれ利用するE3リガーゼや、E3リガーゼに結合する部分構造などに違いがあるようです。
上述のCrews教授らはPROTAC法(Proteolysis Targeting Chimeraの略)と名付け、von Hippel-Lindau (VHL)やcelebron(CRBN)をE3リガーゼとして用いています(Fig.3 参照)。また、Blander博士らはDegronimidと名付け、同じくCRBNを標的E3リガーゼとしていますが、E3リガーゼのリガンドとしてサリドマイド誘導体を用いています。日本では国立医薬品食品衛生え研究所の内藤先生・大岡先生らのグループでE3リガーゼとしてIAPを用いたSNIPER(Specific and Nongenetic Inhibitor of Apoptosis Protein(IAP)-dependent Portein Eraser)という技術の開発が行われています。
新しい創薬の手法としてプロテインノックダウン 法は注目を集めており、今年は第一回目となる国際会議Targeted Protein Degradation Summit 2018なども開かれたそうです。
長所・短所
では、プロテインノックダウン法には従来の創薬手法と比較してどのような長所があるのでしょうか?
長所①
まず標的とできるタンパク質の範囲が広がったということがあげられます。"Drugging Undruggable Targets"というキャッチーなフレーズが表すように、従来の低分子医薬品では標的とすることが難しかったタンパク質を標的とすることができる、と言われています。繰り返しになりますが、酵素として働くわけでも、シグナル伝達に関わるわけでもない、それ自体の機能が明確ではないタンパク質であったとしても、結合する部分構造さえ見つけてしまえば、分解というアプローチで標的とすることができます。
(妥当かどうかは置いておいて)わかりやすい例ではアルツハイマー病との関連が示唆されているアミロイドβや、パーキンソン病と関連するα-シヌクレインがあげられるでしょうか? 両者ともに細胞内に蓄積することで神経細胞死を起こす、といわれているため、分解を誘導し蓄積を解消できれば治療効果が期待できるかもしれません(*4)。
長所②
また、結合の位置がタンパク質の機能と関わりのある場所である必要がないため、高い選択性をもつ化合物が得られる可能性があります。例えばキナーゼを標的とする場合、多くの低分子阻害剤は酵素活性と密接に関係するATP結合サイトを結合サイトとして利用しています。ところが、機能と密接に関係する部位のため、異なるキナーゼ間でもタンパク質の構造の類似性が高く、狙ったキナーゼのみを阻害することが難しくなります。
一方で、プロテインノックダウン 法であれば、目的は酵素機能の阻害ではなく、とにかく結合してユビキチンE3リガーゼへと誘導できればよいわけですから、化合物の結合部位の選択肢が広がります。したがって、狙ったタンパク質に特有の部分構造(あまり進化で保存されて部位など?)を結合部位として利用できる可能性があり、その意味で高い選択性を発揮するポテンシャルがあります。
もちろん長所ばかりではありません。
短所①
Fig. 3のPROTACの分子構造を見ていただければ一目瞭然ですが、二つの構造を繋ぐという手法のため、分子が巨大化しやすいという問題点があります(単純化すれば従来の低分子 x 2以上の分子量)。昨年の創薬アドベントカレンダーの記事(Druglikenessについてのよもやま話 – y__sama – Medium )でもとりあげられていましたが、一般に、分子量の増大は低分子医薬品にとってはできれば避けたいといわれています(Rule of 5)。理由としては細胞膜透過性の低下による経口吸収性の低下や、薬物動態の悪化、などでしょうか?、、、とにかくあんまり良く無いっぽい(適当)
短所②
また、プロテインノックダウン 法の化合物は2種類のタンパク質に同時に結合しなければなりません。したがって、単純に「化合物 対 タンパク質 = 1 対 1」とくらべて結合のために出会う確率が下がります。そのため、細胞系の実験では高濃度にしなければ望みの効果が発揮されないといった問題点も指摘されていました。この点については、より洗練された化合物の開発により低濃度でも薬効を示すものが見出されているそうです。
実用化に向けて
肯定派・否定派さまざまな意見があるようですが、低分子で標的とできるターゲットタンパク質はあらかた出尽くした、とか、ターゲットが枯渇している、などと言われてている医薬品業界で、このように新しい創薬の手法・標的タンパク質の可能性を広げるプロテインノックダウン 法は期待されており、様々なベンチャー企業が開発に乗り出しているようです。例えば、Crews教授らは現在Arvinas 社を設立し、経口投与で薬効を示すPROTAC化合物の臨床試験を準備しているとのことです。また、Blander博士はC4 Therapeutics社を設立しています。他にもkymera 社や、 日本ではFIMECS, Inc. 社が設立されたりと、これからの動向が大変興味深い分野です。(*5)
さて、タンパク質の量を減らす試みを見てきましたが、ついでタンパク質を増やす方向を見てみたいと思います。
②ケミカルシャペロン(ファーマコロジカルシャペロン)
上述のプロテインノックダウン法では生体内の不要なタンパク質を分解する仕組み、ユビキチンープロテアソーム系を利用していました。この不要な"ゴミ"タンパク質の中には、本来の機能を発揮するための3次元構造をとれず、機能の失われたミスフォールドタンパク質も含まれます。
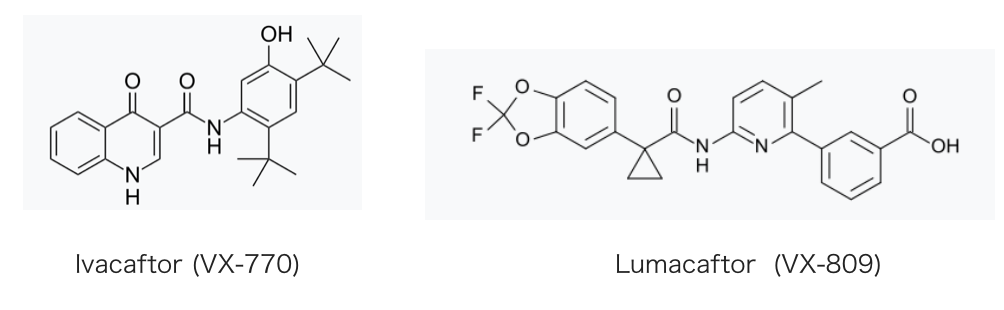
機能しないタンパク質とその治療という話題では、近年、脊髄性筋萎縮症を対象とするSpinrazaや デュシェンヌ型筋ジストロフィーを対象とするExondys 51といった核酸医薬品が注目を集めています。これよりすこし遡りますが2012年、嚢胞性線維症(Cystic Fibrosis)を対象としてKalydeco(一般名; ivacaftor、Vertex社)という低分子医薬品が、ついで2015年Orkambi(ivacaftorとlumacaftorの合剤)がFDAにより承認されました。ivacaftor、lumacafrorは機能するタンパク質を増やすことにより治療効果を発揮する医薬品です。
嚢胞性線維症?
嚢胞性線維症(Cystic fibrosis: 以下CF)は、常染色体劣性遺伝疾患で、日本では非常に稀な疾患ですが、米国では約3万人のCF患者がいるとされています。原因は、塩素イオン・水の輸送を調節する塩素イオンチャネルをコードするCFTR(cystic fibrosis transmembrane conductance regulator)遺伝子の変異で、このイオンチャネルがうまく機能しないことで全身の外分泌機能に異常をきたします。平均生存期間が約20年ともいわれることから、深刻な疾患であることがわかると思います。(*6)
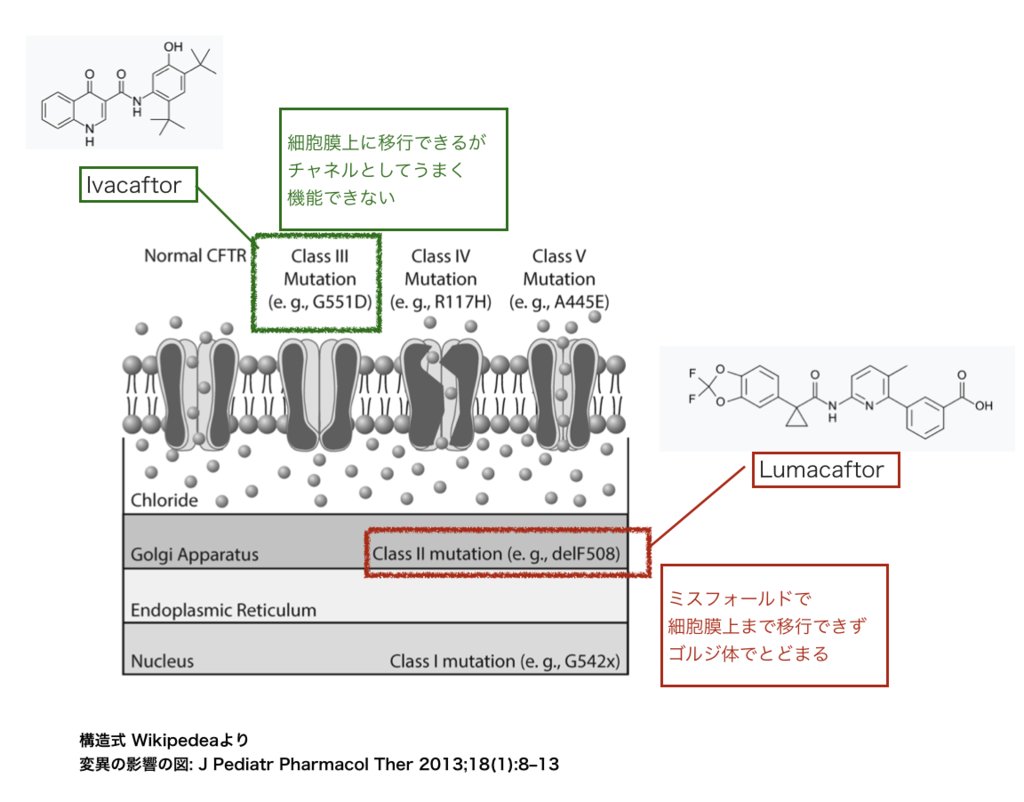
CFにおける変異の分類とタンパク質の機能
CFと一口に言っても様々な遺伝子の変異の種類があり、各変異遺伝子の産物であるイオンチャネルの機能の欠損の度合いには差があります。文献 (*7)を頼りにその分類を見て見ましょう。
CFTRの変異は大きく2つ、
分類①: そもそもイオンチャネルが本来機能する場所である細胞膜上に達することができない(Class I、Class II)
分類②: 細胞膜上には移行するもののイオンチャネルとしての開閉機能に問題がある(Class III、Class IV、Class V)
とに分けることができます。
より詳細には、Class I の変異では核で産生されるタンパク質が短く欠失、Class II はミスフォールドによりゴルジ体にとどまる。Class III、Class IVは全長のCFTRが細胞膜上に発現できるものの、イオンチャネルのゲート開閉機能がうまく働かない(Class III)、イオン透過能(conductivity)が低い(Class IV)といった問題があります。また、Class Vは正常のCFTRですが数が少なくなるという変異です。
各変異をレスキューするための作用機序(MOA: Mode of Action )
では、各変異に対しどのような治療戦略が考えられるでしょうか?
変異の分類①の時は、まずは細胞膜上のCFTRを増やすことが治療の第一歩となります。つまりCFTRの合成を改善(対Class I)、細胞膜への輸送異常を改善(対Class II)となります。一方、分類②では、細胞膜上でCFTRがイオンチャンネルとしてうまく機能できるように手助けしてやる(対Class III)ことがあげられます。
上述の薬剤では、LumacaftorはClass II の変異(delF508)に対して膜移行を助けるケミカルシャペロンとして、Ivacaftor はClass III の変異(G551)に対してイオン透過能を上昇させるポテンシエーターとして働くとされています。
ケミカルシャペロンって?
前置き無しにケミカルシャペロンという言葉を導入しましたが、そもそもシャペロンとは何でしょうか?
シャペロン(英: chaperone)とは、他のタンパク質分子が正しい折りたたみ(フォールディング)をして機能を獲得するのを助けるタンパク質の総称である。分子シャペロン(英: molecular chaperone)、タンパク質シャペロンともいう。 (シャペロン - Wikipedia)(*8)
ケミカルシャペロン(ファーマコロジカルシャペロン)は低分子化合物でありながら、タンパク質のミスフォールドを修正し、適切な高次構造形成の補助・安定化に働くもののことを言います。
Lumacaftorのシャペロン効果の検証
ケミカルシャペロン、コンセプトとしてはわかりますが、その効果はどのように検証すれば良いのでしょうか?2011年Vertex社の論文 (*9)を参照して見ましょう。
まず、ミスフォールドする変異CFTRは、翻訳後修飾(グリコシル化など)がうまく行われず、修飾が正しく行われた正常なCFTR(mature : 成熟)と糖鎖修飾のパターンが異なります(immature : 未成熟)。糖鎖のパターンは細胞内でのタンパク質輸送を左右するため、これが、未成熟な変異CFTRが小胞体-ゴルジ体から細胞膜上へと移行できない理由となります (*10)。
下図は、上記文献Fig. 3Aの引用ですが、左上の図から正常のCFTRと変異CFTRでゲル上の位置が変わってくることがわかります。さらに図中左下から、Lumacaftor処理により濃度依存的に成熟型CFTRの比率が増えている様がわかります。
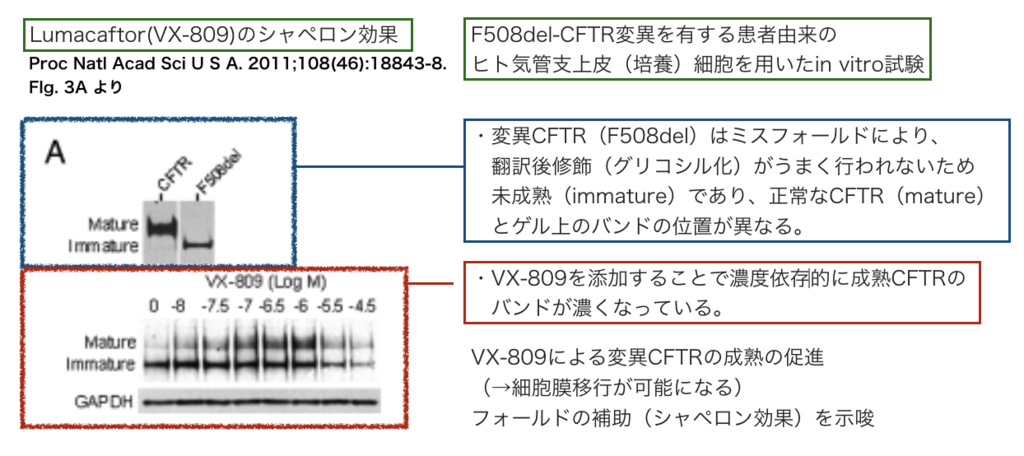
文献中にはCFTRの成熟に関する他の実験、および細胞膜上でのイオンチャネル機能に対する効果の検証もおこなわれており、複数の実験の組合せから、Lumacaftorが変異CFTRのフォールディング補助と、それに続く翻訳語修飾(プロセッシング)を促進する効果が検証されています。オープンアクセスとなっていますのでご興味があるかたは是非元の文献を参照していただければと思います。 私の読解は間違っている可能性が高いので・・・ (*11)
Ivacaftor、Lumacaftorという画期的な医薬品によりCFは完全に治療できるようになったか?というと残念ながらそうではありません。見てきたようにCFには様々な変異の種類があり、これら医薬品で治療可能な範囲には限界があります。Vertex社は継続して様々な変異への適応拡大を目指した臨床試験・新規医薬品の研究開発を進めているようなので、治療環境が改善していくと期待されます。
ケミカルシャペロンの利点?
上記のように遺伝子変異に由来する疾患に非常に有用なことが示唆されるケミカルシャペロンというMOAですが、このような疾患に対する治療戦略としては他にも核酸医薬、ウイルスべクターによる遺伝子治療や酵素補充療法など、様々なアプローチがありうると思います。これらと比較した場合の利点は何でしょうか?よく言われるように低分子はコストが安い、ということでしょうか?
この点については明確にそうだとは私は言い切れません。確かに製造コストは安くなるかもしれませんが、希少疾患である以上、莫大な研究開発費を補うため、一人当たりのCFの治療費は非常に高額なものとなっています。
では低分子ならではのメリットとはなんでしょうか?
長所①:
低分子の他のアプローチに対する利点はまずは薬物動態上のメリットです。核酸医薬では血中の安定性が悪かったり、特定の組織に集積しやすく全身暴露がかかりづらい、といった問題や、酵素補充療法では血液脳関門(BBB)を透過できないため中枢の症状には効果がでない、といった問題があります。低分子では全身暴露・中枢移行性ともに解決可能な可能性が高く、もちろん経口投与も大きなメリットです。
長所②:
また、酵素補充療法(リコンビナントタンパク質補充療法)では、抗タンパク質抗体の出現により、効果がなくなったり、過敏反応、アナフィラキシーショック、半減期の短縮といった問題が生じる可能性があります。この点でもおそらく低分子が優位でしょう。(*12)
もったいぶったわりに普通の結論になってしまった!!
他の事例と今後の期待
これまでVertex社のアプローチをみてきましたが、ここしかないの?と言われるとそんなことはありません。
「小さな命が呼ぶとき」というアメリカ映画のモデルともなったAmicus Therapeuticsという希少疾患をメインターゲットととする製薬会社があります。Amicusの開発したファブリ病(α-ガラクトシダーゼの変異による遺伝性疾患)治療薬、Migalastat(Wikipedia) もその作用機序はケミカルシャペロンで、日本ではガラフォルド®️として今年(2018年3月)製造販売承認が取得されています(*13)。
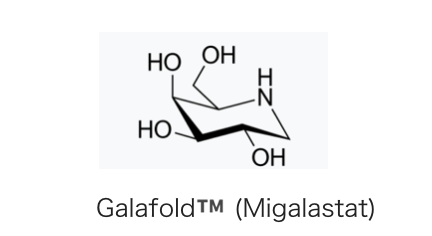
Vertex、Amicusともに遺伝性疾患を対象として、変異により機能障害のおきたタンパク質を低分子により機能回復させています。何故、上述のケミカルシャペロンや、ポテンシエーター(アロステリックモジュレーターに近い?)といった作用機序が魅力があるか? というと、ゲノム解析の進展との関係で興味深いと思うからです。遺伝子レベルの変異の同定と、タンパク質構造・機能の違いの解析が加速した際に、変異し機能が損なわれたタンパク質の機能回復、という選択肢はまだまだポテンシャルがあるのではないかなあ、と思います。・・・こいつ何にもわかってないなと聞き流してください
まとめ
以上、本記事では"タンパク質の量を低分子でコントロールするMOA"という内容で、分解させるプロテインノックダウン法と、増加させるケミカルシャペロンというアプローチについてざっと見てきました。あまり薬理学の教科書でメジャーに扱われる作用機序では無いと思うので、そんなのもあるのかーと思っていただけたら幸いです。
(私が知らないだけで薬学会では一般常識かも・・・)
私自身は専門家では無いため、記事のなかに誤りや不適切な表現が多数あると思います。ご指摘いただければ幸いです。
以上。お付き合いいただきありがとうございました。
~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~
*1
低分子化合物に限っています。DNAと相互作用するシスプラチンのようなものもありますが、話を簡単にするために省いています。
*2
Fig. 1 は以下を引用し、改変して作成
2-1) Wikipedia (https://ja.wikipedia.org/wiki/プロテアソーム )
By Rogerdodd, CC 表示-継承 3.0, https://commons.wikimedia.org/w/index.php?curid=1291009
2-2)
脱ユビキチン化酵素Ubp6はプロテアソームの分子集合を制御する(http://first.lifesciencedb.jp/archives/3121)
佐伯 泰・田中啓二 (東京都医学総合研究所 蛋白質代謝研究室)
ライフサイエンス新着論文レビュー 2011年7月11日 DOI: 107875/first.author.2011.105
http://first.lifesciencedb.jp/archives/3121
© 2011 佐伯 泰・田中啓二 Licensed under CC 表示 2.1 日本
*3
Fig. 3作成にあたって、以下を改変・引用
Proc Natl Acad Sci U S A. 2016 Jun 28; 113(26): 7124–7129.
*4
アミロイド仮説は臨床試験がことごとくうまくいっていないためそれ自体を疑う意見もあるようですが、わかりやすいのであくまで例としてあげました。
*5
その他、プロテインノックダウンに関係して参考にさせていただいたもの
5-1) SNIPERの解説・およびプロテインノックダウン法の日本語によるレビュー
https://www.jstage.jst.go.jp/article/yakushi/138/9/138_18-00113/_pdf/-char/ja
5-2) とてもわかりやすいブログ記事:
タンパク質分解誘導薬とは?これまでの薬と違う新しいタイプの薬 | Luck Is What Happens When Preparation Meets Opportunity
*6
嚢胞性線維症の概要に関しては以下を参考にしました。
https://www.shouman.jp/disease/details/03_06_008/
http://www.nanbyou.or.jp/entry/4532
6-2)
*7
7-1) Ivacaftor: A Novel Gene-Based Therapeutic Approach for Cystic Fibrosis
Condren ME, Bradshaw SM. J. Pediatr. Pharmacol. Ther. 2013;18(1): 8-13
上記文献はFig. 5作成にあたっても改変し引用
Sloane PA, Rowe SM.Curr Opin Pulm Med. 2010;16(6):591-7.
*8
シャペロンの代表的な例としては大腸菌のGroELがよく取り上げられています。
GroELの機能などのわかりやすい解説(日本語)
Journal of Japanese Biochemical Society 87(2): 194-204 (2015)
*9
Correction of the F508del-CFTR protein processing defect in vitro by the investigational drug VX-809
Proc Natl Acad Sci U S A. 2011;108(46):18843-8.
上記文献はFig. 6作成にあたっても改変し引用
*10
糖鎖修飾に関しては以下のページの説明がわかりやすかったです。
梶原研究室HP 研究の背景
*11
なお上記文献およびVertex社の他の文献中で、Lumacaftorはシャペロンとは呼ばれておらず、タンパク質のプロセシングを正常化する"corrector"と呼ばれていました。あえてなのか意図はわかりませんが、Fig. 6からもわかるように、ミスフォールドしたタンパク質を正常な高次構造に戻す様を直接明確に示す実験は困難で、糖鎖修飾やイオンの流出入量の調節といった間接的な機能改善の実験的証拠のみがあげられています。したがってケミカルシャペロンだ、と言い切るのは誤りかもしれませんが、簡単のためケミカルシャペロンの例として今回取り上げさせていただきました。
*12
もちろん低分子でも代謝産物がハプテン化し、過剰な免疫応答を起こすといったリスクはあります。
*13
トランプ大統領が映画のモデルとなったAmicusのCEOと面会したのが承認申請の強い後押しになった、なんて話も・・・