RDKitのファーマコフォアの定義を眺める話
前回の記事で複数の共結晶構造に共通する相互作用をPLIPを使って探してみました。この情報をうまく使えばファーマコフォアが作れるはず・・・
ですがそもそもどうやってファーマコフォアを作っていけばわかりません。
ファーマコフォア?
日本薬学会 薬学用語解説の説明によると以下の通り
「ファーマコフォアとは、医薬品のターゲットとの相互作用に必要な特徴を持つ官能基群と、それらの相対的な立体配置も含めた(抽象的な)概念である・・・(略)・・・同じターゲットの同じ部位に結合する医薬品は活性発現に必須である共通の立体的・電子的特徴を持つ官能基群を持っており、これをファーマコフォアという。」
タンパク質の重要そうな残基と相互作用している、リガンド側の構造の(立体的・電子的)特徴を取り出し、
- 官能基の種類
- 相対的立体配置
をもとに抽象化(モデル化)すれば良いということみたいです。
なるほど!!!
しかし概念はわかりましたがどんな特徴を取り出せばよいかわからない・・・
RDKitを使った場合どのようになるのか調べてみました。参考にしたページは記事末尾に記載しています。*1 *2 *3 *4 *5 *6
RDKitにおけるファーマコフォアの特徴
RDKitでファーマコフォアを構成する特徴(特性基、フィーチャー)をみつけるには、
- 特性基の定義をあつめたフィーチャーファクトリーを作成
- それを用いて実際の分子から該当する特性基を探索
という手順をとるようです。どのような構造を特性基として認識するのでしょうか?
RDKitにおける特性基(化学的特徴/ Chemical Featrues)はSMARTSに基づいた言語で定義づけられています。Feature Definition(FDef)ファイルフォーマットという形式のファイルに、特性基の定義づけに必要な情報が記載されています。
基本となる「BaseFeatures.fdef」というファイルが用意されているのでこちらを利用したいと思います。
フィーチャーファクトリーの作成
ファーマコフォアの認識にFDefファイルの情報を利用するには、こちらを読み込んでフィーチャーファクトリーを作成すれば良いということです。
import os from rdkit import RDConfig from rdkit.Chem import AllChem from rdkit import Chem fdef = AllChem.BuildFeatureFactory(os.path.join(RDConfig.RDDataDir,'BaseFeatures.fdef')) print(fdef.GetNumFeatureDefs()) # 27
用意されているフィーチャーの定義は27個あるようです。
フィーチャーファミリー
フィーチャーをより一般化した分類としてフィーチャーファミリーが用意されており、ファーマコフォマ検索はファミリーを用いて行われます。
print(len(fdef.GetFeatureFamilies())) print(fdef.GetFeatureFamilies()) # 8 # ('Donor', 'Acceptor', 'NegIonizable', 'PosIonizable', 'ZnBinder', 'Aromatic', 'Hydrophobe', 'LumpedHydrophobe')
フィーチャーファミリーは全部で8個あり、水素結合ドナー(Donor)やアクセプター(Acceptor)といった名前から構造が推測できるものから、LumpedHydrophobeといった具体的な例がよくわからない(私だけ?)ものまであります。
フィーチャータイプ
より詳細な記述がされたフィーチャータイプを確認したいと思います。
fdef.GetFeatureDefs()) とすると、各フィーチャーの名前をKey、SMARTSによる定義をvalueとする辞書型で情報を書き出すことができます。
とりあえず名前だけ確認します。
print(fdef.GetFeatureDefs().keys()) # dict_keys(['Donor.SingleAtomDonor', 'Acceptor.SingleAtomAcceptor', 'NegIonizable.AcidicGroup', 'PosIonizable.BasicGroup', 'PosIonizable.PosN', 'PosIonizable.Imidazole', 'PosIonizable.Guanidine', 'ZnBinder.ZnBinder1', 'ZnBinder.ZnBinder2', 'ZnBinder.ZnBinder3', 'ZnBinder.ZnBinder4', 'ZnBinder.ZnBinder5', 'ZnBinder.ZnBinder6', 'Aromatic.Arom4', 'Aromatic.Arom5', 'Aromatic.Arom6', 'Aromatic.Arom7', 'Aromatic.Arom8', 'Hydrophobe.ThreeWayAttach', 'Hydrophobe.ChainTwoWayAttach', 'LumpedHydrophobe.Nitro2', 'LumpedHydrophobe.RH6_6', 'LumpedHydrophobe.RH5_5', 'LumpedHydrophobe.RH4_4', 'LumpedHydrophobe.RH3_3', 'LumpedHydrophobe.tButyl', 'LumpedHydrophobe.iPropyl'])
各フィーチャーは「ファミリー名.xxxx」という形式のようなのでファミリー名を紐づけてDataFrame化してみます。
import pandas as pd family_df = pd.DataFrame(columns=['family', 'definition']) family_names = fdef.GetFeatureFamilies() for k,v in fdef.GetFeatureDefs().items(): for fam in family_names: if fam in k: family_df.loc[k] = [fam, v] family_df.head()

SMARTSの定義の文字列がおかしな感じになっています。$などが文字としてそのまま出力されていないみたいです。
各ファミリーに含まれるフィーチャーの数を確認します。
family_df['family'].value_counts() # LumpedHydrophobe 7 # ZnBinder 6 # Aromatic 5 # PosIonizable 4 # Hydrophobe 2 # NegIonizable 1 # Acceptor 1 # Donor 1
名前だけではわかりづらい「LumpedHydrophobe」や「ZnBinder」の種類が多くなっています。まとまった定義づけが難しいのでしょうか?
SMARTSの解読に挑戦
以前の記事でSMARTS記法について、ちょっと勉強しました。折角なのでSMARTSによる定義を理解できるか、書き下してみたいと思います。
書き下した後でSMARTSviewerという便利すぎるサイトを見つけてしまったので、素人の適当解釈は不要!という方はすっ飛ばして下までスクロールしてください。
SMARTSの説明補足
SMARTSを読み始める前に、前回の記事の補足を少々・・・
- Recursive SMARTS
フィーチャーの定義ではRecursive SMARTSの形式が多く用いられていました。Daylight社のSMARTS記法の説明ページによると、「$(SMARTS)」という表記で注目している原子(atom)の置かれている環境を示す、とのことです。
- 「R<数字>」と「r<数字>」の違い
以前の記事では、無理やりSMARTSの説明を日本語化しようとした結果、逆にわかりづらくなっていたので、Rとrについて英語の説明をそのまま貼っておきます。
| Symbol | Symbol name | Atomic property requirements | Default |
|---|---|---|---|
| R |
ring membership | in |
any ring atom |
| r |
ring size | in smallest SSSR ring of size |
any ring atom |
「幾つのSSSR環に含まれているか?」と、「どんなサイズの環に含まれているか?」ということになりそうです。
ドナー
| family | feature | 定義 SMARTS | 説明 | |
|---|---|---|---|---|
| Donor | Donor.SingleAtomDonor | [$([N&!H0&v3, | Hの数が0ではない、結合次数の総数3の脂肪族N | または |
| N&!H0&+1&v4, | Hの数が0ではない、電荷+1で結合次数の総数4の脂肪族N | または | ||
| n&H1&+0, | Hの数が1で、電荷0の芳香族N | または | ||
| $([$([Nv3](-C)(-C)-C)]), | 脂肪族炭素が3つ結合した、結合次数の総数3の脂肪族N | または | ||
| $([$(n[n;H1]), | 芳香族Nと、Hの数が1の芳香族Nが隣接する並び | または | ||
| $(nc[n;H1])])]), | 芳香族N、芳香族Cと、Hの数が1の芳香族Nが隣接する並び | あるいは | ||
| $([O,S;H1;+0])] | Hの数が1で、電荷0の脂肪族OまたはS |
アクセプター
| family | feature | 定義 SMARTS | 説明 | |
|---|---|---|---|---|
| Acceptor | Acceptor.SingleAtomAcceptor | [$([O;H1;v2]), | Hの数が1で、結合次数の総数が2の脂肪族O | あるいは |
| $([O;H0;v2;!$(O=N-*), | Hの数が0で、結合次数の総数が2の脂肪族Oのうち、 O=N-に連結するものではないもの |
または | ||
| $([O;-;!$(*-N=O)]), | 負電荷(-1)を帯びた脂肪族Oのうち、 O=N-に連結するものではないもの |
または | ||
| $([o;+0])]), | 電荷0の芳香族O | あるいは | ||
| $([n;+0;!X3;!$([n;H1](cc)cc), | 電荷0の芳香族Nで、結合の総数3ではなく、また、 芳香族炭素Cが二つ直列した結合2本とHを1つもつ芳香族Nでもないもの |
または | ||
| $([$[N;H0]#[C&v4])]), | Hの数が0の脂肪族Nと、結合次数の総数4の脂肪族Cが、 3重結合で結合したもの(シアノ基) |
または | ||
| $([N&v3;H0;$(Nc)])]), | 結合次数の総数3で結合するHの数が0の脂肪族Nと、かつ、 芳香族Cに結合する脂肪族N |
あるいは | ||
| $([F;$(F-[#6]);!$(FC[F,Cl,Br,I])])] | 炭素と結合するFで、 ハロゲン原子の結合した脂肪族炭素に結合するFではないもの |
イオン化可能なグループ
| family | feature | 定義 SMARTS | 説明 | |
|---|---|---|---|---|
| NegIonizable | NegIonizable.AcidicGroup | [C,S](=[O,S,P])-[O;H1,H0&-1] | 「O、SまたはP」と二重結合する「CまたはS」に結合した、 OH、あるいはO-(H、0個で負電荷) |
|
| PosIonizable | PosIonizable.BasicGroup | [$([$([N;H2&+0][$([C;!$(C=*)])])]), | 他の脂肪族Cと二重結合しているのではない脂肪族C、 およびH2つと結合する電荷0の脂肪族N |
または |
| $([$([N;H1&+0]([$([C;!$(C=*)])])[$([C;!$(C=*)])])]), | 他の脂肪族Cと二重結合しているのではない脂肪族C2つ、 およびH1つと結合する、電荷0の脂肪族N |
または | ||
| $([$([N;H0&+0]([$([C;!$(C=*)])])([$([C;!$(C=*)])])[$([C;!$(C=*)])])]) | 他の脂肪族Cと二重結合しているのではない脂肪族C3つと 結合する、電荷0の脂肪族N |
かつ | ||
| ;!$(N[a])] | 上記3つのNは芳香族原子と結合していないこと | |||
| PosIonizable.PosN | [#7;+;!$([N+]-[O-])] | 「O-」に結合する「N+」ではない、電荷+1のN | ||
| PosIonizable.Imidazole | c1ncnc1 | イミダゾール骨格 | ||
| PosIonizable.Guanidine | NC(=N)N | グアニジン骨格 |
Zinc Binder
| family | feature | 定義 SMARTS | 説明 |
|---|---|---|---|
| ZnBinder | ZnBinder.ZnBinder1 | [S;D1]-[#6] | Cと結合する1級S |
| ZnBinder.ZnBinder2 | [#6]-C(=O)-C-[S;D1] | C-カルボニル-Cに結合する1級S | |
| ZnBinder.ZnBinder3 | [#6]-C(=O)-C-C-[S;D1] | C-カルボニル-C-Cに結合する1級S | |
| ZnBinder.ZnBinder4 | [#6]-C(=O)-N-[O;D1] | C-カルボニル-Nに結合する1級O | |
| ZnBinder.ZnBinder5 | [#6]-C(=O)-[O;D1] | C-カルボニルに結合する1級O | |
| ZnBinder.ZnBinder6 | [#6]-P(=O)(-O)-[C,O,N]-[C,H] | C-PO2に結合する「C or O or N」に「C or H」が結合 |
芳香族
| family | feature | 定義 SMARTS | 説明(原子環境) | 結合の種類 |
|---|---|---|---|---|
| Aromatic | Aromatic.Arom4 | [$([a;r4,!R1&r3])]1: | 4員環(SSSR size4)の芳香族性原子、または 3員環(size3)で、含まれるSSSR ringが1つではないもの |
芳香属性結合(:) |
| [$([a;r4,!R1&r3])]: | 同上 | 同上 | ||
| [$([a;r4,!R1&r3])]: | 同上 | 同上 | ||
| [$([a;r4,!R1&r3])]:1 | 同上 | 同上 | ||
| 1 | 4員環を閉じる | |||
| Aromatic.Arom5 | [$([a;r5,!R1&r4,!R1&r3])]1: | 5員環(SSSR size5)の芳香族性原子、または 4員環(size4)で、含まれるSSSR ringが1つではないもの、または 3員環(size3)で、含まれるSSSR ringが1つではないもの |
芳香属性結合(:) | |
| [$([a;r5,!R1&r4,!R1&r3])]: | 同上 | 同上 | ||
| [$([a;r5,!R1&r4,!R1&r3])]: | 同上 | 同上 | ||
| [$([a;r5,!R1&r4,!R1&r3])]: | 同上 | 同上 | ||
| [$([a;r5,!R1&r4,!R1&r3])]: | 同上 | 同上 | ||
| 1 | 5員環を閉じる | |||
| Aromatic.Arom6 | [$([a;r6,!R1&r5,!R1&r4,!R1&r3])]1: | 6員環(SSSR size6)の芳香族性原子、または 5員環(size5)で、含まれるSSSR ringが1つではないもの、または 4員環(size4)で、含まれるSSSR ringが1つではないもの、または 3員環(size3)で、含まれるSSSR ringが1つではないもの |
芳香属性結合(:) | |
| [$([a;r6,!R1&r5,!R1&r4,!R1&r3])]: | 同上 | 同上 | ||
| [$([a;r6,!R1&r5,!R1&r4,!R1&r3])]: | 同上 | 同上 | ||
| [$([a;r6,!R1&r5,!R1&r4,!R1&r3])]: | 同上 | 同上 | ||
| [$([a;r6,!R1&r5,!R1&r4,!R1&r3])]: | 同上 | 同上 | ||
| [$([a;r6,!R1&r5,!R1&r4,!R1&r3])]: | 同上 | 同上 | ||
| 1 | 6員環を閉じる | |||
| Aromatic.Arom7 | [$([a;r7,!R1&r6,!R1&r5,!R1&r4,!R1&r3])]1: | 7員環(SSSR size7)の芳香族性原子、または 6員環(size6)で、含まれるSSSR ringが1つではないもの、または 5員環(size5)で、含まれるSSSR ringが1つではないもの、または 4員環(size4)で、含まれるSSSR ringが1つではないもの、または 3員環(size3)で、含まれるSSSR ringが1つではないもの |
芳香属性結合(:) | |
| [$([a;r7,!R1&r6,!R1&r5,!R1&r4,!R1&r3])]: | 同上 | 同上 | ||
| [$([a;r7,!R1&r6,!R1&r5,!R1&r4,!R1&r3])]: | 同上 | 同上 | ||
| [$([a;r7,!R1&r6,!R1&r5,!R1&r4,!R1&r3])]: | 同上 | 同上 | ||
| [$([a;r7,!R1&r6,!R1&r5,!R1&r4,!R1&r3])]: | 同上 | 同上 | ||
| [$([a;r7,!R1&r6,!R1&r5,!R1&r4,!R1&r3])]: | 同上 | 同上 | ||
| [$([a;r7,!R1&r6,!R1&r5,!R1&r4,!R1&r3])]: | 同上 | 同上 | ||
| 1 | 7員環を閉じる | |||
| Aromatic.Arom8 | [$([a;r8,!R1&r7,!R1&r6,!R1&r5,!R1&r4,!R1&r3])]1: | 8員環(SSSR size8)の芳香族性原子、または 7員環(size7)で、含まれるSSSR ringが1つではないもの、または 6員環(size6)で、含まれるSSSR ringが1つではないもの、または 5員環(size5)で、含まれるSSSR ringが1つではないもの、または 4員環(size4)で、含まれるSSSR ringが1つではないもの、または 3員環(size3)で、含まれるSSSR ringが1つではないもの |
芳香属性結合(:) | |
| [$([a;r8,!R1&r7,!R1&r6,!R1&r5,!R1&r4,!R1&r3])]: | 同上 | 同上 | ||
| [$([a;r8,!R1&r7,!R1&r6,!R1&r5,!R1&r4,!R1&r3])]: | 同上 | 同上 | ||
| [$([a;r8,!R1&r7,!R1&r6,!R1&r5,!R1&r4,!R1&r3])]: | 同上 | 同上 | ||
| [$([a;r8,!R1&r7,!R1&r6,!R1&r5,!R1&r4,!R1&r3])]: | 同上 | 同上 | ||
| [$([a;r8,!R1&r7,!R1&r6,!R1&r5,!R1&r4,!R1&r3])]: | 同上 | 同上 | ||
| [$([a;r8,!R1&r7,!R1&r6,!R1&r5,!R1&r4,!R1&r3])]: | 同上 | 同上 | ||
| [$([a;r8,!R1&r7,!R1&r6,!R1&r5,!R1&r4,!R1&r3])]: | 同上 | 同上 | ||
| 1 | 8員環を閉じる |
疎水性基
| family | feature | 定義 SMARTS | 説明 |
|---|---|---|---|
| Hydrophobe | Hydrophobe.ThreeWayAttach | [D3,D4; $([#6;+0;!$([#6;$([#6]~[#7,#8,#9])])])] |
3級、または4級の原子で、かつ 電荷0のCのうち 「N,O,F」のいずれかと結合したCではないもの |
| Hydrophobe.ChainTwoWayAttach | [R0;D2;$([#6;+0; !$([#6;$([#6]~[#7,#8,#9])])])] |
環に含まれていない、2級の原子で 電荷0のCのうち 「N,O,F」のいずれかと結合したCではないもの |
疎水性基の塊?
| family | feature | 定義 SMARTS | 説明 | 結合の種類 | |
|---|---|---|---|---|---|
| LumpedHydrophobe | LumpedHydrophobe.Nitro2 | [N;D3;+](=O)[O-] | 3級で電荷+1のNであり、Oと二重結合、電荷-1のOと単結合するもの | (ニトロ基) | |
| LumpedHydrophobe.RH6_6 | [$([r6,!R1&r5,!R1&r4,!R1&r3]); | 6員環(SSSR size6)に含まれる、または 5員環(size5)で、含まれるSSSR ringが1つではないもの、または 4員環(size4)で、含まれるSSSR ringが1つではないもの、または 3員環(size3)で、含まれるSSSR ringが1つではないもの |
かつ | ||
| $([R; $([c,s,S&H0&v2,Br,I, $([#6;+0;!$([#6;$([#6]~[#7,#8,#9])])])])])]1 |
環に含まれる原子で、かつ 「芳香属性C、芳香属性S、結合次数の総数2でHの数0の脂肪族S、Br、I、または 電荷0のCのうち「N,O,F」のいずれかと結合したCではないもの」 |
単結合or芳香属性結合 | |||
| [$([r6,!R1&r5,!R1&r4,!R1&r3]); $([R;$([c,s,S&H0&v2,Br,I, $([#6;+0;!$([#6;$([#6]~[#7,#8,#9])])])])])] |
同上 | 同上 | |||
| [$([r6,!R1&r5,!R1&r4,!R1&r3]); $([R;$([c,s,S&H0&v2,Br,I, $([#6;+0;!$([#6;$([#6]~[#7,#8,#9])])])])])] |
同上 | 同上 | |||
| [$([r6,!R1&r5,!R1&r4,!R1&r3]); $([R;$([c,s,S&H0&v2,Br,I, $([#6;+0;!$([#6;$([#6]~[#7,#8,#9])])])])])] |
同上 | 同上 | |||
| [$([r6,!R1&r5,!R1&r4,!R1&r3]); $([R;$([c,s,S&H0&v2,Br,I, $([#6;+0;!$([#6;$([#6]~[#7,#8,#9])])])])])] |
同上 | 同上 | |||
| [$([r6,!R1&r5,!R1&r4,!R1&r3]); $([R;$([c,s,S&H0&v2,Br,I, $([#6;+0;!$([#6;$([#6]~[#7,#8,#9])])])])])]1 |
同上 | 6員環を閉じる | |||
| LumpedHydrophobe.RH5_5 | [$([r5,!R1&r4,!R1&r3]); | 5員環(SSSR size5)に含まれる、または 4員環(size4)で、含まれるSSSR ringが1つではないもの、または 3員環(size3)で、含まれるSSSR ringが1つではないもの |
かつ | ||
| $([R; $([c,s,S&H0&v2,Br,I, $([#6;+0;!$([#6;$([#6]~[#7,#8,#9])])])])])]1 |
環に含まれる原子で、かつ 「芳香属性C、芳香属性S、結合次数の総数2でHの数0の脂肪族S、Br、I、または 電荷0のCのうち「N,O,F」のいずれかと結合したCではないもの」 |
単結合 or 芳香属性結合 | |||
| [$([r5,!R1&r4,!R1&r3]); $([R;$([c,s,S&H0&v2,Br,I, $([#6;+0;!$([#6;$([#6]~[#7,#8,#9])])])])])] |
同上 | 同上 | |||
| [$([r5,!R1&r4,!R1&r3]); $([R;$([c,s,S&H0&v2,Br,I, $([#6;+0;!$([#6;$([#6]~[#7,#8,#9])])])])])] |
同上 | 同上 | |||
| [$([r5,!R1&r4,!R1&r3]); $([R;$([c,s,S&H0&v2,Br,I, $([#6;+0;!$([#6;$([#6]~[#7,#8,#9])])])])])] |
同上 | 同上 | |||
| [$([r5,!R1&r4,!R1&r3]); $([R;$([c,s,S&H0&v2,Br,I, $([#6;+0;!$([#6;$([#6]~[#7,#8,#9])])])])])] |
同上 | 5員環を閉じる | |||
| LumpedHydrophobe.RH4_4 | [$([r4,!R1&r3]); | 4員環(SSSR size4)に含まれる、または 3員環(size3)で、含まれるSSSR ringが1つではないもの |
かつ | ||
| $([R; $([c,s,S&H0&v2,Br,I, $([#6;+0;!$([#6;$([#6]~[#7,#8,#9])])])])])]1 |
環に含まれる原子で、かつ 「芳香属性C、芳香属性S、結合次数の総数2でHの数0の脂肪族S、Br、I、または 電荷0のCのうち「N,O,F」のいずれかと結合したCではないもの」 |
単結合 or 芳香属性結合 | |||
| [$([r4,!R1&r3]);$([R; $([c,s,S&H0&v2,Br,I, $([#6;+0;!$([#6;$([#6]~[#7,#8,#9])])])])])] |
同上 | 同上 | |||
| [$([r4,!R1&r3]);$([R; $([c,s,S&H0&v2,Br,I, $([#6;+0;!$([#6;$([#6]~[#7,#8,#9])])])])])] |
同上 | 同上 | |||
| [$([r4,!R1&r3]);$([R; $([c,s,S&H0&v2,Br,I, $([#6;+0;!$([#6;$([#6]~[#7,#8,#9])])])])])]1 |
同上 | 4員環を閉じる | |||
| LumpedHydrophobe.RH3_3 | [$([r3]); | 3員環(SSSR size3)に含まれるもの | かつ | ||
| $([R; $([c,s,S&H0&v2,Br,I, $([#6;+0;!$([#6;$([#6]~[#7,#8,#9])])])])])]1 |
環に含まれる原子で、かつ 「芳香属性C、芳香属性S、結合次数の総数2でHの数0の脂肪族S、Br、I、または 電荷0のCのうち「N,O,F」のいずれかと結合したCではないもの」 |
単結合or芳香属性結合 | |||
| [$([r3]);$([R;$([c,s,S&H0&v2,Br,I, $([#6;+0;!$([#6;$([#6]~[#7,#8,#9])])])])])] |
同上 | 同上 | |||
| [$([r3]);$([R;$([c,s,S&H0&v2,Br,I, $([#6;+0;!$([#6;$([#6]~[#7,#8,#9])])])])])]1 |
同上 | 3員環を閉じる | |||
| LumpedHydrophobe.tButyl | [C;!R](-[CH3])(-[CH3])-[CH3] | 環に含まれていないCで、メチル基3つが結合 | |||
| LumpedHydrophobe.iPropyl | [CH;!R](-[CH3])-[CH3] | 環に含まれていないCで、メチル基2つとH1つが結合 |
SMARTSviewer
さて、つらつらと意味不明な日本語を垂れ流してしまいましたが、上に述べたようにSMARTSviewerという便利すぎるサイトを見つけてしまいました。
こちらは ハンブルク大学 バイオインフォマティクスセンター(Universität Hamburg ZBH-Center for Bioinformatics)が開発・公開しているソフトウェアで SMARTSをとてもわかりやすく可視化してくれます。
まずは見た目を・・・

使い方はとても簡単で、「Try your own SMARTS pattern」と書かれた部分にSMARTSを貼り付け、「Go!」をクリックするだけです。「Donwload」をクリックすればpng形式で綺麗な図が手に入ります。*7
それではフィチャーの中で気になったZnBinderで試してみます。
フィーチャー名「ZnBinder.ZnBinder6」 SMARTS「[#6]-P(=O)(-O)-[C,O,N]-[C,H]」の場合・・・

線形表記と比べて分岐構造の把握がしやすく、またカラフルなので原子の違いもわかりやすいです。
Optionで描画方法をデフォルト「Coplete Visualization」から「Element Symbols」にすると、より2次元構造式に近い見た目となります。

とてもいい感じです。インストール版もありアカデミックフリー、ノンアカデミックはライセンス費用が必要みたいです。SMARTSviewerの開発者はResearch Group for Computational Molecular Design (AMD、Prof. Matthias Rarey)グループで、AMD software serverには他にもSMARTSeditorなど面白そうなソフトウェアが多数ありました。*8
記事の最後にSMARTsviwerに投げ込みやすい形式でRDKitのフィーチャーを出力しておきます。文字書き下しのわかりづらさと図の見易さの違いを比較していただければと思います。
まとめ
以上、今回はファーマコフォア作成に向けた手始めとしてRDKitにおける特徴の定義を眺めてみました。・・・というよりSMARTSを読んだだけで力尽きました・・・
FDefファイルの中身(rdkit/Data/BaseFeatures.fdef)をテキストエディタで開いてみたり、RDKitのDocumentation Feature Definition File Formatを確認した方がわかりやすかったかもしれません。
SMARTSviewerを見つけたのが最大の収穫!!
色々と間違いがあると思いますのでご指摘いただければ幸いです。
SMARTS出力
テーブルでは分けたり、エスケープ記号を入れたりしてしまっているので、そのままのSMARTS出力も載せておきます。
for k, v in fdef.GetFeatureDefs().items(): print(k,':',v)
Donor.SingleAtomDonor : [$([N&!H0&v3,N&!H0&+1&v4,n&H1&+0,$([$([Nv3](-C)(-C)-C)]),$([$(n[n;H1]),$(nc[n;H1])])]),$([O,S;H1;+0])] Acceptor.SingleAtomAcceptor : [$([O;H1;v2]),$([O;H0;v2;!$(O=N-*),$([O;-;!$(*-N=O)]),$([o;+0])]),$([n;+0;!X3;!$([n;H1](cc)cc),$([$([N;H0]#[C&v4])]),$([N&v3;H0;$(Nc)])]),$([F;$(F-[#6]);!$(FC[F,Cl,Br,I])])] NegIonizable.AcidicGroup : [C,S](=[O,S,P])-[O;H1,H0&-1] PosIonizable.BasicGroup : [$([$([N;H2&+0][$([C;!$(C=*)])])]),$([$([N;H1&+0]([$([C;!$(C=*)])])[$([C;!$(C=*)])])]),$([$([N;H0&+0]([$([C;!$(C=*)])])([$([C;!$(C=*)])])[$([C;!$(C=*)])])]);!$(N[a])] PosIonizable.PosN : [#7;+;!$([N+]-[O-])] PosIonizable.Imidazole : c1ncnc1 PosIonizable.Guanidine : NC(=N)N ZnBinder.ZnBinder1 : [S;D1]-[#6] ZnBinder.ZnBinder2 : [#6]-C(=O)-C-[S;D1] ZnBinder.ZnBinder3 : [#6]-C(=O)-C-C-[S;D1] ZnBinder.ZnBinder4 : [#6]-C(=O)-N-[O;D1] ZnBinder.ZnBinder5 : [#6]-C(=O)-[O;D1] ZnBinder.ZnBinder6 : [#6]-P(=O)(-O)-[C,O,N]-[C,H] Aromatic.Arom4 : [$([a;r4,!R1&r3])]1:[$([a;r4,!R1&r3])]:[$([a;r4,!R1&r3])]:[$([a;r4,!R1&r3])]:1 Aromatic.Arom5 : [$([a;r5,!R1&r4,!R1&r3])]1:[$([a;r5,!R1&r4,!R1&r3])]:[$([a;r5,!R1&r4,!R1&r3])]:[$([a;r5,!R1&r4,!R1&r3])]:[$([a;r5,!R1&r4,!R1&r3])]:1 Aromatic.Arom6 : [$([a;r6,!R1&r5,!R1&r4,!R1&r3])]1:[$([a;r6,!R1&r5,!R1&r4,!R1&r3])]:[$([a;r6,!R1&r5,!R1&r4,!R1&r3])]:[$([a;r6,!R1&r5,!R1&r4,!R1&r3])]:[$([a;r6,!R1&r5,!R1&r4,!R1&r3])]:[$([a;r6,!R1&r5,!R1&r4,!R1&r3])]:1 Aromatic.Arom7 : [$([a;r7,!R1&r6,!R1&r5,!R1&r4,!R1&r3])]1:[$([a;r7,!R1&r6,!R1&r5,!R1&r4,!R1&r3])]:[$([a;r7,!R1&r6,!R1&r5,!R1&r4,!R1&r3])]:[$([a;r7,!R1&r6,!R1&r5,!R1&r4,!R1&r3])]:[$([a;r7,!R1&r6,!R1&r5,!R1&r4,!R1&r3])]:[$([a;r7,!R1&r6,!R1&r5,!R1&r4,!R1&r3])]:[$([a;r7,!R1&r6,!R1&r5,!R1&r4,!R1&r3])]:1 Aromatic.Arom8 : [$([a;r8,!R1&r7,!R1&r6,!R1&r5,!R1&r4,!R1&r3])]1:[$([a;r8,!R1&r7,!R1&r6,!R1&r5,!R1&r4,!R1&r3])]:[$([a;r8,!R1&r7,!R1&r6,!R1&r5,!R1&r4,!R1&r3])]:[$([a;r8,!R1&r7,!R1&r6,!R1&r5,!R1&r4,!R1&r3])]:[$([a;r8,!R1&r7,!R1&r6,!R1&r5,!R1&r4,!R1&r3])]:[$([a;r8,!R1&r7,!R1&r6,!R1&r5,!R1&r4,!R1&r3])]:[$([a;r8,!R1&r7,!R1&r6,!R1&r5,!R1&r4,!R1&r3])]:[$([a;r8,!R1&r7,!R1&r6,!R1&r5,!R1&r4,!R1&r3])]:1 Hydrophobe.ThreeWayAttach : [D3,D4;$([#6;+0;!$([#6;$([#6]~[#7,#8,#9])])])] Hydrophobe.ChainTwoWayAttach : [R0;D2;$([#6;+0;!$([#6;$([#6]~[#7,#8,#9])])])] LumpedHydrophobe.Nitro2 : [N;D3;+](=O)[O-] LumpedHydrophobe.RH6_6 : [$([r6,!R1&r5,!R1&r4,!R1&r3]);$([R;$([c,s,S&H0&v2,Br,I,$([#6;+0;!$([#6;$([#6]~[#7,#8,#9])])])])])]1[$([r6,!R1&r5,!R1&r4,!R1&r3]);$([R;$([c,s,S&H0&v2,Br,I,$([#6;+0;!$([#6;$([#6]~[#7,#8,#9])])])])])][$([r6,!R1&r5,!R1&r4,!R1&r3]);$([R;$([c,s,S&H0&v2,Br,I,$([#6;+0;!$([#6;$([#6]~[#7,#8,#9])])])])])][$([r6,!R1&r5,!R1&r4,!R1&r3]);$([R;$([c,s,S&H0&v2,Br,I,$([#6;+0;!$([#6;$([#6]~[#7,#8,#9])])])])])][$([r6,!R1&r5,!R1&r4,!R1&r3]);$([R;$([c,s,S&H0&v2,Br,I,$([#6;+0;!$([#6;$([#6]~[#7,#8,#9])])])])])][$([r6,!R1&r5,!R1&r4,!R1&r3]);$([R;$([c,s,S&H0&v2,Br,I,$([#6;+0;!$([#6;$([#6]~[#7,#8,#9])])])])])]1 LumpedHydrophobe.RH5_5 : [$([r5,!R1&r4,!R1&r3]);$([R;$([c,s,S&H0&v2,Br,I,$([#6;+0;!$([#6;$([#6]~[#7,#8,#9])])])])])]1[$([r5,!R1&r4,!R1&r3]);$([R;$([c,s,S&H0&v2,Br,I,$([#6;+0;!$([#6;$([#6]~[#7,#8,#9])])])])])][$([r5,!R1&r4,!R1&r3]);$([R;$([c,s,S&H0&v2,Br,I,$([#6;+0;!$([#6;$([#6]~[#7,#8,#9])])])])])][$([r5,!R1&r4,!R1&r3]);$([R;$([c,s,S&H0&v2,Br,I,$([#6;+0;!$([#6;$([#6]~[#7,#8,#9])])])])])][$([r5,!R1&r4,!R1&r3]);$([R;$([c,s,S&H0&v2,Br,I,$([#6;+0;!$([#6;$([#6]~[#7,#8,#9])])])])])]1 LumpedHydrophobe.RH4_4 : [$([r4,!R1&r3]);$([R;$([c,s,S&H0&v2,Br,I,$([#6;+0;!$([#6;$([#6]~[#7,#8,#9])])])])])]1[$([r4,!R1&r3]);$([R;$([c,s,S&H0&v2,Br,I,$([#6;+0;!$([#6;$([#6]~[#7,#8,#9])])])])])][$([r4,!R1&r3]);$([R;$([c,s,S&H0&v2,Br,I,$([#6;+0;!$([#6;$([#6]~[#7,#8,#9])])])])])][$([r4,!R1&r3]);$([R;$([c,s,S&H0&v2,Br,I,$([#6;+0;!$([#6;$([#6]~[#7,#8,#9])])])])])]1 LumpedHydrophobe.RH3_3 : [$([r3]);$([R;$([c,s,S&H0&v2,Br,I,$([#6;+0;!$([#6;$([#6]~[#7,#8,#9])])])])])]1[$([r3]);$([R;$([c,s,S&H0&v2,Br,I,$([#6;+0;!$([#6;$([#6]~[#7,#8,#9])])])])])][$([r3]);$([R;$([c,s,S&H0&v2,Br,I,$([#6;+0;!$([#6;$([#6]~[#7,#8,#9])])])])])]1 LumpedHydrophobe.tButyl : [C;!R](-[CH3])(-[CH3])-[CH3] LumpedHydrophobe.iPropyl : [CH;!R](-[CH3])-[CH3]
*2:RDKit Documentation
Chemical Featrues and Pharmacophore
Feature Definition File Format
*3:RDKit Python API rdkit.Chem.rdMolChemicalFeatures module
*4: RDKitブログ記事Using Feature Maps
*7:* Web版のHelepページに図は自由に利用してOKと書いてありました。
citatition:
- SMARTS viewer https://smartsview.zbh.uni-hamburg.de
- Schomburg, H.-C. Ehrlich, K. Stierand, M.Rarey; From Structure Diagrams to Visual Chemical Patterns, J. Chem. Inf. Model., 2010, 50 (9), pp 1529-1535
*8: Prof. RareyはBioSolveITという会社のファウンダーでもあるそうです。
共結晶構造でPLIFを作ろうとした話
これまで化合物ライブラリーの①指標での絞り込み、②部分構造での絞り込みを行なって来ました。今まではリガンド側のことしか考えていなかったので、そろそろタンパク質との関係を考慮に入れたいと思います。ですが、いきなりドッキング(?)はハードルが高いので、まずは共結晶構造をもとに重要な相互作用を確認することから始めたいと思います。
以前、RCSB PDBのviewerを使ってリガンド-受容体相互作用を眺めて遊びました。こんな図が見られます。(PDB id: 5J89)*1

こういう情報をなんとか格好いい感じで利用したい!!ということで色々と検索していると似たような相互作用解析方法を見つけました。
Protein-Ligand Interaction Profiler(PLIP)というものです。 文献はこちらNucleic Acids Res. 2015(43)W443-447
いい感じで相互作用を解析してくれるみたいなのでPLIPで遊んでみたいと思います。
PLIP?
まずはページの見た目から・・・こんなページです。

試しに上の複合体(PDB id: 5J89)を投げ込んで見ます。

相互作用を形式に分けてリストアップしてくれるみたいです。結果をダウンロードしてPyMolで眺めることもできるそうです。プロファイラー格好良い!
認識される相互作用
Supplementary Informationによるとデフォルトで以下のような非共有結合性の相互作用を認識してくれるそうです。
| 相互作用 | 基準 | 変数 | |
|---|---|---|---|
| 疎水性相互作用 | Hydrophobic interacrtions | 距離 (4.0Å以下) | HYDROPH_DIST_MAX |
| 水素結合 | Hydrogen Bonds | 距離 (4.1Å以下) 角度 (100°以上) |
HBOND_DIST_MAX HBOND_DON_ANGLE_MIN |
| π-πスタッキング | Aromatic Stacking | 距離 (7.5Å以下) 角度 (T-stacking 90°±30°、 P-stacking 180°±30°) 環の中心同士の距離 (2.0Å以下) |
PISTACK_DIST_MAX PISTACK_ANG_DEV PISTACK_OFFSET_MAX |
| π-カチオン相互作用 | Pi-Cation interactions | 距離 (6.0Å以下) (3級アミンは角度も) |
PICATION_DIST_MAX |
| 塩橋 | Salt Bridges | 電荷の中心間の距離 (5.5Å以下) | SALTBRIDGE_DIST_MAX |
| 水を介した水素結合 | Water-brodged hydrogen bonds | 水分子の位置 (2.5Å~4.0Å) 角度2つ (75°<ω<140°, 100°<θ) ) |
WATER_BRIDGE_MINDIST, WATER_BRIDGE_MAXDIST WATER_BRIDGE_OMEGA_MIN, WATER_BRIDGE_OMEGA_MAX WATER_BRIDGE_THETA_MIN |
| ハロゲン結合 | Halogen bonds | 距離 (4.0Å以下) 角度 (Donor 165°±30°, Acceptor 120°±30°) |
HALOGEN_DIST_MAX, HALOGEN_DON_ANGLE, HALOGEN_ACC_ANGLE, HALOGEN_ANG_DEV |
自分のPCにインストール
コマンドラインからも使えるそうなのでインストールしてみます。
Githubの説明によると原子の属性の判別にOpenBabelを使っているそうで、OpenBabel(>= 2.3.2)のインストールが必要だそうです。他にはオプションですが
- PyMOL(>=1.7.x with Python bindings)
- Imagemagick(>=1.7.x)
- swig
などに依存しているそうです。
pip install plip
とすることでインストールできますが、Python 2.7.x.で実行してくださいとのことで、pip3としてみたらOpenBabelのwheelがどうのこうのというエラーで止まりました。
解析対象の共結晶構造
今回、相互作用解析を行いたい結晶構造はPD-L1と低分子の共結晶構造です。以前、UniProtの情報をもとに作成した記事から、低分子のものを取り出すと以下となります。
| PDB entry | Resolution (Å) | Chain | Positions | リガンド | リガンドID | 文献 |
|---|---|---|---|---|---|---|
| 5J89 | 2.20 | A/B/C/D | 2-134 | BMS-202 | 6GX | Oncotarget 2016(7)30323 |
| 5J8O | 2.30 | A/B | 18-134 | BMS-8 | 6GZ | Oncotarget 2016(7)30323 |
| 5N2D | 2.35 | A/B/C/D | 2-134 | BMS-37 | 8J8 | J. Med. Chem. 2017(60)5857 |
| 5N2F | 1.70 | A/B | 18-134 | BMS-200 | 8HW | J. Med. Chem. 2017(60)5857 |
| 5NIU | 2.01 | A/B/C/D | 18-134 | BMS-1001 | 8YZ | Oncotarget 2017(8)72167 |
| 5NIX | 2.20 | A/B/C/D | 18-134 | BMS-1166 | 8YQ | Oncotarget 2017(8)72167 |
登録されている低分子の構造がおかしいのではないか?といった問題はありますが、今回はそのあたりは無視します。(ご興味のある方は以前の記事をご参照いただければと思います。記事1 記事2 記事3)
解析実行
PLIPはPDB idさえあればPDBのサーバーからデータをとってきて解析してくれるとのことなので、以下のコマンドをターミナルで実行しました。 (Pythonのパス?の通し方がわからなくてJupyter notebookから使えなかった・・・)
alias plip='python ~/pliptool/plip/plipcmd.py' plip -i 5J89 -x
「-i PDBID」で解析したい構造のIDを、「-x」とすることで結果をXMLレポートファイルで出力できます。
このまま実行しようとしたところ、以下のようなエラーが出て来ました。
File "/usr/local/lib/python3.7/site-packages/plip/modules/supplemental.py", line 388, in read_pdb resource.setrlimit(resource.RLIMIT_STACK, (min(2 ** 28, maxsize), maxsize)) ValueError: current limit exceeds maximum limit
よくわからないので該当の箇所を確認して見ました。
def read_pdb(pdbfname, as_string=False): """Reads a given PDB file and returns a Pybel Molecule.""" pybel.ob.obErrorLog.StopLogging() # Suppress all OpenBabel warnings if os.name != 'nt': # Resource module not available for Windows maxsize = resource.getrlimit(resource.RLIMIT_STACK)[-1] resource.setrlimit(resource.RLIMIT_STACK, (min(2 ** 28, maxsize), maxsize)) sys.setrecursionlimit(10 ** 5) # increase Python recoursion limit return readmol(pdbfname, as_string=as_string)
PDBファイルを読み込むための関数のようです。Pythonライブラリのresourceの説明を見るかぎり、エラーとなっている箇所はプログラムによって使用されているシステムリソースを制限する処理のようです。PDBファイルを読み込むリソースの制限なら外してしまっても良さそうなので、コメントアウトして見ました。
def read_pdb(pdbfname, as_string=False): """Reads a given PDB file and returns a Pybel Molecule.""" pybel.ob.obErrorLog.StopLogging() # Suppress all OpenBabel warnings #if os.name != 'nt': # Resource module not available for Windows #maxsize = resource.getrlimit(resource.RLIMIT_STACK)[-1] #resource.setrlimit(resource.RLIMIT_STACK, (min(2 ** 28, maxsize), maxsize)) sys.setrecursionlimit(10 ** 5) # increase Python recoursion limit return readmol(pdbfname, as_string=as_string)
保存して再度実行したらエラーで止まらずに動き、無事XMLファイルが出力されました。こんな雑なことしていいのかよくわかりませんが、とりあえず動いたので良しとします。
RDKitと組み合わせてPLIFを計算
共結晶構造、全6構造に対して実行し、相互作用の情報を持つXMLファイルを手に入れたもののこれをどうしよう???と思っていたところOxford Protein Informatics Group(OPIG)のブログ記事(How to Calculate PLIFs Using RDKit and PLIP )に行き当たりました。PLIPのデータとRDKitを使ってProtein-Ligand interaction fingerprints (PLIFs)というのを計算できるそうです。
PLIF?
株式会社MOLSISのMOEの機能紹介にPLIFの説明がありました。
「リガンド-受容体間の相互作用の種類と強さをフィンガープリントで表現し、複数の結合状態を統計的に解析します。ドッキング結果や複数の複合体構造に含まれる相互作用を解析することで、活性/不活性に関連する相互作用の検出や、活性/不活性を分類する相互作用組み合わせルールの抽出、ルールに適合する活性ポーズに共通するファーマコフォアの検出などが行えます。」
化合物間の類似性評価にフィンガープリントが用いていましたが、タンパク質との相互作用解析にも拡張した、ということのようです。SAR News No.18の記事「実験と連携できる SBDD へ向けて」によるとPLIFの他にもSIFt(JMC2004(47)337)、aPLIF、aPLIED、Pharm-IF(JCIM2010(50)170)といった手法があるようです。*2
OPIGの真似をする
OPIGで紹介されていた方法は、
- PLIPの解析結果を使って結合サイトに含まれるアミノ酸残基と、リガンドとの相互作用に関わる残基を抜き出す
- 1.の情報をもとにRDKitでフィンガープリントにする
- 2.のフィンガープリントで複合体間の相互作用の類似性を評価
といった流れでした。
早速OPIGのコードをコピペしていきます。(日本語の部分は備忘録として私が加えたものです。)
解析ステップ1
PLIPの解析結果(XMLファイル)から情報を取得します。
# XMLを扱う標準ライブラリ(ElementTree)を使う import xml.etree.ElementTree as ET # 結合サイトのアミノ酸残基情報を取り出すための関数を定義 def generate_plif_lists(report_file, residue_list, lig_ident): # uses report.xml from PLIP to return list of interacting residues # and update list of residues in binding site plif_list_all = [] tree = ET.parse(report_file) #ファイルの読み込み root = tree.getroot() #ルート要素を取得する # list of residue keys that form an interaction for binding_site in root.findall('bindingsite'): #findall()で直接の子要素を検索 nest = binding_site.find('identifiers') #find()で最初の子要素にアクセス lig_code = nest.find('hetid') #<hetid>タグに記載されたリガンドIDを取得 if str(lig_code.text) == str(lig_ident): #関数の引数に与えたlig_identとリガンドIDが等しい時だけ情報を取得 #get the plifs stuff here nest_residue = binding_site.find('bs_residues') #結合サイトに含まれるアミノ酸残基のリストを取得 residue_list_tree = nest_residue.findall('bs_residue') for residue in residue_list_tree: res_id = residue.text dict_res_temp = residue.attrib #要素の属性(attribute)を取得 #結合サイトの残基一覧に含まれていない場合は残基を追加する if res_id not in residue_list: residue_list.append(res_id) #リガンドとの相互作用(contact)TRUEのものだけPLIFリストに追加 if dict_res_temp['contact'] == 'True': if res_id not in plif_list_all: plif_list_all.append(res_id) return plif_list_all, residue_list
では定義した関数を使いましょう。まずは結合サイトに含まれるアミノ酸残基(リガンドとの相互作用の有無は関係なし)を格納するための空のリストを作っておきます。
bs_res_list = []
PDBの6つの構造についてPDB IDと、リガンドIDの組み合わせの辞書を作ります。
P_L_dict = {"5J89":"6GX","5J8O":"6GZ","5N2D":"8J8",
"5N2F":"8HW","5NIU":"8YZ","5NIX":"8YQ"}
contact_res_dict = {}
for pdb_id, lig_id in P_L_dict.items():
pl = "conres_" + pdb_id
# xmlファイルは各PDB ID名のフォルダにそれぞれ格納した
xml_path = "PLIP_results/" + pdb_id + "/report.xml"
pl, bs_res_list = generate_plif_lists(xml_path, bs_res_list, lig_id)
contact_res_dict[pdb_id] = pl
得られた辞書(contact_res_dict)はPDB IDをキーとして、リガンドと接触する残基を値としています。
print(contact_res_dict.keys()) # dict_keys(['5J89', '5J8O', '5N2D', '5N2F', '5NIU', '5NIX']) print(contact_res_dict['5J89']) # ['66B', '56B', '121B', '121A', '56A', '115A', '54B', '123A', '115D', '121D', '115C', '121C', '124C', '54D', '56D', '123C', '20C', '56C', '66D']
結合サイトに含まれるアミノ酸残基の全体はbs_res_listに格納されています。
print(len(bs_res_list)) # 136
136個と非常に多いように思います。今回用いた共結晶構造ではPD-L1の2量体に対してリガンドが1つ結合しています。配列の位置としては等しくても、CHAIN名が異なるものを別の残基としてカウントしているため、多くなっているのではないかと思います。
解析ステップ2
残基のリストが得られたのでRDKitのPLIFとします。
以下の関数では結合サイトに含まれるアミノ酸残基全体の長さの次元のビットベクトルを用意し、相互作用に関わっている残基についてビットを立てます。
from rdkit import Chem, DataStructs from rdkit.DataStructs import cDataStructs def generate_rdkit_plif(residue_list, plif_list_all): #generates RDKit plif given list of residues in binding site and list of interacting residues plif_rdkit = DataStructs.ExplicitBitVect(len(residue_list), False) for index, res in enumerate(residue_list): if res in plif_list_all: # print('here') もともとのコードにはあったが面倒なのでコメントアウト plif_rdkit.SetBit(index) else: continue return plif_rdkit rdkit_plif_dict = {} for pdb_id in contact_res_dict.keys(): con_res = contact_res_dict[pdb_id] generated_plif = generate_rdkit_plif(bs_res_list, con_res) rdkit_plif_dict [pdb_id] = generated_plif print(rdkit_plif_dict.keys()) # dict_keys(['5J89', '5J8O', '5N2D', '5N2F', '5NIU', '5NIX'])
6つとも計算されていそうです。一つ取り出して確認してみます。
print(len(rdkit_plif_dict['5J89'])) # 136 print(rdkit_plif_dict['5J89']) # <rdkit.DataStructs.cDataStructs.ExplicitBitVect object at 0x10d246d88>
長さ136のベクトルが生成されています。ExplictiBitVectオブジェクトの扱い方は「化学の新しいカタチ」さんのこちらの記事RDKitでフィンガープリントを使った分子類似性の判定に記載されていました。
どんな情報が含まれているのか見てみます。
print("全体の長さ:", rdkit_plif_dict['5J89'].GetNumBits()) # 全体の長さ: 136 print("ONビット:", list(rdkit_plif_dict['5J89'].GetOnBits())) print("ONビットの数:", rdkit_plif_dict['5J89'].GetNumOnBits()) print("OFFビットの数:",rdkit_plif_dict['5J89'].GetNumOffBits()) print("相互作用残基の数:",len(contact_res_dict['5J89'])) # ONビット: [5, 9, 14, 17, 20, 23, 33, 36, 45, 46, 49, 58, 64, 73, 77, 79, 80, 82, 84] # ONビットの数: 19 # OFFビットの数: 117 # 相互作用残基の数: 19
PDB id 5J89における相互作用に関わる残基の数とRDKitで変換後のONビットの数が一致しています。
DataFrameとheatmapで可視化
ビットベクトルをもう少しわかりやすく眺めたいと思います。目標は先に見た株式会社MOLSISのページのページのように複数の共結晶構造間におけるONビットの位置の違いを並べて可視化することです。
- BitVectをnumpyのarrayに変換
- データフレームに変換
- heatmapで図示
という手順を行ってみます。
import numpy as np # DataFrameのindex としてPDBのIDを使うためにリストを作成 PDB_id_list = [] finger_print_arrays = [] for pdb_id in rdkit_plif_dict.keys(): PDB_id_list.append(pdb_id) # RDKitのBitVectそのままではDataFrameにできなさそうなのでarrayに変換する。 fp = rdkit_plif_dict[pdb_id] arr = np.zeros((1,)) DataStructs.ConvertToNumpyArray(fp, arr) finger_print_arrays.append(arr) import pandas as pd #DataFrameを作成 FP_df = pd.DataFrame(finger_print_arrays, index=PDB_id_list) FP_df

うまくできていそうです。0ばっかり・・・これがスパースという奴か!!!(適当)
可視化します。
import matplotlib as mpl import matplotlib.pyplot as plt import seaborn as sns %matplotlib inline sns.heatmap(FP_df)

おお!それっぽい!y軸は各複合体(PDB id)で、x軸方向にビットベクトルが並んでいます。各ビットが結合サイトに含まれるアミノ酸残基相当し、黒いところがビットベクトルが立っているリガンドと相互作用する残基となっています。
(私の理解が正しければ・・・X軸方向の並びがアミノ酸配列の並びと正確に一致するかはわかりません。)
各複合体で共通してビットが立っているアミノ酸残基が、複数の複合体で相互作用が維持されている重要な残基と解釈できる・・・はず?
図から雑に解釈
上の図をみると5J8O、5N2FがもっともONビットの分散が小さく、5NIU、5NIXがよりベクトル全体に広がってビットが立っています。
先に、今回のPLIFの方法ではCHAIN毎に区別を行っていると述べました。6つの共結晶構造は全てPD-L1の2量体とリガンド1つとの2対1の複合体ですが、より詳しく見ると
- 5J8O、5N2Fは「PD-L1 2: Ligand 1」
- 5J89、5N2D、5NIU、5NIXは「PD-L1 4: Ligand 2」
という違いがあります。タンパク質側のCHAINが4つ含まれている他の複合体と比較して2つしか含まれていない5J8O、5N2Fではとりうるビットベクトルの範囲がそもそも小さいため以上のような結果となっていそうです。
また、5NIU、5NIXでは結合しているリガンドが、他の複合体よりも大きいものとなっています。リガンドが大きい分相互作用する残基が広がっているのかもしれません。
一覧を再掲します。
| PDB entry | Chain | リガンド | リガンドID | リガンド分子量 |
|---|---|---|---|---|
| 5J89 | A/B/C/D | BMS-202 | 6GX | 419.52 |
| 5J8O | A/B | BMS-8 | 6GZ | 494.42 |
| 5N2D | A/B/C/D | BMS-37 | 8J8 | 448.55 |
| 5N2F | A/B | BMS-200 | 8HW | 497.49 |
| 5NIU | A/B/C/D | BMS-1001 | 8YZ | 594.65 |
| 5NIX | A/B/C/D | BMS-1166 | 8YQ | 643.13 |
だいたい分子量とビットの広がりが相関があるような気もします。なんとなく3つのグループ(①5J89と5N2D、②5J8Oと5N2F、③5NIUと5NIX)に分かれそうです。
図をぼんやりと眺めていても定性的な類似性しかわかりませんので、定量的な評価を行いたいと思います。
解析ステップ3
フィンガープリント間の類似性評価をTanimoto係数を計算して比較します。関数をコピペ・・・
def similarity_plifs(plif_1, plif_2): sim = DataStructs.TanimotoSimilarity(plif_1, plif_2) print(sim) return sim
6つの構造から2つ取り出してTanimoto係数を計算しますが、一つずつやるのは大変なのでitertoolsを使ってみます。
import itertools # 重複無しの順列 itertools.permutations(PDB_id_list, 2) # 重複順列 itertools.product(PDB_id_list, repeat = 2) # 今回は可視化のために重複ありの組み合わせを使う tanimoto_list = [] for v in itertools.combinations_with_replacement(PDB_id_list, 2): #6つから2つ取り出しi, j とする。 i, j = v[0], v[1] plif_i = rdkit_plif_dict[i] plif_j = rdkit_plif_dict[j] sim = similarity_plifs(plif_i, plif_j) #用いた2つの構造とTanimoto係数をもつリストを作成 temp_list = [i, j, sim] #リストのリストを作成 tanimoto_list.append(temp_list) #DataFrameとする tanimoto_df = pd.DataFrame() for k in tanimoto_list: tanimoto_df.at[k[0], k[1]] = k[2] #heatmapで可視化 sns.heatmap(tanimoto_df)


色の濃い組み合わせを見ると
- 5J89、5N2D、0.63
- 5J8O、5N2F、0.42
- 5NIU、5NIZ、0.56
となっています。ビットベクトルをそのまま可視化した際の定性的な解釈と似た結果となっており、うまく数値化できていそうです。
残基番号のみで再解析
ところで、共結晶構造を見る限りリガンドを挟んでPD-L1の二量体が対称的に配置されているように見えます。対称的ならば配列の番号が大事で、CHAINを区別していることでむしろ同じ相互作用を別のものと判断しているかもしれません。そこでCHAINの情報を取り除いて同じことをやってみたいと思います。
・・・といってもStep 1で取り出した残基の情報から最後のアルファベットを除くだけですが。。。
# 結合サイトの残基からCHAINの情報を除く bs_res_list_num = [] for i in bs_res_list: #文字列から最後の一文字の手前まで取り出す j = i[:-1] #後でソートしたいので整数型に変換してからリストに加える bs_res_list_num.append(int(j))
重複している情報を取り除き、ソートします。
bs_res_list_num_unique = sorted(set(bs_res_list_num)) print(len(bs_res_list_num_unique)) print(bs_res_list_num_unique) # 40 # [18, 19, 20, 21, 22, 23, 26, 53, 54, 55, 56, 57, 58, 60, 61, 62, 63, 65, 66, 67, 68, 73, 75, 76, 77, 78, 112, 113, 114, 115, 116, 117, 118, 120, 121, 122, 123, 124, 125, 126]
相互作用の残基からCHAINの情報を除きます。今回は重複も先に除きます。
contact_res_dict_num_unique = {}
for k, v in contact_res_dict.items():
tmp_list = []
for i in v:
j = i[:-1]
tmp_list.append(int(j))
tmp_unique = sorted(set(tmp_list))
contact_res_dict_num_unique[k] = tmp_unique
ビットのリストを取得
RDKitのBitVectを介してからnumpyのarrayに戻すと残基番号の情報が失われてしまうので、OPIGの関数を参考に新しくビットのリストを返す関数を作成してみたいと思います。
def PLIF_list_generator(bs_residues, contact_residues): tmp = [] for i in bs_residues: if i in contact_residues: tmp.append(1) else: tmp.append(0) return tmp
PDB idを行、残基番号を列とするDataFrameを作成します。
FP_df_num = pd.DataFrame(index =PDB_id_list, columns = bs_res_list_num_unique) for k, v in contact_res_dict_num_unique.items(): FP_df_num.loc[k] = PLIF_list_generator(bs_res_list_num_unique, v)

複数の共結晶構造で各残基が相互作用に使われているかを確認したいので、残基ごとのビットの足し算も行っておきます。
FP_df_num.loc['bit_sum']=FP_df_num.sum() %matplotlib inline sns.heatmap(FP_df_num)

なんだかいい感じになってきました!
大事そうな残基を探す
先のBitVectを眺めた際の解釈で、6つの共構造は3つのグループに分かれそうだということでした。ということはbit_sumが5以上のものは3つのグループのすべてで少なくとも一度は使われている残基です。(たぶん・・・)(最初4以上にしていましたが5以上の間違いでした)
取り出して見ましょう。
# queryメソッドは行の抽出なので転置してから使う # bitの合計5以上を取り出し、indexをリスト化 Frequent_residues = list(FP_df_num.T.query('bit_sum >= 5').index) print(Frequent_residues) #[54, 56, 115, 121, 123]
これらは具体的にどんな相互作用なのでしょうか?すべての残基を含んでいそうな「PDB id:5NIX(Ligand:8YQ)」をPLIPのサイトで解析してみましょう。(コマンドラインでの解析を諦めた)
「PD-L1 4: Ligand 2」の複合体なのでリガンドが2つ含まれています。結果をテーブルにまとめました。
| 残基番号 | アミノ酸 | 距離 | 相互作用 | 残基番号 | アミノ酸 | 距離 | 相互作用 | ||
|---|---|---|---|---|---|---|---|---|---|
| 8YQ(chain A/B) | 8YQ(chain C/D) | 54C | ILE | 3.89 | Hydrophobic | ||||
| 56B | TYR | 3.82 | Hydrophobic | 56C | TYR | 3.42/3.62 | Hydrophobic | ||
| 115A/B | MET | 3.70/3.80 | Hydrophobic | 115C/D | MET | 3.85/3.82 | Hydrophobic | ||
| 121A/B | ALA | 3.69/3.75 | Hydrophobic | 121C/D | ALA | 3.96/3.57 | Hydrophobic | ||
| 123A | TYR | 3.80/3.52/3.85 | Hydrophobic | 123D | TYR | 3.72 | Hydrophobic |
リガンドの芳香環が目立っていたのでπ-π相互作用ばかりかと思っていましたが、予想外にメチオニンやアラニンとの相互作用が複数見られました。
もう少し広くカウント数4以上の残基として見ます。
# bitの合計4以上の場合 Frequent_residues4 = list(FP_df_num.T.query('bit_sum >= 4').index) print(Frequent_residues4) # [54, 56, 66, 115, 121, 123, 124]
先の結果に66、124が加わりました。アミノ酸残基でいうと以下となります。
| 残基番号 | アミノ酸 | 距離 | 相互作用 | 残基番号 | アミノ酸 | 距離 | 相互作用 | ||
|---|---|---|---|---|---|---|---|---|---|
| 8YQ(chain A/B) | 66B | GLN | 3.72 | Hydrophobic | 8YQ(chain C/D) | ||||
| 124A | LYS | 5.03/3.18 | π-cation/Salt Bridges | 124D | Lys | 3.70 | Water Bridges/Salt Bridges |
疎水性相互作用ばかりだったところに親水性残基のLysが加わりました。こちらの方がバラエティがあって良さそうです。
PLIPのスナップショットを貼っておきます。こんな相互作用です・・・

まとめ
以上、今回はタンパク質とリガンドの相互作用情報を考察するためPLIP、PLIFといった手法を導入して見ました(コピペですが・・・)。この情報をうまく使えばファーマコフォア(?)を考察できるはず・・・?
色々と間違いがあると思うのでご指摘いただければ幸いです。
*1:AS Rose et al. (2018) NGL viewer: web-based molecular graphics for large complexes. Bioinformatics dio:10.1093/bioinformatics/bty419
オルト位置換基で絞り込む話
前回の記事で、SMARTS記法の扱い方がなんとなくわかりました。本題にもどって、ライブラリをオルト位に置換基の入ったビフェニルで絞り込みたいと思います。
その前に、現在残っている化合物数の確認です。
| Enamine_Premium | Enamie_Advenced | Enamine_HTS | UOS_HTS | total | |
|---|---|---|---|---|---|
| 指標でのフィルタリング後 | 4060 | 37431 | 414562 | 106948 | 563001 |
| ビフェニル有 | 12 | 329 | 3182 | 697 | 4220 (0.7%) |
| ビフェニル無 | 4048 | 37102 | 411380 | 106251 | 558781 (99.3%) |
合計4000個程度残っています。このうちオルト位に置換基が入ったものはいくつあるでしょうか?
オルト位置換基による絞り込み
PandasToolsでSDFをよみこむ
4種類のライブラリから抽出した、ビフェニルを部分構造として含む化合物のリストを連結(extend()を使った)し、SDFファイル「biphenyl_library.sdf」として保存しました。こちらをRDKitのPandasToolsをもちいてDataFrameでよみこみます。
PandasToolsの使い方に関しては化学の新しいカタチさんの記事「RDKitのPandasToolsでデータ分析を加速する」を参考にさせていただきました。
from rdkit import rdBase, Chem from rdkit.Chem import AllChem, Draw from rdkit.Chem import PandasTools import pandas as pd biphenyl_df = PandasTools.LoadSDF('./biphenyl_library.sdf') # 数を確認 len(biphenyl_df) # 4220
こんな感じになりました。

Pandas上で部分構造検索
PandasToolsを使うと「>=(ge比較演算子)」で部分構造検索が行えるとのことですので試しに利用して見ます
ortho_biphenyl= Chem.MolFromSmarts('c1ccc(c(*)c1)c1ccccc1') ortho_df = biphenyl_df[biphenyl_df['ROMol'] >= ortho_biphenyl] len(ortho_df) # 2353
オルト位に置換基を持つ化合物は約半数程度の2353個でした。
一致検索後のDataFrameでは、Molオブジェクトを格納したカラムにおいて、一致した部分構造が赤色でハイライトされて表示されています。

忘れずにSDFに書き出しておきます。SDFに残すプロパティを指定するため、DataFrameのカラム名をリストとして取得します。
property_names = list(biphenyl_df.columns) print(property_names) # ['ID', 'MW', 'MolLogP', 'NumHAcceptors', 'NumHDonors', 'NumRotatableBonds', 'ROMol', 'TPSA', 'original_id']
「ROMol」は不要なのでリストから除きます。
property_names.remove('ROMol') print(property_names) # ['ID', 'MW', 'MolLogP', 'NumHAcceptors', 'NumHDonors', 'NumRotatableBonds', 'TPSA', 'original_id']
出力します。
PandasTools.WriteSDF(ortho_df, 'ortho_biphenyl_library.sdf', properties = property_names)
以上で、オルト位に置換基をもつ部分構造での絞り込みが完了しました。
オルト位の置換基の構造的意義
なぜ「オルト位に置換基を持つ」ということを、絞り込みの基準として重要視するのか?ついでなので「オルト位に置換基を持つ」ということの意味について考えたいと思います。
・・・といっても立体障害でビフェニル骨格が捻れるというだけなのですが、、、、
3次元構造を眺める
オルト位の置換基の有無が3次元構造(コンフォメーション)にどのような影響を与えるか?ということを眺めて見ます。3次元構造の生成は「化学の新しいカタチ」さんの記事「RDKitによる3次元構造の生成」を参考にしました。
まずは構造を準備。無置換、オルト位にメチル基を1つもつもの、2つ持つものの3つを作りました。
biPh = Chem.MolFromSmiles('c1ccc(cc1)c1ccccc1') ortho_Me_biPh = Chem.MolFromSmiles('c1ccc(c(C)c1)c1ccccc1') ortho_diMe_biPh = Chem.MolFromSmiles('c1cc(C)c(c(C)c1)c1ccccc1') biPh_list = [biPh, ortho_Me_biPh, ortho_diMe_biPh] Draw.MolsToGridImage(biPh_list)

水素原子を付加し、ETKDG法で3次元構造を生成します。
biPh_3D_list = [] for mol in biPh_list: mol_H = AllChem.AddHs(mol) AllChem.EmbedMolecule(mol_H, AllChem.ETKDG()) biPh_3D_list.append(mol_H)
生成した構造を眺めます。
from rdkit.Chem.Draw import IPythonConsole import py3Dmol v = py3Dmol.view(width=600, height=300, linked=False, viewergrid=(1,3)) for m, i in zip(biPh_3D_list, [(0,0), (0,1), (0,2)]): mb = Chem.MolToMolBlock(m) v.addModel(mb, 'sdf', viewer=i) v.setBackgroundColor('0xeeeeee') v.setStyle({'stick':{}}) v.zoomTo() v.show()
無置換、モノ置換、ジ置換の順にねじれ角度が大きくなっているのがわかります。

もっとわかりやすくするために、重ねて見ます。
アラインメントをとって重ねる
RDKitブログの記事を参考にして3つとも重ねて見ます。
rdMolAlign.AlignMolを使って部分構造のアラインメントを取り3次元構造を重ね合わせます。重ねたい部分構造(template)を明示的に与えるため引数atomMapに無置換のベンゼン環に相当するatom numberを与えます。
こちらや、こちらを参考にエラーに対処していたらやたらとlist()を使うことになってしまった・・・
from rdkit.Chem import rdMolAlign # 無置換フェニルのテンプレートをSmartsから作成 template = Chem.MolFromSmarts('[cH1]1[cH1][cH1][cH1][cH1]c1-c') # テンプレートに一致する無置換ビフェニルの原子を基準にする # タプルのタプルで得られる template_atoms = biPh_3D_list[0].GetSubstructMatches(template) # アラインメントを取る for m in biPh_3D_list: matched_atoms = m.GetSubstructMatches(template) rdMolAlign.AlignMol(m, biPh_3D_list[0], atomMap = list(zip(list(matched_atoms[0]), list(template_atoms[0])))) # 描く v = py3Dmol.view(width=300, height=300) for m in biPh_3D_list: mb = Chem.MolToMolBlock(m) v.addModel(mb, 'sdf') v.setBackgroundColor('0xeeeeee') v.setStyle({'stick':{}}) v.zoomTo() v.show()


静止画ではわかりにくいですが、無置換、モノ置換、ジ置換の順に二面角(Torsion Angle)が大きくなっているのがわかります。
二面角を計算してみる
それぞれ一つずつコンフォマーを取得して二面角を計算して見ます。
まず二面角を計算するために、目的の部分のatom numberを確認します。無置換ビフェニルの場合・・・
Draw.MolToImage(biPh_3D_list[0], includeAtomNumbers = True)

atom numberがわかったので目的の4原子を指定して、2面角を求めます。
from rdkit.Chem import rdMolTransforms conf=biPh_3D_list[0].GetConformer(0) rdMolTransforms.GetDihedralDeg(conf, 2,3,6,7) # 148.88976772600805
モノ置換、ジ置換も同様に計算し、0° - 90°の範囲に値を修正すると以下のようになります。
| 無置換 | モノ置換 | ジ置換 | |
|---|---|---|---|
| 二面角 | 31° | 50° | 81° |
順に二面角が大きくなり、特にジ置換ではほぼ直角近くになっています。
フィンガープリントではどう認識される?
上記の二面角の差異がフィンガープリントとしてはどのように認識できるか、少し見てみたいと思います。Atom PairsとTopological Torsionsでそれぞれ類似性を比較した際にどのようになるか試してみます。
この二つを選んだ理由は名前の雰囲気です・・・(計算方法を理解していません。すみません)。
from rdkit import DataStructs from rdkit.Chem.AtomPairs import Pairs pair_fps = [Pairs.GetAtomPairFingerprint(mol) for mol in biPh_3D_list] from rdkit.Chem.AtomPairs import Torsions tts_fps = [Torsions.GetTopologicalTorsionFingerprintAsIntVect(mol) for mol in biPh_3D_list] # Tanimoto係数、Dice係数をそれぞれの組み合わせで計算 pair_Dice_0_1 = DataStructs.DiceSimilarity(pair_fps[0], pair_fps[1]) pair_Tani_0_1 = DataStructs.TanimotoSimilarity(pair_fps[0], pair_fps[1]) # 残りの組み合わせ、TorsionFingerprintについても同じ計算をしたので省略
結果をまとめると以下になりました。
| 無置換-モノ置換 | 無置換-ジ置換 | モノ置換-ジ置換 | |
|---|---|---|---|
| AtomPair_Tanimoto係数 | 0.76 | 0.59 | 0.77 |
| AtomPair_Dice係数 | 0.86 | 0.75 | 0.87 |
| Torsion_Tanimoto係数 | 0.87 | 0.77 | 0.88 |
| Torsion_Dice係数 | 0.93 | 0.86 | 0.94 |
Torsionの方が差異が大きくなると面白いと思っていたのですが、今回の結果ではAtom Pairの方が置換の数の差を反映しているようにも見えます。いずれにせよ、メチルの数が1つ増えるか2つ増えるかで類似度が下がっていくことに代わりわなさそうです。
オルト位置換を創薬の話に繋げようとする
オルト位に置換基を加えることで分子がねじれ、立体的な構造が変化するということがわかりました。では、このねじれを医薬品に活用した事例は???ということでFasiglifam(TAK-875)の構造を眺めて見たいと思います。2型糖尿病を適応として開発が行われていた武田薬品の化合物で、GPCRの一つGPR40の作動薬です。残念ながら臨床試験第三相で肝臓における安全性の懸念から開発中止となり、上市にはいたらなかったそうです。
Fasiglifam(TAK-875)の構造
なぜこちらを取り上げるのか? 構造を見ていただくとお分かりいただけると思います。 印象的なビフェニル、オルト-ジ置換構造を持っています。
Fasiglifam_mol = Chem.MolFromSmiles('CC1=CC(=CC(=C1C2=CC(=CC=C2)COC3=CC4=C(C=C3)[C@@H](CO4)CC(=O)O)C)OCCCS(=O)(=O)C') from rdkit.Chem.Draw import rdMolDraw2D from IPython.display import SVG highlights = Fasiglifam_mol.GetSubstructMatch(ortho_diMe_biPh) view = rdMolDraw2D.MolDraw2DSVG(500,500) pre_mol = rdMolDraw2D.PrepareMolForDrawing(Fasiglifam_mol) view.DrawMolecule(pre_mol, highlightAtoms=highlights) view.FinishDrawing() svg = view.GetDrawingText() SVG(svg.replace('svg:,',''))

Fasiglifamを見出した構造活性相関の研究に関していくつかの論文が出されています。ずいぶんと以前に読んだ論文のため引用しようにも忘れてしまいました(おそらくJ.Med.ChemやACS Med. Chem. Lett.だったような・・・)。その中でビフェニル構造に置換基を加えることで2面角が変化すること、その構造の変化が活性に大きな影響を与えたことが議論されていたように思います。(いろいろぼんやりとした記憶で不確かですみません・・・)
では立体的にはどのような構造なのか?眺めて見ます。PubChemのID「cid: 24857286」をつかってpy3Dmolで描画して見ます。
化学の新しいカタチさんの記事「py3Dmolを使って化学構造をJupyter上で美しく表示する」を参考にさせていただきました。
view = py3Dmol.view(width= 800, height=300, viewergrid=(1,3), query='cid:24857286') view.setStyle({'cartoon': {}}, viewer=(0,0)) view.setStyle({'stick': {}}, viewer=(0,1)) view.setStyle({'sphere': {}}, viewer=(0,2)) view.show()

ビフェニル構造がほぼ直角にねじれています。
受容体との共結晶構造
FasiglifamとGPR40との共結晶構造が解かれています(PDB id: 4PHU*1)。ついでに眺めて見ます。 *2*3
view = py3Dmol.view(width=680, height=480, query='pdb:4PHU', viewergrid=(1,1)) view.setStyle({'cartoon': {'color': 'spectrum'}}, viewer=(0,0)) view.setStyle({'resn': '2YB'}, {'sphere': {'colorsheme': 'default'}}, viewer=(0,0)) view.addSurface(py3Dmol.SES, {'opacity': 0.6, 'color': '#FADBD8'}, viewer=(0,0)) view.show()

とても興味深いことに、Fasiglifamはその構造の半分程度がGPR40のヘリックスの合間から外に向かって飛び出しています。
上の図では少しわかりにくいのでRCSD PDBのビューワーでリガンドと受容体の相互作用部位を拡大して眺めます。
こんな感じ・・・

ちょうど直角にねじれたビフェニル構造の部分が受容体内-外の境目に来ています。最近ではGPCRの構造が多数解かれるようになってきますが、この構造が公開された当時(2014年)はまだそこまで多くなく、さらにリガンドの結合様式も見たことがないものでこんな結合の仕方あるのか!!!と、とても驚いたのを覚えています。
まとめ
以上、今回はオルト位に置換基を含む化合物のライブラリからの抽出と、その構造的意義についてざっと眺めて見ました。オルト位に置換基を加えることでライブラリ化合物は2400弱にまで絞り込むことができました。後半、古い記憶を辿ってFasiglifamの話などをし始めたのは、ライブラリスクリーニングの方法に行き詰まったからです・・・困難から逃走するタイプ・・・
*1:Crystal structure of Human GPR40 bound to allosteric agonist TAK-875 DOI: 10.2210/pdb4PHU/pdb
*2:High-resolution structure of the human GPR40 receptor bound to allosteric agonist TAK-875. (2014) Nature 513: 124-127
*3:AS Rose et al. (2018) NGL viewer: web-based molecular graphics for large complexes. Bioinformatics dio:10.1093/bioinformatics/bty419
SMARTS記法とRDKitの化学反応で遊んだ話
前回の記事で、ビフェニル部分構造によるフィルタリングを行いましたが、オルト位に置換基の入ったビフェニルについてはうまく実行することができませんでした。@iwatobipen先生、@mojaie先生にSMARTSを使えば良いというご指摘をいただきました。ありがとうございました!
まずは前回失敗した処理を再掲します。
活性化合物のデータセット (共闘用シェアデータ )から取り出した以下の分子に対し、

RDKitの部分構造検索(HasSubstructMatch)を用い、オルト位に置換基の入った構造を取り出そうとしました。
from rdkit import rdBase, Chem
from rdkit.Chem import AllChem, Draw
ortho_biphenyl = Chem.MolFromSmiles('c1ccc(c(*)c1)c1ccccc1')
Draw.MolToImage(ortho_biphenyl)

検索したい部分構造は上のものです。(* はワイルドカード)
test_mol.HasSubstructMatch(ortho_biphenyl)
# False
上記のようにFalseとなってしまうというのが前回の失敗でした。
まずは@iwatobipen先生に教えていただいたSMARTSを用いた方法を試します。
ortho_biphenyl_2 = Chem.MolFromSmarts('c1cccc([!H])c1-c1ccccc1')
Draw.MolToImage(ortho_biphenyl_2)

SMARTSから構築したMOlオブジェクトは、先に作成したものとは異なる形で認識されているようです。(見た感じ・・・)
こちらを使って部分構造の一致を検証します。
test_mol.HasSubstructMatch(ortho_biphenyl_2)
# True
True! うまく構造の一致を認識できました!
次に@mojaie先生にご指摘いただいた事項。
私が失敗したコードでは、Chem.MolFromSmiles()としてしまっているから上手くいかないのではないか、ということでした。「ワイルドカードの*は検索条件なので、本来はSMARTSの表記」のため、Chem.MolFromSmarts()とするのが適切、ということだそうです。
ortho_biphenyl_3 = Chem.MolFromSmarts('c1ccc(c(*)c1)c1ccccc1') test_mol.HasSubstructMatch(ortho_biphenyl_3) # True
True! こちらでもうまく認識できました!
先生方ありがとうございました!
今回失敗した原因は、SMILESとSMARTSの違いを理解せずに適当に使ってしまっていたことでした。
RDKitのドキュメンテーションsubstructure_searchingによると、部分構造検索のクエリ分子はSMILESからでもSMARTSからでも良いが、二つの記法で違いがあるから注意してください、といった指摘がなされていました。
そこで、両者の違いについて少し勉強してみることとしました。
SMILESとSMARTSの違い
SMARTSとは?
そもそもSMARTSって何の略なんだろう?と思っていたのですが、株式会社MOLSISさんのページにわかりやすくまとめられていたので転記させていただきます。 (同社はTwitterで何度かお見かけしたドッキングに使われていたソフト、MOEも製品として取り扱われているそうです)
SMILES(Simplified Molecular Input Line Entry System) : 分子記述言語
- 分子の二次元構造を文字列として記述
- 情報をコンパクトに保存
- 原子座標の羅列と違い、ユーザーにも理解しやすい
SMARTS(SMiles ARbitrary Target Specification): パターン記述言語
- SMILES言語を検索クエリ用に拡張したパターン記述言語
- ワイルドカードや条件指定といった構造検索条件を表記可能
つまり、SMILESを構造検索に用いるため発展させたもの、ということのようです。
Daylight社 の解説ページでSMILES / SMARTSを勉強
SMARTSの詳細については、SMILESの考案者Dr. David Weiningerの設立したDaylight C.I.S. 社のページ を参照してください、と「化学の新しいカタチ」さんのこちらの記事(SMILES記法は化学構造の線形表記法)でオススメされていたので早速眺めて見たいと思います。
部分構造検索とは?
- 分子(グラフ)から特定のパターン(サブグラフ)を見つけ出すプロセス
部分構造検索におけるSMILESの特徴
SMILESを直接拡張したSMARTSではさらに・・・・
- 論理演算子(logical operator)
- より一般的な原子、結合を表現するための特別な原子記号(atomic symbol)、結合記号(bond symbol)が使える
ようになっているそうです。
基本的にはSMILESの拡張のため、SMARTSでもSMILESの表現が使えるそうですが、SMILESは分子記述、SMARTSはパターン記述という違いから同じ文字列でも意味合いが異なってくることに注意が必要、とのこと。Daylight社のページでは以下の具体例が挙げられていました。
例1: 分子とパターン
例2: 原子を括弧 () を使わないで指定した場合
記法のルールを写経
具体的にはどのような記法になるのか、ざっくりテーブルを日本語にします。順番など少し入れ替えています。
SMILES記法同様、角括弧「」は原子の境界を、丸括弧「()」は分岐構造を示します。
原子
| 記号 | 意味 | デフォルト |
|---|---|---|
| * | ワイルドカード(どの原子でも良い) | |
| a | 芳香属性原子 | |
| A | 脂肪族原子 | |
| D<数字> | 明示的な結合の数(級数:degree)が<数字> | ちょうど1 |
| H<数字> | 結合している水素原子が<数字>個 | ちょうど1 |
| h<数字> | 暗に示された水素原子が<数字>個 | 少なくとも1 |
| R<数字> | <数字>のSSSR環構造に含まれる(ring membership) | 環の構成原子どれでも |
| r<数字> | <数字>のサイズのSSSR環構造に含まれる(ring size) | 環の構成原子どれでも |
| v<数字> | 結合次数の総数が<数字> | ちょうど1 |
| X<数字> | 結合の総数が<数字> | ちょうど1 |
| x<数字> | 環構造の結合の総数が<数字>(ring connectivity) | 少なくとも1 |
| -<数字> | マイナス <数字> の電荷 | -1 電荷、--なら-2のチャージ |
| +<数字> | プラス <数字> の形式電荷 | +1 電荷、++なら+2のチャージ |
| #<数字> | 原子番号<数字> | |
| @ | 不斉、反時計回り(@@は時計回り) | |
| <数字> | 明示的な原子量 |
結合
| 記号 | 意味 |
|---|---|
| - | 単結合(脂肪族) |
| = | 2重結合 |
| # | 3重結合 |
| : | 芳香族性結合 |
| ~ | ワイルドカード(どのような結合形式でも良い) |
| @ | 環結合ならなんでもよい |
| / | 方向性のある結合 アップ |
| |方向性性のある結合 ダウン | |
| /? | アップあるいは指定されていない |
| \? | ダウンあるいは指定されていない |
二重結合の幾何異性体は、例えば「 /C=C/ : trans型2重結合(E)」「/C=C\ : cis型2重結合(Z)」と言ったように表すことができるそうです(SMILES記法の場合)。
論理演算子
| 記号 | 意味 |
|---|---|
| ! | not |
| & | and |
| , | or |
| ; | and |
andを意味するのに「&」と「;」の2つがありますが、「&」が優先されるそうです。
論理演算子を使って原子のパターンを記述するには、角括弧「[]」内部にその原子と条件指定全体を含めることに注意が必要とのことです。
Reaction SMILES / SMARTS
SMILESには他にも拡張として、化学反応を記述できるようにしたReaction SMILESというものもあるそうです。 どのような拡張がなされているのか、「化学の新しいカタチ」さんのこちらの記事RDKitで化学反応:ケモインフォマティクスにおける反応式の扱い方 にわかりやすく記載されているのでご参照ください。
折角なので遊んで見たいと思います。取り上げる反応はC-H結合活性化!(・・・私が学生の頃とても流行ってた)
Murai reaction
まずは村井眞二先生らによる世界初の実用的触媒的C-H結合活性化型反応から*1*2

「原料・試薬 >> 生成物」とのことなのでReaction SMILESは以下のようになります。*3
O=C-C1=CC=CC=C1.CCO[Si](OCC)(OCC)C=C>>CCO[Si](CCC1=C(C=O)C=CC=C1)(OCC)OCC
二つの原料はSMILES同士が「.」でつながれています。
肝心の触媒を忘れました。
RuH2(CO)(PPh3)3を二つの「>」の間に挿入しなければいけません。PubChemによるとこの触媒のSMILESは「[C-]#[O+].C1=CC=C(C=C1)P(C2=CC=CC=C2)C3=CC=CC=C3.C1=CC=C(C=C1)P(C2=CC=CC=C2)C3=CC=CC=C3.C1=CC=C(C=C1)P(C2=CC=CC=C2)C3=CC=CC=C3.[RhH2]」だそうです。長すぎる・・・
O=C-C1=CC=CC=C1.CCO[Si](OCC)(OCC)C=C>[C-]#[O+].C1=CC=C(C=C1)P(C2=CC=CC=C2)C3=CC=CC=C3.C1=CC=C(C=C1)P(C2=CC=CC=C2)C3=CC=CC=C3.C1=CC=C(C=C1)P(C2=CC=CC=C2)C3=CC=CC=C3.[RhH2]>CCO[Si](CCC1=C(C=O)C=CC=C1)(OCC)OCC
訳が分からなくなってしまいました。
RDKitの反応に使うにはReaction SMARTSにするとのことなので触媒を省略した上で以下のように書いてみました。
[O:1]=[C:2]-[c:3][c:4].[C:5]=[C:6]-[Si:7] >> [O:1]=[C:2]-[c:3][c:4]-[C:5]=[C:6]-[Si:7]
では実際に反応を試します。反応させるMolオブジェクト、反応パターン(Reaction SMARTS)を用意した上でRunReactantsを実行します。
reactant_1 = Chem.MolFromSmiles('O=C-C1=CC=CC=C1') reactant_2 = Chem.MolFromSmiles('CCO[Si](OCC)(OCC)C=C') reaction_pattern = '[O:1]=[C:2]-[c:3][c:4].[C:5]=[C:6]-[Si:7] >> [O:1]=[C:2]-[c:3][c:4]-[C:5]=[C:6]-[Si:7]' rxn = AllChem.ReactionFromSmarts(reaction_pattern) x = rxn.RunReactants([reactant_1, reactant_2]) print(x) # ((<rdkit.Chem.rdchem.Mol object at 0x115f19fa8>,), (<rdkit.Chem.rdchem.Mol object at 0x115f19e48>,))
結果はMolオブジェクトを要素とするタプルのタプルとして与えられるようです。外側のタプルはとりうる反応点の組み合わせに相当し、内部のタプルは一つの反応点の組み合わせあたりに複数の生成物ができる場合(加水分解など)に生成物を格納するためにあるようです。
今回は同一の分子が2つ生成されていました。一方を取り出して見ます。
x_0 = x[0][0] Draw.MolToImage(x_0)

アルデヒドの酸素原子が水になっています!どうしてこうなった。
RDKitオンラインドキュメンテーションのChemical-reactionsによると、生成される分子はサニタイゼーションされていないので、サニタイゼーションが必要とのことです。
Chem.SanitizeMol(x_0)
# rdkit.Chem.rdmolops.SanitizeFlags.SANITIZE_NONE と返ってきた
Draw.MolToImage(x_0)

今度は酸素原子がうまく認識されています。よかった。
Fagnouの添加剤
だいたい使い方がわかってきました。ちょっと添加剤にもこだわってみましょう。
Keith Fagnouらによって見出された条件、ピバル酸の添加を行ってみます*4
こちらの論文ではピパル酸を添加することで、触媒的C-H活性化反応が加速するということを見出しています。メカニズムとして、パラジウム触媒に配位し、協奏的メタル化-脱プロトン化反応機構を加速するのではないかということが提唱されています。
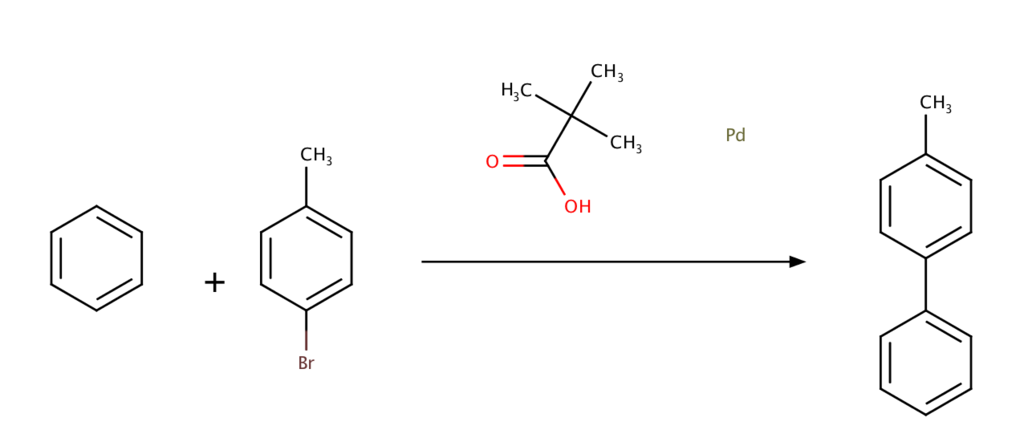
上記のReaction SMILESは以下のようになります。
C1=CC=CC=C1.CC1=CC=C(Br)C=C1>CC(C)(C)C(O)=O.[Pd]>CC1=CC=C(C=C1)C1=CC=CC=C1
手抜きして酢酸パラジウム(II)をパラジウム単体にしてしまっていますがご容赦ください。
reactant_1 = Chem.MolFromSmiles('C1=CC=CC=C1') reactant_2 = Chem.MolFromSmiles('CC1=CC=C(Br)C=C1') reaction_pattern = '[c:1].[c:2]-[Br:3] >CC(C)(C)C(O)=O.[Pd]> [c:1]-[c:2]' rxn = AllChem.ReactionFromSmarts(reaction_pattern) x = rxn.RunReactants([reactant_1, reactant_2]) len(x) # 6
・・・添加剤とか記載しなくても結果変わらなかった気がする。まあ、いいや。
ベンゼンを原料としてのC-H活性化反応なので6つの化合物が生成されました。重複を取り除きたいと思います。
手順としては、
- 空の辞書を作成
- サニタイゼーションしてからSMILESに変換(あとで描画したいのでサニタイゼーションもここで行なった)
- 辞書にSMILESをkeyとして、valueにMolオブジェクトを渡す。
これで重複した分子は、keyのSMILESが一致するため複数格納されることはなく、ユニークな分子のみとなります。
uniqps = {}
for p in x:
Chem.SanitizeMol(p[0])
smi = Chem.MolToSmiles(p[0])
uniqps[smi] = p[0]
print(uniqps)
# {'Cc1ccc(-c2ccccc2)cc1': <rdkit.Chem.rdchem.Mol object at 0x115f28138>}
分子一つだけが残りました。生成物を確認します。
Draw.MolToImage(uniqps['Cc1ccc(-c2ccccc2)cc1'])

団子・・・
Baranの溶媒
今度は溶媒にもこだわってみましょう。C-H活性化の溶媒と言えば・・・ウーロン茶ですよね!
- ウーロン茶の中でも医薬品の化学合成が可能に | Chem-Station (ケムステ)
- 【反応】Practical and innate carbon–hydrogen functionalization of heterocycles : ChemASAP
さまざまなインパクトのある全合成、反応開発を行い、近年では電気化学の合成反応への応用にも力を入れている様子のPhil S. Baranらにより2012年Natureに報告された反応です。*5

上記のReaction SMILESは以下のようになります。
CN1C=NC2=C1C(=O)N(C)C(=O)N2C.FC(F)(F)S(=O)O[Zn]OS(=O)C(F)(F)F>>CN1C(=NC2=C1C(=O)N(C)C(=O)N2C)C(F)(F)F
まずは溶媒を指定せずに行います。
reaction_pattern = '[n:1][cH1:2][n:3]>>[n:1][c:2](C(F)(F)F)[n:3]' reactant = Chem.MolFromSmiles('CN1C=NC2=C1C(=O)N(C)C(=O)N2C') rxn = AllChem.ReactionFromSmarts(reaction_pattern) x = rxn.RunReactants([reactant,]) uniqps = {} for p in x: Chem.SanitizeMol(p[0]) smi = Chem.MolToSmiles(p[0]) uniqps[smi] = p[0] print(uniqps) # {'Cc1ccc(-c2ccccc2)cc1': <rdkit.Chem.rdchem.Mol object at 0x115f28138>} Draw.MolToImage(uniqps['Cn1c(=O)c2c(nc(C(F)(F)F)n2C)n(C)c1=O'])

うまくカフェインのトリフルオロメチル化ができました。
それではウーロン茶(Oolong tea)を溶媒として反応を仕込みましょう。
reaction_pattern = '[n:1][cH1:2][n:3]>Oolong tea>[n:1][c:2](C(F)(F)F)[n:3]' rxn = AllChem.ReactionFromSmarts(reaction_pattern) x = rxn.RunReactants(reactant) --------------------------------------------------------------------------- ValueError Traceback (most recent call last) <ipython-input-150-2ccb3852d288> in <module>() ----> 1 rxn = AllChem.ReactionFromSmarts(reaction_pattern) 2 x = rxn.RunReactants(reactant) ValueError: ChemicalReactionParserException: Problems constructing agent from SMARTS: Oolong tea
怒られました。そんなSMARTSは無い!!とのこと・・・ふざけてすみません。
MacMillanによるLate-Stage Trifluoromethylation
少し真面目な話に戻して、、、
C-H活性化反応ですが、合成終盤に分子の多様性を増加させるLate-Stage Functionalizationとしての使い方、とくに医薬品低分子への適用が、その有用性を示すデモンストレーションとして行われることが多いです。*6 「創薬化学で多用される部分構造であるトリフルオロメチル基を、医薬品分子そのものに1ステップで直接導入できれば、新しいプロファイルの有用な分子をできるのではないないか?それに複数の生成物を一回で作ることができれば、有望な分子のバリエーションを増やせて良いよね!」というのが、彼らの主張です。
反応形式としては芳香環のC-H結合のトリフルオロメチル基への変換です。

光触媒としてRu(phen)3Cl2が用いられていますが、SMILESに直すと大変そうなので省いてあります。
上記のReaction SMILESは以下の通り。
FC(F)(F)S(Cl)(=O)=O.C1=CC=CC=C1>>FC(F)(F)C1=CC=CC=C1
Reaction SMARTSは [c:1][cH1:2][c:3]>>[c:1][c:2](C(F)(F)F)[c:3] としておけば良さそうです。
論文中ではLipitor(Atrovastatin)を基質として反応を行い、トリフルオロメチル化されたCF3-Lipitorの合成を行なっています。
基質のLipitorを描くのが面倒なのでSMILESをPubChemから取得します。
import pubchempy as pcp import pandas as pd Lipitor_df = pcp.get_properties('canonical_smiles', 'Lipitor', 'name', as_dataframe=True)

Lipitorのフリー体はPubChemの「CID : 60823」のようなのでこちらのSMILESを使います。
Lipitor_smi = Lipitor_df.loc[60823, 'CanonicalSMILES'] Lipitor_mol = Chem.MolFromSmiles(Lipitor_smi) Draw.MolToImage(Lipitor_mol)

Lipitorの構造は上記のようなものです。
Lipitorには3つのベンゼン環があり、反応点としては多数ありますが、MacMillanらは3つの主要なトリフルオロメチル化生成物を、1:1:1の位置異性体混合物(収率74%)で取得しています。
反応させましょう。
CF3_reaction = '[c:1][cH1:2][c:3]>>[c:1][c:2](C(F)(F)F)[c:3]' rxn = AllChem.ReactionFromSmarts(CF3_reaction) x = rxn.RunReactants([Lipitor_mol,]) # ユニークな分子を取り出す CF3_Lipitors = {} for p in x: Chem.SanitizeMol(p[0]) smi = Chem.MolToSmiles(p[0]) CF3_Lipitors[smi] = p[0] print(len(CF3_Lipitors)) # 8個の分子が生成された # 辞書をリストに変換したうえで描画 CF3_Lipitor_list = list(CF3_Lipitors.values()) Draw.MolsToGridImage(CF3_Lipitor_list, molsPerRow=4, subImgSize=(200,200))

このままではどこにCF3が入ったか分からないので、Lipitorの骨格でアラインメントを取ります。
AllChem.Compute2DCoords(Lipitor_mol) for m in CF3_Lipitor_list: AllChem.GenerateDepictionMatching2DStructure(m, Lipitor_mol)
ついでにCF3をハイライトするため、部分構造検索と一致した原子の番号を取得します。
CF3_list = [] CF3_subst = Chem.MolFromSmarts('C(F)(F)F') for mol in CF3_Lipitor_list: matched_atoms = mol.GetSubstructMatches(CF3_subst) #取得した原子番号はタプルのタプルの型となっているのでindex[0]で最初のものを取り出す CF3_list.append(matched_atoms[0])
レジェンドをつけて描画します。
#レジェンドはintではダメだったのでstringに変換 legends = [str(i) for i in range(8)] Draw.MolsToGridImage(CF3_Lipitor_list, molsPerRow=4, subImgSize=(200,200), legends=legends, highlightAtomLists=CF3_list)

以上のようになりました。整列してハイライトするととてもわかりやすくなった気がします。
上の構造のうち、実際に論文中で生成が確認された3つの分子は4番(4'-CF3-Lipitor, 単離収率22%)、5番(2-CF3-Lipitor, 単離収率25%)、7(4-CF3-Lipitor, 単離収率27%)です。
フルオロフェニルで反応が進行していないこと、アニリンでの反応位置をみると、このトリフルオロメチル化の反応の選択性は、電子豊富な環、立体的に空いている位置が優先するとなりそうです。
まとめ
以上、今回はSMILESとSMARTSの違いを勉強するということから始めて、SMILESのもう一つの拡張、Reaction SMILES/SMARTS を用いたRDKitの化学反応を使用してみました。通常、有機合成反応では高い選択性かつクリーンな反応が好まれますが、Late-Stage Functionalizationの観点からはむしろ多様な分子を作り出すため、選択性があまりでないことが重要となっています。このような反応形式を検証するにあたって、生成物としてできうる構造をルールに従って全て書き出すことができる、というのはとても便利だなという印象を受けました。反応開発や副生成物の解析(不純物のプロファイリング)といった用途にも色々と応用できそうです。
*3:反応の構造式、Reaction SMILESともにMarvinSketch 18.24.0を用いて作成
*4:Lafrance, M. and Fagnou, K. J.Am.Chem.Soc.,2006,128,16496
*6:最近では、Late-Stage Diversificationともいうのでしょうか?(合成後期多様化法)))
そこで最後に2011年David W. C. MacMillanらがNature誌に報告した光酸化還元触媒(Photoredox Catalyst)を用いたトリフルオロメチル化反応を眺めて見たいと思います。((Nature volume2012(480)224
部分構造で絞り込む話
Twitterで拝見した以下の処理の手順を参考に、指標を用いた粗い絞り込みまでを実施しました。
処理の手順
| 処理 | 意図 | |
|---|---|---|
| ① | 分子量の範囲を設定 | 粗い絞り込み |
| ② | 部分構造で絞り込み | 活性化合物を参照したLBVS(?) |
| ③ | 粗いドッキング | タンパク質との相互作用を予測するSBVS(?) |
| ④ | ファーマコフォアを指定したより詳細なドッキング | SBVS 2回目(?) |
指標による絞り込み基準
| 指標 | 分子量 | LogP | 水素結合供与体数 | 水素結合受容体数 | 回転可能結合数 | 極性表面積 |
|---|---|---|---|---|---|---|
| Rule of 5 | ≦500 | ≦5 | ≦5 | ≦10 | ||
| 基準 | >300 | >3 | >3 | >3 | >60 |
今回は指標による絞り込みの次の段階、部分構造での絞り込みを行いたいと思います。
参考にさせていただいている処理の方法では、オルト位に置換基の入ったビフェニルで絞り込んだとのことでしたが、いきなり置換基なども含めた処理を行うのは難しいので、まずはビフェニル構造での絞り込みを行いたいと思います。
活性化合物のデータセットでビフェニルを探す
そもそもなぜビフェニルなのか? 活性化合物のデータセット (共闘用シェアデータ )中の分子(マクロサイクル型を除く)が、ビフェニル構造をもつのか検証してみます。
「化学の新しいカタチ」さんのこちらの記事「 RDKitを用いた部分構造検索とMCSアルゴリズム 」 を参考にRDKitの部分構造検索メソッドを利用します。
まずは必要なものをimport・・・
from rdkit import rdBase, Chem from rdkit.Chem import AllChem, Draw
RDKitで部分構造検索を行う際、検索構造(query)はMolオブジェクトにする必要があるということなので、まずはビフェニル構造のMolオブジェクトをSMILESから作成します。
biphenyl = Chem.MolFromSmiles('c1ccc(cc1)c1ccccc1')
Draw.MolToImage(biphenyl)

きちんとビフェニル構造のMolオブジェクトが作られていそうです。こちらをテンプレートとして用い、部分構造検索を行います。
#以前取り出した鎖状分子のSDFを使用する chain_compounds_suppl = Chem.ForwardSDMolSupplier('chain_compounds.sdf') mols = [mol for mol in chain_compounds_suppl if mol is not None]
HasSubstructMatchをもちいて、ビフェニル構造を持つ分子と持たない分子を区別し、それぞれ別々のリストに入れていきます。
#ビフェニルありのリスト biphenyl_compounds = [] #ビフェニルなしのリスト w_o_biphenyl = [] for mol in mols: if mol.HasSubstructMatch(biphenyl): biphenyl_compounds.append(mol) else: w_o_biphenyl.append(mol) print('Number of compounds with biphenyl', len(biphenyl_compounds)) # Number of compounds with biphenyl 29 print('Number of compounds without biphenyl', len(w_o_biphenyl)) # Number of compounds without biphenyl 10
39個の分子のうち、29個にビフェニルが含まれました。確かに活性化合物は多くがビフェニル構造を持つようです。
最初の一つの分子を取り出して、ビフェニル構造の確認を行います。
GetSubstructMatchesでマッチした複数の原子インデックスを取得できるそうです。
test_mol = biphenyl_compounds[0] matched_atoms = test_mol.GetSubstructMatches(biphenyl) print(matched_atoms) # ((23, 17, 18, 20, 21, 22, 24, 25, 26, 27, 28, 29),)
原子インデックスのタプルのタプルとして得られました。
原子インデックスを描画してみます。
RDKitのメーリングリストのディスカッション によると、オプションを「Draw.DrawingOptions.includeAtomNumbers=True」と設定することで、表示できるようです。少し書き方を変えて以下のようにしました。
Draw.MolToImage(test_mol, includeAtomNumbers = True)

構造式の上の方に、認識された原子インデックスがあるのがわかります。
こちらをハイライトしてみます。
#タプルのタプルなのでインデックス[0]が必要 Draw.MolToImage(test_mol, highlightAtoms = matched_atoms[0])

順番に眺めます。
from ipywidgets import interact,fixed,IntSlider import ipywidgets def biphenyl_viewer(idx): mol = biphenyl_compounds[idx] matched_atoms = mol.GetSubstructMatches(biphenyl) return(Draw.MolToImage(mol, highlightAtoms = matched_atoms[0])) interact(biphenyl_viewer, idx=ipywidgets.IntSlider(min=0,max=len(biphenyl_compounds)-1, step=1));

ビフェニル構造は末端近くにあるものが多いようですが、いくつかは分子の中央部分にもあります。共通構造といっても分子の中で占める位置が異なるものもあるようです。
中央部分にビフェニルを含むものは、ビフェニルを中心とした対称構造のようにもみえるので、結合様式等(?)の知見を反映してデザインされた化合物なのかもしれません。
ライブラリ化合物で部分構造による絞り込み
それでは、ライブラリ化合物の中からビフェニル構造を持つ化合物を取り出してみたいと思います。
まずは指標を計算済みのEnamine_Premium_collectionについて、指標でのフィルタリングを実施します。
EPc_suppl = Chem.ForwardSDMolSupplier('./Enamine_Premium_collection.sdf') EPc_mols_filt = [] for x in EPc_suppl: if x is not None: mol = x MW_value = mol.GetDoubleProp('MW') MolLogP_value = mol.GetDoubleProp('MolLogP') NumHDonors_value = mol.GetDoubleProp('NumHDonors') NumHAcceptors_value = mol.GetDoubleProp('NumHAcceptors') NumRotatableBonds_value = mol.GetDoubleProp('NumRotatableBonds') TPSA_value = mol.GetDoubleProp('TPSA') #フィルタリング基準を満たす場合のみリストに追加する if MW_value > 300 and MW_value <= 500 \ and MolLogP_value > 3 and MolLogP_value <=5\ and NumHDonors_value <= 5 \ and NumHAcceptors_value > 3 and NumHAcceptors_value <= 10 \ and NumRotatableBonds_value > 3 \ and TPSA_value > 60 : filtered_mols.append(mol)
フィルタリングを満たした化合物のリストを、活性化合物群に対して行なったのと同様にビフェニルの有無で別々のリストにします。
biphenyl = Chem.MolFromSmiles('c1ccc(cc1)c1ccccc1') biphenyl_compounds = [] w_o_biphenyl = [] <200b> for mol in filtered_mols: if mol.HasSubstructMatch(biphenyl): biphenyl_compounds.append(mol) else: w_o_biphenyl.append(mol) print(len(biphenyl_compounds)) #12 print(len(w_o_biphenyl)) #4048
Enamine_Premium_collectionで条件を満たすものは12個となりました。 構造を眺めてみます。
Draw.MolsToGridImage(biphenyl_compounds, molsPerRow=4, subImgSize=(200,200))

なかなかPremium感のあるいい感じの化合物が残っている気がします。
他のライブラリに適用した結果とあわせてまとめます。
| Enamine_Premium | Enamie_Advenced | Enamine_HTS | UOS_HTS | total | |
|---|---|---|---|---|---|
| 指標でのフィルタリング後 | 4060 | 37431 | 414562 | 106948 | 563001 |
| ビフェニル有 | 12 | 329 | 3182 | 697 | 4220 (0.7%) |
| ビフェニル無 | 4048 | 37102 | 411380 | 106251 | 558781 (99.3%) |
全体を合わせて 4220 個の分子が残りました。まだまだ多いですが、かなり数を絞り込めてきているように思います。
部分構造検索をKNIMEで
Twitter話題のKNIME・・・、@sumatat_ さんのブログ非プログラマーのためのインフォマティクス 入門。(仮) のコンテンツが素晴らしいのでKNIMEブームに便乗することにしました。
(ブログタイトルがまさにこれを求めていた!!って感じです)
今回行った流れは、
- 活性化合物群をビフェニルの有無で分ける
- フィンガープリントを計算し、PCAで次元圧縮したうえでプロット
- インタラクティブに遊ぶ
です。
以下の記事を参考にさせていただきました
- @Mochimasa さんの記事 化合物をベクトルにして比較しプロットする
- @sumatat_ さんの記事 KNIMEで化合物をクラスタリング&可視化してみよう
- @iwatobipen先生の記事 Make interactive plot with Knime
- @PKさんの記事 【KNIME】CellProfilerの画像解析結果を可視化する
早速ワークフロー全体です。2通りの可視化を行うため、プロットのためののメタノード(灰色)が2つあります。

KNIMEの視認性の高さはすごいですね!何となくやっていることがわかります。 Jupyter NoteBookは3日経つと自分の操作の意味すらわからなくなります(私だけ?)が、これなら思い出しやすそうです。
一応ワークフローの説明をすると
- SDFの読み込み(SDF Reader)
- SDFの構造ををRDKitのMolオブジェクトに変換(RDKit To Molecule)
- ビフェニル構造の有無で2つに分割(RDKit Substructure Filter)
- 分割したテーブルにクラス名のカラムを追加(Constant Value Column)
- 2つのテーブルを一つに再結合(Concatenate)
- フィンガープリント(Morgan/ 1024ビット/ 半径2)を計算(RDKit Fingerprint)
- フィンガープリントのビットベクトルをPCAに渡すため整数値のカラムに分割(Expand Bit Vector)
- PCA(PCA)
- 分子の構造を描画のためSVGに変換(Renderer to Image)
- クラスに応じた色分け(Color Manager)
- プロット
プロットでは、ビフェニルの有無での色分けと、各化合物の出元(特許の出願人(applicant))での色分けの2通りを行っています。 前者ではTile View (KNIME 3.7.1 ではCard Viewの名称が変更となったそうです)、後者ではTable Viewを使用しました。
メタノードの中身はこちら


実行した結果はこのようになります。 まずはビフェニルの有無で色分けした場合・・・

ビフェニルを含まない構造(赤色)の多くは、プロットの右下に集まっています。これらは全てapplicantがAurigeneとなっています。他のapplicantとは系統の異なる化合物を報告しているようです。 また、ビフェニルを含む構造(緑色)でも左下には中国医学科学院の化合物群、中央上部にはIncyteの化合物群が集まっているように見えます。興味深いことに、Morganフィンガープリントにもとづくプロットで化合物の出所がうまく分かれているようです。
applicantによる色分けを眺めてみます。

先のプロットで想定した通り、applicantごとにプロット上でうまく分かれていそうです。
見よう見まねでも何となく使えてしまうとはKNIMEの完成度高い!
オルト位置換の認識に失敗
元々の部分構造フィルタリングの目標は、オルト位に置換基の入ったビフェニルだったので、SMILESのワイルドカード(*)をつかって、以下のコードを実施しました。
ortho_biphenyl = Chem.MolFromSmiles('c1ccc(c(*)c1)c1ccccc1')
Draw.MolToImage(ortho_biphenyl)

オルト位置換(なんでも良い)が再現できていそうです。 以下のテスト分子で試します。

test_mol.HasSubstructMatch(ortho_biphenyl)
# Flase
・・・ダメでした。置換基あるのに・・・
まとめ
今回、「ビフェニル構造を部分構造としてもつか否か」を基準としてライブラリのさらなる絞り込みを実施しました。その結果、約4000 個にまで化合物数を絞り込むことができました。また、KNIMEをもちいた可視化を実施し、フィンガープリントを持ちいることで化合物群を出所(出願者)に応じてうまく識別できる可能性があることがわかりました。
残念ながら、オルト位に置換基の入ったビフェニル、というフィルタリングはうまくできませんでした。私、応用力無さすぎ・・・
Lipinski先生に従うことにした話
以前の記事 でライブラリの指標の計算を行いました。その結果、さらに前の記事 で活性化合物群から求めた指標の閾値では、全く化合物の数が絞れないことがわかりました。
やはり、素人が付け焼き刃で基準値を考えるのは無理がある・・・ここは偉大な先人の後についていくしかない・・・ということでまずは、以下の2つのフィルタリングで化合物数が絞り込めるか試してみたいと思います。
- LipinskiのRule of 5を満たすもの
- フラグメント指標 Rule of Threeを元にしたフラグメントの削除
1.のLipinskiのRule of 5 に加えて、2.の基準を加えた理由は、
「今回の目標はフラグメントレベルではなくある程度の活性を達成できるような化合物を見出すことなので、むしろフラグメント以下の分子は除いてしまった方が良い」
ということです。
LipinskiのRule of 5の適用
Lipinskiの法則に関しては「化学の新しいカタチ」さんのこちらの記事( RDKitにおける記述子の扱い方をリピンスキーの法則を通して学ぶ )を参考にさせていただきました。
| 指標 | 分子量 | LogP | 水素結合供与体数 | 水素結合受容体数 |
|---|---|---|---|---|
| Rule of 5 | ≦500 | ≦5 | ≦5 | ≦10 |
指標の計算値のみを集めたcsvファイルを出力しておいたのでこちらを利用して、Rule of 5を満たす化合物の数を取得します。
まずはPandasのDataFrameで読み込みます。
df_EAc = pd.read_csv('./Enamine_Advanced_collection_desc.csv')
Lipinskiの法則を判断する関数を作成し、DataFrameにapplyで適用しようとしたのですが、どうしてもうまくできませんでした。また、mapでは遅いという記事を見かけたので、こちら(pandasで複数カラムを参照して高速に1行1行値を調整する際のメモ)を参考に処理を行いました。
PandasのDataFrameにapplyで関数を適用すると、Seriesとして1行ずつ処理することになるようですが、こちらのSeriesへのアクセスが遅いのが問題とのことです。解決策として、Pythonの辞書へのアクセスが高速であることを利用すれば良い、とのことでした。
# Lipinskiの判別に用いる指標を辞書として取り出す。(to_dict関数) MW_dict = df_EAc['MW'].to_dict() MolLogP_dict = df_EAc['MolLogP'].to_dict() NumHDonors_dict = df_EAc['NumHDonors'].to_dict() NumHAcceptors_dict = df_EAc['NumHAcceptors'].to_dict() # 辞書のKeyをDataFrameのindexの値としているので、indexの値を格納するカラムをDataFrameに追加する。 df_EAc['index_val'] = df_EAc.index # Lipinskiのルールを判別する関数を作成 def lipinski(index): # indexの分子の指標を取り出す MW = MW_dict[index] MolLogP = MolLogP_dict[index] NumHDonors = NumHDonors_dict[index] NumHAcceptors = NumHAcceptors_dict[index] # Lipinskiのルールに合致するならTrue、しないならFalseとする if MW <= 500 \ and MolLogP <=5\ and NumHDonors <=5 \ and NumHAcceptors <=10: return True else: return False # 上記関数を適用し、新しいカラム(Lipinski)に当てはめる df_EAc['Lipinski'] = df_EAc['index_val'].apply(lipinski)
以上で、Lipinskiのルールの判別が完了しました。あとはTrueの数を数えれば合致した分子の数がわかります。
Trueは1、Falseは0なので、そのままsum()を実行することで条件を満たす要素の数が得られるそうです。(参考記事)
EAc_Lipinski_True = df_EAc['Lipinski'].sum() print(EAc_Lipinski_True)
483858となりました。Enamine_Advanced_collectionは元々が486322だったので、2千個程度Lipinskiの法則を満たさないものが含まれているようです。
他のライブラリの計算結果と合わせると下記の通りです。
| Lipinski | Enamine_Premium (分子量300以上) |
Enamine_Advenced | Enamine_HTS | UOS_HTS |
|---|---|---|---|---|
| 総数 | 109602 | 486322 | 1921489 | 516664 |
| True | 109590 | 483858 | 1843248 | 437821 |
| False | 12 | 2464 | 78241 | 78843 |
Enamine_Premium_collectionのみFalseとなった数が12と少ないのが興味深い結果です。Premiumだけに何らかのプレミアムな基準で選ばれた優良な化合物たちなのでしょうか??
Enamine_Premium_collectionだけでも11万個の化合物があるので、こちらのみから絞り込むのでも良い気がしてきました・・・
フラグメントライクな化合物
Lipinskiの基準は、ある閾値以下の化合物を選抜するものです。前回参照したSAR News Np.19の記事には、フラグメント指標「Rule of Three」が記載されていました。
| 指標 | 分子量 | LogP | 水素結合供与体数 | 水素結合受容体数 | 回転可能結合数 | 極性表面積 |
|---|---|---|---|---|---|---|
| 基準 | ≦300 | ≦3 | ≦3 | ≦3 | ≦3 | ≦60 |
こちらを頼りにしてフラグメントライクな分子を取り除こうと思います。
以下に、一つずつの指標でフィルタリングした場合と、全てを満たす場合の数をまとめました。
| 指標 | 総分子数 | 分子量 | LogP | 水素結合供与体数 | 水素結合受容体数 | 回転可能結合数 | 極性表面積 | すべて満たすもの |
|---|---|---|---|---|---|---|---|---|
| 基準 | >300 | >3 | >3 | >3 | >3 | >60 | ||
| Enamine_Premium (分子量300以上) |
109602 | 109602 (100%) |
13327 (12%) |
49 (0.04%) |
99362 (91%) |
78633 (72%) |
84269 (77%) |
0 |
| Enamine_Advenced | 486322 | 305230 (63%) |
129679 (27%) |
902 (0.2%) |
325234 (67%) |
305416 (63%) |
277665 (57%) |
64 (0.01%) |
| Enamine_HTS | 1921489 | 1609119 (84%) |
904582 (47%) |
6847 (0.4%) |
1492627 (78%) |
1547785 (81%) |
1319649 (69%) |
87 (0.05%) |
| UOS_HTS | 516664 | 439751 (85%) |
284727 (55%) |
2461 (0.5%) |
400416 (78%) |
433231 (84%) |
351715 (68%) |
746 (0.14%) |
6つの指標すべてでフラグメント指標 Rule of Threeよりも大きいものの和集合、という基準(一番右の列)にしてしまうと、ほとんどの分子が除外されてしまいます。
特にプレミアム感のある期待のライブラリEnamine_Premium_collection がすべてなくなってしまいます。これではちょっとやりすぎ感があります。
上記の表を見ると、特に水素結合供与体数(>3)が削減率が高く、ついで LogP(>3) で削られているものも多そうです。
計算方法が間違っているといけないので、以下にコードを転記しておきます。(かなり冗長です)
#一つずつ計算する場合 df_MW_300 = df_EAc['MW'] > 300 df_MolLogP_3 = df_EAc['MolLogP'] > 3 df_NumHD_3 = df_EAc['NumHDonors'] > 3 df_NumHA_3 = df_EAc['NumHAcceptors'] > 3 df_NumRB_3 = df_EAc['NumRotatableBonds'] > 3 df_TPSA_60 = df_EAc['TPSA'] > 60 print(df_MW_300.sum()) print((df_MW_300.sum() / len(df_EAc))*100) print(df_MolLogP_3.sum()) print((df_MolLogP_3.sum() / len(df_EAc))*100) print(df_NumHD_3.sum()) print((df_NumHD_3.sum() / len(df_EAc))*100) print(df_NumHA_3.sum()) print((df_NumHA_3.sum() / len(df_EAc))*100) print(df_NumRB_3.sum()) print((df_NumRB_3.sum() / len(df_EAc))*100) print(df_TPSA_60.sum()) print((df_TPSA_60.sum() / len(df_EAc))*100) # すべての基準の和集合 df_all =((df_EAc['MW'] > 300) & \ (df_EAc['MolLogP'] > 3) & \ (df_EAc['NumHDonors'] > 3) & \ (df_EAc['NumHAcceptors'] > 3) & \ (df_EAc['NumRotatableBonds'] > 3) & \ (df_EAc['TPSA'] > 60)) print(df_all.sum()) print((df_all.sum() / len(df_EAc)) * 100)
PCAを用いた次元圧縮
フラグメントライブラリの指標をすべて逆転して用いてしまうと、化合物数を減らしすぎてしまう・・・でもどの基準を残せば良いかわからない・・・。
指標を選択する基準はないか???ということでPCAを用いた次元圧縮を行ってみたいと思います。 (たぶん使い方間違ってる)
まずはすべてのDataframeを統合します。Enamine_Advanced_collectionのMolLogPの計算値には欠損値(NaN)があるのでこれは除いておきます。
# NaNを含む列の削除 df_EAc_w_o_NaN = df_EAc.dropna() # 確認(isnullでTrueとなる数をカウントして合計が0ならNaNは無い) print(df_EAc_w_o_NaN.isnull().values.sum() == 0) # True # 4つのライブラリを統合 df_all = pd.concat([df_EPc, df_EAc_w_o_NaN, df_EHc, df_UH]) # 化合物総数 print(len(df_all)) #3034076
全部で約 300万個あります。
これにPCAを行ってみます。
from sklearn.preprocessing import StandardScaler from sklearn.decomposition import PCA import matplotlib.pyplot as plt sc = StandardScaler() # 指標の値をPandasからnumpyのndarrayに変換する (values) X = df_all[descriptors].values # 標準化 X_std = sc.fit_transform(X) # PCA(2成分) pca = PCA(n_components =2) X_pca = pca.fit_transform(X_std) #可視化 plt.figure() plt.scatter(X_pca[:, 0], X_std[:, 1]) plt.xlabel('PC1') plt.ylabel('PC2') plt.show() #次元ごとの寄与率 print(pca.explained_variance_ratio_)

コピペしてみたが、何がおきているかよくわからない!!
とりあえず第1主成分と、第2主成分をあわせても累積寄与率(?)は7割以下みたいです。
因子負荷量の計算
よくわからないが理解するのを待っていたらいつまでかかるかわからない!突き進むしかない!
各主成分に対してどの指標がどの程度相関しているかを眺めるため、因子負荷量を計算します。
import numpy as np # 因子負荷量を計算 pca_components = pca.components_*np.c_[np.sqrt(pca.explained_variance_)] # 眺めるための新しいDataFrameを作成 column_names = descriptors PCA_df = pd.DataFrame([pca_components[0], pca_components[1]], columns=column_names)
以下のようになりました。

ついでにグラフ化しておきます。(グラフでは絶対値にしています)
labels = ['MW', 'MolLogP', 'NumHD', 'NumHA', 'NumRB', 'TPSA'] # NumPyのfabsを使って絶対値に修正 PCA_df_abs = np.fabs(PCA_df) height_0=PCA_df_abs.loc[0] height_1=PCA_df_abs.loc[1] left= np.arange(len(height_1)) width=0.3 plt.bar(left, height_0, color='r', width =width) plt.bar(left+width, height_1, color='b', width =width) plt.xticks(left, labels) plt.show()

第1主成分(赤色)は、分子量(MW)、水素結合受容体数(NumHA)、回転可能結合数(NumRB)、極性表面積(TPSA)との相関が、第2主成分(青色)はLogP(MolLogP)との相関が強そうです。
水素結合供与体数(NumHD)は第1主成分よりも第2主成分との相関が強そうですが、MolLogPと比べると見劣りがします。
もう、NumHDを削ってしまっても良さそうな気がしてきました。(・・・無理やりこじつけた)
# NumHDonor以外の基準の和集合 df_all_w_o_NumHD =((df_all['MW'] > 300) & \ (df_all['MolLogP'] > 3) & \ (df_all['NumHAcceptors'] > 3) & \ (df_all['NumRotatableBonds'] > 3) & \ (df_all['TPSA'] > 60)) print(df_all_w_o_NumHD.sum()) print((df_all_w_o_NumHD.sum() / len(df_all)) * 100)
フラグメント指標から水素結合供与体数を除いた残りの5つを使って、5つ全てを満たす化合物数を求めると645704個(21%)となりました。
これならまだマシそうなので、こちらを用いたいと思います。
二つの基準を満たす分子の数
以上、見てきた内容をまとめると適用する基準は下記となります。
| 指標 | 分子量 | LogP | 水素結合供与体数 | 水素結合受容体数 | 回転可能結合数 | 極性表面積 |
|---|---|---|---|---|---|---|
| Lipinskiより | ≦500 | ≦5 | ≦5 | ≦10 | ||
| フラグメント指標より | >300 | >3 | >3 | >3 | >60 |
2つの基準を満たす分子はどの程度あるでしょうか?
df_all_both = ((df_all['MW'] > 300) & (df_all['MW'] <= 500) & \ (df_all['MolLogP'] > 3) & (df_all['MolLogP'] <= 5) &\ (df_all['NumHDonors'] <= 5) & \ (df_all['NumHAcceptors'] > 3) & (df_all['NumHAcceptors'] <= 10) &\ # 5以下になっていたので10以下に修正(01/28) (df_all['NumRotatableBonds'] > 3) & \ (df_all['TPSA'] > 60)) print(len(df_all)) # 3034076 print(df_all_both.sum()) # 563000 print((df_all_both.sum() / len(df_all)) * 100) # 18.55589642447981
元々の分子の総数 3034076、両基準を満たす分子の数563000(19%)となりました。
(最初の投稿では NumHA 10以下ではなく間違って5以下としていたため、このさらに半分の約30万個にまで減っていました。(01/28修正))
両基準で絞り込んだ化合物数は約56万個で、 もともと300万個の化合物があったので、1/5程度にまで減らすことができたことになります。
かなり無茶苦茶な話をしているので怒られてしまいそうですが、とりあえずの結果としてこの基準をもとに進めていきたいと思います。
解釈や使用方法等に誤りがたくさんあると思うのでご指摘いただければ幸いです。
SSSRを使って環状化合物と鎖状化合物を区別しようとする話
以前の記事で、創薬レイドバトルの共闘用シェアデータを2つにクラスタリングした後、環状ペプチドとそれ以外の分子の集まりとに手作業で分類しました。これでは数増えた時に大変ですし、見落としもありそうです。そこで今回はRDKit オンラインドキュメンテーションに記載のあったSSSRという考え方を頼りに同様の分類できるかどうか試してみたいと思います。
RDKitオンラインドキュメンテーションの該当箇所は下記です。
1. Ring Information
2. Ring Finding and SSSR
SSSR(smallest set of smallest rings; SSSR)については @mojaie さんのこちらの記事 化学構造情報とグラフ理論の説明が非常にわかりやいのでご参照ください。
とにかく、分子に含まれる環構造のうち「環の個数と環の大きさが最小になるような組み合わせ」だそうです。(ふわっとした理解)
環の情報がわかるなら、大員環とそれ以外の分子と区別することもできるのでは??? というのが今回の記事の目的です。
まずはデータの準備
from rdkit import rdBase, Chem from rdkit.Chem import AllChem, Draw, Descriptors, PandasTools import pandas as pd df = pd.read_csv('../pd1_inhibitor_dataset-master/PD1_inhibitor_dataset.csv') PandasTools.AddMoleculeColumnToFrame(df, 'smiles')
GetSSSR関数
RDKitのドキュメンテーションによると、SSSRに関してはGetSSSRとGetSymmSSSRの2つの関数があるそうです。
まずはGetSSSR関数を用いて見ます。こちらはSSSRの数を取得することができる関数です。
データセットの最初の分子をテスト化合物としてどのような結果が出るのか眺めて見ます。
最初の分子の構造はこのようなものです。
test_mol = df.loc[0, 'ROMol'] Draw.MolToImage(test_mol)

この分子のSSSRの数は???
test_SSSR = Chem.GetSSSR(test_mol) print(test_SSSR) # 8
8個だそうです!
・・・わからん。
SSSRの数だけではよくわかりません。
GetSymmSSSR関数
もう一方の関数、GetSymmSSSRを使うと、認識された各環構造の情報を取得することもできるそうです。
test_SymmSSSR = Chem.GetSymmSSSR(test_mol) print(type(test_SymmSSSR)) # <class 'rdkit.rdBase._vectNSt3__16vectorIiNS_9allocatorIiEEEE'> print(type(test_SSSR)) # <class 'int'>
GetSSSRで得た結果は整数値(int)ですが、GetSymmSSSRで得た結果はrdkitのオブジェクトでSSSRについてより詳細な情報をもっていそうです。
GetSymmSSSRの結果から、SSSRの数を取得するには長さを求めれば良いそうです。
print(len(test_SymmSSSR)) #8
先ほどの結果と一致しました。
最大の環構造を探す
ドキュメンテーションの説明をみる限り、GetSymmSSSRで得られるものは、認識された環構造のリストとなっており、リストの要素は各環構造に含まれる原子(atom index)のリストとなっているようなオブジェクトのようです(リストのリスト)。
8個の各環構造のサイズを求めてみます。
for i in range(len(test_SymmSSSR)): print('{}番目の環のサイズ:{}'.format(i, len(test_SymmSSSR[i]))) """ 出力結果 0番目の環のサイズ:45 1番目の環のサイズ:5 2番目の環のサイズ:5 3番目の環のサイズ:6 4番目の環のサイズ:5 5番目の環のサイズ:6 6番目の環のサイズ:5 7番目の環のサイズ:6 """
0番目が環員数 45となっています。ぼんやりと眺めていましたが環状ペプチドの長さはこんなにも長いものなんですね。具体的な数値を見るとあらためてびっくりしました。
この最大の環構造を構成する原子の番号(atom index)を取得して見ます。
largest_ring_member = list(test_SymmSSSR[0]) print(largest_ring_member) # [4, 5, 7, 8, 9, 11, 12, 13, 14, 15, 16, 18, 19, 20, 22, 23, 24, 26, 27, 28, 30, 34, 35, 37, 38, 39, 41, 42, 43, 45, 46, 47, 49, 50, 51, 53, 54, 55, 57, 58, 59, 61, 62, 63, 65]
構成要素の原子がわかったので、この情報を用いて分子のどの原子が該当の環構造に含まれるのか、ハイライトして描画して見ました。

期待通り、環状ペプチドの骨格となる構造を認識できていそうです。
環構造を順番に眺める
ipywidgetsを利用して各環構造を順番に眺めて見ます。
# 描画用 from rdkit.Chem.Draw import rdMolDraw2D from IPython.display import SVG from matplotlib.colors import ColorConverter # インタラクティブなビューワー from ipywidgets import interact,fixed,IntSlider import ipywidgets # ビューワーに与える関数を作成 def SSSR_viwer(idx): ring_member_atoms = list(test_SymmSSSR[idx]) prepro_test_mol = rdMolDraw2D.PrepareMolForDrawing(test_mol) target_ring_atoms = list(test_SymmSSSR[idx]) #デフォルトの設定は見づらいので色を変える color_dict = {} radius_dict = {} for i in target_ring_atoms: color_dict[i] = ColorConverter().to_rgb('khaki') radius_dict[i] = 0.7 #コンテナとなるオブジェクトの作成 view = rdMolDraw2D.MolDraw2DSVG(600,600) #コンテナの描画設定 option = view.drawOptions() option.circleAtoms=False option.continuousHighlights=False #最大の環構造に含まれる原子をハイライトに設定 view.DrawMolecule(prepro_test_mol, highlightAtoms=target_ring_atoms, highlightAtomColors= color_dict, highlightAtomRadii= radius_dict, highlightBonds=[]) #コンテナをファイナライズ view.FinishDrawing() #コンテナに書き込んだデータを取り出す svg = view.GetDrawingText() #データを描画 return(SVG(svg.replace('svg:,',''))) #ビューワーを実行 interact(SSSR_viwer, idx=ipywidgets.IntSlider(min=0,max=len(test_SymmSSSR)-1, step=1));

順番に表示させることでSSSRがどのような環構造を認識するのか、非常にわかりやすくなりました。
SSSRを利用して大員環を区別
大体SSSRでどのような情報が得られるのかわかったので、これを用いてマクロサイクル型の化合物とそれ以外を区別できるか検証して見たいと思います。
Wikipediaによると大員環化合物(macrocyclic compound)は「概ね10個以上の原子からなる環状構造を持つ有機化合物」ということなので、これを基準として用います。SSSRで得た環構造のうち、最大の大きさの環構造を構成する原子数が10以上の場合、大員環化合物とすることとしたいと思います。
まずは、各化合物のSSSRをもとめ、最大の環構造の情報を取得します。
関数を作成し、Molオブジェクトを含むカラムに適用し、得た値を新しいカラムへと格納します。
def largest_ring_member_count(mol): SymmSSSR = Chem.GetSymmSSSR(mol) size_list = [] # 各環の大きさを順番にリストに追加 for i in range(len(SymmSSSR)): size_list.append(len(SymmSSSR[i])) # リストの最大値をとりだす max_size = max(size_list) return(max_size) # 最大の環の情報をもつ新しいカラムを作成 df['largest_ring_size'] = df['ROMol'].map(largest_ring_member_count)
環のサイズが10以上の時はマクロサイクル型(macro)、それより小さい場合は鎖型(chain)として分類してみます。
#classifierとか一回言ってみたかった def macro_classifier(size): if size >= 10: return('macro') elif size < 10: return('chain') df['macro_chain'] = df['largest_ring_size'].map(macro_classifier)
どのような分類になったか数を確認してみます。
df['macro_chain'].value_counts() """ chain 39 macro 14 Name: macro_chain, dtype: int64 """
マクロサイクル型 14個、鎖状型39個、となりました。
前回の分類では、外れ値としたマクロサイクル型化合物1つの除いた、合計52個を、マクロサイクル型13個と鎖状型39個とに分けました。数を見る限り同様の結果が得られていそうです。
それぞれ別のDataFrameに分けたあと、構造を確認します。
マクロサイクル型・・・
df_macro = df[df['macro_chain']== 'macro'] macro_list = list(df_macro['ROMol']) def macro_viewer(idx): mol = macro_list[idx] return(display(Draw.MolToImage(mol))) interact(class_1_viewer, idx=ipywidgets.IntSlider(min=0,max=len(macro_list)-1, step=1));

鎖状型・・・
df_chain = df[df['macro_chain']== 'chain'] chain_list = list(df_chain['ROMol']) def chain_viewer(idx): mol = chain_list[idx] return(display(Draw.MolToImage(mol))) interact(chain_viewer, idx=ipywidgets.IntSlider(min=0,max=len(chain_list)-1, step=1));

しっかりと望み通りの分類ができていそうです。
まとめ
今回はSSSRを使って、大員環化合物とそれ以外の化合物を分類できるか?ということを行ってみました。
RDKitのドキュメンテーションを読んでいる時は、どのような目的で使えば良いものかさっぱりわからず、どうして The SSSR Problem という節を設けるほど解説してあるのか疑問に思っていたのですが、実際に使って見るととても使い勝手の良い重要な考え方ということがわかりました。
かなり適当に使ってしまったので、概念の理解の誤り等ございましたらご指摘いただければ幸いです。