SMARTS記法とRDKitの化学反応で遊んだ話
前回の記事で、ビフェニル部分構造によるフィルタリングを行いましたが、オルト位に置換基の入ったビフェニルについてはうまく実行することができませんでした。@iwatobipen先生、@mojaie先生にSMARTSを使えば良いというご指摘をいただきました。ありがとうございました!
まずは前回失敗した処理を再掲します。
活性化合物のデータセット (共闘用シェアデータ )から取り出した以下の分子に対し、

RDKitの部分構造検索(HasSubstructMatch)を用い、オルト位に置換基の入った構造を取り出そうとしました。
from rdkit import rdBase, Chem
from rdkit.Chem import AllChem, Draw
ortho_biphenyl = Chem.MolFromSmiles('c1ccc(c(*)c1)c1ccccc1')
Draw.MolToImage(ortho_biphenyl)

検索したい部分構造は上のものです。(* はワイルドカード)
test_mol.HasSubstructMatch(ortho_biphenyl)
# False
上記のようにFalseとなってしまうというのが前回の失敗でした。
まずは@iwatobipen先生に教えていただいたSMARTSを用いた方法を試します。
ortho_biphenyl_2 = Chem.MolFromSmarts('c1cccc([!H])c1-c1ccccc1')
Draw.MolToImage(ortho_biphenyl_2)

SMARTSから構築したMOlオブジェクトは、先に作成したものとは異なる形で認識されているようです。(見た感じ・・・)
こちらを使って部分構造の一致を検証します。
test_mol.HasSubstructMatch(ortho_biphenyl_2)
# True
True! うまく構造の一致を認識できました!
次に@mojaie先生にご指摘いただいた事項。
私が失敗したコードでは、Chem.MolFromSmiles()としてしまっているから上手くいかないのではないか、ということでした。「ワイルドカードの*は検索条件なので、本来はSMARTSの表記」のため、Chem.MolFromSmarts()とするのが適切、ということだそうです。
ortho_biphenyl_3 = Chem.MolFromSmarts('c1ccc(c(*)c1)c1ccccc1') test_mol.HasSubstructMatch(ortho_biphenyl_3) # True
True! こちらでもうまく認識できました!
先生方ありがとうございました!
今回失敗した原因は、SMILESとSMARTSの違いを理解せずに適当に使ってしまっていたことでした。
RDKitのドキュメンテーションsubstructure_searchingによると、部分構造検索のクエリ分子はSMILESからでもSMARTSからでも良いが、二つの記法で違いがあるから注意してください、といった指摘がなされていました。
そこで、両者の違いについて少し勉強してみることとしました。
SMILESとSMARTSの違い
SMARTSとは?
そもそもSMARTSって何の略なんだろう?と思っていたのですが、株式会社MOLSISさんのページにわかりやすくまとめられていたので転記させていただきます。 (同社はTwitterで何度かお見かけしたドッキングに使われていたソフト、MOEも製品として取り扱われているそうです)
SMILES(Simplified Molecular Input Line Entry System) : 分子記述言語
- 分子の二次元構造を文字列として記述
- 情報をコンパクトに保存
- 原子座標の羅列と違い、ユーザーにも理解しやすい
SMARTS(SMiles ARbitrary Target Specification): パターン記述言語
- SMILES言語を検索クエリ用に拡張したパターン記述言語
- ワイルドカードや条件指定といった構造検索条件を表記可能
つまり、SMILESを構造検索に用いるため発展させたもの、ということのようです。
Daylight社 の解説ページでSMILES / SMARTSを勉強
SMARTSの詳細については、SMILESの考案者Dr. David Weiningerの設立したDaylight C.I.S. 社のページ を参照してください、と「化学の新しいカタチ」さんのこちらの記事(SMILES記法は化学構造の線形表記法)でオススメされていたので早速眺めて見たいと思います。
部分構造検索とは?
- 分子(グラフ)から特定のパターン(サブグラフ)を見つけ出すプロセス
部分構造検索におけるSMILESの特徴
SMILESを直接拡張したSMARTSではさらに・・・・
- 論理演算子(logical operator)
- より一般的な原子、結合を表現するための特別な原子記号(atomic symbol)、結合記号(bond symbol)が使える
ようになっているそうです。
基本的にはSMILESの拡張のため、SMARTSでもSMILESの表現が使えるそうですが、SMILESは分子記述、SMARTSはパターン記述という違いから同じ文字列でも意味合いが異なってくることに注意が必要、とのこと。Daylight社のページでは以下の具体例が挙げられていました。
例1: 分子とパターン
例2: 原子を括弧 () を使わないで指定した場合
記法のルールを写経
具体的にはどのような記法になるのか、ざっくりテーブルを日本語にします。順番など少し入れ替えています。
SMILES記法同様、角括弧「」は原子の境界を、丸括弧「()」は分岐構造を示します。
原子
| 記号 | 意味 | デフォルト |
|---|---|---|
| * | ワイルドカード(どの原子でも良い) | |
| a | 芳香属性原子 | |
| A | 脂肪族原子 | |
| D<数字> | 明示的な結合の数(級数:degree)が<数字> | ちょうど1 |
| H<数字> | 結合している水素原子が<数字>個 | ちょうど1 |
| h<数字> | 暗に示された水素原子が<数字>個 | 少なくとも1 |
| R<数字> | <数字>のSSSR環構造に含まれる(ring membership) | 環の構成原子どれでも |
| r<数字> | <数字>のサイズのSSSR環構造に含まれる(ring size) | 環の構成原子どれでも |
| v<数字> | 結合次数の総数が<数字> | ちょうど1 |
| X<数字> | 結合の総数が<数字> | ちょうど1 |
| x<数字> | 環構造の結合の総数が<数字>(ring connectivity) | 少なくとも1 |
| -<数字> | マイナス <数字> の電荷 | -1 電荷、--なら-2のチャージ |
| +<数字> | プラス <数字> の形式電荷 | +1 電荷、++なら+2のチャージ |
| #<数字> | 原子番号<数字> | |
| @ | 不斉、反時計回り(@@は時計回り) | |
| <数字> | 明示的な原子量 |
結合
| 記号 | 意味 |
|---|---|
| - | 単結合(脂肪族) |
| = | 2重結合 |
| # | 3重結合 |
| : | 芳香族性結合 |
| ~ | ワイルドカード(どのような結合形式でも良い) |
| @ | 環結合ならなんでもよい |
| / | 方向性のある結合 アップ |
| |方向性性のある結合 ダウン | |
| /? | アップあるいは指定されていない |
| \? | ダウンあるいは指定されていない |
二重結合の幾何異性体は、例えば「 /C=C/ : trans型2重結合(E)」「/C=C\ : cis型2重結合(Z)」と言ったように表すことができるそうです(SMILES記法の場合)。
論理演算子
| 記号 | 意味 |
|---|---|
| ! | not |
| & | and |
| , | or |
| ; | and |
andを意味するのに「&」と「;」の2つがありますが、「&」が優先されるそうです。
論理演算子を使って原子のパターンを記述するには、角括弧「[]」内部にその原子と条件指定全体を含めることに注意が必要とのことです。
Reaction SMILES / SMARTS
SMILESには他にも拡張として、化学反応を記述できるようにしたReaction SMILESというものもあるそうです。 どのような拡張がなされているのか、「化学の新しいカタチ」さんのこちらの記事RDKitで化学反応:ケモインフォマティクスにおける反応式の扱い方 にわかりやすく記載されているのでご参照ください。
折角なので遊んで見たいと思います。取り上げる反応はC-H結合活性化!(・・・私が学生の頃とても流行ってた)
Murai reaction
まずは村井眞二先生らによる世界初の実用的触媒的C-H結合活性化型反応から*1*2

「原料・試薬 >> 生成物」とのことなのでReaction SMILESは以下のようになります。*3
O=C-C1=CC=CC=C1.CCO[Si](OCC)(OCC)C=C>>CCO[Si](CCC1=C(C=O)C=CC=C1)(OCC)OCC
二つの原料はSMILES同士が「.」でつながれています。
肝心の触媒を忘れました。
RuH2(CO)(PPh3)3を二つの「>」の間に挿入しなければいけません。PubChemによるとこの触媒のSMILESは「[C-]#[O+].C1=CC=C(C=C1)P(C2=CC=CC=C2)C3=CC=CC=C3.C1=CC=C(C=C1)P(C2=CC=CC=C2)C3=CC=CC=C3.C1=CC=C(C=C1)P(C2=CC=CC=C2)C3=CC=CC=C3.[RhH2]」だそうです。長すぎる・・・
O=C-C1=CC=CC=C1.CCO[Si](OCC)(OCC)C=C>[C-]#[O+].C1=CC=C(C=C1)P(C2=CC=CC=C2)C3=CC=CC=C3.C1=CC=C(C=C1)P(C2=CC=CC=C2)C3=CC=CC=C3.C1=CC=C(C=C1)P(C2=CC=CC=C2)C3=CC=CC=C3.[RhH2]>CCO[Si](CCC1=C(C=O)C=CC=C1)(OCC)OCC
訳が分からなくなってしまいました。
RDKitの反応に使うにはReaction SMARTSにするとのことなので触媒を省略した上で以下のように書いてみました。
[O:1]=[C:2]-[c:3][c:4].[C:5]=[C:6]-[Si:7] >> [O:1]=[C:2]-[c:3][c:4]-[C:5]=[C:6]-[Si:7]
では実際に反応を試します。反応させるMolオブジェクト、反応パターン(Reaction SMARTS)を用意した上でRunReactantsを実行します。
reactant_1 = Chem.MolFromSmiles('O=C-C1=CC=CC=C1') reactant_2 = Chem.MolFromSmiles('CCO[Si](OCC)(OCC)C=C') reaction_pattern = '[O:1]=[C:2]-[c:3][c:4].[C:5]=[C:6]-[Si:7] >> [O:1]=[C:2]-[c:3][c:4]-[C:5]=[C:6]-[Si:7]' rxn = AllChem.ReactionFromSmarts(reaction_pattern) x = rxn.RunReactants([reactant_1, reactant_2]) print(x) # ((<rdkit.Chem.rdchem.Mol object at 0x115f19fa8>,), (<rdkit.Chem.rdchem.Mol object at 0x115f19e48>,))
結果はMolオブジェクトを要素とするタプルのタプルとして与えられるようです。外側のタプルはとりうる反応点の組み合わせに相当し、内部のタプルは一つの反応点の組み合わせあたりに複数の生成物ができる場合(加水分解など)に生成物を格納するためにあるようです。
今回は同一の分子が2つ生成されていました。一方を取り出して見ます。
x_0 = x[0][0] Draw.MolToImage(x_0)

アルデヒドの酸素原子が水になっています!どうしてこうなった。
RDKitオンラインドキュメンテーションのChemical-reactionsによると、生成される分子はサニタイゼーションされていないので、サニタイゼーションが必要とのことです。
Chem.SanitizeMol(x_0)
# rdkit.Chem.rdmolops.SanitizeFlags.SANITIZE_NONE と返ってきた
Draw.MolToImage(x_0)

今度は酸素原子がうまく認識されています。よかった。
Fagnouの添加剤
だいたい使い方がわかってきました。ちょっと添加剤にもこだわってみましょう。
Keith Fagnouらによって見出された条件、ピバル酸の添加を行ってみます*4
こちらの論文ではピパル酸を添加することで、触媒的C-H活性化反応が加速するということを見出しています。メカニズムとして、パラジウム触媒に配位し、協奏的メタル化-脱プロトン化反応機構を加速するのではないかということが提唱されています。
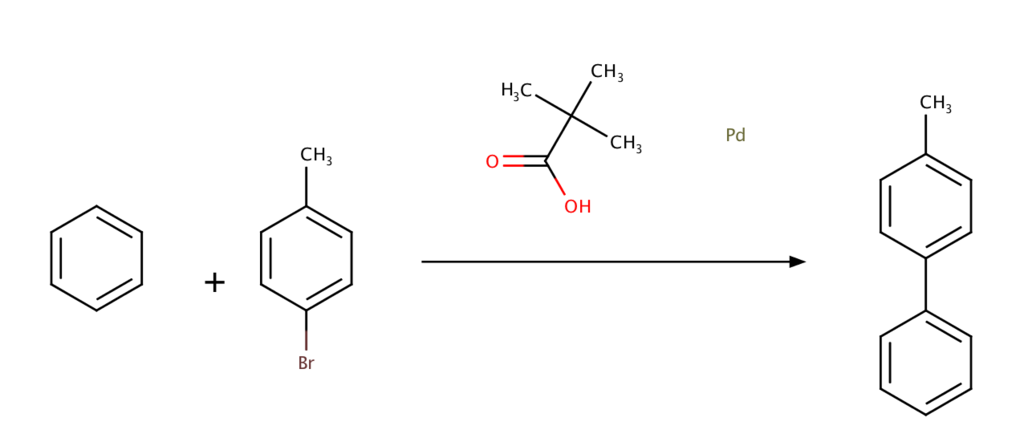
上記のReaction SMILESは以下のようになります。
C1=CC=CC=C1.CC1=CC=C(Br)C=C1>CC(C)(C)C(O)=O.[Pd]>CC1=CC=C(C=C1)C1=CC=CC=C1
手抜きして酢酸パラジウム(II)をパラジウム単体にしてしまっていますがご容赦ください。
reactant_1 = Chem.MolFromSmiles('C1=CC=CC=C1') reactant_2 = Chem.MolFromSmiles('CC1=CC=C(Br)C=C1') reaction_pattern = '[c:1].[c:2]-[Br:3] >CC(C)(C)C(O)=O.[Pd]> [c:1]-[c:2]' rxn = AllChem.ReactionFromSmarts(reaction_pattern) x = rxn.RunReactants([reactant_1, reactant_2]) len(x) # 6
・・・添加剤とか記載しなくても結果変わらなかった気がする。まあ、いいや。
ベンゼンを原料としてのC-H活性化反応なので6つの化合物が生成されました。重複を取り除きたいと思います。
手順としては、
- 空の辞書を作成
- サニタイゼーションしてからSMILESに変換(あとで描画したいのでサニタイゼーションもここで行なった)
- 辞書にSMILESをkeyとして、valueにMolオブジェクトを渡す。
これで重複した分子は、keyのSMILESが一致するため複数格納されることはなく、ユニークな分子のみとなります。
uniqps = {}
for p in x:
Chem.SanitizeMol(p[0])
smi = Chem.MolToSmiles(p[0])
uniqps[smi] = p[0]
print(uniqps)
# {'Cc1ccc(-c2ccccc2)cc1': <rdkit.Chem.rdchem.Mol object at 0x115f28138>}
分子一つだけが残りました。生成物を確認します。
Draw.MolToImage(uniqps['Cc1ccc(-c2ccccc2)cc1'])

団子・・・
Baranの溶媒
今度は溶媒にもこだわってみましょう。C-H活性化の溶媒と言えば・・・ウーロン茶ですよね!
- ウーロン茶の中でも医薬品の化学合成が可能に | Chem-Station (ケムステ)
- 【反応】Practical and innate carbon–hydrogen functionalization of heterocycles : ChemASAP
さまざまなインパクトのある全合成、反応開発を行い、近年では電気化学の合成反応への応用にも力を入れている様子のPhil S. Baranらにより2012年Natureに報告された反応です。*5

上記のReaction SMILESは以下のようになります。
CN1C=NC2=C1C(=O)N(C)C(=O)N2C.FC(F)(F)S(=O)O[Zn]OS(=O)C(F)(F)F>>CN1C(=NC2=C1C(=O)N(C)C(=O)N2C)C(F)(F)F
まずは溶媒を指定せずに行います。
reaction_pattern = '[n:1][cH1:2][n:3]>>[n:1][c:2](C(F)(F)F)[n:3]' reactant = Chem.MolFromSmiles('CN1C=NC2=C1C(=O)N(C)C(=O)N2C') rxn = AllChem.ReactionFromSmarts(reaction_pattern) x = rxn.RunReactants([reactant,]) uniqps = {} for p in x: Chem.SanitizeMol(p[0]) smi = Chem.MolToSmiles(p[0]) uniqps[smi] = p[0] print(uniqps) # {'Cc1ccc(-c2ccccc2)cc1': <rdkit.Chem.rdchem.Mol object at 0x115f28138>} Draw.MolToImage(uniqps['Cn1c(=O)c2c(nc(C(F)(F)F)n2C)n(C)c1=O'])

うまくカフェインのトリフルオロメチル化ができました。
それではウーロン茶(Oolong tea)を溶媒として反応を仕込みましょう。
reaction_pattern = '[n:1][cH1:2][n:3]>Oolong tea>[n:1][c:2](C(F)(F)F)[n:3]' rxn = AllChem.ReactionFromSmarts(reaction_pattern) x = rxn.RunReactants(reactant) --------------------------------------------------------------------------- ValueError Traceback (most recent call last) <ipython-input-150-2ccb3852d288> in <module>() ----> 1 rxn = AllChem.ReactionFromSmarts(reaction_pattern) 2 x = rxn.RunReactants(reactant) ValueError: ChemicalReactionParserException: Problems constructing agent from SMARTS: Oolong tea
怒られました。そんなSMARTSは無い!!とのこと・・・ふざけてすみません。
MacMillanによるLate-Stage Trifluoromethylation
少し真面目な話に戻して、、、
C-H活性化反応ですが、合成終盤に分子の多様性を増加させるLate-Stage Functionalizationとしての使い方、とくに医薬品低分子への適用が、その有用性を示すデモンストレーションとして行われることが多いです。*6 「創薬化学で多用される部分構造であるトリフルオロメチル基を、医薬品分子そのものに1ステップで直接導入できれば、新しいプロファイルの有用な分子をできるのではないないか?それに複数の生成物を一回で作ることができれば、有望な分子のバリエーションを増やせて良いよね!」というのが、彼らの主張です。
反応形式としては芳香環のC-H結合のトリフルオロメチル基への変換です。

光触媒としてRu(phen)3Cl2が用いられていますが、SMILESに直すと大変そうなので省いてあります。
上記のReaction SMILESは以下の通り。
FC(F)(F)S(Cl)(=O)=O.C1=CC=CC=C1>>FC(F)(F)C1=CC=CC=C1
Reaction SMARTSは [c:1][cH1:2][c:3]>>[c:1][c:2](C(F)(F)F)[c:3] としておけば良さそうです。
論文中ではLipitor(Atrovastatin)を基質として反応を行い、トリフルオロメチル化されたCF3-Lipitorの合成を行なっています。
基質のLipitorを描くのが面倒なのでSMILESをPubChemから取得します。
import pubchempy as pcp import pandas as pd Lipitor_df = pcp.get_properties('canonical_smiles', 'Lipitor', 'name', as_dataframe=True)

Lipitorのフリー体はPubChemの「CID : 60823」のようなのでこちらのSMILESを使います。
Lipitor_smi = Lipitor_df.loc[60823, 'CanonicalSMILES'] Lipitor_mol = Chem.MolFromSmiles(Lipitor_smi) Draw.MolToImage(Lipitor_mol)

Lipitorの構造は上記のようなものです。
Lipitorには3つのベンゼン環があり、反応点としては多数ありますが、MacMillanらは3つの主要なトリフルオロメチル化生成物を、1:1:1の位置異性体混合物(収率74%)で取得しています。
反応させましょう。
CF3_reaction = '[c:1][cH1:2][c:3]>>[c:1][c:2](C(F)(F)F)[c:3]' rxn = AllChem.ReactionFromSmarts(CF3_reaction) x = rxn.RunReactants([Lipitor_mol,]) # ユニークな分子を取り出す CF3_Lipitors = {} for p in x: Chem.SanitizeMol(p[0]) smi = Chem.MolToSmiles(p[0]) CF3_Lipitors[smi] = p[0] print(len(CF3_Lipitors)) # 8個の分子が生成された # 辞書をリストに変換したうえで描画 CF3_Lipitor_list = list(CF3_Lipitors.values()) Draw.MolsToGridImage(CF3_Lipitor_list, molsPerRow=4, subImgSize=(200,200))

このままではどこにCF3が入ったか分からないので、Lipitorの骨格でアラインメントを取ります。
AllChem.Compute2DCoords(Lipitor_mol) for m in CF3_Lipitor_list: AllChem.GenerateDepictionMatching2DStructure(m, Lipitor_mol)
ついでにCF3をハイライトするため、部分構造検索と一致した原子の番号を取得します。
CF3_list = [] CF3_subst = Chem.MolFromSmarts('C(F)(F)F') for mol in CF3_Lipitor_list: matched_atoms = mol.GetSubstructMatches(CF3_subst) #取得した原子番号はタプルのタプルの型となっているのでindex[0]で最初のものを取り出す CF3_list.append(matched_atoms[0])
レジェンドをつけて描画します。
#レジェンドはintではダメだったのでstringに変換 legends = [str(i) for i in range(8)] Draw.MolsToGridImage(CF3_Lipitor_list, molsPerRow=4, subImgSize=(200,200), legends=legends, highlightAtomLists=CF3_list)

以上のようになりました。整列してハイライトするととてもわかりやすくなった気がします。
上の構造のうち、実際に論文中で生成が確認された3つの分子は4番(4'-CF3-Lipitor, 単離収率22%)、5番(2-CF3-Lipitor, 単離収率25%)、7(4-CF3-Lipitor, 単離収率27%)です。
フルオロフェニルで反応が進行していないこと、アニリンでの反応位置をみると、このトリフルオロメチル化の反応の選択性は、電子豊富な環、立体的に空いている位置が優先するとなりそうです。
まとめ
以上、今回はSMILESとSMARTSの違いを勉強するということから始めて、SMILESのもう一つの拡張、Reaction SMILES/SMARTS を用いたRDKitの化学反応を使用してみました。通常、有機合成反応では高い選択性かつクリーンな反応が好まれますが、Late-Stage Functionalizationの観点からはむしろ多様な分子を作り出すため、選択性があまりでないことが重要となっています。このような反応形式を検証するにあたって、生成物としてできうる構造をルールに従って全て書き出すことができる、というのはとても便利だなという印象を受けました。反応開発や副生成物の解析(不純物のプロファイリング)といった用途にも色々と応用できそうです。
*3:反応の構造式、Reaction SMILESともにMarvinSketch 18.24.0を用いて作成
*4:Lafrance, M. and Fagnou, K. J.Am.Chem.Soc.,2006,128,16496
*6:最近では、Late-Stage Diversificationともいうのでしょうか?(合成後期多様化法)))
そこで最後に2011年David W. C. MacMillanらがNature誌に報告した光酸化還元触媒(Photoredox Catalyst)を用いたトリフルオロメチル化反応を眺めて見たいと思います。((Nature volume2012(480)224
部分構造で絞り込む話
Twitterで拝見した以下の処理の手順を参考に、指標を用いた粗い絞り込みまでを実施しました。
処理の手順
| 処理 | 意図 | |
|---|---|---|
| ① | 分子量の範囲を設定 | 粗い絞り込み |
| ② | 部分構造で絞り込み | 活性化合物を参照したLBVS(?) |
| ③ | 粗いドッキング | タンパク質との相互作用を予測するSBVS(?) |
| ④ | ファーマコフォアを指定したより詳細なドッキング | SBVS 2回目(?) |
指標による絞り込み基準
| 指標 | 分子量 | LogP | 水素結合供与体数 | 水素結合受容体数 | 回転可能結合数 | 極性表面積 |
|---|---|---|---|---|---|---|
| Rule of 5 | ≦500 | ≦5 | ≦5 | ≦10 | ||
| 基準 | >300 | >3 | >3 | >3 | >60 |
今回は指標による絞り込みの次の段階、部分構造での絞り込みを行いたいと思います。
参考にさせていただいている処理の方法では、オルト位に置換基の入ったビフェニルで絞り込んだとのことでしたが、いきなり置換基なども含めた処理を行うのは難しいので、まずはビフェニル構造での絞り込みを行いたいと思います。
活性化合物のデータセットでビフェニルを探す
そもそもなぜビフェニルなのか? 活性化合物のデータセット (共闘用シェアデータ )中の分子(マクロサイクル型を除く)が、ビフェニル構造をもつのか検証してみます。
「化学の新しいカタチ」さんのこちらの記事「 RDKitを用いた部分構造検索とMCSアルゴリズム 」 を参考にRDKitの部分構造検索メソッドを利用します。
まずは必要なものをimport・・・
from rdkit import rdBase, Chem from rdkit.Chem import AllChem, Draw
RDKitで部分構造検索を行う際、検索構造(query)はMolオブジェクトにする必要があるということなので、まずはビフェニル構造のMolオブジェクトをSMILESから作成します。
biphenyl = Chem.MolFromSmiles('c1ccc(cc1)c1ccccc1')
Draw.MolToImage(biphenyl)

きちんとビフェニル構造のMolオブジェクトが作られていそうです。こちらをテンプレートとして用い、部分構造検索を行います。
#以前取り出した鎖状分子のSDFを使用する chain_compounds_suppl = Chem.ForwardSDMolSupplier('chain_compounds.sdf') mols = [mol for mol in chain_compounds_suppl if mol is not None]
HasSubstructMatchをもちいて、ビフェニル構造を持つ分子と持たない分子を区別し、それぞれ別々のリストに入れていきます。
#ビフェニルありのリスト biphenyl_compounds = [] #ビフェニルなしのリスト w_o_biphenyl = [] for mol in mols: if mol.HasSubstructMatch(biphenyl): biphenyl_compounds.append(mol) else: w_o_biphenyl.append(mol) print('Number of compounds with biphenyl', len(biphenyl_compounds)) # Number of compounds with biphenyl 29 print('Number of compounds without biphenyl', len(w_o_biphenyl)) # Number of compounds without biphenyl 10
39個の分子のうち、29個にビフェニルが含まれました。確かに活性化合物は多くがビフェニル構造を持つようです。
最初の一つの分子を取り出して、ビフェニル構造の確認を行います。
GetSubstructMatchesでマッチした複数の原子インデックスを取得できるそうです。
test_mol = biphenyl_compounds[0] matched_atoms = test_mol.GetSubstructMatches(biphenyl) print(matched_atoms) # ((23, 17, 18, 20, 21, 22, 24, 25, 26, 27, 28, 29),)
原子インデックスのタプルのタプルとして得られました。
原子インデックスを描画してみます。
RDKitのメーリングリストのディスカッション によると、オプションを「Draw.DrawingOptions.includeAtomNumbers=True」と設定することで、表示できるようです。少し書き方を変えて以下のようにしました。
Draw.MolToImage(test_mol, includeAtomNumbers = True)

構造式の上の方に、認識された原子インデックスがあるのがわかります。
こちらをハイライトしてみます。
#タプルのタプルなのでインデックス[0]が必要 Draw.MolToImage(test_mol, highlightAtoms = matched_atoms[0])

順番に眺めます。
from ipywidgets import interact,fixed,IntSlider import ipywidgets def biphenyl_viewer(idx): mol = biphenyl_compounds[idx] matched_atoms = mol.GetSubstructMatches(biphenyl) return(Draw.MolToImage(mol, highlightAtoms = matched_atoms[0])) interact(biphenyl_viewer, idx=ipywidgets.IntSlider(min=0,max=len(biphenyl_compounds)-1, step=1));

ビフェニル構造は末端近くにあるものが多いようですが、いくつかは分子の中央部分にもあります。共通構造といっても分子の中で占める位置が異なるものもあるようです。
中央部分にビフェニルを含むものは、ビフェニルを中心とした対称構造のようにもみえるので、結合様式等(?)の知見を反映してデザインされた化合物なのかもしれません。
ライブラリ化合物で部分構造による絞り込み
それでは、ライブラリ化合物の中からビフェニル構造を持つ化合物を取り出してみたいと思います。
まずは指標を計算済みのEnamine_Premium_collectionについて、指標でのフィルタリングを実施します。
EPc_suppl = Chem.ForwardSDMolSupplier('./Enamine_Premium_collection.sdf') EPc_mols_filt = [] for x in EPc_suppl: if x is not None: mol = x MW_value = mol.GetDoubleProp('MW') MolLogP_value = mol.GetDoubleProp('MolLogP') NumHDonors_value = mol.GetDoubleProp('NumHDonors') NumHAcceptors_value = mol.GetDoubleProp('NumHAcceptors') NumRotatableBonds_value = mol.GetDoubleProp('NumRotatableBonds') TPSA_value = mol.GetDoubleProp('TPSA') #フィルタリング基準を満たす場合のみリストに追加する if MW_value > 300 and MW_value <= 500 \ and MolLogP_value > 3 and MolLogP_value <=5\ and NumHDonors_value <= 5 \ and NumHAcceptors_value > 3 and NumHAcceptors_value <= 10 \ and NumRotatableBonds_value > 3 \ and TPSA_value > 60 : filtered_mols.append(mol)
フィルタリングを満たした化合物のリストを、活性化合物群に対して行なったのと同様にビフェニルの有無で別々のリストにします。
biphenyl = Chem.MolFromSmiles('c1ccc(cc1)c1ccccc1') biphenyl_compounds = [] w_o_biphenyl = [] <200b> for mol in filtered_mols: if mol.HasSubstructMatch(biphenyl): biphenyl_compounds.append(mol) else: w_o_biphenyl.append(mol) print(len(biphenyl_compounds)) #12 print(len(w_o_biphenyl)) #4048
Enamine_Premium_collectionで条件を満たすものは12個となりました。 構造を眺めてみます。
Draw.MolsToGridImage(biphenyl_compounds, molsPerRow=4, subImgSize=(200,200))

なかなかPremium感のあるいい感じの化合物が残っている気がします。
他のライブラリに適用した結果とあわせてまとめます。
| Enamine_Premium | Enamie_Advenced | Enamine_HTS | UOS_HTS | total | |
|---|---|---|---|---|---|
| 指標でのフィルタリング後 | 4060 | 37431 | 414562 | 106948 | 563001 |
| ビフェニル有 | 12 | 329 | 3182 | 697 | 4220 (0.7%) |
| ビフェニル無 | 4048 | 37102 | 411380 | 106251 | 558781 (99.3%) |
全体を合わせて 4220 個の分子が残りました。まだまだ多いですが、かなり数を絞り込めてきているように思います。
部分構造検索をKNIMEで
Twitter話題のKNIME・・・、@sumatat_ さんのブログ非プログラマーのためのインフォマティクス 入門。(仮) のコンテンツが素晴らしいのでKNIMEブームに便乗することにしました。
(ブログタイトルがまさにこれを求めていた!!って感じです)
今回行った流れは、
- 活性化合物群をビフェニルの有無で分ける
- フィンガープリントを計算し、PCAで次元圧縮したうえでプロット
- インタラクティブに遊ぶ
です。
以下の記事を参考にさせていただきました
- @Mochimasa さんの記事 化合物をベクトルにして比較しプロットする
- @sumatat_ さんの記事 KNIMEで化合物をクラスタリング&可視化してみよう
- @iwatobipen先生の記事 Make interactive plot with Knime
- @PKさんの記事 【KNIME】CellProfilerの画像解析結果を可視化する
早速ワークフロー全体です。2通りの可視化を行うため、プロットのためののメタノード(灰色)が2つあります。

KNIMEの視認性の高さはすごいですね!何となくやっていることがわかります。 Jupyter NoteBookは3日経つと自分の操作の意味すらわからなくなります(私だけ?)が、これなら思い出しやすそうです。
一応ワークフローの説明をすると
- SDFの読み込み(SDF Reader)
- SDFの構造ををRDKitのMolオブジェクトに変換(RDKit To Molecule)
- ビフェニル構造の有無で2つに分割(RDKit Substructure Filter)
- 分割したテーブルにクラス名のカラムを追加(Constant Value Column)
- 2つのテーブルを一つに再結合(Concatenate)
- フィンガープリント(Morgan/ 1024ビット/ 半径2)を計算(RDKit Fingerprint)
- フィンガープリントのビットベクトルをPCAに渡すため整数値のカラムに分割(Expand Bit Vector)
- PCA(PCA)
- 分子の構造を描画のためSVGに変換(Renderer to Image)
- クラスに応じた色分け(Color Manager)
- プロット
プロットでは、ビフェニルの有無での色分けと、各化合物の出元(特許の出願人(applicant))での色分けの2通りを行っています。 前者ではTile View (KNIME 3.7.1 ではCard Viewの名称が変更となったそうです)、後者ではTable Viewを使用しました。
メタノードの中身はこちら


実行した結果はこのようになります。 まずはビフェニルの有無で色分けした場合・・・

ビフェニルを含まない構造(赤色)の多くは、プロットの右下に集まっています。これらは全てapplicantがAurigeneとなっています。他のapplicantとは系統の異なる化合物を報告しているようです。 また、ビフェニルを含む構造(緑色)でも左下には中国医学科学院の化合物群、中央上部にはIncyteの化合物群が集まっているように見えます。興味深いことに、Morganフィンガープリントにもとづくプロットで化合物の出所がうまく分かれているようです。
applicantによる色分けを眺めてみます。

先のプロットで想定した通り、applicantごとにプロット上でうまく分かれていそうです。
見よう見まねでも何となく使えてしまうとはKNIMEの完成度高い!
オルト位置換の認識に失敗
元々の部分構造フィルタリングの目標は、オルト位に置換基の入ったビフェニルだったので、SMILESのワイルドカード(*)をつかって、以下のコードを実施しました。
ortho_biphenyl = Chem.MolFromSmiles('c1ccc(c(*)c1)c1ccccc1')
Draw.MolToImage(ortho_biphenyl)

オルト位置換(なんでも良い)が再現できていそうです。 以下のテスト分子で試します。

test_mol.HasSubstructMatch(ortho_biphenyl)
# Flase
・・・ダメでした。置換基あるのに・・・
まとめ
今回、「ビフェニル構造を部分構造としてもつか否か」を基準としてライブラリのさらなる絞り込みを実施しました。その結果、約4000 個にまで化合物数を絞り込むことができました。また、KNIMEをもちいた可視化を実施し、フィンガープリントを持ちいることで化合物群を出所(出願者)に応じてうまく識別できる可能性があることがわかりました。
残念ながら、オルト位に置換基の入ったビフェニル、というフィルタリングはうまくできませんでした。私、応用力無さすぎ・・・
Lipinski先生に従うことにした話
以前の記事 でライブラリの指標の計算を行いました。その結果、さらに前の記事 で活性化合物群から求めた指標の閾値では、全く化合物の数が絞れないことがわかりました。
やはり、素人が付け焼き刃で基準値を考えるのは無理がある・・・ここは偉大な先人の後についていくしかない・・・ということでまずは、以下の2つのフィルタリングで化合物数が絞り込めるか試してみたいと思います。
- LipinskiのRule of 5を満たすもの
- フラグメント指標 Rule of Threeを元にしたフラグメントの削除
1.のLipinskiのRule of 5 に加えて、2.の基準を加えた理由は、
「今回の目標はフラグメントレベルではなくある程度の活性を達成できるような化合物を見出すことなので、むしろフラグメント以下の分子は除いてしまった方が良い」
ということです。
LipinskiのRule of 5の適用
Lipinskiの法則に関しては「化学の新しいカタチ」さんのこちらの記事( RDKitにおける記述子の扱い方をリピンスキーの法則を通して学ぶ )を参考にさせていただきました。
| 指標 | 分子量 | LogP | 水素結合供与体数 | 水素結合受容体数 |
|---|---|---|---|---|
| Rule of 5 | ≦500 | ≦5 | ≦5 | ≦10 |
指標の計算値のみを集めたcsvファイルを出力しておいたのでこちらを利用して、Rule of 5を満たす化合物の数を取得します。
まずはPandasのDataFrameで読み込みます。
df_EAc = pd.read_csv('./Enamine_Advanced_collection_desc.csv')
Lipinskiの法則を判断する関数を作成し、DataFrameにapplyで適用しようとしたのですが、どうしてもうまくできませんでした。また、mapでは遅いという記事を見かけたので、こちら(pandasで複数カラムを参照して高速に1行1行値を調整する際のメモ)を参考に処理を行いました。
PandasのDataFrameにapplyで関数を適用すると、Seriesとして1行ずつ処理することになるようですが、こちらのSeriesへのアクセスが遅いのが問題とのことです。解決策として、Pythonの辞書へのアクセスが高速であることを利用すれば良い、とのことでした。
# Lipinskiの判別に用いる指標を辞書として取り出す。(to_dict関数) MW_dict = df_EAc['MW'].to_dict() MolLogP_dict = df_EAc['MolLogP'].to_dict() NumHDonors_dict = df_EAc['NumHDonors'].to_dict() NumHAcceptors_dict = df_EAc['NumHAcceptors'].to_dict() # 辞書のKeyをDataFrameのindexの値としているので、indexの値を格納するカラムをDataFrameに追加する。 df_EAc['index_val'] = df_EAc.index # Lipinskiのルールを判別する関数を作成 def lipinski(index): # indexの分子の指標を取り出す MW = MW_dict[index] MolLogP = MolLogP_dict[index] NumHDonors = NumHDonors_dict[index] NumHAcceptors = NumHAcceptors_dict[index] # Lipinskiのルールに合致するならTrue、しないならFalseとする if MW <= 500 \ and MolLogP <=5\ and NumHDonors <=5 \ and NumHAcceptors <=10: return True else: return False # 上記関数を適用し、新しいカラム(Lipinski)に当てはめる df_EAc['Lipinski'] = df_EAc['index_val'].apply(lipinski)
以上で、Lipinskiのルールの判別が完了しました。あとはTrueの数を数えれば合致した分子の数がわかります。
Trueは1、Falseは0なので、そのままsum()を実行することで条件を満たす要素の数が得られるそうです。(参考記事)
EAc_Lipinski_True = df_EAc['Lipinski'].sum() print(EAc_Lipinski_True)
483858となりました。Enamine_Advanced_collectionは元々が486322だったので、2千個程度Lipinskiの法則を満たさないものが含まれているようです。
他のライブラリの計算結果と合わせると下記の通りです。
| Lipinski | Enamine_Premium (分子量300以上) |
Enamine_Advenced | Enamine_HTS | UOS_HTS |
|---|---|---|---|---|
| 総数 | 109602 | 486322 | 1921489 | 516664 |
| True | 109590 | 483858 | 1843248 | 437821 |
| False | 12 | 2464 | 78241 | 78843 |
Enamine_Premium_collectionのみFalseとなった数が12と少ないのが興味深い結果です。Premiumだけに何らかのプレミアムな基準で選ばれた優良な化合物たちなのでしょうか??
Enamine_Premium_collectionだけでも11万個の化合物があるので、こちらのみから絞り込むのでも良い気がしてきました・・・
フラグメントライクな化合物
Lipinskiの基準は、ある閾値以下の化合物を選抜するものです。前回参照したSAR News Np.19の記事には、フラグメント指標「Rule of Three」が記載されていました。
| 指標 | 分子量 | LogP | 水素結合供与体数 | 水素結合受容体数 | 回転可能結合数 | 極性表面積 |
|---|---|---|---|---|---|---|
| 基準 | ≦300 | ≦3 | ≦3 | ≦3 | ≦3 | ≦60 |
こちらを頼りにしてフラグメントライクな分子を取り除こうと思います。
以下に、一つずつの指標でフィルタリングした場合と、全てを満たす場合の数をまとめました。
| 指標 | 総分子数 | 分子量 | LogP | 水素結合供与体数 | 水素結合受容体数 | 回転可能結合数 | 極性表面積 | すべて満たすもの |
|---|---|---|---|---|---|---|---|---|
| 基準 | >300 | >3 | >3 | >3 | >3 | >60 | ||
| Enamine_Premium (分子量300以上) |
109602 | 109602 (100%) |
13327 (12%) |
49 (0.04%) |
99362 (91%) |
78633 (72%) |
84269 (77%) |
0 |
| Enamine_Advenced | 486322 | 305230 (63%) |
129679 (27%) |
902 (0.2%) |
325234 (67%) |
305416 (63%) |
277665 (57%) |
64 (0.01%) |
| Enamine_HTS | 1921489 | 1609119 (84%) |
904582 (47%) |
6847 (0.4%) |
1492627 (78%) |
1547785 (81%) |
1319649 (69%) |
87 (0.05%) |
| UOS_HTS | 516664 | 439751 (85%) |
284727 (55%) |
2461 (0.5%) |
400416 (78%) |
433231 (84%) |
351715 (68%) |
746 (0.14%) |
6つの指標すべてでフラグメント指標 Rule of Threeよりも大きいものの和集合、という基準(一番右の列)にしてしまうと、ほとんどの分子が除外されてしまいます。
特にプレミアム感のある期待のライブラリEnamine_Premium_collection がすべてなくなってしまいます。これではちょっとやりすぎ感があります。
上記の表を見ると、特に水素結合供与体数(>3)が削減率が高く、ついで LogP(>3) で削られているものも多そうです。
計算方法が間違っているといけないので、以下にコードを転記しておきます。(かなり冗長です)
#一つずつ計算する場合 df_MW_300 = df_EAc['MW'] > 300 df_MolLogP_3 = df_EAc['MolLogP'] > 3 df_NumHD_3 = df_EAc['NumHDonors'] > 3 df_NumHA_3 = df_EAc['NumHAcceptors'] > 3 df_NumRB_3 = df_EAc['NumRotatableBonds'] > 3 df_TPSA_60 = df_EAc['TPSA'] > 60 print(df_MW_300.sum()) print((df_MW_300.sum() / len(df_EAc))*100) print(df_MolLogP_3.sum()) print((df_MolLogP_3.sum() / len(df_EAc))*100) print(df_NumHD_3.sum()) print((df_NumHD_3.sum() / len(df_EAc))*100) print(df_NumHA_3.sum()) print((df_NumHA_3.sum() / len(df_EAc))*100) print(df_NumRB_3.sum()) print((df_NumRB_3.sum() / len(df_EAc))*100) print(df_TPSA_60.sum()) print((df_TPSA_60.sum() / len(df_EAc))*100) # すべての基準の和集合 df_all =((df_EAc['MW'] > 300) & \ (df_EAc['MolLogP'] > 3) & \ (df_EAc['NumHDonors'] > 3) & \ (df_EAc['NumHAcceptors'] > 3) & \ (df_EAc['NumRotatableBonds'] > 3) & \ (df_EAc['TPSA'] > 60)) print(df_all.sum()) print((df_all.sum() / len(df_EAc)) * 100)
PCAを用いた次元圧縮
フラグメントライブラリの指標をすべて逆転して用いてしまうと、化合物数を減らしすぎてしまう・・・でもどの基準を残せば良いかわからない・・・。
指標を選択する基準はないか???ということでPCAを用いた次元圧縮を行ってみたいと思います。 (たぶん使い方間違ってる)
まずはすべてのDataframeを統合します。Enamine_Advanced_collectionのMolLogPの計算値には欠損値(NaN)があるのでこれは除いておきます。
# NaNを含む列の削除 df_EAc_w_o_NaN = df_EAc.dropna() # 確認(isnullでTrueとなる数をカウントして合計が0ならNaNは無い) print(df_EAc_w_o_NaN.isnull().values.sum() == 0) # True # 4つのライブラリを統合 df_all = pd.concat([df_EPc, df_EAc_w_o_NaN, df_EHc, df_UH]) # 化合物総数 print(len(df_all)) #3034076
全部で約 300万個あります。
これにPCAを行ってみます。
from sklearn.preprocessing import StandardScaler from sklearn.decomposition import PCA import matplotlib.pyplot as plt sc = StandardScaler() # 指標の値をPandasからnumpyのndarrayに変換する (values) X = df_all[descriptors].values # 標準化 X_std = sc.fit_transform(X) # PCA(2成分) pca = PCA(n_components =2) X_pca = pca.fit_transform(X_std) #可視化 plt.figure() plt.scatter(X_pca[:, 0], X_std[:, 1]) plt.xlabel('PC1') plt.ylabel('PC2') plt.show() #次元ごとの寄与率 print(pca.explained_variance_ratio_)

コピペしてみたが、何がおきているかよくわからない!!
とりあえず第1主成分と、第2主成分をあわせても累積寄与率(?)は7割以下みたいです。
因子負荷量の計算
よくわからないが理解するのを待っていたらいつまでかかるかわからない!突き進むしかない!
各主成分に対してどの指標がどの程度相関しているかを眺めるため、因子負荷量を計算します。
import numpy as np # 因子負荷量を計算 pca_components = pca.components_*np.c_[np.sqrt(pca.explained_variance_)] # 眺めるための新しいDataFrameを作成 column_names = descriptors PCA_df = pd.DataFrame([pca_components[0], pca_components[1]], columns=column_names)
以下のようになりました。

ついでにグラフ化しておきます。(グラフでは絶対値にしています)
labels = ['MW', 'MolLogP', 'NumHD', 'NumHA', 'NumRB', 'TPSA'] # NumPyのfabsを使って絶対値に修正 PCA_df_abs = np.fabs(PCA_df) height_0=PCA_df_abs.loc[0] height_1=PCA_df_abs.loc[1] left= np.arange(len(height_1)) width=0.3 plt.bar(left, height_0, color='r', width =width) plt.bar(left+width, height_1, color='b', width =width) plt.xticks(left, labels) plt.show()

第1主成分(赤色)は、分子量(MW)、水素結合受容体数(NumHA)、回転可能結合数(NumRB)、極性表面積(TPSA)との相関が、第2主成分(青色)はLogP(MolLogP)との相関が強そうです。
水素結合供与体数(NumHD)は第1主成分よりも第2主成分との相関が強そうですが、MolLogPと比べると見劣りがします。
もう、NumHDを削ってしまっても良さそうな気がしてきました。(・・・無理やりこじつけた)
# NumHDonor以外の基準の和集合 df_all_w_o_NumHD =((df_all['MW'] > 300) & \ (df_all['MolLogP'] > 3) & \ (df_all['NumHAcceptors'] > 3) & \ (df_all['NumRotatableBonds'] > 3) & \ (df_all['TPSA'] > 60)) print(df_all_w_o_NumHD.sum()) print((df_all_w_o_NumHD.sum() / len(df_all)) * 100)
フラグメント指標から水素結合供与体数を除いた残りの5つを使って、5つ全てを満たす化合物数を求めると645704個(21%)となりました。
これならまだマシそうなので、こちらを用いたいと思います。
二つの基準を満たす分子の数
以上、見てきた内容をまとめると適用する基準は下記となります。
| 指標 | 分子量 | LogP | 水素結合供与体数 | 水素結合受容体数 | 回転可能結合数 | 極性表面積 |
|---|---|---|---|---|---|---|
| Lipinskiより | ≦500 | ≦5 | ≦5 | ≦10 | ||
| フラグメント指標より | >300 | >3 | >3 | >3 | >60 |
2つの基準を満たす分子はどの程度あるでしょうか?
df_all_both = ((df_all['MW'] > 300) & (df_all['MW'] <= 500) & \ (df_all['MolLogP'] > 3) & (df_all['MolLogP'] <= 5) &\ (df_all['NumHDonors'] <= 5) & \ (df_all['NumHAcceptors'] > 3) & (df_all['NumHAcceptors'] <= 10) &\ # 5以下になっていたので10以下に修正(01/28) (df_all['NumRotatableBonds'] > 3) & \ (df_all['TPSA'] > 60)) print(len(df_all)) # 3034076 print(df_all_both.sum()) # 563000 print((df_all_both.sum() / len(df_all)) * 100) # 18.55589642447981
元々の分子の総数 3034076、両基準を満たす分子の数563000(19%)となりました。
(最初の投稿では NumHA 10以下ではなく間違って5以下としていたため、このさらに半分の約30万個にまで減っていました。(01/28修正))
両基準で絞り込んだ化合物数は約56万個で、 もともと300万個の化合物があったので、1/5程度にまで減らすことができたことになります。
かなり無茶苦茶な話をしているので怒られてしまいそうですが、とりあえずの結果としてこの基準をもとに進めていきたいと思います。
解釈や使用方法等に誤りがたくさんあると思うのでご指摘いただければ幸いです。
SSSRを使って環状化合物と鎖状化合物を区別しようとする話
以前の記事で、創薬レイドバトルの共闘用シェアデータを2つにクラスタリングした後、環状ペプチドとそれ以外の分子の集まりとに手作業で分類しました。これでは数増えた時に大変ですし、見落としもありそうです。そこで今回はRDKit オンラインドキュメンテーションに記載のあったSSSRという考え方を頼りに同様の分類できるかどうか試してみたいと思います。
RDKitオンラインドキュメンテーションの該当箇所は下記です。
1. Ring Information
2. Ring Finding and SSSR
SSSR(smallest set of smallest rings; SSSR)については @mojaie さんのこちらの記事 化学構造情報とグラフ理論の説明が非常にわかりやいのでご参照ください。
とにかく、分子に含まれる環構造のうち「環の個数と環の大きさが最小になるような組み合わせ」だそうです。(ふわっとした理解)
環の情報がわかるなら、大員環とそれ以外の分子と区別することもできるのでは??? というのが今回の記事の目的です。
まずはデータの準備
from rdkit import rdBase, Chem from rdkit.Chem import AllChem, Draw, Descriptors, PandasTools import pandas as pd df = pd.read_csv('../pd1_inhibitor_dataset-master/PD1_inhibitor_dataset.csv') PandasTools.AddMoleculeColumnToFrame(df, 'smiles')
GetSSSR関数
RDKitのドキュメンテーションによると、SSSRに関してはGetSSSRとGetSymmSSSRの2つの関数があるそうです。
まずはGetSSSR関数を用いて見ます。こちらはSSSRの数を取得することができる関数です。
データセットの最初の分子をテスト化合物としてどのような結果が出るのか眺めて見ます。
最初の分子の構造はこのようなものです。
test_mol = df.loc[0, 'ROMol'] Draw.MolToImage(test_mol)

この分子のSSSRの数は???
test_SSSR = Chem.GetSSSR(test_mol) print(test_SSSR) # 8
8個だそうです!
・・・わからん。
SSSRの数だけではよくわかりません。
GetSymmSSSR関数
もう一方の関数、GetSymmSSSRを使うと、認識された各環構造の情報を取得することもできるそうです。
test_SymmSSSR = Chem.GetSymmSSSR(test_mol) print(type(test_SymmSSSR)) # <class 'rdkit.rdBase._vectNSt3__16vectorIiNS_9allocatorIiEEEE'> print(type(test_SSSR)) # <class 'int'>
GetSSSRで得た結果は整数値(int)ですが、GetSymmSSSRで得た結果はrdkitのオブジェクトでSSSRについてより詳細な情報をもっていそうです。
GetSymmSSSRの結果から、SSSRの数を取得するには長さを求めれば良いそうです。
print(len(test_SymmSSSR)) #8
先ほどの結果と一致しました。
最大の環構造を探す
ドキュメンテーションの説明をみる限り、GetSymmSSSRで得られるものは、認識された環構造のリストとなっており、リストの要素は各環構造に含まれる原子(atom index)のリストとなっているようなオブジェクトのようです(リストのリスト)。
8個の各環構造のサイズを求めてみます。
for i in range(len(test_SymmSSSR)): print('{}番目の環のサイズ:{}'.format(i, len(test_SymmSSSR[i]))) """ 出力結果 0番目の環のサイズ:45 1番目の環のサイズ:5 2番目の環のサイズ:5 3番目の環のサイズ:6 4番目の環のサイズ:5 5番目の環のサイズ:6 6番目の環のサイズ:5 7番目の環のサイズ:6 """
0番目が環員数 45となっています。ぼんやりと眺めていましたが環状ペプチドの長さはこんなにも長いものなんですね。具体的な数値を見るとあらためてびっくりしました。
この最大の環構造を構成する原子の番号(atom index)を取得して見ます。
largest_ring_member = list(test_SymmSSSR[0]) print(largest_ring_member) # [4, 5, 7, 8, 9, 11, 12, 13, 14, 15, 16, 18, 19, 20, 22, 23, 24, 26, 27, 28, 30, 34, 35, 37, 38, 39, 41, 42, 43, 45, 46, 47, 49, 50, 51, 53, 54, 55, 57, 58, 59, 61, 62, 63, 65]
構成要素の原子がわかったので、この情報を用いて分子のどの原子が該当の環構造に含まれるのか、ハイライトして描画して見ました。

期待通り、環状ペプチドの骨格となる構造を認識できていそうです。
環構造を順番に眺める
ipywidgetsを利用して各環構造を順番に眺めて見ます。
# 描画用 from rdkit.Chem.Draw import rdMolDraw2D from IPython.display import SVG from matplotlib.colors import ColorConverter # インタラクティブなビューワー from ipywidgets import interact,fixed,IntSlider import ipywidgets # ビューワーに与える関数を作成 def SSSR_viwer(idx): ring_member_atoms = list(test_SymmSSSR[idx]) prepro_test_mol = rdMolDraw2D.PrepareMolForDrawing(test_mol) target_ring_atoms = list(test_SymmSSSR[idx]) #デフォルトの設定は見づらいので色を変える color_dict = {} radius_dict = {} for i in target_ring_atoms: color_dict[i] = ColorConverter().to_rgb('khaki') radius_dict[i] = 0.7 #コンテナとなるオブジェクトの作成 view = rdMolDraw2D.MolDraw2DSVG(600,600) #コンテナの描画設定 option = view.drawOptions() option.circleAtoms=False option.continuousHighlights=False #最大の環構造に含まれる原子をハイライトに設定 view.DrawMolecule(prepro_test_mol, highlightAtoms=target_ring_atoms, highlightAtomColors= color_dict, highlightAtomRadii= radius_dict, highlightBonds=[]) #コンテナをファイナライズ view.FinishDrawing() #コンテナに書き込んだデータを取り出す svg = view.GetDrawingText() #データを描画 return(SVG(svg.replace('svg:,',''))) #ビューワーを実行 interact(SSSR_viwer, idx=ipywidgets.IntSlider(min=0,max=len(test_SymmSSSR)-1, step=1));

順番に表示させることでSSSRがどのような環構造を認識するのか、非常にわかりやすくなりました。
SSSRを利用して大員環を区別
大体SSSRでどのような情報が得られるのかわかったので、これを用いてマクロサイクル型の化合物とそれ以外を区別できるか検証して見たいと思います。
Wikipediaによると大員環化合物(macrocyclic compound)は「概ね10個以上の原子からなる環状構造を持つ有機化合物」ということなので、これを基準として用います。SSSRで得た環構造のうち、最大の大きさの環構造を構成する原子数が10以上の場合、大員環化合物とすることとしたいと思います。
まずは、各化合物のSSSRをもとめ、最大の環構造の情報を取得します。
関数を作成し、Molオブジェクトを含むカラムに適用し、得た値を新しいカラムへと格納します。
def largest_ring_member_count(mol): SymmSSSR = Chem.GetSymmSSSR(mol) size_list = [] # 各環の大きさを順番にリストに追加 for i in range(len(SymmSSSR)): size_list.append(len(SymmSSSR[i])) # リストの最大値をとりだす max_size = max(size_list) return(max_size) # 最大の環の情報をもつ新しいカラムを作成 df['largest_ring_size'] = df['ROMol'].map(largest_ring_member_count)
環のサイズが10以上の時はマクロサイクル型(macro)、それより小さい場合は鎖型(chain)として分類してみます。
#classifierとか一回言ってみたかった def macro_classifier(size): if size >= 10: return('macro') elif size < 10: return('chain') df['macro_chain'] = df['largest_ring_size'].map(macro_classifier)
どのような分類になったか数を確認してみます。
df['macro_chain'].value_counts() """ chain 39 macro 14 Name: macro_chain, dtype: int64 """
マクロサイクル型 14個、鎖状型39個、となりました。
前回の分類では、外れ値としたマクロサイクル型化合物1つの除いた、合計52個を、マクロサイクル型13個と鎖状型39個とに分けました。数を見る限り同様の結果が得られていそうです。
それぞれ別のDataFrameに分けたあと、構造を確認します。
マクロサイクル型・・・
df_macro = df[df['macro_chain']== 'macro'] macro_list = list(df_macro['ROMol']) def macro_viewer(idx): mol = macro_list[idx] return(display(Draw.MolToImage(mol))) interact(class_1_viewer, idx=ipywidgets.IntSlider(min=0,max=len(macro_list)-1, step=1));

鎖状型・・・
df_chain = df[df['macro_chain']== 'chain'] chain_list = list(df_chain['ROMol']) def chain_viewer(idx): mol = chain_list[idx] return(display(Draw.MolToImage(mol))) interact(chain_viewer, idx=ipywidgets.IntSlider(min=0,max=len(chain_list)-1, step=1));

しっかりと望み通りの分類ができていそうです。
まとめ
今回はSSSRを使って、大員環化合物とそれ以外の化合物を分類できるか?ということを行ってみました。
RDKitのドキュメンテーションを読んでいる時は、どのような目的で使えば良いものかさっぱりわからず、どうして The SSSR Problem という節を設けるほど解説してあるのか疑問に思っていたのですが、実際に使って見るととても使い勝手の良い重要な考え方ということがわかりました。
かなり適当に使ってしまったので、概念の理解の誤り等ございましたらご指摘いただければ幸いです。
ライブラリの指標計算結果まとめ
前回の記事で、活性化合物群の情報をもとにライブラリから候補化合物を絞り込むため、指標の閾値(フィルター)を決めようと試みました。とりあえずの結果として、以下の値を求めました。
| 指標 | small_molecules min |
small_molecules max |
フィルタリング目安 | ||
|---|---|---|---|---|---|
| ① | 分子量 | MW | 215 | 835 | > 300 |
| ② | cLogP | MolLogP | -2.5 | 9.5 | -3 ~ 10 |
| ③ | 水素結合供与体数 | NumHDonoes | 1 | 7 | < 10 |
| ④ | 水素結合受容体数 | NumHAcceptors | 3 | 9 | < 10 |
| ⑤ | 回転可能結合数 | NumRotatableBonds | 2 | 15 | < 15 |
| ⑥ | 極性表面積 | TPSA | 48 | 250 | < 250 |
こちらのフィルタリング目安を、前処理済みのライブラリEnamine_Premium_collection(分子量300より小さいものは除去済み)に適用すべく、各化合物の指標を計算、その統計値を求めました。
結果は以下の通り・・・

なんと、欲張って幅を広くとりすぎたためか、分子量以外の指標では化合物の数をうまく減らせそうにありません。
絞り込み対象のライブラリにおける指標の値の分布を検証せずに、手抜きして活性化合物群のみから閾値を決めようとしたことに原因があるかもしれません。
まずは、相手を知る必要がありそうです。
そこで各指標値の値を残りのライブラリ3つ(Enamine_Advanced_collection、Enamine_HTS_collection、UOS_HTS)に対しても求め分布を把握してみることにしました。 これらのライブラリに対しては分子量 300以下を除く処理はしていません。
まずは結果から・・・
① Enamine_Advanced_collection

② Enamine_HTS_collection

③ UOS_HTS

分子量300以下を除く処理をしていないためか、分子量の最小値の小ささにすこしびっくりしました。
このままではよくわからないので、値のばらつきを見るため箱ひげ図を用いてばらつきを可視化しました。 以下は、Enamine_Advanced_collectionの場合です。

こちらを見ると先日設定したフィルタリング目安は、最大値よりも大きく、外れ値とも言えそうな化合物も全て残してしまう、ということがわかります。
先にこっちを見ておくべきだった!!
以下に、上記を求めた際につまづいた点などを記載しておきます。
MolLogPの計算
Enamine_Premium_collectionを計算する際には問題とならなかったのですが、Enamine_Advanced_collectionを処理する際に、MolLogPの計算でエラーが生じました。指標の要約統計量を確認するとEnamine_Advanced_collectionのMolLogPのみ、count(値の総数:486321)が分子の総数(486322)よりも1小さくなっています。
エラーの内容は以下の通り。
---> 42 MolLogP_value = Chem.Descriptors.MolLogP(mol_neu) 43 mol_neu.SetDoubleProp('MolLogP', MolLogP_value) 44 MolLogP_list.append(MolLogP_value) /root/miniconda/lib/python3.6/site-packages/rdkit/Chem/Crippen.py in <lambda>(*x, **y) 168 _pyMolMR.version = "1.1.0" 169 --> 170 MolLogP = lambda *x, **y: rdMolDescriptors.CalcCrippenDescriptors(*x, **y)[0] 171 MolLogP.version = rdMolDescriptors._CalcCrippenDescriptors_version 172 MolLogP.__doc__ = """ Wildman-Crippen LogP value ValueError: Sanitization error: Explicit valence for atom # 6 B, 5, is greater than permitted
Value Error と書かれたエラー内容から推測すると、この分子はホウ素(B : 原子番号 6)を含み、その原子に対する結合の数が、通常よりも多いため分子としてうまく認識できなくなっているようです。構造は以下の通り。

上記のようにホウ素に3つのフッ素原子とピロールが結合した分子のカリウム塩となっています。4本の結合+負電荷で 「valence # 6 B, 5」となりエラーとなったようです。どうやらこのライブラリにはクロスカップリング反応の試薬(有機トリフルオロボレート塩 (Chem-Station さんより))となるような化合物も含まれているようです。
前処理のコード全体
上記エラーのためforループが途中でとまってしまい、前処理ができなくなってしまいました。そこで、MolLogPの計算については例外処理(try…except…)を追加しました。前処理のコード全体としては下記となります。
from rdkit import Chem import gzip from rdkit.Chem import Descriptors, MolStandardize EAc_gz = gzip.open('Enamine_Advanced_collection.sdf.gz') EAc_suppl = Chem.ForwardSDMolSupplier(EAc_gz) #Molオブジェクト、および各指標を計算し格納するためのからのリストを作成 EAc_mols = [] MW_list = [] MolLogP_list = [] NumHDonors_list = [] NumHAcceptors_list = [] NumRotatableBonds_list = [] TPSA_list = [] for x in EAc_suppl: if x is not None: mol = x #処理の前にidnumberを取り出しておく ID = mol.GetProp('idnumber') #構造の標準化 normalizer =MolStandardize.normalize.Normalizer() mol_norm = normalizer.normalize(mol) #一番大きいサイズのフラグメントのみ残す lfc = MolStandardize.fragment.LargestFragmentChooser() mol_desalt = lfc.choose(mol_norm) #電荷の中和 uc = MolStandardize.charge.Uncharger() mol_neu = uc.uncharge(mol_desalt) #処理後のMolオブジェクトに、取り出しておいた元々のidnumberをoriginal_idとして付与 mol_neu.SetProp('original_id', ID) #分子量を計算 MW_value = Chem.Descriptors.MolWt(mol_neu) #分子量をプロパティとして持たせる。(小数点を含むfloat型) mol_neu.SetDoubleProp('MW', MW_value) #値のみのリストにも追加する MW_list.append(MW_value) #MolLogPを計算(MolLogPは対応しない元素を含むとエラーを返すので例外処理として記入) try: MolLogP_value = Chem.Descriptors.MolLogP(mol_neu) mol_neu.SetDoubleProp('MolLogP', MolLogP_value) MolLogP_list.append(MolLogP_value) except: MolLogP_list.append(None) #NumHDonorsでも同じことをする。 NumHDonors_value = Chem.Descriptors.NumHDonors(mol_neu) #非負の整数値(int型)のプロパティなので SetUnsignedIntProp でも良いが面倒なのでSetDoublePropにする。 #たぶんメモリの無駄遣い。 mol_neu.SetDoubleProp('NumHDonors', NumHDonors_value) NumHDonors_list.append(NumHDonors_value) #NumHAcceptors NumHAcceptors_value = Chem.Descriptors.NumHAcceptors(mol_neu) mol_neu.SetDoubleProp('NumHAcceptors', NumHAcceptors_value) NumHAcceptors_list.append(NumHAcceptors_value) #NumRotatableBonds NumRotatableBonds_value = Chem.Descriptors.NumRotatableBonds(mol_neu) mol_neu.SetDoubleProp('NumRotatableBonds', NumRotatableBonds_value) NumRotatableBonds_list.append(NumRotatableBonds_value) #TPSA TPSA_value = Chem.Descriptors.TPSA(mol_neu) mol_neu.SetDoubleProp('TPSA', TPSA_value) TPSA_list.append(TPSA_value) #Molオブジェクトのリストも忘れずに EAc_mols.append(mol_neu)
以後の処理をできるだけ簡単にするため、指標の数値のみを含むcsvファイルを作成しました。
import pandas as pd property_names = ['MW', 'MolLogP', 'NumHDonors', 'NumHAcceptors', 'NumRotatableBonds', 'TPSA'] property_values = [MW_list, MolLogP_list, NumHDonors_list, NumHAcceptors_list, NumRotatableBonds_list, TPSA_list] df = pd.DataFrame(index=[], columns=property_names) for i, j in zip(property_names, property_values): df[i] = j df.to_csv('Enamine_Advanced_collection_desc.csv', sep=',')
また別途、構造情報と各指標の値を紐づけた上でsdfを作成しました。
#構造と各種プロパティを残したsdfファイルを作成 writer = Chem.SDWriter('Enamine_Advanced_collection_desc.sdf') writer.SetProps(property_names) #01/29追記 このままだとidが抜けるので適当に追加してください。またやってしまった・・・ for mol in EAc_mols: writer.write(mol) writer.close()
以上が、各ライブラリファイルに対して実行した前処理の全体の流れとなります。
処理に失敗した分子の確認
先の処理でMolLogPの計算ができなかったものについてはNoneを格納するように例外処理を指定しました。このリストをpandasで読み込むとNoneは自動的にNaNとしてDataFrameに取り込まれるそうです。
どのような化合物が処理に失敗したかを確認するため、
- 上記で出力したcsvファイルをPandasのDataFrameとして読み込む
- MolLogPの列で値がNaNとなっている行のindexを取得する
- 取得したindexに相当する分子の構造を確認する という手順を実施しました。
(上述のValue Errorとなった構造の描画のために行った処理です)
# csvをDataFrameとする df_EAc = pd.read_csv('./Enamine_Advanced_collection_desc.csv') # MolLogPの列がNaNとなっている(query)行番号(index)を取り出し、リスト化する(list) df_EAc_MolLogP_None = list(df_EAc.query('MolLogP == "NaN"').index) print(df_EAc_MolLogP_None) # [8135] と出力された
index 8135の分子一つのみが、処理に失敗したもののようです。コードは省略しますが、この分子をsdfから取り出し描画することで、先の構造(ピロールのトリフルオロボレート塩)を確認しました。
SDFの持つ情報をとりあえず確認する方法
上記、前処理ですが Enamine~~ と名前のついた3つのsdfではうまくいったものの、UOS_HTSではうまくいきませんでした。原因はidnumber というプロパティが存在しなかったことです。どうやらこちらのライブラリのみ、化合物のIDが別の名称の属性として格納されているようです。
TextエディタやMarvinViewなどで開いてSDFの持つ構造情報以外の属性を確認する、という手段もありますが、なにぶんファイルサイズが大きく開くだけでも大変です。ここは RDKitをうまく使いたい・・・ということで、とりあえずsdfから最初の1分子のみを読み込んでみることとしました。
以前こちらの記事で検証したように、ForwardSDMolSupplierで読み込んだSDFからはnext()を使うことで順番に一つずつ取り出すことができます。
UH_suppl = Chem.ForwardSDMolSupplier('./UOS_HTS.sdf') #組み込み関数next()を使って最初の一つの分子を取り出す test_mol = next(UH_suppl) #どんなPropertyが含まれているか取り出し、リスト化、出力 prop_names = list(test_mol.GetPropNames()) print(prop_names) #['ID']と出力された
UOS_HTSについては、化合物のIDはIDという属性に格納されているようです。 これでidnumberの代わりにIDとして、他のライブラリに行ったのと同じ処理を行えば良いことがわかりました。
なんどもクドイようですが、上記処理に続けて前処理のコードを実行すると失敗します。
理由は属性の確認のため、ForwardSDMolSupplierのオブジェクトから最初の一つをとりだしてしまったため、再度sdfを読み込むところから始めないと、最初の一つを捨ててしまったことになるからです。(私は以前記事を書いておきながらもこの失敗をまた繰り返しました・・・学習能力低すぎ・・・)
統計量の計算
pandasのdescribeを用いると項目数が増えるので、総数(count)、最小値(min)、平均値(mean)、最大値(max)を別個に計算しました。
df_count = df_EAc[descriptors].count() df_min = df_EAc[descriptors].min() df_mean = df_EAc[descriptors].mean() df_max = df_EAc[descriptors].max() df_desc_view_EAc = pd.DataFrame([df_count, df_min, df_mean, df_max], index=['count', 'min', 'mean', 'max']) df_desc_view_EAc.round(2)
これで冒頭の統計量を出しました。
箱ひげ図を用いたばらつきの可視化
要約統計量のみでは値の広がりがわからないので箱ひげ図を用いてばらつきを可視化しました。
import matplotlib as mpl import matplotlib.pyplot as plt %matplotlib inline
一つのプロットに全ての指標を乗せてしまうと、値の範囲の違い(分子量と水素結合受容体の数では値の幅が全然違う)のため、潰れてしまったのでsubplotを指定しました。
descriptors = ['MW', 'MolLogP', 'NumHDonors', 'NumHAcceptors', 'NumRotatableBonds', 'TPSA'] df_EAc[descriptors].plot(kind = 'box', subplots=True, figsize=(20, 6))
これで冒頭の図を得ました。
以上です。
活性化合物群を分析して指標の閾値を決めようとする話
前回、約13万個の化合物を含むEnamine_Premium_collectionから、分子量300を基準とした分子の絞り込みにより約11万個まで化合物数を減らすことができました。
まだまだ多い・・・と思っていたら@iwatobipen先生に分子を絞り込む指標を考える上でとても参考になる記事 SAR News Np.19 をご紹介いただきました。
SAR newsは日本薬学会 構造活性相関部会のHPの発行しているニュースレターで、だいたい年に2本のペースで発行されており、部会に所属していなくても過去のニュースレターを含めて自由に閲覧可能となっています(バックナンバーはこちらから閲覧可能です)。
- SAR Newsで用いられている指標
- PD/PD-L1阻害活性 化合物群の指標の分析
- クラスタリングによる分類
- 各クラスターに含まれる構造のインタラクティブな描画
- 環状ペプチドと低分子とで2つに再度クラス分け
- まとめ
SAR Newsで用いられている指標
さて、ご紹介いただいたニュースレターの 株式会社理論創薬研究所 吉森篤史 氏による記事では「標的タンパク質指向型フラグメントライブラリの構築」を適用例として、RDKitを用いたフィルタリングの方法が実際のコード含めて解説されています。
用いられているフィルタリング基準は、フラグメント指標「Rule of Three」で、具体的には以下となっています。
| 指標 | 基準 | 記事中のコード | 現在のコード? | |
|---|---|---|---|---|
| ① | 分子量 | ≦300 | AvailDescriptors.descDict['MolWt'](mol) | Descriptors.MolWt(mol) |
| ② | cLogP | ≦3 | AvailDescriptors.descDict['MolLogP'](mol) | Descriptors.MolLogP(mol) |
| ③ | 水素結合供与体数 | ≦3 | AvailDescriptors.descDict['NumHDonors'](mol) | Descriptors.NumHDonors(mol) |
| ④ | 水素結合受容体数 | ≦3 | AvailDescriptors.descDict['NumHAcceptors'](mol) | Descriptors.NumHAcceptors(mol) |
| ⑤ | 回転可能結合数 | ≦3 | AvailDescriptors.descDict['NumRotatableBonds'](mol) | Descriptors.NumRotatableBonds(mol) |
| ⑥ | 極性表面積 | ≦60 | AvailDescriptors.descDict['TPSA'](mol) | Descriptors.TPSA(mol) |
記事が2010年のもののためか、現在のRDKitのモジュールを眺めてもAvailDescriptorsというものが見つけられませんでした。「化学の新しいカタチ」さんの記事「 RDKitにおける記述子の扱い方をリピンスキーの法則を通して学ぶ」 に同じ目的と思われるモジュール(Descriptors)が紹介されていましたので、こちらも上記の表に追加しました。
上記、指標の基準はフラグメント("活性が弱いながらも適切な結合を形成できる小分子")を対象としているためか、かなり小さな値となっているように見えます。今回の創薬レイドバトルの目標は、ある程度活性が強いものを取得することなので、この基準をそのまま適用するのは良くなさそうです。
また、他の指標としてリピンスキーの法則を用いる、というのも良さそうですが、前回活性化合物群の分子量のデータを眺めた印象では、分子量 500以上の大きなものも多く、すでにRule of Five を外れてしまっています。
一般的な指標をそのまま適用するだけでは、既知活性化合物群ですら外れてしまいそうなので、まずは活性化合物群の値がどのようになっているかを検証したいと思います。
PD/PD-L1阻害活性 化合物群の指標の分析
指標の計算
まずは必要なものをimportします。
from rdkit import rdBase, Chem from rdkit.Chem import AllChem, Draw, Descriptors, PandasTools import pandas as pd print('rdkit version: ',rdBase.rdkitVersion) print('pandas version: ', pd.__version__) #rdkit version: 2018.09.1 #pandas version: 0.23.4
前回同様、創薬レイドバトルの共闘用シェアデータを使用します。
#csv形式のデータをPandasのDataFrameで読み込む df = pd.read_csv('../pd1_inhibitor_dataset-master/PD1_inhibitor_dataset.csv') #smilesカラムのSMILES情報を使ってをMolオブジェクトを追加する PandasTools.AddMoleculeColumnToFrame(df, 'smiles') #ROMolカラムに追加されているMolオブジェクトを使って記述子を計算し、値を追加する。 #①分子量 df['MW'] = df.ROMol.map(Descriptors.MolWt) #②LogP df['MolLogP'] = df.ROMol.map(Descriptors.MolLogP) #③水素結合供与体数 df['NumHDonors'] = df.ROMol.map(Descriptors.NumHDonors) #④水素結合受容体数 df['NumHAcceptors'] = df.ROMol.map(Descriptors.NumHAcceptors) #⑤回転可能結合数 df['NumRotatableBonds'] = df.ROMol.map(Descriptors.NumRotatableBonds) #⑥極性表面積 df['TPSA'] = df.ROMol.map(Descriptors.TPSA)
計算した指標の要約統計量を求めてみます。
#計算した指標のリスト Descriptors_list = ['MW', 'MolLogP', 'NumHDonors', 'NumHAcceptors', 'NumRotatableBonds', 'TPSA'] #要約統計量を計算。小数点以下の桁数が多いので丸める df[Descriptors_list].describe().round(2)

相関係数は以下のようになっています。
#相関係数の計算 df[Descriptors_list].corr().round(2)

ざっと見る限りMolLogPのみが他の指標との相関係数が負かつ絶対値が小さくなっています。
指標をプロット
数字を見てもイマイチ感触がつかめません。前回分子量を眺めた際に描画すると値の分布がわかりやすかったので今回も描画を行います。一つ一つみるのは面倒なのでSeabornを使ってペアプロットを作成します。
import matplotlib import matplotlib.pyplot as plt import seaborn as sns %matplotlib inline #Seabornを用いたペアプロットの作成 sns.pairplot(df[Descriptors_list])

確かに、MolLogPだけが他の指標とのプロットした際に相関が悪そうです。
見やすさのためMolLogPを外して再度プロットして見ます。
#計算した指標のリストから MolLogPを除いたもの Descriptors_list_2 = ['MW', 'NumHDonors', 'NumHAcceptors', 'NumRotatableBonds', 'TPSA'] sns.pairplot(df[Descriptors_list_2])

残りの指標間は直線上に乗っているようにみえわかりやすくなりました。
外れ値を取り除く
各プロットを見ると、最大値となっている分子が1つだけ他の分子から遠く、外れ値のようになっています。特にNumRotatableBondsで顕著にみえるので、こちらの最大値がどのような分子なのか眺めて見たいと思います。
#NumRotatableBondsが最大値となる化合物を眺める。 #まずは最大値となる化合物の行番号を取得する print(df['NumRotatableBonds'].idxmax()) #5と出力 #Molオブジェクトの格納されているcolumnの番号を確認 print(df.columns.get_loc('ROMol')) #6と出力
DatafFrameの(5, 6) がみたい構造のMOlオブジェクトがある場所のようです。これを描画します。描画の手順は省きます。

環状ペプチドにさらに長い鎖状構造がつながっています。他の構造と比べてかなり特異な構造なので、こちらは外れ値として省いてしまっても良さそうです。
新しいDataFrameを作成し、再度ペアプロットを作成します。
#行番号5を除去する df_new = df.drop(5, axis=0) sns.pairplot(df_new[Descriptors_list_2])

かなり分布がわかりやすくなってきました。いずれの指標においても大きく2つのグループに分子を分けることができそうです。
これは・・・クラスタリングというやつが使えるのではないか???
クラスタリングによる分類
Morganフィンガープリントとタニモト係数による類似性評価
「化学の新しいカタチ」さんのこちらの記事(RDKitとk平均法による化合物の非階層型クラスタリング )を参考に化合物を2つのクラスターに分けたいと思います。以下の操作は上記記事の処理手順をほぼそのまま使用させていただいています。
各分子のMorganフィンガープリントを計算し、分子間の類似性をタニモト係数によって比較、クラスタリングに用いるための距離行列を作成します。
from rdkit import DataStructs from rdkit.Chem import AllChem import numpy as np #計算に使用するためMolオブジェクトのリストを作成 mol_list = list(df_new['ROMol']) print(len(mol_list)) #52個の分子を含む #Morganフィンガープリントの計算 morgan_fp = [AllChem.GetMorganFingerprintAsBitVect(x,2,2048) for x in mol_list] #総数52の分子間で距離行列の計算 dis_matrix = [DataStructs.BulkTanimotoSimilarity(morgan_fp[i], morgan_fp[:52],returnDistance=True) for i in range(52)] ### numpy.ndarrayへの変換 dis_array = np.array(dis_matrix) dis_array.shape
52 x 52 の距離行列が得られました。
k平均法によるクラスタリング
k平均法で2つのクラスターに分類します。
from sklearn.cluster import KMeans import pandas as pd kmeans = KMeans(n_clusters=2) kmeans.fit(dis_array) pd.value_counts(kmeans.labels_)

10個の分子を含むクラス0と、42個の分子を含むクラス1に分類できました。 このクラス分類もDataFrameに追加しておきます。
class_list = [] for i in range(len(kmeans.labels_)): if kmeans.labels_[i] == 0: class_list.append('class_0') elif kmeans.labels_[i] == 1: class_list.append('class_1') df_new['kmeans_class'] = class_list
ペアプロットへのクラス分類の反映
先に作成したpairplotをkmeans法で分類したクラスに応じて色分けするとどうなるか、見て見たいと思います。
もともと他の指標との相関が悪そうだったMolLogPについても、色分けしたらどうなるか気になるので、今回はpairplotに加えたいと思います。
#計算した指標のリスト(MolLogPを含む)にkmeansで形成したクラスも加える Descriptors_class = ['MW', 'MolLogP', 'NumHDonors', 'NumHAcceptors', 'NumRotatableBonds', 'TPSA', 'kmeans_class'] sns.pairplot(df_new[Descriptors_class], hue='kmeans_class')

Morganフィンガープリントをつかって2つのクラスターに分けた結果で色分けすると、指標の分布でも比較的良好に2つのクラスターに分かれているように見えます。
とくに興味深いのが、MolLogPで、ヒストグラムでは2つのクラスターが重なっていますが、ペアプロット上では割と良好に分布が分かれているように見えます。
Morganフィンガープリントがどのように計算しているのか、まだ理解していないのですが指標の分布をうまく説明するようなクラスタリングができているのは面白い結果です。
各クラスターに含まれる構造のインタラクティブな描画
プロット上での描画
各点の構造がどのようなものか、分布の広がりがありつつ2つのクラスターが綺麗に分かれているMolLogPとTPSAの2軸によるプロットを用いて可視化して見たいと思います。
@iwatobipen先生の記事 Plot Chemical space with d3js based libraryを参考にさせていただきました。
import mpld3 #別途インストールしておかないとimportできない from mpld3 import plugins mpld3.enable_notebook() #描画を作成するためのモジュール from rdkit.Chem.Draw import rdMolDraw2D from IPython.display import SVG #svgを格納するための空のリスト svgs_list = [] for i in range(len(df_new)): view = rdMolDraw2D.MolDraw2DSVG(300,300) #Molオブジェクトを含むカラムの番号 ROMol_col = df_new.columns.get_loc('ROMol') #i行目のMolオブジェクトを取得 mol = df_new.iloc[i, ROMol_col] #描画する分子の前処理 prepro_mol = rdMolDraw2D.PrepareMolForDrawing(mol) #分子をコンテナに追加 view.DrawMolecule(prepro_mol) #コンテナをファイナライズ view.FinishDrawing() #コンテナに書き込んだデータを取り出す svg = view.GetDrawingText() #データをリストに追加 svgs_list.append(svg.replace('svg:,',''))
以上で、見たい化合物の構造情報(svg形式)を含むリストが作成できました。あとは、プロットと組み合わせて、プロット上の各点がどのような構造に相当するのかを眺めます。
fig, ax = plt.subplots() ax.set_xlabel('MolLogP') ax.set_ylabel('TPSA') points = ax.scatter(df_new['MolLogP'], df_new['TPSA']) tooltip = plugins.PointHTMLTooltip(points, svgs_list) plugins.connect(fig, tooltip)

y軸(TPSA)値が 500よりも上に位置するのがclass_1、500よりも下に位置するのがclass_0となっています。
カーソルをうまくGIFに写すことができずわかりづらくなってしまいましたが、プロット上を右上から反時計回りに順にカーソルを移動させ、構造を表示させています。 class_1は全て環状ペプチド、class_0はほとんどが環状ペプチドではない低分子ですが、左下に位置する3つは環状ペプチド構造となっています。
スライドバー形式での描画
少しわかりづらかったので、もうひとつのインタラクティブな描画方法を試します。こちらはRDKitのBlogで紹介されていたものです。
まずは class_0 のみを要素とするDataFrameを作成し、さらにMolオブジェクトのリストとします。
df_class_0 = df_new[df_new['kmeans_class']== 'class_0'] class_0_list = list(df_class_0['ROMol'])
以下の方法ではipywidgetsを使って可視化します。
from ipywidgets import interact,fixed,IntSlider import ipywidgets #class_0 に分類された構造を順番に眺めていく def class_0_viewer(idx): mol = class_0_list[idx] return(display(Draw.MolToImage(mol))) interact(class_0_viewer, idx=ipywidgets.IntSlider(min=0,max=len(class_0_list)-1, step=1));

class_0 のうち idx = [29, 30, 32] は環状ペプチド構造となっており、その他の構造とは少し違った形となっています。
ついでにclass_1もやってみます。同様の操作なのでコードは省略します。

順番に構造をおくるとかなりわかりやすくなりました。
環状ペプチドと低分子とで2つに再度クラス分け
環状ペプチドと、それ以外で分けた方がより良いクラス分けとなりそうです。 そこでclass_0 に含まれる環状構造 をclass_1に移動し、新しいデータフレームを2つ作成します。
df_peptides = pd.concat([df_class_1, df_class_0.iloc[[29,30,32]]]) df_small_molecules = df_class_0.drop([40,41,43]) # idx=[29, 30, 32]番目の行のindexは[40,41,43] print(len(df_peptides)) # 13と出力 print(len(df_small_molecules)) # 39と出力
合計52(13 + 39)個の2つのデータフレームに分けることができました。 処理の最初の段階で、外れ値とも思える大きな分子を一つ取り除いているので、もともとのデータセット(53個)よりも一つ少なくなっています。
活性化合物群に基づき指標の閾値を考察
さて、いろいろと操作してきたため、本来の目的を見失ってきました。
もともとの目的は、化合物ライブラリから化合物の数を絞り込むための指標の閾値を、活性があると報告されている化合物群のデータをもとに決めよう、というものでした。
活性化合物群を可視化した結果、2つのクラスターに分類することができ、これをもとに環状ペプチドと、それ以外の低分子との2つに分けることができました。このうち、「PD/PD-L1の相互作用を阻害する低分子を見つける」という目標として参照するのに適しているのは、後者となりそうです。
以下のテーブルでは、低分子化合物クラスター(small_molecules)と環状ペプチドクラスター(peptides)における各指標の最小値と最大値、およびLipinski の Rule of Five の値を並べています。
化合物を絞り込む際に用いる指標の閾値としては、値の範囲よりも少し広めにとって最右列のフィルタリング目安とするのが手かもしれません。
| 指標 | small_molecules min |
small_molecules max |
peptides min |
peptides max |
Lipinski Rule of 5 |
フィルタリング目安 | ||
|---|---|---|---|---|---|---|---|---|
| ① | 分子量 | MW | 215 | 835 | 491 | 2096 | <= 500 | > 300 (実施済) |
| ② | cLogP | MolLogP | -2.5 | 9.5 | -6.35 | 3.20 | <= 5 | -3 ~ 10 |
| ③ | 水素結合供与体数 | NumHDonoes | 1 | 7 | 9 | 22 | <= 5 | < 10 |
| ④ | 水素結合受容体数 | NumHAcceptors | 3 | 9 | 10 | 26 | <= 10 | < 10 |
| ⑤ | 回転可能結合数 | NumRotatableBonds | 2 | 15 | 3 | 39 | < 15 | |
| ⑥ | 極性表面積 | TPSA | 48 | 250 | 237 | 713 | < 250 |
念のため今回クラス分類した2つのデータフレームを出力しておきます。
properties = df_small_molecules.columns.values Chem.PandasTools.WriteSDF(df_small_molecules, 'small_molecules.sdf', molColName='ROMol', idName='compound_id', properties=properties) Chem.PandasTools.WriteSDF(df_peptides, 'peptides.sdf', molColName='ROMol', idName='compound_id', properties=properties)
まとめ
以上、本記事では分子量以外の化合物絞り込みの基準を探すべく、活性化合物群の情報を分析して見ました。
Morganフィンガープリントとk平均法を組み合わせた分類では、環状ペプチドのような構造もいくつかは鎖状低分子と同一のクラスにふくまれるなど、想定外のクラス分けがされて興味深い結果になりました。 また、ある程度構造が限られた化合物セットであれば順番に表示させるような機能を利用することで、化合物セットの雰囲気をつかむことができることがわかりました。
本記事を作成するにあたって、さまざまなページを参照させていただきました。また、多くのコードを拝借させていただいております。誤り等ございましたらご指摘いただければ幸いです。
追記
前回の記事で分子量で絞り込んだあとの化合物群(約11万個)の指標を計算してみたら、上記のテーブルのフィルタリング目安ではほとんど絞り込みにならなさそうです。 もっとちゃんと考えないとダメそうです・・・私の休日・・・

~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~
参考記事
① SAR News No.19
Chemoinformatics Toolkits を用いた創薬システム開発におけるラピッドラタイピング 吉森 篤史
http://bukai.pharm.or.jp/bukai_kozo/SARNews/SARNews_19.pdf
② 化学の新しいカタチ
RDKitにおける記述子の扱い方をリピンスキーの法則を通して学ぶ
③ 化学の新しいカタチ
RDKitとk平均法による化合物の非階層型クラスタリング
④ IS LIFE WORTH LIVING? Plot Chemical space with d3js based library #RDKit #Chemoinformatics | Is life worth living?
⑤ The RDKit Blog
RDKit: Using the new fingerprint bit rendering code
⑥ ipywidgetsのインストール方法
ipywidgetsの使い方
⑦ ipywidgets の User Guide
ipywidgets — Jupyter Widgets 7.4.2 documentation
分子量で絞り込もうとした話
なんとか化合物を読み込むということはできましたが、総数は200万以上・・・
このままでは全くどうして良いかわかりません。
どうしたものかと思っていたらTwitterで処理の方法を紹介してくださっている方がいらっしゃいました。
計算するのに化合物が多すぎたので
— mayudqx (@mayudqx) 2019年1月15日
1.分子量300以下は削除
2.オルト位に置換基をもつビフェニルに限定
3.それでも多かったのでざーっとドッキングして-8でカット
4.ファーマコフォアをビフェニルに指定して試行回数増やして再計算
@mayudqxさんのされていた処理の順序は、
| 処理 | 意図 | |
|---|---|---|
| ① | 分子量の範囲を設定 | 粗い絞り込み |
| ② | 部分構造で絞り込み | 活性化合物を参照したLBVS(?) |
| ③ | 粗いドッキング | タンパク質との相互作用を予測するSBVS(?) |
| ④ | ファーマコフォアを指定したより詳細なドッキング | SBVS 2回目(?) |
という感じです。
このうち①と②なら、RDKitを使って行うことができそうです。とりあえずプロの真似をしよう!
今回は、まずは①分子量での絞り込みに挑戦したいと思います。
絞りこみの対象となる化合物群の構造式をいくつか眺めた感想として、確かにとても小さな分子(試薬??)もライブラリに含まれていることが意外でした。今回の目標は、生理活性のある分子を予測しよう、というものなので、@mayudqxさんがされているように、あまりにも小さく活性の期待できなさそうなものを省くというのは、最初に行う絞り込みとして良さそうです。
ところで、絞り込みの閾値を分子量300とされていましたが、こういう値をどうやって設定して良いものかわかりません。
何か参考になるデータはないか・・・と思っていたら創薬レイドバトルのgithubページ に「共闘用シェアデータ」という、活性化合物群のデータを掲載してくださっていました!
様々な特許から「低分子PD-1阻害剤と思われる 低分子化合物の構造を抽出し」たもので、「各特許の記述に基づいて最も高活性とされる化合物を1例ずつ抽出し」たものだそうです。
こんな大変そうなデータセットを公開してくださるとは・・・。ありがとうございます!
活性のある化合物は閾値を決める根拠としてとても良さそうです。これを使わない手はない!ということでこちらを参照して見たいと思います。
前回に引き続き、RDKitのPandasToolsを使って解析を行って見たいと思います。「化学の新しいカタチ」さんのこちらの記事 を参考にさせていただきました。
まずは色々必要なものをimportします。
from rdkit import rdBase, Chem from rdkit.Chem import AllChem, Draw, Descriptors, PandasTools import pandas as pd print('rdkit version: ',rdBase.rdkitVersion) print('pandas version: ', pd.__version__) #rdkit version: 2018.09.1 #pandas version: 0.23.4
データはcsv形式で提供されているので、まずはpandasで読み込みます。
githubからダウンロードしてくるとpd1_inhibitor_dataset-masterというフォルダの中にPD1_inhibitor_dataset.csvという名前でデータが保存されていました。
df = pd.read_csv('../pd1_inhibitor_dataset-master/PD1_inhibitor_dataset.csv') print(df.shape) #(53, 6) print(df.columns) #Index(['compound_id', 'patent_no', 'example_no', 'schembl_id', 'applicant', 'smiles'], dtype='object')

全部で53個の化合物が含まれており、構造情報はSMILESとして格納されています。
SMILESのままではよくわからないので、構造式を眺めるためにMolオブジェクトに変換します。
PandasTools.AddMoleculeColumnToFrame(df, 'smiles')

ROMolという列にMolオブジェクトが保存されており、小さくて見づらいですが構造式として表示されます。 最初の8個を構造式だけを取り出して並べて見ます。
PandasTools.FrameToGridImage(df[:8], column='ROMol', legendsCol='compound_id', molsPerRow=4, subImgSize=(300,300))

BMS-##というcompoun_idのついた化合物はマクロサイクル(環状ペプチド)のような分子も含まれているようです。このあたりは分子量が大きそうです。
今回知りたいのは、これらの活性化合物の分子量がどのような値となっているか?、なので分子量を計算しDataframeに加えます。
df['MW'] = df.ROMol.map(Descriptors.MolWt)

分子量の分布がどのようになっているか知りたいので、まずはpandasのdescribe()で要約統計量を取得します。
df['MW'].describe() ''' count 53.000000 mean 819.726075 std 626.128326 min 215.213000 25% 419.525000 50% 574.899000 75% 753.340000 max 2699.165000 Name: MW, dtype: float64 '''
平均値 820 で最大値 2699 ! 予想以上に分子量が大きいです。分子量の大きそうな巨大なマクロサイクルで、分子量の統計量が大きな値へと引っ張られていそうです。
要約統計量だけから判断すると、大きな値に引きづられてしまいそうなので、念のためヒストグラムを作成し、分子量の値の広がりを確認したいと思います。
import matplotlib.pyplot as plt #notebookにグラフを描くためのマジックコマンド %matplotlib inline #MWのMaxが約2700なので 0 から 2800 までの幅50のヒストグラムを作成 edges = range(0,2800,50) n, bins, patches = plt.hist(df['MW'], bins=edges)

分子量500前後と分子量2000を超える分子の大きく2つに山が分かれています。 今回の創薬レイドバトルの目標は低分子のPD/PD-L1の阻害剤を見つけるということですから、分子量500前後の山に相当するような分子を見つけ出すことが目的になりそうです。(そもそも分子量2000を超える分子がライブラリに含まれているか疑問・・・)
分子量が小さい山から、分子量による絞り込みの閾値を決めたいと思います。より具体的な値をみるため各ビンに含まれる数を取り出して見ます。
hist_df= pd.DataFrame(n, bins[1:]).T

分子量300以下は一つしかないため、300を足切りにして絞り込むというのは非常に妥当な値のように見えます。閾値を一瞬で見定めるとは・・・プロすごい・・・
それでは、スクリーニング対象の化合物を検証したいと思います。
データサイズが大きいのでまたしてもGoogle colaboratoryを利用したいと思います。
ちょうど以前の記事で前処理の際に分子量を計算し、圧縮ファイル(Enamine_Premium_collection_pro_id_MW_sdf.gz)としてGoogleドライブに保存していたので、こちらを利用します。
もろもろの設定は省略し、SDFを読み込むところから始めます。
from rdkit import Chem import gzip from rdkit.Chem import Descriptors EPc_gz = gzip.open('Enamine_Premium_collection_pro_id_MW_sdf.gz') #ForwardSDMolSupplierで読み込む。 EPc_suppl = Chem.ForwardSDMolSupplier(EPc_gz) #molオブジェクトのリストを作る段階で、分子量を取得して分子量のみのリストを作成する。 #空のリストを作成 EPc_MW_list = [] EPc_mols = [] #ループを回せ!!! for x in EPc_suppl: if x is not None: mol = x #MolオブジェクトにMWという名前で格納している分子量の値(プロパティ)を取得し、リストに追加する。 #小数点を含むfloat型のプロパティなので GetDoubleProp を使う mw = mol.GetDoubleProp('MW') EPc_MW_list.append(mw) #Molオブジェクトのリストも作成 EPc_mols.append(mol) #総数を確認 len(EPc_MW_list) # 128816と出力された
分子量のリスト(EPc_MW_list)が作成できたので、pandasで処理します。
import pandas as pd df_MW = pd.DataFrame(EPc_MW_list, columns=['MW']) df_MW['MW'].describe() """ count 128816.000000 mean 332.064740 std 32.450643 min 204.277000 25% 312.377000 50% 332.426000 75% 352.398000 max 527.150000 Name: MW, dtype: float64 """
平均 332 で 204 から 527 の範囲の分子量となっているようです。
ヒストグラムを描いてみます。
import matplotlib.pyplot as plt %matplotlib inline #分子量 100 から 600 までの幅50のヒストグラムを作成 edges = range(100,600,50) n, bins, patches = plt.hist(df_MW['MW'], bins=edges)

分子量が300を超えるものについてのみのMolオブジェクトのリストを作成します。
EPc_mols_300 = [] for mol in EPc_mols: MW = mol.GetDoubleProp('MW') if MW > 300: EPc_mols_300.append(mol) len(EPc_mols_300) #109602
残った分子の総数は約11万個。元々のEnamine_Premium_collectionには約13万の分子が含まれていたので、とりあえず2万個程度の分子を減らすことができたようです!
粗い絞り込みでまだまだ残った分子の数は多いですが、最初のフィルタリングとしてはそれなりにうまくいったのではないでしょうか。
sdfとして出力したいと思いますが、前回気づかぬ間にMolオブジェクトのプロパティが失われているというミスを犯したので、まずはプロパティを確認します。
#今あるプロパティのリスト prop_names = [p for p in EPc_mols_300[0].GetPropNames()] print(prop_names) # ['idnumber', 'original_id', 'MW']
きちんとプロパティが保持されていそうです。安心してSDFを作成できます。
#構造と各種プロパティを残したsdfファイルを作成 writer = Chem.SDWriter('Enamine_Premium_collection_MW_filter.sdf') writer.SetProps(prop_names) for mol in EPc_mols_300: writer.write(mol) writer.close() #大きいので圧縮 !gzip -c Enamine_Premium_collection_MW_filter.sdf > Enamine_Premium_collection_MW_filter.sdf.gz #Googleドライブへ出力 upload_file = drive.CreateFile() upload_file.SetContentFile('Enamine_Premium_collection_MW_filter.sdf.gz') upload_file.Upload()
以上、最初のステップとして分子量による絞り込みでした。
まだまだ分子数多い・・・しかもこれより多いデータセットがまだ3つある・・・
締め切りに間に合うのでしょうか???っていうか締め切りいつ???