ChEMBLのHELMモノマーライブラリーを解析した話 〜XMLをDataFrameに変換して部分構造検索〜
マクロ分子の表現HELMについて調べています。HELMは階層的な構造をとっており、モノマー (ex. アミノ酸)を組み合わせてポリマー (ex.ペプチド)を表現します。
HELMの特徴はその表現の拡張性にあり、モノマーライブラリーにオリジナルなモノマーを追加することで非天然な構造を表現することもできます。
一方で、HELM表現は略記(ID)を用いるので、モノマーライブラリーが共有されていない場合、同一のIDで異なるモノマーを指定する危険性があるため、モノマーライブラリを把握しておくことが重要となりそうです。
そこで今回はHELMも扱っているおなじみのデータベース、ChEMBLでどのようなモノマー情報が格納されているか、探ってみたいと思います。
具体的にはXMLファイルで提供されているモノマーライブラリーを読み込んでPandasDataFrameに変換することを目指します。
つまりXML良くわからない私の単なるお勉強メモ!!
- ChEMBL HELM モノマーライブラリーの取得
- XMLの中身
- XML宣言と名前空間
- ElementTreeでXMLを解析
- モノマー情報の取得
- DataFrameへの変換
- DataFrame内の構造式検索
- まとめ
ChEMBL HELM モノマーライブラリーの取得
ChEMBL のHELM モノマーライブラリーをはHELM表記のある化合物のCompound Report Cardから取得することができます。
例えば、GnRH(性腺刺激ホルモン放出ホルモン)アンタゴニストであるアバレリクス(Abarelix)の場合、Compound Report CardにおけるHELM表記は以下のようになっています。
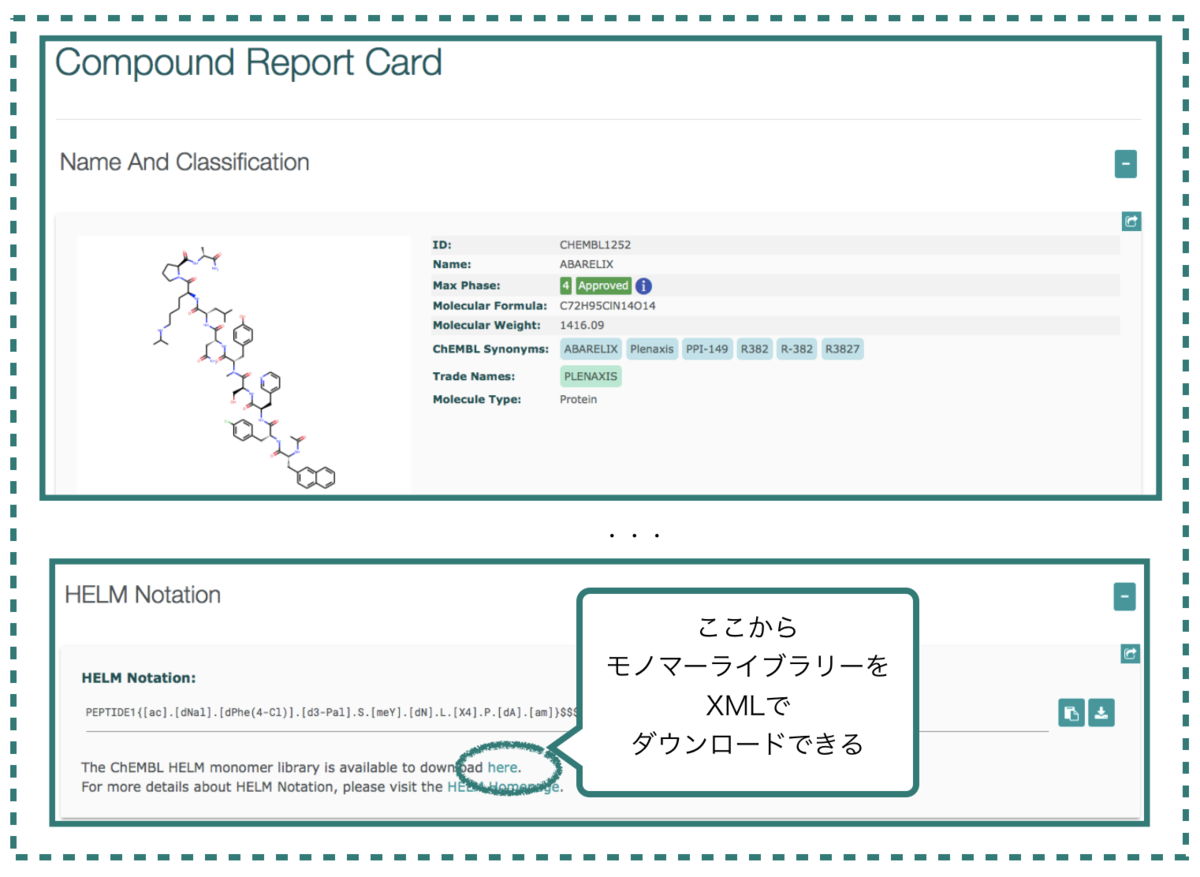
図の真ん中あたりの「here」 というところをクリックするとxmlファイルのダウンロードが始まります。
2020/11/23現在「chembl_27_monomer_library.xml」というファイルが提供されています。
XMLの中身
XMLについてよくわかっていないので、順番にファイルの中身を見ていきたいと思います。
話がそれますがこちらの記事(Macs in Chemistry 「Determining the Amino Acids in a collection of peptides」)でChEMBLモノマーライブラリーXMLの解析事例が紹介されているので、正しい情報が欲しい方はご参照ください。
閑話休題。
とりあえずテキストエディタで開くと中身はこんな感じの構造になっています。

HELMの記載方法同様、モノマーライブラリも階層的な構造となっているようです。
- <PolymerList>
- <Polymer>
- <monomer>
という順にタグが入れ子構造(ネスト)になっており、<monomer> ~ </monomer>ごとに各モノマーの情報が入っているようです。
monomerに記載されている情報は、Pistoia Alliance HPのHELMの解説におけるMonomerと見比べてみると分かりやすいですね。

上図のテーブルに該当するタグがXMLに含まれているようです。
それでは具体的にどのようなモノマーが含まれているのか、その総数や各モノマーの特徴解析を進めていきたいと思います。
・・・・が、早速冒頭2行から意味がわかりませんでした。
XML宣言と名前空間
ファイルの冒頭2行、以下のように記載されています。
<?xml version="1.0" encoding="UTF-8"?> <MonomerDB xmlns="lmr" xmlns:xsi="http://www.w3.org/2001/XMLSchema-instance">
1行目はXML宣言というもので、使用するXMLのバージョンや文字コードについて記述してあるそうです。*1
この場合バージョン1.0、文字コードUTF-8となっているようです。
2行目、タグの中にある「xmlns="lmr"」という箇所は、XMLの名前空間を表しているようです。*2
XMLマスターというこちらのページの説明が分かりやすかったです。
XML名前空間とは、XML文書内にある「定義内容は異なるが同じ名前の要素名または属性名」を区別し、名前の衝突を回避するための仕組みです。(XMLマスターポイントレッスン 〜ベーシック編〜 第11回 XML名前空間 より引用)
ここでは接頭辞(prefix)を使わずに名前空間宣言が行われているため、デフォルトの名前空間に相当する記載となっています。
従って、MonomerDBという要素に対して、lmrをデフォルトの名前空間識別子(URI: Uniform Resource Indentifier)として宣言していることになり、
基本的には全てのタグが{lmr}タグという形に展開されることになります。
一方で、2行目後半「xmlns:xsi="http://www.w3.org/2001/XMLSchema-instance"」 という箇所はXMLのスキーマ定義に関わっているようです。xsiという接頭辞にXML Schema instanceをURIとして宣言しています。*3
どういうこっちゃ?
ElementTreeでXMLを解析
「入門 Python3」によると、XMLの解析にはとりあえずElementTreeを使えば良いとのことなので、試してみます。
ライブラリをインポートした後、まずはparseでファイルを読み込みます。その後getroot()でルートとなるノードを取得して処理を行えば良いそうです。*4
import xml.etree.ElementTree as ET tree = ET.parse('./chembl_27_monomer_library.xml') root = tree.getroot()
タグはtag、属性はattribに入っている、とのことなので確認してみます。
print(root.tag) # {lmr}MonomerDB print(root.attrib) # {}
先に見た通り、デフォルトの名前空間がlmrとなっているため、タグのURIとして{lmr}が付与されています。
また、MonomerDBタグは名前空間の宣言以外の属性がないのでattribは空となっているようです。
子ノードを取得してみます。*5イテレート可能、とのことなのでforループでタグを表示させてみます。
for child in root: print('タグ: ', child.tag, '属性: ', child.attrib) # タグ: {lmr}PolymerList 属性: {} # タグ: {lmr}AttachmentList 属性: {}
PolymerListの他にAttachmentListというタグがあるようです。
PolmerLystのもう一つ下の階層にアクセスします。インデックスを利用してみます。
polymerlist = root[0] print(len(polymerlist)) for child in polymerlist: print(child.tag, child.attrib) # 1 # {lmr}Polymer {'polymerType': 'PEPTIDE'}
PolymerTypeはPEPTIDEのみでした。HELMには他に RNA、SAC、CHEMといったPolymerTypeがありますが、ChEMBLではPEPTIDEのみしか含まれていないようです。
モノマーライブラリーがペプチドのみ、ということがわかったので含まれるモノマーは全てアミノ酸(誘導体)となっていると思われます。
いくつのモノマーが含まれているでしょうか?名前空間のURIに気をつけて{lmr}Monomerの全ての要素をfindall()で取得します。
polymer = root[0][0] monomer_elements = polymer.findall('{lmr}Monomer') print(len(monomer_elements)) # 2851
全部で2851個のモノマーが登録されているようです。PEPTIDEのモノマーってそんなに種類あるんですか???
上記はindexで階層を辿っていますがXPath機能を使うこともできるそうです。.//を使うことで木全体から要素を選択することができます。*6
monomer_elements_2 = root.findall(".//{lmr}Monomer") print(monomer_elements == monomer_elements_2) # True
先に取り出したものと一致することが確認できました。
モノマー情報の取得
ElementとしてMonomerを取り出すことができました。その中身を表示して見ます。
タグで囲まれた内容は.textで確認することができます。
mon = monomer_elements[0] monomer_tag_list = [] for m in mon: monomer_tag_list.append(m.tag) print(m.tag, m.text) # {lmr}MonomerID X2484 # {lmr}MonomerSmiles CC(=O)SCCCC[C@H](N[*])C([*])=O |$;;;;;;;;;;_R1;;_R2;$| # {lmr}MonomerMolFile H4sIAAAAAAAAAI2TTU7DMBCF9z7FSHTLaMb/swbUFQW1iAtUCPX+F2BsN7ZTUYgVRe/Fn1/G48QAnM6Xj6/vy5mYLQcmS/bZGGAP7ACoXlzvY4gIfFoiMsUxOpVNSWiKkMqzJ2gRNxxh+oMjtJFiUxzITdzbzFmMtEoZ6jRzDmXTewPae3krLmLYlJcwlUb9mydInbuTp5tdxFgTc+uCDZSnNYc5OyOFhcte/MQdHwbHhG5LrYN75NturfIYvZPryQWR9PsZct0XN6PKtX024/uMqtCNqtiNqtSNqtyNLCZWw7TMqGKeMO4VqCqf/DD+Ws4rwHH/3mplqEQBwZaZl4P+Ljsd5gd2o4VdTQMAAA== # {lmr}MonomerType Backbone # {lmr}PolymerType PEPTIDE # {lmr}NaturalAnalog X # {lmr}MonomerName X2484 # {lmr}Attachments
モノマーを表す特徴として8つの情報が含まれています。先に図で示したXMLファイルの中身と同様の内容がtextで取り出されているようです。
最後、Attachmentは更に深い階層に情報が書かれています。一つ一つ階層を辿るのが面倒なので手っ取り早くiter()での処理を試してみます。
Elementオブジェクトはiter()で現在の要素を根とする木のイテレータが作成できるそうです。*7
Att = mon.find('{lmr}Attachments') for Att_el in Att.iter(): print(Att_el.tag, Att_el.text) # {lmr}Attachments # {lmr}Attachment # {lmr}AttachmentID R1-H # {lmr}AttachmentLabel R1 # {lmr}CapGroupName H # {lmr}CapGroupSmiles [*][H] |$_R1;$| # {lmr}Attachment # {lmr}AttachmentID R2-OH # {lmr}AttachmentLabel R2 # {lmr}CapGroupName OH # {lmr}CapGroupSmiles O[*] |$;_R2$|
2箇所のAttachmentについてそれぞれの下の階層の情報まで取得することができました。
IDやLabel、CapGroupの情報が含まれているようです。ここでもSMILESはCXSMILESのようですね。
DataFrameへの変換
Monomer情報の取得ができるようになったので、より扱いやすくするためモノマーライブラリーをDataFrameに変換してみたいと思います。
具体的にはRDKitのPandasTools*8を利用して、各行に各Monomerの情報とMolオブジェクトを含むDataFrameを作成します。作成したDataFrameは部分構造検索などが利用できるので、簡易的なデータベースとして使えるはず!
まず、8つの特徴のうち、MonomerMolFileとAttachmentsを除いた以下の6つを対象として、DataFrameのカラムとします。
- MonomerID
- MonomerSmiles
- MonomerType
- PolymerType
- NaturalAnalog
- MonomerName
monomer_tag_list.remove('{lmr}MonomerMolFile') monomer_tag_list.remove('{lmr}Attachments') # 見やすさのため{lmr}を取り除いて列名とする column_names = [s.replace('{lmr}', '') for s in monomer_tag_list]
必要なライブラリを読み込みます。
from rdkit import rdBase, Chem from rdkit.Chem import AllChem, Draw, rdDepictor, PandasTools from rdkit.Chem.Draw import IPythonConsole import pandas as pd # DataFrameの準備 df = pd.DataFrame(columns = column_names )
モノマーごとに取得した情報をリストに格納後、pd.Seriesに変換して、DataFrameに追加していきます。
for m in monomer_elements: data = [] for tag in monomer_tag_list: e = m.find(tag) data.append(e.text) s = pd.Series(data, index=column_names) df = df.append(s, ignore_index=True) print(df.shape) # (2851, 6)
2851個のモノマーを含むDataFrameが作成できました!
中身を確認してみます。
df.head()
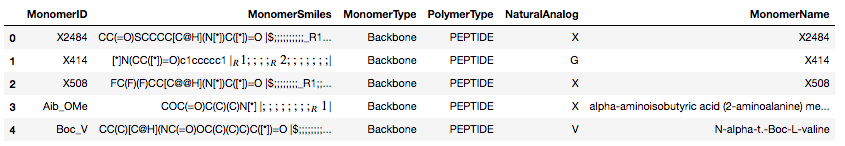
きちんと情報が入っていそうです。
RDKitのPandasToolsを使って各行のMonomerSMILESからROMolオブジェクトを作成します。
PandasTools.AddMoleculeColumnToFrame(df, 'MonomerSmiles')
df.head()
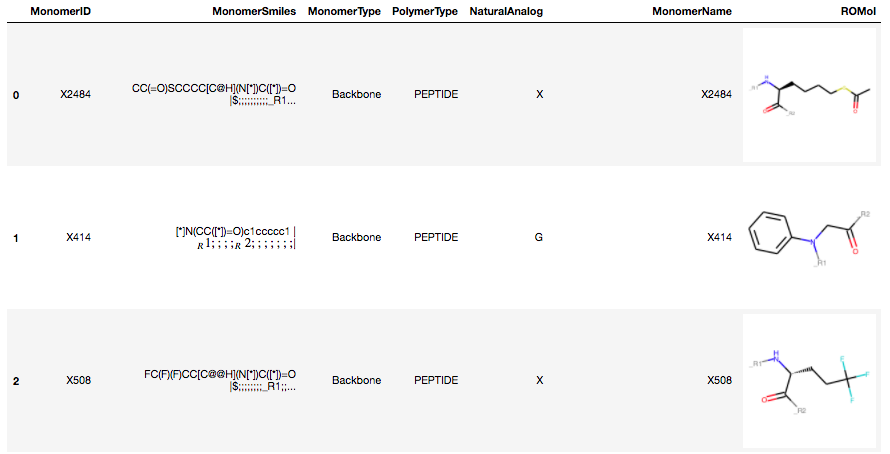
構造式データを含むDataFrameが無事作成できました!
DataFrame内の構造式検索
ではPandasToolsで作成したDataFrameをが構造式データベースとして利用可能か? 部分構造検索等、試していきましょう!
例としてチロシンの誘導体にどのような構造が含まれているか検索してみます。
まずは簡単にNaturalAnalogとしてYが設定されているものを抜き出してみます。
Y_derivatives = df.query('NaturalAnalog == "Y"') print(Y_derivatives.shape) # (176, 7)
176個のチロシン関連構造が含まれているようです。L-チロシンそのものの情報を確認してみます。
Y_derivatives.query('MonomerName == "Tyrosine"')
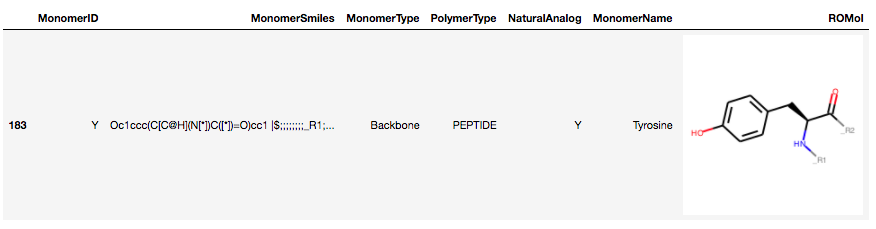
L-Tyrosineがどのように格納されているかがわかったので、この構造情報をつかって部分構造検索を行ってみます。
元のDataFrameから「>= (ge比較演算子)」を使って検索します。
# L-tyrosineのROMolオブジェクトをDataFrameから取得 L_ty = df.iat[183, 6] # ge比較演算子で部分構造マッチ L_ty_sub = df[df.ROMol >= L_ty] print(L_ty_sub.shape) # (77, 7) L_ty_sub.head()
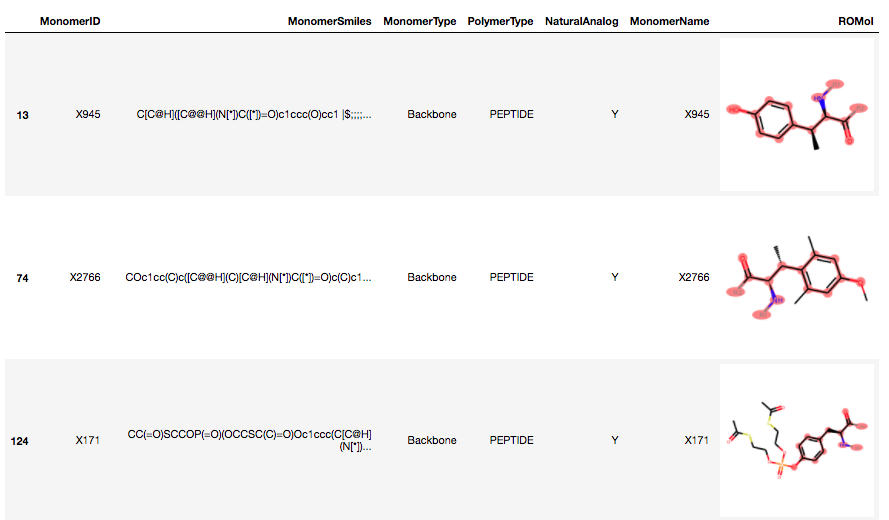
L-tyrosineを部分構造として含むものは77個見つかりました。先にNaturalAnalog == Yとしたものは176個でしたので、チロシンのアナログのなかには元の部分構造自体にも変化が加わっているものがあるようです。
では逆にL-tyrosineと部分構造が一致したもののNaturalAnalogは全てYとなっているのでしょうか?
Analog_vc = L_ty_sub['NaturalAnalog'].value_counts() Analog_vc # Y 72 # X 5 # Name: NaturalAnalog, dtype: int64
NaturalAnalogがXとなっているものも5つあるようです。
L_ty_sub_X = L_ty_sub.query('NaturalAnalog == "X"')
L_ty_sub_X.head()

NaturalAnalogがXとなっていたものは、L-tyrosineと一致する構造をもちつつ、ビシクロ環構造となっているもののようです。
こんな構造もモノマーに入ってるんですね。予想以上に非天然です。。。
試しに一番下のモノマー「MonomerID X316」を含む構造にどのようなものがあるかChEMBLで検索してみました。
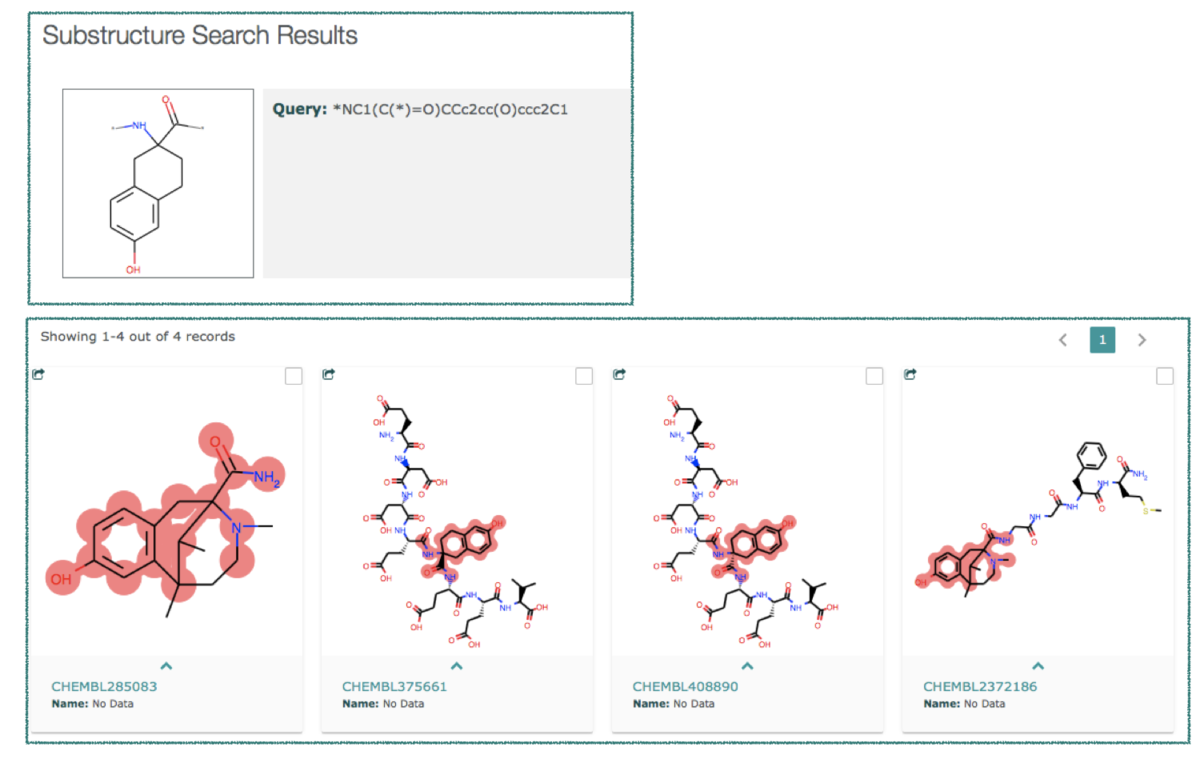
左から2番目、化合物「CHEMBL375661」の情報元の文献は以下でした。
Spatial Conformation and Topography of the Tyrosine Aromatic Ring in Substrate Recognition by Protein Tyrosine Kinases
J. Med. Chem. 2006, 49, 6, 1916–1924
この部分構造が含まれているのも納得なタイトルですね!
まとめ
以上、今回はChEMBLデータベースが提供しているHELMのモノマーライブラリーの中身を解析してみました。
アミノ酸の誘導体だけで3000個近くものモノマーがあるというのはびっくりです。
また、XMLの解析の練習をかねてDataFrameへの変換を行ってみました。RDKitのPandasToolsを使うことで部分構造検索もできるので、うまく使うことができれば簡単なデータベース感覚で利用できそうです。比較演算子が使えることがここまで便利とは思いませんでした。もうちょっと良い使い方があるか考えてみよう。
今回も色々と間違いが多そうです。ご指摘いただければ幸いです。
*1:Let'sプログラミング 「XML宣言を記述する」
*2:ELementTree XML API 「名前空間のあるXMLの解析」
*3:都元ダイスケ IT-PRESS 「XMLのスキーマの定義の仕方(2)」
*4:参考 LIFE WITH PYTHON 「ライブラリ:ElementTree」
*6:ELementTree XML API 「XPath サポート 」
*7:ELementTree XML API 「Element オブジェクト 」
*8:化学の新しいカタチ 「RDKitのPandasToolsでデータ分析を加速する 」