Non-Chemical Functionality 〜RDKit 直訳 Day21〜
(12/30追記)試訳をまとめたテストサイトを作成しました。よろしければご参照ください。
こちらはRDKit直訳 Advent Calendar 2018 - Adventar 21日目の記事です。基本的な進め方は1日目の記事をご覧ください。
本日の訳出に困った用語
binary value: 2進値、バイナリ値
sparsely: まばらな、低密度の
negate the vector: ベクトルの符号を反転する
completion: 補完
tooltip: ツールチップ
以下、訳
化学以外の機能(Non-Chemical Functionality)
[Link] https://www.rdkit.org/docs/GettingStartedInPython.html#non-chemical-functionality
ビットベクトル(Bit vetors)
[Link] https://www.rdkit.org/docs/GettingStartedInPython.html#bit-vectors
ビットベクトルは多数のバイナリ値(例えばフィンガープリントを表すバイナリ値)を効率的に格納するための入れ物です。RDKitには2つのタイプのフィンガープリントがあり、値を内部に格納する方法に違いがあります。2つのタイプは簡単に互いに変換することができますが、それぞれ最適な使用目的は異なっています:
- SparseBitVectsはベクトルに含まれるビットセットのリストだけを保存します。ファーマコフォアフィンガープリントのように非常に大きく、低密度のベクトルを保存するのにとても適しています。立っているビットのリストを取得するといった操作は非常に速いです。一方でベクトルの符号を反転するといった操作はとても、とても遅いです。
- ExplicitBitVectsは立っているビットも立っていないビットも両方の情報を記録します。一般にSparseBitVectsよりも速いですが、保存するのに必要なメモリーが大きくなります。
DIscrete value vectors
[Link] https://www.rdkit.org/docs/GettingStartedInPython.html#discrete-value-vectors
3D grids
[Link] https://www.rdkit.org/docs/GettingStartedInPython.html#d-grids
Points
[Link] https://www.rdkit.org/docs/GettingStartedInPython.html#points
ヘルプの使い方(Getting Help)
[Link] https://www.rdkit.org/docs/GettingStartedInPython.html#getting-help
RDKitのdocistringにはたくさんのドキュメントが含まれており、利用することができます。Pythonのヘルプコマンドを使って参照することができます。
>>> m = Chem.MolFromSmiles('Cc1ccccc1') >>> m.GetNumAtoms() 7 >>> help(m.GetNumAtoms) Help on method GetNumAtoms: GetNumAtoms(...) method of rdkit.Chem.rdchem.Mol instance GetNumAtoms( (Mol)arg1 [, (int)onlyHeavy=-1 [, (bool)onlyExplicit=True]]) -> int : Returns the number of atoms in the molecule. ARGUMENTS: - onlyExplicit: (optional) include only explicit atoms (atoms in the molecular graph) defaults to 1. NOTE: the onlyHeavy argument is deprecated C++ signature : int GetNumAtoms(RDKit::ROMol [,int=-1 [,bool=True]]) >>> m.GetNumAtoms(onlyExplicit=False) 15
コマンドの補完やツールチップを使うことができる環境で作業する場合、利用可能なメソッドをとても簡単に確認することができます。Jupyternotebookをつかった使用例のスクリーンショットはこのようになります:
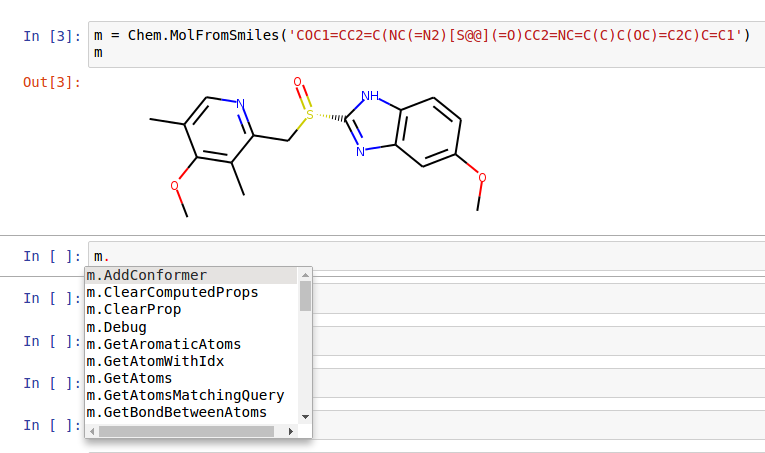
picutre_6.png
発展的なトピック/注意点(Advanced Topics/Warnings)
[Link] https://www.rdkit.org/docs/GettingStartedInPython.html#advanced-topics-warnings
分子の編集(Editing Molecules)
[Link] https://www.rdkit.org/docs/GettingStartedInPython.html#editing-molecules
RDKitで提供されているいくつかの機能では、"その場で"分子を編集することができます。
>>> m = Chem.MolFromSmiles('c1ccccc1') >>> m.GetAtomWithIdx(0).SetAtomicNum(7) >>> Chem.SanitizeMol(m) rdkit.Chem.rdmolops.SanitizeFlags.SANITIZE_NONE >>> Chem.MolToSmiles(m) 'c1ccncc1'
サニタイゼーションを行うのを忘れないようにしてください。忘れると、(深く考えない限り)一見うまくいったかのような結果を返しますが、
>>> m = Chem.MolFromSmiles('c1ccccc1') >>> m.GetAtomWithIdx(0).SetAtomicNum(8) >>> Chem.MolToSmiles(m) 'c1ccocc1'
この結果はもちろん完全にナンセンスで、サニタイゼーションをしようとすれば分かります:
>>> Chem.SanitizeMol(m) Traceback (most recent call last): File "/usr/lib/python2.6/doctest.py", line 1253, in __run compileflags, 1) in test.globs File "<doctest default[0]>", line 1, in <module> Chem.SanitizeMol(m) ValueError: Sanitization error: Can't kekulize mol
より複雑な変換はrdkit.Chem.rdchem.RWMolクラスを使うことで実行できます:
[link] rdkit.Chem.rdchem.RWMol
>>> m = Chem.MolFromSmiles('CC(=O)C=CC=C') >>> mw = Chem.RWMol(m) >>> mw.ReplaceAtom(4,Chem.Atom(7)) >>> mw.AddAtom(Chem.Atom(6)) 7 >>> mw.AddAtom(Chem.Atom(6)) 8 >>> mw.AddBond(6,7,Chem.BondType.SINGLE) 7 >>> mw.AddBond(7,8,Chem.BondType.DOUBLE) 8 >>> mw.AddBond(8,3,Chem.BondType.SINGLE) 9 >>> mw.RemoveAtom(0) >>> mw.GetNumAtoms() 8
RWMolはROMolと同じように使うことができます:
>>> Chem.MolToSmiles(mw) 'O=CC1=NC=CC=C1' >>> Chem.SanitizeMol(mw) rdkit.Chem.rdmolops.SanitizeFlags.SANITIZE_NONE >>> Chem.MolToSmiles(mw) 'O=Cc1ccccn1'
標準的な分子を扱うよりも、RWMolを使った方がナンセンスな結果を出しやすいです。化学的に妥当な結果が必要ならば、必ず結果にサニタイゼーションを行うようにしてください。
12/21/2018
このブログ記事のライセンス
以上はRDKit Documentationの試訳です。
ライセンスはCC BY-SA 4.0で、Greg Landrum氏の功績の元に作成しています。
Creative Commons — Attribution-ShareAlike 4.0 International — CC BY-SA 4.0