LBVSしようとする話
さて、ファーマコフォアを用いた化合物の絞り込みも終わりました・・・と言いたい所ですが、またしても間違いに気づきました。ファーマコフォアサーチに用いたsdfは、オルト位置換基で絞り込む前の、ビフェニルで絞り込んだ段階のものでした!!!
順番は入れ替わってしまいますが、ファーマコフォアスクリーニング後の化合物群に対して部分構造(オルト位置換基)で絞り込みをかけても結果は変わらない、、、はず。。。ということで、改めて部分構造のフィルターをかけます。
from rdkit import Chem from rdkit.Chem import AllChem ortho_biphenyl= Chem.MolFromSmarts('c1ccc(c(*)c1)c1ccccc1') P_Matched2_suppl = Chem.ForwardSDMolSupplier('./P_Matched2.sdf') P_Matched2_mols = [mol for mol in P_Matched2_suppl if mol is not None] print(len(P_Matched2_mols)) #997 #オルト置換ありのリスト ortho_cpds = [] #オルト置換なしのリスト w_o_ortho = [] for mol in P_Matched2_mols: if mol.HasSubstructMatch(ortho_biphenyl): ortho_cpds.append(mol) else: w_o_ortho.append(mol) print('Number of ortho substituted compounds', len(ortho_cpds)) # Number of ortho substituted compounds 494 print('Number of ortho unsubstituted compounds', len(w_o_ortho)) # Number of ortho unsubstituted compounds 503
ファーマコフォアスクリーニングで残った化合物(997個)のうち、オルト位に置換基を有するものは約半数の494個でした。こちらを「P_Matched_ortho.sdf」という名前で保存しておきました。
さて、ようやく今回の本題に、、、
これまで既知の活性化合物の情報をもとに
- 部分構造(オルト置換ビフェニル)の有無
- ファーマコフォア(Aromatic x2、Acceptor x1)を満たすか否か
と、すこしずつ構造を抽象化(?)しながら数を絞り込んできたことになります。次のステップとして、さらに抽象化した基準で、スコアとして評価する、ということを目指したいと思います。
具体的には
- フィンガープリントの使用(複数種類)
- Tanimoto係数による評価
を行います。複数のフィンガープリントを使用するのは、前回の記事で参照した DeNA 望月氏の資料で単一のものを用いるよりも、統合した方が良い結果が出た、との記載があったからです。
1. フィンガープリントによるスコア化
1-1. スコア化手順
具体的な手順としては以下とします。
- 活性化合物群からファーマコフォア基準を満たすものを選ぶ
- フィンガープリントを計算
- 複数の活性化合物で共通するビットを重要なビットとして抜き出す
- 3.で抜き出したビットのベクトルと比較した類似度をスコアとする
- 複数のフィンフガープリントにおけるスコアを統合
1-2. step 1. 活性化合物の選別
まずは活性化合物群から、前回スクリーニングに用いたファーマコフォア基準を満たすものを選別します。すべての化合物を用いないのは、すでにライブラリ側の絞り込みをおこなっているため、条件を等しくした方がファーマコフォアに明示的に取り込めなかった重要な情報を反映させることができるのではないか、と考えたからです。
(ちょっと自分でも何を言っているかよく分からないですが、そんな気がしました。)
同じことの繰り返しなので詳細は省きますが、共闘用シェアデータから抜き出した、環状ペプチドを除く低分子 39個のうち、24個の分子で2段階のファーマコフォアスクリーニングの条件を満たしました。
この化合物群に、共結晶構造から抜き出したリガンド6個を加えて、合計30個をフィンガープリントを計算する対象の活性化合物群とします。(fp_molsというリストに格納しました)
1-3. step 2. フィンガープリントを計算
つづいてフィンガープリントを計算します。以下の6つを使います(Morganを2種類使用)。
- MACCS keys RDKit documentation / Python API
- RDKit RDKit documentation / Python API / Python API
- Morgan(Circular) RDKit documentation / Python API
- AtomPairs RDKit documentation / Python API
- Avalon Python API
これ以外に、Topological Torsions ( RDKit documentation / Python API )も候補にあったのですが、ビット数が多すぎて異音、発熱とともにjupyter notebookのカーネルが落ちたので省きました。
計算にあたっては以下の記事、資料を参考とさせていただきました。
- 参考1: 化合物をベクトルにして比較しプロットする *1
- 参考2: AI創薬のためのケモインフォマティクス入門 6章 *2
- 参考3: 化学の新しいカタチ RDKitでフィンガープリントを使った分子類似性の判定 *3
maccs_fps = [AllChem.GetMACCSKeysFingerprint(mol) for mol in fp_mols] rdkit_fps = [AllChem.RDKFingerprint(mol) for mol in fp_mols] ECFP_fps = [AllChem.GetMorganFingerprintAsBitVect(mol, 2, 2048) for mol in fp_mols] FCFP_fps = [AllChem.GetMorganFingerprintAsBitVect(mol, 2, 2048, useFeatures=True) for mol in fp_mols] Apair_fps = [AllChem.GetAtomPairFingerprint(mol) for mol in fp_mols] from rdkit.Avalon import pyAvalonTools avalon_fps = [pyAvalonTools.GetAvalonFP(mol) for mol in fp_mols]
1-4. step 3. 共通するビットの抜き出し
ついで各フィンガープリントから複数の活性化合物で共通するビットを抜き出します。
まずはフィンガープリントをnumpyのarrayに変換します。(上記、参考記事1 をご参照ください)
MACCS keysを使って、試し実験を行います。
import numpy as np #関数作成(コピペ) def fp2arr(fp): from rdkit import DataStructs arr = np.zeros((1,)) DataStructs.ConvertToNumpyArray(fp, arr) return arr #MACCS keysについてアレイを作成、行列化する maccs_fpMtx = np.array([fp2arr(fp) for fp in maccs_fps])
この行列を使って、以前の記事でPLIFを作成した時と同様の可視化を行おうと思います。
import pandas as pd #行列からDataFrameへ maccs_df = pd.DataFrame(maccs_fpMtx) #各列の合計値をもとめ、DataFrameに追加 sums = maccs_df.sum() maccs_df = maccs_df.append(sums, ignore_index=True) #Heatmapで可視化 %matplotlib inline import matplotlib as mpl import matplotlib.pyplot as plt import seaborn as sns plt.figure(figsize=(20, 10)) sns.heatmap(maccs_df)

少々見づらいですが、縦軸上から「0 ~ 29」が化合物(30個)、一番下の行「30」が各列の合計値です。横軸はMACCS keyの各ビット(167個)です。数に応じて着色されていて、一番下の行をみるとMACCS keyのビットの番号が大きいもので、特に多数の化合物でビットが立っている傾向があることがわかります。
予想以上にわかりにくいので、最後の行を抜き出して棒グラフにします。
plt.figure(figsize=(20, 10)) sums.plot(kind='bar', color='k', alpha=0.7)

要約統計量を眺めて見ます。
sums.describe() """ count 167.000000 mean 10.377246 std 11.409474 min 0.000000 25% 0.000000 50% 3.000000 75% 21.000000 max 30.000000 dtype: float64 """
各ビットは30個の化合物のうち、平均して10個でビットがたっているようです。10個以上の活性化合物でビットが立っていることを基準として、重要なビット、と捉えることにしてみます。
maccs_common_bit = [] for x in sums: if x >= 10: maccs_common_bit.append(1) else: maccs_common_bit.append(0) print(sum(maccs_common_bit)) #75
MACCS keysフィンガープリントのうち、活性化合物群で重要と考えられるビットのベクトルが手に入りました。75 個 (167 個中)から構成されているようです。
上記処理を関数としますが、いちいちデータフレームにするのは意味がなさそうなので合計値と、ビットのアレイをそれぞれ作ることとします。まずはMACCS keysを例に手順を確認してみます。
maccs_size = len(fp2arr(maccs_fps[0])) #合計値を格納するnumpyのarrayを用意 sum_arr = np.zeros(maccs_size) #numpy行列の足し算 for i in maccs_fps: arr = fp2arr(i) sum_arr += arr print(sum_arr.mean()) #10.377245508982035
先ほどと同じ平均値を返しました。DataFrameを介さずにすみそうです。
平均値の小数点以下を切り捨てた整数部分を閾値とするため、mathのfloorを使います
import math maccs_threshold = math.floor(sum_arr.mean()) print(maccs_threshold) #10
閾値を求めるところまで確認できたので関数とします。フィンガープリントのサイズと、閾値、各ビットで30個の化合物で立っている数、そして平均以上の数立っているビットのベクトルの4つを戻り値とします。
def common_bit(fps): fp_size = len(fp2arr(fps[0])) sumarr = np.zeros(fp_size) for i in fps: arr = fp2arr(i) sumarr += arr mean = sumarr.mean() threshold = math.floor(mean) common_bit = [] for x in sumarr: if x >= threshold: common_bit.append(1) else: common_bit.append(0) return fp_size, threshold, sumarr, common_bit
フィンガープリント6種に対して適用します。
fingerprints = [maccs_fps, rdkit_fps, ECFP_fps, FCFP_fps, Apair_fps, avalon_fps] fp_names = ['maccs_fps', 'rdkit_fps', 'ECFP_fps', 'FCFP_fps', 'Apair_fps', 'avalon_fps'] #各返り値を格納するためのリストを用意 fp_size_list = [] threshold_list = [] sumarr_list = [] common_bit_list = [] #計算実行 for fps in fingerprints: sz, th, ar, cb = common_bit(fps) fp_size_list.append(sz) threshold_list.append(th) sumarr_list.append(ar) common_bit_list.append(cb)
各フィンガープリントのサイズと閾値を確認してみます。
for name, size, threshold in zip(fp_names, fp_size_list, threshold_list): print(name, ':', size, ':', threshold) """ maccs_fps : 167 : 10 rdkit_fps : 2048 : 14 ECFP_fps : 2048 : 0 FCFP_fps : 2048 : 0 Apair_fps : 8388608 : 0 avalon_fps : 512 : 10 """
ECFP、FCFP、Atom_pairでは閾値が0となってしまっています。ビットベクトルのサイズの大きさの割に立っているビットが少なく(スパース?)、平均値がとても小さな値となってしまっているのでしょうか???
これでは閾値として不適合なので、フィンガープリント全体ではなく、合計値がゼロではないビットのみについての平均値を代わりに用いたいと思います。
MACCS keysで実験してみます。
#np.nonzero()は戻り値がインデックスであることに注意 non_zero_sum = sum_arr[np.nonzero(sum_arr)] print(non_zero_sum.mean()) #16.349056603773583
今度は閾値が16という結果となりました。MACCS keyにおいて非ゼロのビットのみを考慮すると、30個の化合物群のうち、平均して半数で同一のビットが立っているということになります。(この解釈で正しいでしょうか?自信がありません。)
上記の新しい閾値をとりいれた関数に書き換えます。
def common_bit2(fps): fp_size = len(fp2arr(fps[0])) sumarr = np.zeros(fp_size) for i in fps: arr = fp2arr(i) sumarr += arr NonZeroArr = sumarr[np.nonzero(sumarr)] NonZeroMean = NonZeroArr.mean() threshold = math.floor(NonZeroMean) common_bit = [] for x in sumarr: if x >= threshold: common_bit.append(1) else: common_bit.append(0) return fp_size, threshold, sumarr, common_bit
実行の部分は同じなので省略し、結果のみ記載します。
for name, size, threshold in zip(fp_names, fp_size_list2, threshold_list2): print(name, ':', size, ':', threshold) """ maccs_fps : 167 : 16 rdkit_fps : 2048 : 14 ECFP_fps : 2048 : 4 FCFP_fps : 2048 : 5 Apair_fps : 8388608 : 14 avalon_fps : 512 : 11 """
全て閾値が0ではなくなりました!スパース性を解決した!
レイドバトルは非ゼロサムゲーム!(知らんけど)
くどいですが再度MACCS keysの値を確認します。
print('MACCS NonZero: ', len(non_zero_sum)) #MACCS NonZero: 106 print('MACCS base bits: ', sum(common_bit_list2[0])) #MACCS base bits: 61
MACCS keyに関しては「非ゼロのビットが106個で、このうち61個で閾値よりを満たす」、となりました。先の棒グラフを見てもそこまでおかしな値とはなっていなさそうです。
取り急ぎ、手に入れたビットベクトルを基準としてもちいることとし、辞書に格納しておきます。
base_BitVect_dict={}
for k, v in zip(fp_names, common_bit_list2):
base_BitVect_dict[k] = v
1-5. step 4. 類似度の指標を作成
基準となるビットベクトルが用意できたので、このベクトルに対してスクリーニング対象の化合物のビットベクトルの類似度を計算したいと思います。
タニモト係数を使いたいのですが、numpyのarrayとしており、RDKitのオブジェクトではなくなってしまったのでDataStructs.TanimotoSimilarity()を使えるかわかりません。
Tanimoto類似度の説目を見る限り、ビットが立っている要素について積集合を和集合で割れば良いということのようですから、比較対象のビットベクトル同士を足し合わせて、「2の要素数」を「1 or 2の要素数」で割ってしまえば良さそうです。
まずはファーマコフォアで絞り込んだスクリーニング対象の化合物(オルト位置換も考慮済みのもの)をSDFから読み込み、scr_molsというリストに格納しました。
一番最初の分子をテスト化合物として取り出し、各フィンガープリントを計算したのち、アレイに変換し、辞書型として格納します。
test_mol = scr_mols[0] #関数を作成 def array_dict(mol): maccsFP_arr = fp2arr(AllChem.GetMACCSKeysFingerprint(mol)) rdkitFP_arr = fp2arr(AllChem.RDKFingerprint(mol)) ECFP_arr = fp2arr(AllChem.GetMorganFingerprintAsBitVect(mol, 2, 2048)) FCFP_arr = fp2arr(AllChem.GetMorganFingerprintAsBitVect(mol, 2, 2048, useFeatures=True)) Apair_arr = fp2arr(AllChem.GetAtomPairFingerprint(mol)) avalon_arr = fp2arr(pyAvalonTools.GetAvalonFP(mol)) tmp_dict = {} fp_names = ['maccs_fps', 'rdkit_fps', 'ECFP_fps', 'FCFP_fps', 'Apair_fps', 'avalon_fps'] fp_arrs = [maccsFP_arr, rdkitFP_arr, ECFP_arr, FCFP_arr, Apair_arr, avalon_arr] for k, v in zip(fp_names, fp_arrs): tmp_dict[k] = v return tmp_dict #テスト分子で実行 test_array_dict = array_dict(test_mol)
MACCS key に関して、テスト分子と、先に作成した基準ビットベクトルでTanimoto係数を計算してみます。
#それぞれのビットベクトル(array)を読み込む base_maccsAR = base_BitVect_dict['maccs_fps'] test_maccsAR = test_array_dict['maccs_fps'] #array同士を足し算 maccsAR_sum = base_maccsAR + test_maccsAR #各値の数を確認 test_count_1 = np.sum(maccsAR_sum == 1) print('1 :', test_count_1) # 1 : 32 test_count_2 = np.sum(maccsAR_sum == 2) print('2 :', test_count_2) # 2 : 36 #Tanimoto類似度を計算 test_Tanimoto = test_count_2 / (test_count_1 + test_count_2) print('Tamimoto :', test_Tanimoto) #Tamimoto : 0.5294117647058824
うまくいったようです。関数化します。
def TaniSimCal(arr1, arr2): arr_sum = arr1 + arr2 count1 = np.sum(arr_sum == 1) count2 = np.sum(arr_sum == 2) Tanimoto = count2 / (count1 + count2) return Tanimoto
スクリーニング対象の化合物について、6種類のフィンガープリントのそれぞれのTanimoto類似度を計算します。
Scores = [MACCS_Scores, RDkit_Scores, ECFP_Scores, FCFP_Scores, Apair_Scores, Avalon_Scores] MACCS_Scores = [] RDkit_Scores = [] ECFP_Scores = [] FCFP_Scores = [] Apair_Scores = [] Avalon_Scores = [] for mol in scr_mols: #スクリーニング対象のFPのアレイを作成(辞書) fp_arr_dict = array_dict(mol) #基準の各ビットベクトルに対するTanimoto類似度を計算 for fp, sc in zip(fp_names, Scores): base_arr = base_BitVect_dict[fp] scr_arr = fp_arr_dict[fp] TaniScore = TaniSimCal(scr_arr, base_arr) #各フィンガープリントのスコアリストに追加 sc.append(TaniScore)
スクリーニング対象494化合物の、6種類のフィンガープリントについて、活性化合物群から導いた基準ビットベクトルに対する類似度をプロットしてみました。
for i in range(6): plt.subplot(2, 3, i+1) plt.hist(Scores[i], bins=20, range=(0, 1)) plt.title(fp_names[i])

1-6. step 5. 統合したスコアの作成
上記で求めた値を統合して、各化合物の統合スコアとしたいのですが、このままではフィンガープリントごとにスコアの分布のばらつきが大きいです。Tanimoto類似度にこのような処理をするのが適切かわかりませんが、各フィンガープリントにおいてスコアを標準化(平均 0, 分散 1)したのち、和をとった値を統合したスコアとしたいと思います*4。
import statistics Scores_STD = [] for l in Scores: l_mean = statistics.mean(l) l_stdev = statistics.stdev(l) s = [(i-l_mean) / l_stdev for i in l] Scores_STD.append(s)
標準化したスコアを先と同様にプロットします。
for i in range(6): plt.subplot(2, 3, i+1) plt.hist(Scores_STD[i], bins=20) plt.title(fp_names[i])

標準化が完了したので、各スコアを足しあわせて統合スコアとします。なんとなく0以上の値にしたいので最小値分シフトさせます。
Integrated_SC_arr = np.zeros(494) for i in Scores_STD: Integrated_SC_arr += np.array(i) #最小値を求める int_min = min(Integrated_SC_arr) #最小値の絶対値分シフト Integrated_SC = [i + abs(int_min) for i in Integrated_SC_arr] #グラフ化 plt.hist(Integrated_SC, bins=20)

スコア化が完了したので、SetDoublePropを使って、Molオブジェクトにプロパティ(Similarity_Score)として持たせ、SDFで出力しました(SCR_compounds_SimScore.sdf)。
2. 上位化合物の検証
スコアの付与とデータの書き出しが完了しました。今回設定したスコアで、上位となった化合物はどのようなものか、確認して見たいと思います。PandasToolsでDataFrameに読み込みます。
from rdkit.Chem import PandasTools SIM_df = PandasTools.LoadSDF('./SCR_compounds_SimScore.sdf')
スコアの順に並べ替えたいので、typeをstrからfloatに変換しておきます。
SIM_df['Similarity_Score'] = SIM_df['Similarity_Score'].astype(float)
新しく追加したスコアをつかって降順にソートします。
SIM_df_st = SIM_df.sort_values('Similarity_Score', ascending=False)
スコアの良いものから10個取り出してスコアととともに描画します。
from rdkit.Chem import Draw #Molオブジェクトの取り出し Top_10 = SIM_df_st.iloc[:10, 8] #スコアの取り出しとstrへの変換 Top_10_scores = SIM_df_st['Similarity_Score'][:10] legends = [str(i) for i in Top_10_scores] Draw.MolsToGridImage(Top_10, legends = legends)

かなりそれらしい化合物が選択できてきているのではないでしょうか?
少し想定外だったのは、オルト位に置換基の入ったビフェニル構造でスクリーニングした際に、置換基の大きさ(原子数?)をしていなかったためか、オルト位からそのまま分子が伸長したような化合物が上位にヒットしてきているということです。
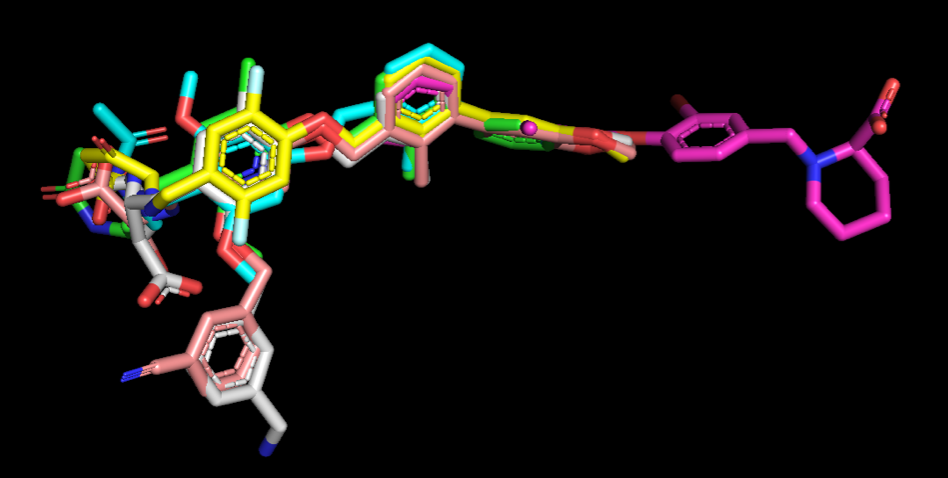
例えばこれまで何度かみてきた下の構造のように、オルト位置換基をもちつつ、他の位置(メタ位など)からさらに分子が伸長している、というのがぼんやりとした理想だったのですが、、、

3. まとめ
今回は、リガンドをベースにしたスクリーニングを行うにあたって、これまでよりも抽象化し、フィンガープリントを使って見ました。また、使用にあたって
- 複数の活性化合物の情報の取り組み
- 複数の種類のフィンガープリントの組み合わせ
というのを試みて見ました。
上位ランク化合物は、なんとなく頭の中で「こういう化合物がひっかかったらいいな」と思っていた構造とはことなりました。適切な情報にまで落とし込むことと、それを表現することがまだまでできていないようです。
ひとまずLBVSっぽいということにしたい!
ところで、創薬レイドバトルの候補化合物絞り込みトップ500と、トップ10の提出みたいなのですが、すでに500個ない。 どうしよう・・・
それっぽいことをやって見たいというだけで、スコアの標準化や統合(アンサンブル?)などかなり適当なことをしています。間違っている点等あればご指摘いただければ幸いです。
*1:化合物をベクトルにして比較しプロットする https://qiita.com/Mochimasa/items/f1b60246ece7da46f6a9
*2: AI創薬のためのケモインフォマティクス入門 https://github.com/Mishima-syk/py4chemoinformatics/blob/
*3: 化学の新しいカタチ「RDKitでフィンガープリントを使った分子類似性の判定」https://future-chem.com/rdkit-fingerprint/
*4: Pythonで正規化・標準化(リスト、NumPy配列、pandas.DataFrame) https://note.nkmk.me/python-list-ndarray-dataframe-normalize-standardize/
リガンドを重ね合わせてファーマコフォアを作る話
前回の記事でファーマコフォアスクリーニングを実施し、約2500個の化合物までリストを絞り込みました。思った以上に減らなかったので、さらなる絞り込みのため、ファーマコフォアを作成した流れを振り返りたいと思います。
- リガンド-タンパク共結晶構造を取得(PDB, 6構造)
- PLIPを使って相互作用を解析
- RDKitでPLIF作成
- 複数の構造で相互作用に関与する残基に着目
- 4.の残基と近接するリガンドの部分構造を選定(フィーチャー、3つ)
- 5.のフィーチャーの位置関係からファーマコフォアを作成
このうち、Step 5、Step 6の部分では、共結晶構造1つ(PDB id: 5NIX)をもとに作成をおこなっています。課題として「フィーチャー3つの選択基準がかなり恣意的だった」、ということが挙げられると思います。そこで今回はもう少し、根拠(?)をもってフィーチャーを選択できるか、残りの共結晶構造5つを眺めたいと思います。
1. 今回の流れ
といっても、ただ単に眺めるだけでは仕方ないので、今回は「複数の構造を重ね合わせて眺める」という点に主眼を置きたいと思います。
モチベーションは2つ・・・
- AI創薬のためのケモインフォマティクス入門 *1 6章、SBDDの項目で異なる化合物の複合体結晶構造の重ね合わせが紹介されていたこと
- ファーマコフォアについてのよもやま話 *2 において、結晶構造以外のアプローチとしてリガンド重ね合わせのアプローチが紹介されていたこと
です。折角、異なるリガンドを含む共結晶構造が複数あるのだから、タンパクごと重ね合わせて、そこに含まれているリガンドの重なりを比較してみれば、リガンドのみの重ね合わせよりもより実際の結合のポーズを意識したヒントが得られるかもしれません。
ということで、まずは
- Pymolで複合体構造をアラインメント
- アラインメント後のリガンドの座標を出力
をやってみます。
2. Pymolでアラインメント
用いる複合体構造を再掲します。①、②、③のグループは以前の記事でPLIFをベースに分けてみたものです。
| Group | PDB entry | Chain | リガンド | リガンドID | リガンド分子量 | 文献 |
|---|---|---|---|---|---|---|
| ① | 5J89 | A/B/C/D | BMS-202 | 6GX | 419.52 | Oncotarget 2016(7)30323 |
| 5N2D | A/B/C/D | BMS-37 | 8J8 | 448.55 | J. Med. Chem. 2017(60)5857 | |
| ② | 5J8O | A/B | BMS-8 | 6GZ | 494.42 | Oncotarget 2016(7)30323 |
| 5N2F | A/B | BMS-200 | 8HW | 497.49 | J. Med. Chem. 2017(60)5857 | |
| ③ | 5NIU | A/B/C/D | BMS-1001 | 8YZ | 594.65 | Oncotarget 2017(8)72167 |
| 5NIX | A/B/C/D | BMS-1166 | 8YQ | 643.13 | Oncotarget 2017(8)72167 |
pymolの設定などに関しては或る化みす途さんのブログ*3を参考にさせてきただきました。
具体的には、以下3ステップです。
- PDBファイル6つをpymolで読み込む
- chain C/Dを除いてA/Bのみにする
「remove (chain C,D)」と打ち込む - アラインメントをとる
例)「align 5j89, 5nix」
意) 5j89を5nixに重ね合わせる
そのままの重ね合わせではうまくいかなかったため、ステップ2(chain A/Bのみを残す)を行なっています。全て5nixに重ね合わせました。
次いで、下記の設定で描画を行いました。
- set cartoon_transparency, 0.8
- リガンドを中心に持ってくる(Command+左クリック)
- 水を消す(object panelのallでH(hide)からwatersを選択)
以下のような見た目となりました。比較は後回しにして、座標の出力へと進みます。

3. リガンドの座標を出力
ついで、重ね合わせからリガンドの座標のみを取り出します。以下のように一つずつpdbファイルで出力しました。
「aligned_5nix.pdb」という名前で、5nixのリガンドのみを含むpdbファイルが出力されました。
出力の方法に関してはPymolWikiの Save *4を参照しました。
- 「save file [,(selection) [,state [,format]] ]」 という形でつかう
- formatとしてpdb以外にもmol、sdfなど多数選択可能
- stateは「Default = -1」(現在の状態のみを保存)
stateに関しては、formatを指定する場合に省略すると、引数がおかしいと怒られたので、Defaultで良くても書き加える必要がありそうです。
出力するリガンドを選択する際に、pymolのwindow上でクリックすると、意図しない残基を選択してしまうことがあったので、「Sequence」でリガンドIDを選択した方がやりやすいと思います。

出力したpdbファイルを再度読み込んで確認します。
アラインメントの情報(変換された座標)を保ったまま、構造が出力されていそうです。
今度はまとめてsdfで出力して見ます。拡張子をファイル名に記載していれば自動でformatを認識して処理してくれる、ということだったので
「save aligned_ligands.sdf」
とだけ、打ち込みました。
ちなみに、pymol上で座標を確認するには目的の構造を選択した後、
- xyz = cmd.get_coords('sele', 1)
- print xyz
とすることで、各原子のxyz座標の3要素のリストを要素とする、リストのリストとして座標が出力されました。PyMolWikiの Get Coordinates I *5によると、他にも色々と方法があるそうです。
4. 重なりの比較
準備ができたのでリガンドの重なりの比較を行なっていきたいと思います。
4-1. 5J89と5N2D (グループ1)
5J89(リガンドID: 6GX)と 5N2D(リガンドID: 8J8)を表示させました。前回もちいたファーマコフォアの3点をメインとする構造のように見えます。

芳香族の重なりはかなり良いようですが、末端の親水性領域についてはズレが大きくなっています。この辺りは溶媒露出面にもなっていそうなので、許容される動きの幅が大きいのかもしれません。
・・・ということは、この親水性部位の座標を狭い範囲で厳格に決めすぎてファーマコフォアを作ることはよくない・・・、ということでしょうか?フィーチャー3点の一つとして3角形をつくる前にもう少し慎重になった方が良かったかもしれません。
4-2. 5N2D (グループ1) と5N2F (グループ2)
同一の論文で報告されている5N2D と 5N2F(リガンドID: 8HW)を表示させました。

5N2Dと5N2Fの大きな違いは、後者において図右端のフェニル環にさらに環構造が付加していることです。これにともない、タンパク側の chain A 残基56 Tyrの位置が大きく動いています。押しのけられた、、、という感じでしょうか?(Induced Fit?)
残念ながら構造が報告されている論文(J. Med. Chem. 2017(60)5857)*6は読めなかったのですが、オープンアクセスの Oncotarget 2017(8)72167で、構造の伸長に伴うTyrの動きが議論されていました。
4-3. 5J89(グループ1) と5J8O (グループ2)
もう一つのペア、5j89 と 5j8O(リガンドID: 6GZ)を表示させました。
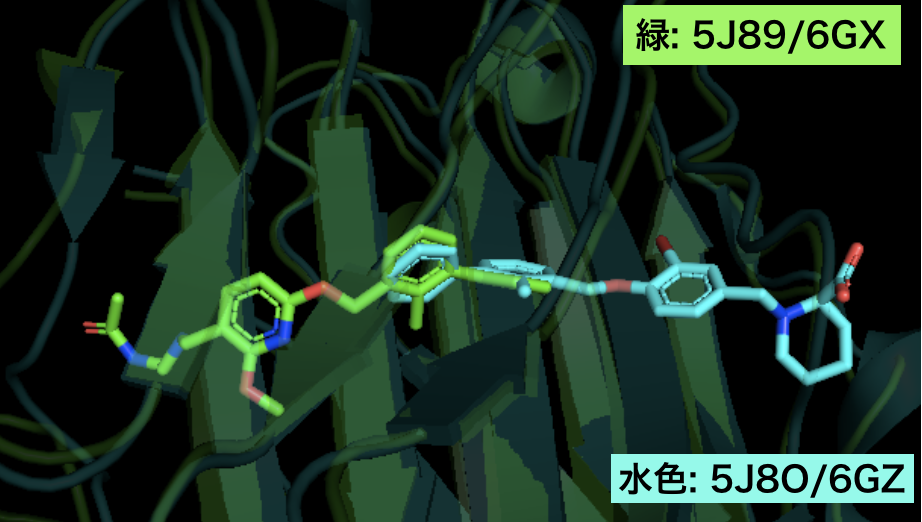
興味深いことに、このペアではビフェニル骨格で重なっているものの対称的な位置に広がっており、リガンド分子全体としての重なりはとても悪くなっています。リガンドベースではなく、タンパク質を基準としたアラインメントになっているため、このような結果になっていると思われます。共結晶構造がPD-L1の対称的な二量体となっていることを踏まえると、確かに対照的な位置に結合してもおかしくないという気もします。(結晶の群とか対称とかわかりません。すみません)
予期せぬ重ね合わせ構造でびっくりしましたが、これはこれで示唆に富む結果だと思います。なぜかというと、共闘用シェアデータに抜き出されている特許構造をながめても、最近のものに移るにつれて、中心に疎水性、両末端に親水性基をもつ対称性のある構造へと変化しているように見えるからです。(構造式は MarvinSketch 18.24.0で作成)

Twitterで拝見したDeNA 望月氏の講演資料「コンペティションから見るAI創薬」において、創薬レイドバトルに関して化合物の線対称に近い構造に着目しているとのコメントがありました(スライド p27)。*7 ご自身で記述子を開発されているとのこと、一体どんな工夫をされているのか?とても結果が楽しみです。
"コンペティションから見るAI創薬" というタイトルで DeNA の AIエンジニアが日本オミックス医学会シンポジウムに登壇しました。
— DeNA Tech (@DeNAxTech) 2019年3月20日
コンペという軸で、AI創薬の近年の動向について紹介しています!https://t.co/EQx48elkL6
対称性のある構造の医薬品
ちょっと脱線しますが、上述の対称性のある構造を見た際にC型肝炎治療薬を思い出しました。C型肝炎治療薬といえば、ハーボニー配合錠が薬価や偽造品流通などで話題となりました。こちらはNS5A阻害剤 レジパスビル と NS5B阻害剤 ソホスブビルを含む合剤となっています。 NS5A阻害剤は複数ありますが、例えば以下のように、分子量が大きく対称的な構造をとっている、という印象です。

経口薬というのが驚きの巨大分子ばかりです。
Wikipedia(英語)の記事「Discovery and development of NS5A inhibitors」に、BMSの研究者によるNS5A 結晶構造の論文(PDB id: 4CL1) (Protein Sci. 2014(23)723)が引用されていました。共結晶構造ではなかったのですが、
- 2量体の構造解析
- ドッキングシミュレーション
等の結果を示すFigureがありました(・・・詳しく読んでません。すみません)。
2量体の形成する疎水性の溝に、対称性の高いリガンド(Daclatasvir : DCV)が結合している図は、これまで見てきたPD-L1の共結晶構造を彷彿とさせるもので、ひょっとしてNS5B阻害剤、PD-L1にもヒットするのでは???という気もしてきます。・・・まあ、無理か。。。
当てずっぽうはさておき、LipinskiのRule of 5からは真っ先に弾かれてしまいそうなこれらの分子が経口薬として上市に至っている、という成功例がPD-1/PD-L1 結合阻害剤においても巨大化、対称化の流れを進める背景にもあったりするのかな、と想像してしまいます。創薬の現場からみたらどうなんでしょう???
4-4. 5J89(グループ1) と5NIU、5NIX (グループ3)
閑話休題。
残りの5NIU(リガンドID: 8YZ)、5NIX(リガンドID: 8YQ)を5J89と重ね合わせます。
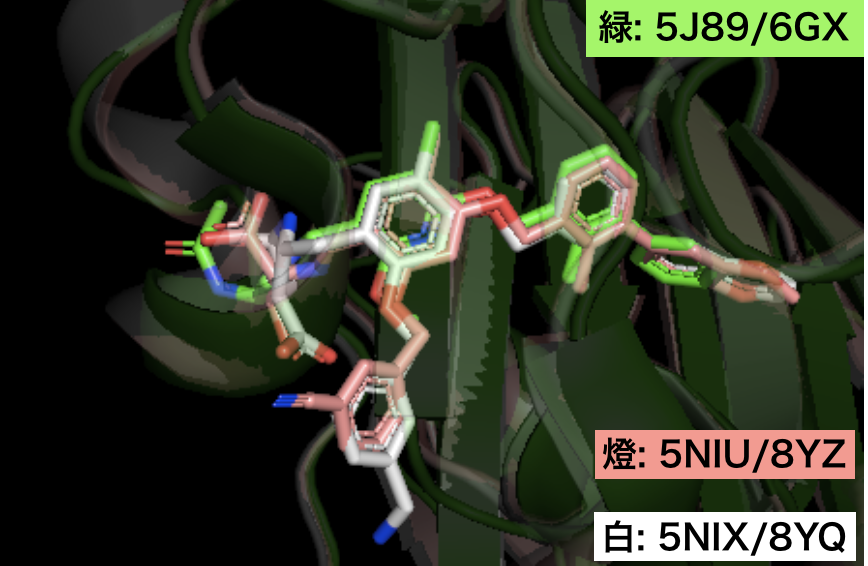
全体として重なりがよく、グループ3の大きな違いはグループ1、2と異なりベンジル基が左下方向にむかって付け足されていることです。文献(Oncotarget 2017(8)72167)*8中では、この置換基が新しい相互作用の獲得に寄与しているとの指摘もなされていました。
重ね合わせ結果の考察
以上、重ねあわせ構造を順番にながめてみました。6つの構造で、前回ファーマコフォアとして選択した3つのフィーチャーを満たしているようにみえ、とりあえずの選択としては十分要件を満たしていたのではないかな?という印象です。
・・・つまり、選択に根拠はあった!(・・・後付け)
共結晶構造中のリガンドをベースに考える場合、更なるポイントとして前回のファーマコフォアに加えて、
- 右末端フェニルからの置換基伸長(疎水性ポケット)
- 左下方向への芳香環伸長
- 左端、親水性基は座標の自由度がある
といった点を考慮していくと良いかもしれません。
5. ファーマコフォアを再作成
5-1. 今回の目標
観察をもとに、更なる絞り込みのためのファーマコフォアを作りたいと思います。
2.左下への芳香環伸長は新しいポケットを埋めるという意味で魅力的ですが、「共闘用シェアデータ」の活性化合物群の流れをみる限り、こちらのポケットは活用しない方向に流れがシフトしているように見えます。そこで、今回は1.右端疎水性ポケットと3. 親水性基の自由度を考慮したファーマコフォアを作成したいと思います。
5-2. Pymolで出力したSDFをRDKitで読み込む
まずは、Pymolで出力したファイルをRDkitで読み込めるか確認して見ます。
from rdkit import Chem aligned_lig_sup = Chem.SDMolSupplier('./aligned_ligands.sdf') aligned_ligs = [x for x in aligned_lig_sup if x is not None]
以下のようなエラーがたくさん出てきました。
RDKit ERROR: [20:29:00] ERROR: non-ring atom 14 marked aromatic RDKit ERROR: [21:10:06] non-ring atom 4 marked aromatic RDKit ERROR: [21:10:06] ERROR: Could not sanitize molecule ending on line 72
MarvinViewでは開き、構造を確認することができたので、どうもRDKitとpymolの相性が悪そうです。
RDKitのエラーメッセージによると、環に含まれていない原子(non-ring atom)に芳香族性が紐づけられていることが問題のようです。SDFの中身を色々とみた結果、エラーがでた構造ではすべてカルボキシ基を有しており、結合表(bond block)においてこの官能基を構成する原子間の結合タイプが「4 = aromatic」となっていることが原因(?)、という結論に達しました。*9
aromatic bondと紐づけられているためaromatic atomと認識された原子が、ring atomではないためエラーとなり、sanitizeもできなかったと思われます。(推定)
5-3. Pymolで出力したpdbをRDKitで読み込む
SDFの中身を修正するのは私には難しいので、PDBファイルとして出力した個々の構造の検証に移りたいと思います。
from rdkit.Chem import Draw pdb_5j8o = Chem.MolFromPDBFile('./aligned_ligands/aligned_5j8o.pdb') Draw.MolToImage(pdb_5j8o)

こちらは読み込み自体はうまくいきましたが、PDBフォーマットに結合次数が記載されていないため、全て単結合、カルボキシル基に至ってはジェミナルジオール構造のように認識されています。
RDKitメーリングリストのディスカッション *10を参考に結合次数を割り当てたいと思います。
手順としては
- smilesからMolオブジェクトを作成(テンプレート)
- AssignBondOrdersFromTemplate(refmol, mol)でテンプレートから次数を割り当て
となります。
from rdkit.Chem import AllChem smi_5j8o = 'Cc1c(COc2ccc(CN3CCCC[C@@H]3C(O)=O)cc2Br)cccc1-c1ccccc1' template_5j8o = Chem.MolFromSmiles(smi_5j8o) mol_5j8o = AllChem.AssignBondOrdersFromTemplate(template_5j8o, pdb_5j8o) Draw.MolToImage(mol_5j8o)

うまくいったようです。残りの5つも同様に処理します。
#pdb idをkey、smilesをvalueとする辞書を作成 smi_dict = {} smi_dict['5j89'] = 'COc1nc(OCc2cccc(c2C)-c2ccccc2)ccc1CNCCNC(C)=O' smi_dict['5n2d'] = 'COc1cc(OCc2cccc(c2C)-c2ccccc2)cc(OC)c1CNCCNC(C)=O' smi_dict['5n2f'] = 'Cc1c(COc2cc(F)c(CNCC(=O)CC(O)=O)cc2F)cccc1-c1ccc2OCCOc2c1' smi_dict['5niu'] = 'Cc1cc(CN[C@H](CO)C(O)=O)c(OCc2cccc(c2)C#N)cc1OCc1cccc(c1C)-c1ccc2OCCOc2c1' smi_dict['5nix'] = 'Cc1c(COc2cc(OCc3cccc(CN)c3)c(C[C@H]3N[C@H](O)C[C@H]3C(O)=O)cc2Cl)cccc1-c1ccc2OC=COc2c1' mol_5j8o.SetProp('PDB_id', '5j8o') #結合次数を割り当てたMolオブジェクトを格納するリストを作成 mols = [mol_5j8o] for k, v in smi_dict.items(): # aligned_xxxx.pdb という名前(xxxxはPDB id)のファイルから作成 path = './aligned_ligands/aligned_' + k + '.pdb' tmp = Chem.MolFromPDBFile(path) template = Chem.MolFromSmiles(v) mol = AllChem.AssignBondOrdersFromTemplate(template, tmp) # PDB idをプロパティとして持たせる mol.SetProp('PDB_id', k) mols.append(mol) #確認 Draw.MolsToGridImage(mols)

#SDFとして書き出しておきます writer=Chem.SDWriter('aligned_pdb_molecules.sdf') writer.SetProps(['PDB_id']) for mol in mols: writer.write(mol) writer.close()
5-4. 重ね合わせでフィーチャーを眺める
出力したSDFをつかって、pymol + RDKitでフィーチャーを描画して見ます。
import os from rdkit import RDConfig fdef = AllChem.BuildFeatureFactory(os.path.join(RDConfig.RDDataDir,'BaseFeatures.fdef')) from rdkit.Chem.Features import ShowFeats from rdkit.Chem import PyMol import subprocess from IPython import display showfeatpath = os.path.join(RDConfig.RDCodeDir, 'Chem/Features/ShowFeats.py') v = PyMol.MolViewer() v.DeleteAll() process = subprocess.Popen(['python', showfeatpath, '--writeFeats', 'aligned_pdb_molecules.sdf'], stdout=subprocess.PIPE) stdout = process.communicate()[0] png=v.GetPNG() display.display(png)

この構造をもとに、5-1. 今回のファーマコフォアの狙いと合わせると、下図のようなフィーチャーの選択が考えられそうです。

"Aromatic" 2つと "Acceptor" 1つの3点という点は以前と同じですが、選択する場所を変えています。
- 右側疎水性ポケットを意識した"Aromatic"の選択
- 複数のリガンドの親水性基が集まる場所を意識した"Acceptor"の選択
としています。
Acceptorの位置を決めるにあたっては、複数のリガンドの親水性基の中心にPseudoatomを作成することとしました。具体的には各リガンドで、該当部位の近辺の親水性基をなす原子を選択し、すべて選んだあとで「pseudoatom hyd, sele」とコマンドを打ち込みました。
Aromatic 2点についてもそれぞれ中心にPseudoatomを作成し、meshで表示すると以下の図のようになりました。

「Wizard → Measurement」で測定した距離と、別に取得した座標情報「例) xyz = cmd.get_coords('hyd', 1)」とを合わせてまとめると以下のようになりました。

なんだか格好良くなってきました!それっぽいぞ!(意味不明)
この情報を使って、再度ファーマコフォアサーチを行います。
5-5. ファーマコフォアを作成
前回と設定値以外変わってませんが念のため・・・
from rdkit.Chem import ChemicalFeatures from rdkit.Chem.Pharm3D import Pharmacophore from rdkit import Geometry feat_1=ChemicalFeatures.FreeChemicalFeature('Aromatic',Geometry.Point3D(-11.796, 10.999, -22.536)) feat_2=ChemicalFeatures.FreeChemicalFeature('Aromatic',Geometry.Point3D(-7.625, 10.407, -21.653)) feat_3=ChemicalFeatures.FreeChemicalFeature('Acceptor',Geometry.Point3D(1.222, 9.145, -28.891)) feats = [feat_1,feat_2,feat_3] pcophore = Pharmacophore.Pharmacophore(feats) # ファーマコフォアの設定 d_upper = 1.5 # 特性基間の距離の許容範囲(上限値) d_lower = 0.5 # 特性基間の距離の許容範囲(下限値) # feat_1とfeat_2の距離 4.3Åを基準に、下限と上限を設定 pcophore.setLowerBound(0,1, 4.3-d_lower) pcophore.setUpperBound(0,1, 4.3+d_upper) # feat_2とfeat_3の距離 11.5Åを基準に、下限と上限を設定 pcophore.setLowerBound(1,2, 11.5-d_lower) pcophore.setUpperBound(1,2, 11.5+d_upper) # feat_1とfeat_3の距離 14.6Åを基準に、下限と上限を設定 pcophore.setLowerBound(0,2, 14.6-d_lower) pcophore.setUpperBound(0,2, 14.6+d_upper) print(pcophore) """ Aromatic Aromatic Acceptor Aromatic 0.000 5.800 16.100 Aromatic 3.800 0.000 13.000 Acceptor 14.100 11.000 0.000 """
新しいファーマコフォアの準備ができました。 関数は前回作成したPSearch3というものを用います。
6. ファーマコフォアサーチ
6-1. サーチ
前回、ファーマコフォアサーチを実施したSDFファイルを読み込みます。
PSerched_suppl = Chem.SDMolSupplier('./PSearched.sdf') PSerched_mols = [x for x in PSerched_suppl if x is not None] print(len(PSerched_mols )) #4220
Embedできた数をPScoreというプロパティに格納しているので、こちらの値が1以上のものを、ファーマコフォアにマッチしたもの(P_Matched)、0のものをマッチしなかったもの(P_MissMatched)として別々のリストに分けます。
P_Matched = [] P_MissMatched = [] for x in PSerched_mols : PScore = int(x.GetProp('PScore')) if PScore >= 1: P_Matched.append(x) else: P_MissMatched.append(x) print('P_Matched:', len(P_Matched)) #2463 print('P_MissMatched:', len(P_MissMatched)) #1757
前回のファーマコフォアにマッチした約2500個について、今回新しく作成したファーマコフォアスクリーニングを適用します。
for x in P_Matched: num_embeds = PSearch3(x) x.SetIntProp('PScore2', num_embeds)
マッチしたものを取り出します。(上と同じなので省略)
#取り出したリストをP_Matched_2、P_MissMatched_2としている。 print('P_Matched:', len(P_Matched_2)) #997 print('P_MissMatched:', len(P_MissMatched_2)) #1466
前回と今回のファーマコフォア両者を満たすものは約1000個でした。 「P_Matched2.sdf」という名前でSDFを出力しました。
6-2. 確認
PandasのDataFrameで読み込んで見ます。
from rdkit.Chem import PandasTools import pandas as pd PSearched2_df = PandasTools.LoadSDF('./P_Matched2.sdf')
今回のスクリーニング(PScore2)の値の分布を取得して見ます。
print(PSearched2_df['PScore2'].describe()) """ count 997 unique 16 top 1 freq 254 Name: PScore2, dtype: object """
最大値や最小値が返ってくるのを期待していたのですが、どうも様子がおかしいです。データ型を確認して見ます。
PSearched2_df.dtypes """ ID object MW object MolLogP object NumHAcceptors object NumHDonors object NumRotatableBonds object PScore object PScore2 object ROMol object TPSA object original_id object dtype: object """
PandasToolsで読み込んだものは全てobject型になっています。こちらの記事「pandasのデータ型dtype一覧とastypeによる変換*11」によると、要素に文字列を含むDataFrameの列はobject型となるらしく、また、化学の新しいカタチさんの記事RDKitのPandasToolsでデータ分析を加速する*12にもPandasToolsで読み込んだ場合は「プロパティが全て「文字列」として認識」されると記載されていました。
astypeで整数型(int)に変換します。
PSearched2_df = PSearched2_df.astype({'PScore': int, 'PScore2': int})
PSearched2_df.describe()
"""
PScore PScore2
count 997.000000 997.000000
mean 14.343029 3.289870
std 12.517565 2.394612
min 1.000000 1.000000
25% 5.000000 1.000000
50% 10.000000 3.000000
75% 20.000000 4.000000
max 99.000000 31.000000
"""
無事出力できました。それぞれの最大値の構造を確認して見ます。
#最大値のindexを取得 PScore_max = PSearched2_df['PScore'].idxmax() PScore2_max = PSearched2_df['PScore2'].idxmax() #Molオブジェクトを取り出し max_mol = PSearched2_df.loc[PScore_max, 'ROMol'] max_mol2 = PSearched2_df.loc[PScore2_max, 'ROMol'] #スコアを構造とともにlegendとして描画する準備 legends = [] for x in [PScore_max, PScore2_max]: score1 = str(PSearched2_df.loc[x, 'PScore']) score2 = str(PSearched2_df.loc[x, 'PScore2']) legend = 'PScore:' + score1 + '/ PScore2:' + score2 legends.append(legend) Draw.MolsToGridImage([max_mol, max_mol2], legends = legends)

前回と今回で同じものがEmbed数最大となっていたようです。
7. まとめ
今回は、より良いファーマコフォアの作成を目指して複数の共結晶構造を重ねあわせる、というステップを加えて見ました。タンパク質をあつかうにはPyMolが非常に使いやすく、Wikiの充実しているので少しずつ機能がわかってきました。
今回のファーマコフォアマッチングの課題としてはEmbedの数しか考慮しておらず、リガンドの3次元構造を実際に発生、最適化させることまでできていないことです。3次元構造発生 → ドッキング みたいな感じに持っていけると良いのですが、、、進捗が悪い。。。
行ったり来たりあちらこちらで躓いていたらとても冗長になってしまいました。おかしな点が多数あると思いますのでご指摘いただければ幸いです。
*1:AI創薬のためのケモインフォマティクス入門 https://github.com/Mishima-syk/py4chemoinformatics/blob/master/pdf/py4chemoinformatics.pdf
*2:ファーマコフォアについてのよもやま話 http://blog.kzfmix.com/entry/1552452149
*3: 話題のXofluzaの変異をPymolで見てみる https://keetaneblog.hatenablog.com/entry/2019/02/08/210016
*4:PymolWiki Save https://pymolwiki.org/index.php/Save
*5:PyMolWiki Get Coordinates I https://pymolwiki.org/index.php/Get_Coordinates_I
*6:https://pubs.acs.org/doi/abs/10.1021/acs.jmedchem.7b00293
*7:コンペティションから見るAI創薬 https://speakerdeck.com/m_mochizuki/ai-drug-discovery-in-the-view-of-competitions
*8:Oncotarget 2017(8)72167 http://www.oncotarget.com/index.php?journal=oncotarget&page=article&op=view&path%5B%5D=20050
*9:化学構造の表記 http://www.metabolomics.jp/wiki/Aritalab:Lecture/Compchem/MOL
*10: https://sourceforge.net/p/rdkit/mailman/message/31844451/
*11:pandasのデータ型dtype一覧とastypeによる変換 https://note.nkmk.me/python-pandas-dtype-astype/
*12: RDKitのPandasToolsでデータ分析を加速する https://future-chem.com/rdkit-pandastools/
ファーマコフォアスクリーニングをする話
前回の記事で共結晶構造を元にしたRDKitのファーマコフォアの作成を実施しました。いよいよこちらを用いてスクリーニングを実施したいと思います。
ファーマコフォアの準備
前回の再掲ですが、SAR News No.19の吉森氏による記事「Chemoinformatics Toolkits を用いた創薬システム開発における ラピッドプロトタイピング」での流れを確認します。
- 座標を使って特性基を定義
- 距離情報を使ってファーマコフォアを設定
- ファーマコフォアサーチ
- 対象のフィーチャーをそもそも含んでいるか?
- フィーチャー間の距離が条件を満たすか?
- 距離情報を拘束条件にして3D構造を発生させる。
このうちファーマコフォアの設定までは、前回のものをそのまま利用します。

import os from rdkit import RDConfig from rdkit.Chem import AllChem from rdkit import Chem from rdkit.Chem import ChemicalFeatures from rdkit.Chem.Pharm3D import Pharmacophore from rdkit import Geometry from rdkit.Chem.Pharm3D import EmbedLib from rdkit.Chem import rdDistGeom Ffactory = AllChem.BuildFeatureFactory(os.path.join(RDConfig.RDDataDir,'BaseFeatures.fdef')) feat_1=ChemicalFeatures.FreeChemicalFeature('Aromatic',Geometry.Point3D(-7.625, 10.407, -21.653)) feat_2=ChemicalFeatures.FreeChemicalFeature('Aromatic',Geometry.Point3D(-1.851, 11.051, -24.019)) feat_3=ChemicalFeatures.FreeChemicalFeature('Acceptor',Geometry.Point3D(-0.100, 13.586, -28.137)) feats = [feat_1,feat_2,feat_3] pcophore = Pharmacophore.Pharmacophore(feats) # ファーマコフォアの設定 d_upper = 1.5 # 特性基間の距離の許容範囲(上限値) d_lower = 0.5 # 特性基間の距離の許容範囲(下限値) # feat_1とfeat_2の距離 6.3Åを基準に、下限と上限を設定 pcophore.setLowerBound(0,1, 6.3-d_lower) pcophore.setUpperBound(0,1, 6.3+d_upper) # Acceptor(feat_3)の代表点の選び方によってfeat_2との距離は[5.0~5.6]の値をとる pcophore.setLowerBound(1,2, 5.0-d_lower) pcophore.setUpperBound(1,2, 5.6+d_upper) # 同様にfeat1とfeat_3は[9.8~11.2]の値をとる pcophore.setLowerBound(0,2, 9.8-d_lower) pcophore.setUpperBound(0,2, 11.2+d_upper)
ファーマコフォアをpcophore、フィーチャーファクトリーをFfactoryとして用意しました。molオブジェクトが与えられた場合に、ファーマコフォアサーチを行う関数を作成します。
ファーマコフォアサーチの関数作成(関数1)
距離情報を拘束条件にした3D構造がうまく発生できた場合、その構造をファーマコフォア合致構造とすることとします。また、合致構造間での順位づけとして、構造がうまく生成できた数(条件を満たすコンフォマーの数)をスコアとしてもちいることとしたいと思います。生成させる構造の最大数(count)は10としました。
具体的な関数を以下のように作成しました。
def PSearch(mol): #水素原子を付加(3次元構造利用のため) molH = AllChem.AddHs(mol) #水素原子を綺麗に整える座標計算 AllChem.Compute2DCoords(molH) #フィーチャーをそもそも持っているか? match, mList = EmbedLib.MatchPharmacophoreToMol(molH, Ffactory, pcophore) print(match) if match == True: #距離の検証 #距離行列の取得 bounds = rdDistGeom.GetMoleculeBoundsMatrix(molH) #ファーマコフォアのマッチング pList = EmbedLib.GetAllPharmacophoreMatches(mList,bounds,pcophore) #pListのそれぞれについてFeatureとマッチする原子のidを取得する num_match = len(pList) phMatches = [] for i in range(num_match): num_feature = len(pList[i]) phMatch = [] for j in range(num_feature): phMatch.append(pList[i][j].GetAtomIds()) phMatches.append(phMatch) #原子のidとファーマコフォアをもとに3次元構造を埋め込む Generated_embeds = [] for phMatch in phMatches: bm,embeds,nFail =EmbedLib.EmbedPharmacophore(molH, phMatch, pcophore,count=5, silent=1) Generated_embeds.append(len(embeds)) return sum(Generated_embeds) else: return 0
関数1の検証とエラー
この関数をもちいて動作確認のため、共闘用シェアデータ ) から取り出した分子のうち、前回用いなかったものを含めて検証します*1。
chain_suppl = Chem.SDMolSupplier('./chain_compounds.sdf', removeHs=False) chain_mols = [x for x in chain_suppl if x is not None]
この化合物群に適用したところ早速以下のエラーで止まりました。
--------------------------------------------------------------------------- KeyError Traceback (most recent call last) <ipython-input-40-74609285009c> in <module>() ----> 1 PSearch(test_m3) <ipython-input-38-904fad3ab3c2> in PSearch(mol) 34 35 for phMatch in phMatches: ---> 36 bm,embeds,nFail =EmbedLib.EmbedPharmacophore(molH, phMatch, pcophore,count=20, silent=1) 37 38 Generated_embeds.append(embeds) ~/.pyenv/versions/anaconda3-4.4.0/lib/python3.6/site-packages/rdkit/Chem/Pharm3D/EmbedLib.py in EmbedPharmacophore(mol, atomMatch, pcophore, randomSeed, count, smoothFirst, silent, bounds, excludedVolumes, targetNumber, useDirs) 427 for idx, stereo in mol._chiralCenters: 428 if stereo in ('R', 'S'): --> 429 vol = ComputeChiralVolume(m2, idx) 430 if (stereo == 'R' and vol >= 0) or (stereo == 'S' and vol <= 0): 431 keepIt = False ~/.pyenv/versions/anaconda3-4.4.0/lib/python3.6/site-packages/rdkit/Chem/Pharm3D/EmbedLib.py in ComputeChiralVolume(mol, centerIdx, confId) 1241 nbrRanks = [] 1242 for nbr in nbrs: -> 1243 rank = int(nbr.GetProp('_CIPRank')) 1244 pos = conf.GetAtomPosition(nbr.GetIdx()) 1245 nbrRanks.append((rank, pos)) KeyError: '_CIPRank'
EmbedPharmacophoreの部分で止まっているようです。ComputeChiralVolumeという箇所のようです。
エラーの出た構造はこちら、
不斉点をもつ構造でエラーが出たようです。
エラーの原因は不斉点?
pythonファイルの該当部分の周辺をみると、nbrは不斉中心の近隣(neigbhbor)の原子を指定しているように見えました。
エラー箇所、
1243 rank = int(nbr.GetProp('_CIPRank'))
は、近隣原子の特性(atom property)を取得した際に、マジックプロパティの一つ_CIPRankの情報が見当たらないと言う内容のようです。
不斉に関する設定なので、CIPはカーン・インゴルド・プレローグ順位則(wikipedia: Cahn–Ingold–Prelog priority rulesの略と推測できます。不斉中心の近隣の原子のCIPRank、ということですから、CIP順位則に従って各原子に割り当てられた番号(rank)、を意味していそうです。
描画を見る限り光学活性体として認識されているのに、この情報が見当たらない、設定されていないというのはなぜ???
おそらくですが、一番最初に水素原子を付加(AllChem.AddHs())した際に、不斉中心(3級炭素)の周りの原子のプロパティ情報(_CIPRank)失われているように見えます。
検証してみたいと思います。
水素原子の取り扱いが原因?
まずは水素原子を付与する前のMolオブジェクトでプロパティを確認します。
不斉中心の情報を取得します。
Chem.FindMolChiralCenters(test_m3)
#[(31, 'S')]
AtomID: 31の原子に不斉 S が割り当てられています。GetNeighbors()を使って近隣の原子のidを取得します。
chiral_center = test_m3.GetAtomWithIdx(31)
nbrs = chiral_center.GetNeighbors()
取得した近隣の原子について、AtomIDと元素記号、問題の_CIPrankを出力します。
for x in nbrs: print('AtomID: ', x.GetIdx(), 'Symbol: ', x.GetSymbol(), 'CIPRank: ', x.GetProp('_CIPRank')) #AtomID: 30 Symbol: C CIPRank: 4 #AtomID: 32 Symbol: C CIPRank: 39 #AtomID: 26 Symbol: N CIPRank: 42
水素原子を付与する前のMolオブジェクトでは問題なくCIPRankが割り当てられています。値の意味はわかりません・・・・(1,2,3,4の順位かと思っていましたが、なんらかの基準でもっと広い範囲の値が割り当てられ大小を比較していそう・・・)
続いて水素を付与した場合の確認をします。
test_m3H = AllChem.AddHs(test_m3)
AllChem.Compute2DCoords(test_m3H)
Chem.FindMolChiralCenters(test_m3H)
#[(31, 'S')]
不斉中心を持つと言うことは認識されていそうです。
chiral_centerH = test_m3H.GetAtomWithIdx(31)
nbrsH = chiral_centerH.GetNeighbors()
for i in nbrsH:
print('AtomID: ', i.GetIdx(), 'Symbol: ', i.GetSymbol())
#AtomID: 30 Symbol: C
#AtomID: 32 Symbol: C
#AtomID: 26 Symbol: N
#AtomID: 79 Symbol: H
CIPRankを確認します。
nbrsH[0].GetProp('_CIPRank') """ --------------------------------------------------------------------------- KeyError Traceback (most recent call last) <ipython-input-99-89378a1786f7> in <module>() ----> 1 nbrsH[0].GetProp('_CIPRank') KeyError: '_CIPRank' """
付与した水素原子だけでなく、もともとあった炭素原子からも_CIPRankの情報が失われていました。
立体化学を再度割り当てれば良いのかと思い、Chem.AssignStereochemistry()を使ってみましたが、_CIPRankの情報は付与されていませんでした。
そもそも水素原子を付加していたのは、3次元構造を扱うためでした。今回、embedできたか否かを判定するだけ(3次元構造の最適化といった処理をしない)ですので、水素原子付加をしなくても良さそうです。
したがって、最初の2つの処理
molH = AllChem.AddHs(mol) AllChem.Compute2DCoords(molH)
を除いてしまえば良さそうです。
エラーを回避した関数作成(関数2)
上記2つを除いた関数をPSearch2として作成しました。(コード省略)
この関数を用いたところエラーで止まることなく最後まで計算できました。
PScore2_list = [] for x in chain_mols: num_embeds = PSearch2(x) PScore2_list.append(num_embeds) print(num_embeds) x.SetIntProp('PScore', num_embeds) print(PScore2_list) #[4, 62, 0, 7, 7, 2, 11, 78, 0, 0, 0, 0, 0, 60, 0, 2, 0, 0, 51, 0, 75, 0, 0, 29, 157, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 14, 0, 0, 20]
0の数が多いようなので確認してみます。
#リストから要素が0となっている数を求める print( len([i for i, x in enumerate(PScore2_list) if x == 0]) ) #24
活性化合物群のなかでもembedできた数が0となる構造が多数ありました(24個)。
実行結果で気になる点として、
RDKit DEBUG: [22:55:56] DEBUG: Removed embedding due to chiral constraints.
という表示が多く出力されました。不斉の制限のせいで弾かれている構造が多そうです。かなり荒い3次元構造の取り扱いをしているので、不斉点にそこまで強い制約を加えない方が良い気がします。
不斉点を考慮しない関数の作成(関数3)
今回の計算にあたって、不斉点を無視して処理したいと思います。具体的にはSMILESに変換(オプションでisomericSmiles=Falseとする)して不斉の情報を除いてからMolオブジェクトに戻すという処理を加えます。
#そのままSMILESに変換した場合 test_iso_smi = Chem.MolToSmiles(test_m3) print('chiral:', test_iso_smi) #出力: chiral: Cc1c(COc2cc(OCc3cncc(C#N)c3)c(CN3CCCC[C@H]3C(=O)O)cc2Cl)cccc1-c1cccc(OCCCN2CCC(O)CC2)c1C #立体の情報を除いてSMILESに変換した場合 test_smi = Chem.MolToSmiles(test_m3, isomericSmiles=False) print('achiral: ', test_smi) #出力: achiral: Cc1c(COc2cc(OCc3cncc(C#N)c3)c(CN3CCCCC3C(=O)O)cc2Cl)cccc1-c1cccc(OCCCN2CCC(O)CC2)c1C #もう一度Molオブジェクトに戻す test_mol_re = Chem.MolFromSmiles(test_smi) AllChem.Compute2DCoords(test_mol_re) #立体の確認 print(Chem.FindMolChiralCenters(test_mol_re)) #空のリストが出力された
isomericSmiles=Falseとしたものでは@Hがなくなっており、またChem.FindMolChiralCenters()が空のリストを返すことから、不斉の情報がなくなっていることが確認できました。
上記の処理を加えた関数を再定義します。
def PSearch3(mol): #水素原子を付加しない smi = Chem.MolToSmiles(mol, isomericSmiles=False) mol_re = Chem.MolFromSmiles(smi) AllChem.Compute2DCoords(mol_re) #フィーチャーをそもそも持っているか? match, mList = EmbedLib.MatchPharmacophoreToMol(mol_re, Ffactory, pcophore) if match == True: #距離の検証 #距離行列の取得 bounds = rdDistGeom.GetMoleculeBoundsMatrix(mol_re) #ファーマコフォアのマッチング pList = EmbedLib.GetAllPharmacophoreMatches(mList,bounds,pcophore) #pListのそれぞれについてFeatureとマッチする原子のidを取得する num_match = len(pList) phMatches = [] for i in range(num_match): num_feature = len(pList[i]) phMatch = [] for j in range(num_feature): phMatch.append(pList[i][j].GetAtomIds()) phMatches.append(phMatch) #原子のidとファーマコフォアをもとに3次元構造を埋め込む Generated_embeds = [] for phMatch in phMatches: bm,embeds,nFail =EmbedLib.EmbedPharmacophore(mol_re, phMatch, pcophore,count=5, silent=1) Generated_embeds.append(len(embeds)) return sum(Generated_embeds) else: return 0
関数3の検証
活性化合物群に適用します。
PScore3_list = [] for x in chain_mols: num_embeds = PSearch3(x) PScore3_list.append(num_embeds) #Molオブジェクトのプロパティに結果を保持させる x.SetIntProp('PScore', num_embeds)
今回はRDKit DEBUGは出ませんでした。期待した挙動を示していそうです。 embedできなかった数を数えます。
False_list = [i for i, x in enumerate(PScore3_list) if x == 0] print(False_list) #出力: [8, 14, 21, 25, 27, 28, 29, 30, 31, 32, 33, 34] print(len(False_list)) #出力: 12
今回はembedできた数が0となった構造は12個でした。キラルの制約を無くしたことで、活性化合物群のファーマコフォアベースのヒット率が上がったようです。
embedできなかった化合物について具体的な構造を一つ見てみます。
test_m8 = chain_mols[8] print(test_m8.GetProp('PScore')) # 0 Draw.MolToImage(test_m8)

Property「'PScore'」に値が保持されていることが確認できました。
ipywidgetsをつかって順番に眺めてみます。
from ipywidgets import interact,fixed,IntSlider import ipywidgets def False_mols(idx): mol_idx = False_list[idx] mol = chain_mols[mol_idx] return(display(Draw.MolToImage(mol))) interact(False_mols, idx=ipywidgets.IntSlider(min=0,max=len(False_list)-1, step=1));
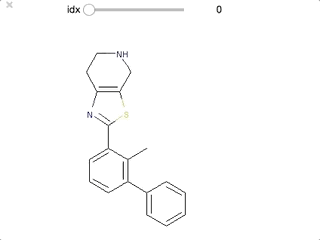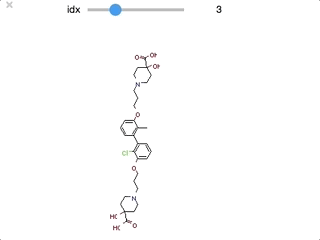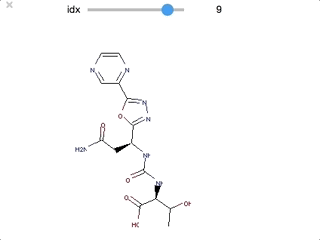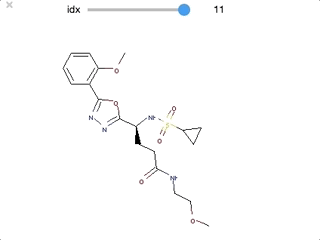
最初のいくつかはファーマコフォアを満たすとして認識されても良さそうですが、後半は芳香環を一つしかもたないなど、確かに要件を満たしていない、ということがわかります。
スクリーニングの実施
スクリーニング対象の再確認
関数の動作確認ができたので、いよいよ本題のスクリーニングに進みたいと思います。
スクリーニング対象の化合物群に関しては、以下の処理をこれまでに行いました。(だいぶ日があいたので再確認)
| 処理 | 内容 | |
|---|---|---|
| ① | 指標による絞り込み | Rule of Five & フラグメントライクな化合物の除去 |
| ② | 部分構造で絞り込み-1 | ビフェニル構造をもつもの |
| ③ | 部分構造で絞り込み-2 | オルト位に置換基を持つもの |
指標としては以下を用いています。
| 指標 | 分子量 | LogP | 水素結合供与体数 | 水素結合受容体数 | 回転可能結合数 | 極性表面積 |
|---|---|---|---|---|---|---|
| Rule of 5 | ≦500 | ≦5 | ≦5 | ≦10 | ||
| 基準 | >300 | >3 | >3 | >3 | >60 |
上記の結果として4220個の化合物まで絞り込み、biphenyl_library.sdfというSDFで保存しています。
スクリーニング本番
この化合物群にファーマコフォアサーチを実施します。
biphenyl_suppl = Chem.SDMolSupplier('./biphenyl_library.sdf') biphenyl_mols = [x for x in biphenyl_suppl if x is not None] biphenyl_PScores = [] for x in biphenyl_mols: num_embeds = PSearch3(x) biphenyl_PScores.append(num_embeds) x.SetIntProp('PScore', num_embeds)
ヒット(一つ以上ファーマコフォアを満たす構造がembedできた化合物)の数を確認します。
Hit_list = [i for i, x in enumerate(biphenyl_PScores) if x != 0] print(len(Hit_list)) # 2463
合計2463個の化合物が残りました。約半数に絞り込めたようです。
上記の計算過程で、
RDKit ERROR: [23:37:56] DEBUG: Removed embedding due to c[00:09:28] UFFTYPER: Unrecognized charge state for atom: 1
といったエラーが出ましたが、4つだったので今回は深追いするのをやめようと思います。(困難から逃げるダメな大人)
embedの数が最も多かった化合物を確認してみます。
print([i for i, x in enumerate(biphenyl_PScores) if x == max(biphenyl_PScores)]) # [1351] max_mol = biphenyl_mols[1351] max_mol.GetProp('PScore') #'99' Draw.MolToImage(max_mol)
数は99で、構造は以下でした。
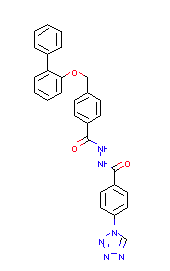
・・・ほとんどsp2です。溶解度悪そう、、、
とりあえず結果をSDFで出力しておきます。
#保持するためのpropertyを確認 check_mol = biphenyl_mols[0] prop_names = [p for p in check_mol.GetPropNames()] print(prop_names) # ['original_id', 'MW', 'MolLogP', 'NumHDonors', 'NumHAcceptors', 'NumRotatableBonds', 'TPSA', 'PScore'] writer = Chem.SDWriter('PSearched.sdf') writer.SetProps(prop_names) for mol in biphenyl_mols: writer.write(mol) writer.close()
おしまい。
まとめ
今回は、前回作成したファーマコフォアをもちいて、スクリーニングを実施してみました。不斉点を持つ化合物に対して期待した挙動をしめさず、いくつか修正が必要となりましたが、最終的に約4000個の化合物から、2500個程度に絞ることができました。思っていたほど減りませんでしたが、ファーマコフォアの設定ポイント(フィーチャー)として、ビフェニル骨格の芳香環2つを選んでいることに由来しているかもしれません。すでに「ビフェニル構造を部分構造として持つ」という絞り込みを行なったリストに対してスクリーニングを実施しているので、「より数を絞り込む」と言う観点からはビフェニル以外のフィーチャーを選択すべきであったかもしれません。
ファーマコフォアを作ろうとする話
前回の記事でRDKitのファーマコフォアがどのような化学的構造を認識しているのか、その定義を眺めました。実際の化合物に適応するとどうなるのか、活性化合物のデータセット (共闘用シェアデータ ) から取り出した分子(マクロサイクル型を除いたもの)で試したいと思います。
- 1. 活性化合物で検証
- 2. 共結晶構造をつかって検証
- 3. 共結晶構造をもとにファーマコフォアを作成
- 4. RDKitでファーマコフォアを設定
- 5. ファーマコフォアを基準に共結晶構造に重ね合わせ
- まとめ
1. 活性化合物で検証
1-1. フィーチャーの探索
まずは活性化合物群を読み込みます。
import os from rdkit import RDConfig from rdkit.Chem import AllChem from rdkit import Chem chain_suppl = Chem.SDMolSupplier('./chain_compounds.sdf', removeHs=False) chain_mols = [x for x in chain_suppl if x is not None]
FDefファイルを読み込んでフィーチャーファクトリーを作成します。
fdef = AllChem.BuildFeatureFactory(os.path.join(RDConfig.RDDataDir,'BaseFeatures.fdef')) print(fdef.GetFeatureFamilies()) #('Donor', 'Acceptor', 'NegIonizable', 'PosIonizable', 'ZnBinder', 'Aromatic', 'Hydrophobe', 'LumpedHydrophobe')
一つ目の化合物を取り出し、特性基(フィーチャー)の探索を行います。
test_m = chain_mols[0] # そのままfeatureを検索 rawFeats = fdef.GetFeaturesForMol(test_m) len(rawFeats) #17
17個のフィーチャーが認識されました。各フィーチャーがどのフィーチャーファミリーのものか確認します。
feat_fam = [] for feat in rawFeats: feat_fam.append(feat.GetFamily())
Python標準ライブラリcollectionsを使って、上記のリストの各要素の個数を確認します。*1
import collections c = collections.Counter(feat_fam) print(c) # Counter({'Hydrophobe': 5, 'Acceptor': 4, 'Aromatic': 3, 'Donor': 2, 'LumpedHydrophobe': 2, 'PosIonizable': 1})
用意されている8種類ファミリーのうち「'Hydrophobe', 'LumpedHydrophobe', 'ZnBinder'」の3つは特殊なように思えたので、より基本的な残りの5種類「'Donor', 'Acceptor', 'NegIonizable', 'PosIonizable', 'Aromatic'」を使うことにします。*2
keep = ('Donor','Acceptor','NegIonizable','PosIonizable','Aromatic') featLists = [] for feat in rawFeats: # ファミリーを取得し、keepに含まれている時のみリストに追加 if feat.GetFamily() in keep: featLists.append(feat) len(featLists) # 10
Hydrophobe(5個)とLumpedHydrophobe(2個)を除くと、基本的なフィーチャーは10個となりました。
1-2. フィーチャーの可視化
@iwatobipen先生の記事visualize-pharmacophore-in-rdkitを参考に、フィーチャーとして認識された構造を眺めます。
atom_ids_list = [] legend_list = [] for feat in featLists: # feat_family_list.append(feat.GetFamily()) atom_ids_list.append(feat.GetAtomIds()) # feat_type_list.append(feat.GetType()) legend_list.append(feat.GetFamily() + ':' + feat.GetType()) from rdkit.Chem.Draw import IPythonConsole from rdkit.Chem import Draw Draw.MolsToGridImage([test_m]*10, molsPerRow=5, subImgSize=(200, 200), legends=legend_list, highlightAtomLists=atom_ids_list)

水素結合ドナー(Donor)としてはアミンやアミドのNH、アクセプターとしてはアミド結合のカルボニル酸素、エーテル結合の酸素原子が認識されています。また、ピリジンのNは塩基性があるので、プラス電荷を帯びる塩基性グループ(PosIonizable:BasicGroup)としても良さそうですが、デフォルトのフィーチャーファクトリーでは、水素結合アクセプターとして認識されるようです(下段左端)。
1-3. SMARTSの表現を再確認
前回フィーチャーのSMARTSを眺めましたが、ピリジンのNを認識していると思われる部分を再度確認しましょう。 AcceptorファミリーのフィーチャーAcceptor.SingleAtomAcceptor、「[n;+0;!X3;!\$([n;H1](cc)cc)]」あたりでしょうか?
SMARTSviewerを使ってみます。*3

「1」で囲まれた芳香族NH(ピロールのような構造?)ではない(「!:否定」)、芳香族Nと解釈できそうなので、ピリジンNを認識しそうです。
もっとかっこよく眺めたい!ということで、RDKitとPyMolの組み合わせを使ってみたいと思います。
1-4. PyMol x RDKit
@iwatobipen先生の記事Visualize pharmacophore with RDKitとGreg先生のノートShow_Ph4_Features_in_PyMOL.ipynbを参考にさせていただきました。
まず、ターミナルでpymolを xml-rpc(remote procedure call)サーバーモードで起動します。
# ターミナル pymol -R
jupyter notebookに戻って以下を実行します。
from IPython import display from rdkit.Chem import PyMol IPythonConsole.ipython_useSVG=True # viewerを作成 v = PyMol.MolViewer() # viewerを一旦綺麗に掃除 v.DeleteAll() # 先の構造を表示 v.ShowMol(test_m)
先に起動していたpymolを眺めると構造式が出ていました。グリグリ回してポーズを決めてから次のコマンドをjupyter notebookで実行するとノート上に静止画(PNG)が表示されました。
png=v.GetPNG() display.display(png)

PyMol綺麗・・・
PyMolが無事動いたので、フィーチャーの可視化に進みます。
1-5. PyMolでフィーチャーを可視化
フィーチャーを表示させるための関数を作成します。さっぱりわからないのでGreg先生のipynbからコピペさせていただきます。
RDKitのFeatures.ShowFeatsモジュールを使っているようですが、CGOなど全くわからない・・・
# コピペ from rdkit.Chem.Features import ShowFeats def showMolFeats(mol,factory,viewer,name="mol",showOnly=True): featLabel = f'{name}-feats' dirLabel = f'{name}-feats-dirs' if(showOnly): ShowFeats._globalSphereCGO = [] ShowFeats._globalArrowCGO = [] # workaround for what is either a bug in the way pymol handles CGOs # or a gap in my understanding of how it should work viewer.server.resetCGO(featLabel) viewer.server.resetCGO(dirLabel) viewer.server.sphere((0, 0, 0), .01, (1, 0, 1), featLabel) viewer.server.cylinder((0, 0, 0), (.01, .01, .01), .01, (1, 0, 1), dirLabel) ShowFeats.ShowMolFeats(mol,factory,v,name=name,featLabel=featLabel,dirLabel=dirLabel, useFeatDirs=False,showOnly=showOnly) viewer.server.renderCGO(ShowFeats._globalSphereCGO,featLabel,1) viewer.server.renderCGO(ShowFeats._globalArrowCGO,dirLabel,1)
フィーチャーを眺めたい分子(molオブジェクト)はtest_m、フィーチャーファクトリーはfdef、ビューワーはv、という名前にしているので、これらを引数にして上の関数を使います。
showMolFeats(test_m,fdef,v)
pymolに移動して回転してから、jupyter notebookに表示します。
png=v.GetPNG() display.display(png)

格好いい!!
認識されたフィーチャーが球として表示されています。
先にMolsToGridImageで並べて見ていた時との違いは、黄土色の球の部位です。
表示されないようにFeatureFamilyから除いた「Hydrophobe」や「LumpedHydrophobe」のようです。
2. 共結晶構造をつかって検証
2-1. 共結晶構造のリガンドでフィーチャーを確認
かなりいい感じでフィーチャーを眺められるようになってきたので、PLIPを検証した記事で眺めた共結晶構造(PDB id: 5NIX)のリガンド(ID: 8YQ)でも、同じことをしてみようと思います。
まず、PDBからリガンドの構造をSDFでとってきて、Chain Aに含まれるリガンドの座標のみ残して保存しました(8YQ_noH.sdf)。
suppl_8YQ = Chem.SDMolSupplier('./8YQ_noH.sdf') mol_8YQ = [x for x in suppl_8YQ if x is not None][0] showMolFeats(mol_8YQ,fdef,v) png=v.GetPNG() display.display(png)

なかなかごつい感じになりました。ほとんどの原子がフィーチャーに認識されていて、単なるBall&Stick表示のようにも見えてしまいます。
このフィーチャーのなかから、PLIPで探した「複数の共結晶構造に共通する相互作用」に該当する「フィーチャー」を取り出して組み合わせればファーマコフォアのモデルができるはず!
2-2. 共結晶構造の相互作用をPyMolで眺める
まずは、取り出した相互作用形式を再度確認します。
「PDB id:5NIX(Ligand:8YQ)」の場合・・・
| 残基番号 | アミノ酸 | 距離 | 相互作用 | 残基番号 | アミノ酸 | 距離 | 相互作用 | ||
|---|---|---|---|---|---|---|---|---|---|
| 8YQ(chain A/B) | 8YQ(chain C/D) | 54C | ILE | 3.89 | Hydrophobic | ||||
| 56B | TYR | 3.82 | Hydrophobic | 56C | TYR | 3.42/3.62 | Hydrophobic | ||
| 66B | GLN | 3.72 | Hydrophobic | ||||||
| 115A/B | MET | 3.70/3.80 | Hydrophobic | 115C/D | MET | 3.85/3.82 | Hydrophobic | ||
| 121A/B | ALA | 3.69/3.75 | Hydrophobic | 121C/D | ALA | 3.96/3.57 | Hydrophobic | ||
| 123A | TYR | 3.80/3.52/3.85 | Hydrophobic | 123D | TYR | 3.72 | Hydrophobic | ||
| 124A | LYS | 5.03/3.18 | π-cation/Salt Bridges | 124D | LYS | 3.70 | Water Bridges/Salt Bridges |
このうちChain A/B の残基(左半分)についてpymol上でハイライトして眺めて見ます。
pymolの設定などに関しては或る化みす途さんのブログ話題のXofluzaの変異をPymolで見てみるを参考にさせてきただきました。
「5nix.pdb」を表示させた後、
- set cartoon_transparency, 0.8
- リガンドを中心に持ってくる(Command+左クリック)
- 水を消す(object panelのallでH(hide)からwatersを選択)
- 見たい残基を選択(sequenceを表示「右下Sをクリック」して順番に選択)
- 残基をLine表示(object panelの(sele)でS(show)からas→wire→lines)
とします。スナップショットを保存するには、右上の「Draw/ray」というボタンから出力したいサイズを選んでDraw→save image to fileとしてやれば良いそうです。以下のようになりました。

2-3. ファーマコフォア作成に向けた偽アトムの設定
ファーマコフォアを作るには、フィーチャー間の距離や角度といった情報をつかって定義するようです。 フィーチャーが原子一つに帰着できるなら良いですが、芳香環みたいな複数の原子からなるグループ(官能基)の場合は、どの点を基準にして距離計測を行うかで結果が変わりそうです。
先に描画した図では芳香環(Aromatic フィーチャー)の真ん中に球 が表示されていました。同じような点(偽原子)を作って見たいと思います。
PyMolWikiを参考にPseudoatomというのを作って見ました。
- 右下緑文字のSelectingをクリックしてAtomsに変更
- 芳香環の各原子を選択
- pseudoatom arom1, sele
と打ち込みました。これで真ん中の芳香環の中に、新しい点(arom1という名前のobjectにしました)ができました

同様にして他の環にもpseudoatomを作っていきました。
2-4. 相互作用を順番に
~ Tyr / Gln ~
だいたい準備ができた感じがするので一つ一つの相互作用部位を見ていきます。
| 残基番号 | アミノ酸 | 距離 | 相互作用 | |
|---|---|---|---|---|
| 8YQ(chain A/B) | 56B | TYR | 3.82 | Hydrophobic |
| 66B | GLN | 3.72 | Hydrophobic |

距離(点線)はWizardのMeasurementを選択してから、atomをクリックしていくと表示されました。 Chain Bの残基56 Tyrと66 GLNは芳香環と疎水性相互作用をしているようです。
~ Ala / Met ~
| 残基番号 | アミノ酸 | 距離 | 相互作用 | |
|---|---|---|---|---|
| 8YQ(chain A/B) | 115A/B | MET | 3.70/3.80 | Hydrophobic |
| 121A/B | ALA | 3.69/3.75 | Hydrophobic |

Chain Aの残基115 METとChain Bの残基121 ALA、Chain Bの残基115 METとChain Aの残基121 ALAがそれぞれ組み合わせとなって別々の芳香環と疎水性相互作用をしているようです。
~ Tyr / Lys ~
| 残基番号 | アミノ酸 | 距離 | 相互作用 | |
|---|---|---|---|---|
| 8YQ(chain A/B) | 123A | TYR | 3.80/3.52/3.85 | Hydrophobic |
| 124A | LYS | 5.03/3.18 | π-cation/Salt Bridges |

Chain Aの残基123 TYR(黄色)は芳香環と疎水性相互作用、Chain Aの残基124 LYS(オレンジ色)は芳香環との相互作用に加えて、末端カルボキシ基とも相互作用が見られるようです。
3. 共結晶構造をもとにファーマコフォアを作成
3-1. フィーチャーの選択と相対配置
タンパク質残基との相互作用を眺めることで、リガンド側で大事そうな部位がわかってきました。
SAR news No.19の記事を参考に、aromatic 2つとacceptor 1つの3点のフィーチャーをファーマコフォアとして選んで見ました。
(大事な点が3つあります!っていうと格好いいから3にした)

距離をPymol上で確認して見ます。
- Mouse Modeを3-Button Editingに変更
- 2点を選ぶと距離、3点では角度、4点で2面角が表示される

結果をまとめると以下のようです。

便宜的にAcceptorはカルボキシ基の炭素原子との距離を測りました。 酸素原子を使った場合の距離はそれぞれ、5.1Åが[5.0, 5.6] 、10.4Åが[9.8, 11.2]となります。
着目するフィーチャー3点と、その位置関係が決まりました。このファーマコフォアを定義するため、フィーチャーの座標(位置情報)を確認します。
3-2. 共結晶構造におけるフィーチャーの座標の取得
pymolでshowfeatを使う際に、--writeFeatsのオプションをつかうことで、フィーチャーの座標を出力できるようになるそうです。@iwatobipen先生の記事Visualize pharmacophore with RDKitのコードをまたしてもそのままコピペさせていただきました。
(Greg先生の先の関数「showMolFeats」のどこかに--writeFeatsを書き込めばよかったのかもしれませんが私の能力ではわかりませんでした・・・)
import subprocess import pprint showfeatpath = os.path.join(RDConfig.RDCodeDir, 'Chem/Features/ShowFeats.py') v = PyMol.MolViewer() v.DeleteAll() process = subprocess.Popen(['python', showfeatpath, '--writeFeats','./8YQ_noH.sdf'], stdout=subprocess.PIPE) stdout = process.communicate()[0] res = stdout.decode('utf-8').replace('\t', ' ').split('\n') pprint.pprint(res)
下の図のように、座標が出力されます。(一部抜粋。全フィーチャーが並んでいる。)

ここから必要なフィーチャーの座標を抜き出します。フィーチャーを間違えないようにAtom_Idを確認しておきます。
Draw.MolToImage(mol_8YQ, includeAtomNumbers = True)

Atomid と Familyから該当の座標を取り出してきます。Draw.MolToImageで表示させたAtomNumberと、ShowFeatのAtom Idは何故か1ずつ番号がずれていました。理由はよくわかりません・・・とりあえず先に進みます。
芳香環2つの座標は以下・・・
- 'Aromatic -7.625 10.407 -21.653 1.0 # 1 10 11 36 35 34'
- 'Aromatic -1.851 11.051 -24.019 1.0 # 6 8 37 38 39 19'
アクセプター2点(カルボキシル基酸素原子)の間が、ちょうど'NegIonizable'として認識されているのでこちらの座標を使いたいと思います。
- 'NegIonizable -0.100 13.586 -28.137 1.0 # 45 28 20'
- 'Acceptor 0.862 14.067 -27.897 1.0 # 20', 'Acceptor -1.171 13.517 -28.363 1.0 # 28'
フィーチャーの座標位置とその位置関係が決まりました。いよいよRDKitを使ってファーマコフォアを設定したいと思います。
4. RDKitでファーマコフォアを設定
4-1. ファーマコフォア設定方法の流れ
SAR News No.19の吉森氏による記事「Chemoinformatics Toolkits を用いた創薬システム開発における ラピッドプロトタイピング」での流れを確認します。
- 座標を使って特性基を定義
- 距離情報を使ってファーマコフォアを設定
- ファーマコフォアサーチ
- 対象のフィーチャーをそもそも含んでいるか?
- フィーチャー間の距離が条件を満たすか?
- 距離情報を拘束条件にして3D構造を発生させる。
以上の流れを辿って見ます。テストとして、記事冒頭に用共闘用シェアデータから取り出した分子を用います。
4-2. ファーマコフォアの設定を実践
フィーチャーの定義にはChemicalFeaturesモジュールを使うようです。
ChemicalFeatures.FreeChemicalFeature(Feature Family, Feature Type, 位置) とすることで、フィーチャーを定義できます(Typeは省略可能)。 位置の指定にはGeometryのPoint3Dをもちいます。
from rdkit.Chem import ChemicalFeatures from rdkit.Chem.Pharm3D import Pharmacophore from rdkit import Geometry feat_1=ChemicalFeatures.FreeChemicalFeature('Aromatic',Geometry.Point3D(-7.625, 10.407, -21.653)) feat_2=ChemicalFeatures.FreeChemicalFeature('Aromatic',Geometry.Point3D(-1.851, 11.051, -24.019)) feat_3=ChemicalFeatures.FreeChemicalFeature('Acceptor',Geometry.Point3D(-0.100, 13.586, -28.137)) feats = [feat_1,feat_2,feat_3]
Aromatic 2つとAcceptor1つが定義できました。
続いてPharm3D.Pharmacophoreモジュールを使って距離情報を設定します。先に定義したフィーチャーのリストでファーマコフォアを設定した後、フィーチャー間の距離のとりうる範囲を下限値(setLowerBound)、上限値(setUpperBound)という形で与えます。
SAR Newsの記事では距離の許容範囲の値として、下限値に0.5(d_lower)、上限値に1.5(d_upper)が用いられていました。
pcophore = Pharmacophore.Pharmacophore(feats) # ファーマコフォアの設定 d_upper = 1.5 # 特性基間の距離の許容範囲(上限値) d_lower = 0.5 # 特性基間の距離の許容範囲(下限値) # feat_1とfeat_2の距離 6.3Åを基準に、下限と上限を設定 pcophore.setLowerBound(0,1, 6.3-d_lower) pcophore.setUpperBound(0,1, 6.3+d_upper) # Acceptor(feat_3)の代表点の選び方によってfeat_2との距離は[5.0~5.6]の値をとる pcophore.setLowerBound(1,2, 5.0-d_lower) pcophore.setUpperBound(1,2, 5.6+d_upper) # 同様にfeat1とfeat_3は[9.8~11.2]の値をとる pcophore.setLowerBound(0,2, 9.8-d_lower) pcophore.setUpperBound(0,2, 11.2+d_upper) print(pcophore)

以上でファーマコフォアの設定が終わりました。printで出力したPharmacophoreを見てみると、距離行列となっており、対角線右上三角に上限値、左下三角に下限値の情報を持つようです。
4-3. ファーマコフォアサーチ ~step 1. フィーチャーの有無~
それではテスト分子を使ってファーマコフォアサーチを行います。3次元構造を扱うので水素原子を付加しておきます。
test_mH = AllChem.AddHs(test_m) AllChem.Compute2DCoords(test_mH)
まずはフィーチャーをそもそも持っているか?
Parm3D.EmbedLibモジュールのMatchPharmacophoreToMolを使います。
EmbedLib.MatchPharmacophoreToMol(molオブジェクト,フィーチャーファクトリー, ファーマコフォア)と、することで対象のmolオブジェクトにファーマコフォアのマッピングを行い、可能なマッピングのリストを作成します。
戻り値は2要素のタプルで、
- 全てのフィーチャーが見つけられたか否かのブール値
- フィーチャーの並びのリスト
となっています。
テスト分子をtest_mH、フィーチャーファクトリーをfdef、ファーマコフォアをpcophoreとして実行します。
from rdkit.Chem.Pharm3D import EmbedLib match, mList = EmbedLib.MatchPharmacophoreToMol(test_mH, fdef, pcophore) print(match) # True
どうやら全てのフィーチャーのマッチングはOKだったみたいです。確認のためリストからフィーチャーの情報を取り出して見ます。
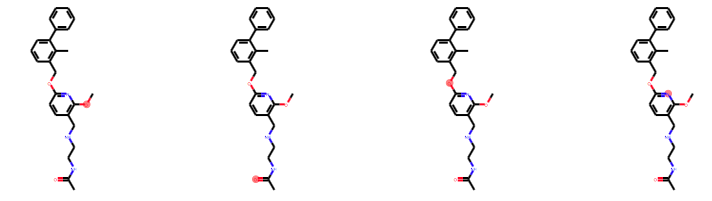
print(len(mList)) #3 print(len(mList[2])) #4 print(mList[2][0].GetFamily()) #Acceptor print(mList[2][0].GetType()) #SingleAtomAcceptor print(mList[2][2].GetAtomIds()) #(15,)
心配なのでAcceptorについて描画して見ます。
highlight_list = [mList[2][x].GetAtomIds() for x in range(len(mList[2]))] Draw.MolsToGridImage([test_m]*4, molsPerRow=4, subImgSize=(200, 200), highlightAtomLists=highlight_list)
うまく機能していそうです。
4-4. ファーマコフォアサーチ ~step 2. 距離条件~
ついで距離条件の検証を行います。
まず、rdDistGeomモジュールのGetMoleculeBoundsMatrixを使って分子の距離行列を取得します。
Parm3D.EmbedLibモジュールのGetAllPharmacophoreMatchesを使って、先に取得した分子の距離行列が、ファーマコフォアで定義されている距離の条件を満たすか否かを判定します。
from rdkit.Chem import rdDistGeom # 距離行列の取得 bounds = rdDistGeom.GetMoleculeBoundsMatrix(test_mH) #ファーマコフォアのマッチング pList =EmbedLib.GetAllPharmacophoreMatches(mList,bounds,pcophore) print(len(pList)) # 4
ファーマコフォア(3つのフィーチャー)の条件を満たす組み合わせは4つあったようです。一つ目の組み合わせのフィーチャーを構成する原子のIDを取得して見ます。
AtomIds_list = [] for i in range(len(pList[0])): p = pList[0][i] print(p.GetFamily(), ':', p.GetAtomIds()) AtomIds_list.append(p.GetAtomIds())

Draw.MolsToGridImage([test_m]*3, subImgSize=(200, 200), highlightAtomLists=AtomIds_list)
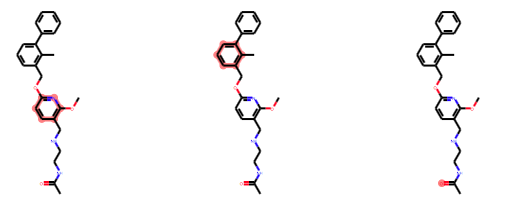
他の組み合わせでは以下になりました。

想定していたのと異なり、一番遠くの芳香環を使った組み合わせとなっていました。
ところで、このマッチングは距離を条件につかっているのですが、検索対象としているmolオブジェクトは3次元構造を生成させていません(pymolで眺めた時もぺちゃんこだった)。
SAR Newsの記事の該当部分を引用すると
一般的なファーマコフォアサーチにおいては、分子の3D構造生成後、ファーマコフォアのサーチを行うが、RDKitにおいては、事前に分子の3D構造を生成させるのではなく、ファーマコフォアを満たす3D構造が生成できるか否かを判定基準として、ファーマコフォアのサーチを行うことができる。
とのことだそうです。それでは 距離条件をみたすような3次元構造が本当に作れるのかどうか、検証したいと思います。
4-5. ファーマコフォアサーチ ~step 3. 3D構造の発生~
距離情報を拘束条件にして3D構造を発生させます。まずはマッチしたフィーチャーの原子IDのリストを作成します。コピペ・・・
num_match = len(pList) phMatches = [] for i in range(num_match): num_feature = len(pList[i]) phMatch = [] for j in range(num_feature): phMatch.append(pList[i][j].GetAtomIds()) phMatches.append(phMatch)
距離情報を拘束条件にした3D構造の発生にはPharm3D.EmbedLibのEmbedPharmacophoreを使います。
引数がたくさんありますが、対象のmolオブジェクト(mol)に対して、ファーマコフォアのフィーチャーを構成する原子のid(atomMatch)をつかって、ファーマコフォア(pcophore)の条件を満たすように3次元構造を生成(embedding)することができるようです。 生成させる構造の最大数をcountで設定します。(silentはよくわかりません・・・)
戻り値は3要素のタプルで、
- ファーマコフォアに合うように調整された拘束条件の距離行列
- 3次元構造(コンフォマー1つを有する分子)のリスト
- 3次元構造の生成に失敗した回数
となっています。
生成させたのち、ETKDG法を使って3次元構造の最適化を実施します。
confs = [] for phMatch in phMatches: bm,embeds,nFail =EmbedLib.EmbedPharmacophore(test_mH, phMatch, pcophore,count=20, silent=1) print("Generated embeds: ",len(embeds)) print("Failed attempts: ",nFail) for embed in embeds: AllChem.EmbedMolecule(embed, AllChem.ETKDG()) confs.append(embed)

フィーチャーの組み合わせによって、構造生成の成否回数が異なっているようです。
SAR newsの記事ではUniversal Force Field法を使って構造最適化を行うと記載してあったのですが、以下のようなエラーメッセージが出てしまいました。
RuntimeError Traceback (most recent call last) <ipython-input-254-6895836e8c95> in <module>() 8 print("Failed attempts: ",nFail) 9 for embed in embeds: ---> 10 AllChem.UFFOptimizeMolecule(embed) 11 confs.append(embed) RuntimeError: Invariant Violation bad direction in linearSearch Violation occurred on line 234 in file Code/Numerics/Optimizer/BFGSOpt.h Failed Expression: status >= 0 RDKIT: 2018.09.1 BOOST: 1_65_1
よくわからない・・・。飛ばそう・・・
念のためETKDGで最適化した3次元構造を一つ眺めて見ます。
from rdkit.Chem.Draw import IPythonConsole import py3Dmol IPythonConsole.drawMol3D(confs[0])

いい感じにできていそうです。
5. ファーマコフォアを基準に共結晶構造に重ね合わせ
活性化合物に関して、ファーマコフォアの基準を満たす3次元構造の取得までが確認できました。この3D構造は、共結晶構造のリガンド構造と類似しているはず・・・なので、共結晶構造に重ね合わせてタンパク質と一緒に描いて見たいと思います。
5-1. PDB座標にアラインメント
ファーマコフォア作成に用いたリガンドの座標はPDBの共結晶構造を元にしているので、この座標をテンプレートとしてアライメントをとります。
Numerics.rdAlignmentモジュールのGetAlignmentTransformを使って、参照分子の座標(refPoints)と目的の分子の座標(probePoints)とで、RMSDが最小となる最適なアラインメントを計算します。
rdkit.Numerics.rdAlignment.GetAlignmentTransform(refPoints, probePoints)の戻り値として、SSD値(Sum of Squared Difference)と、4x4変換行列(transform matrix)のアレイを要素として持つ、2-タプルが得られます。
得た変換をAllChem.TransformMolを使って、適応することで、アラインメントをとった座標を持つmolオブジェクトを得ます。*4
# 生成した構造のフィーチャーを取得 match, mList_0 = EmbedLib.MatchPharmacophoreToMol(confs[0], fdef, pcophore) bounds_0 = rdDistGeom.GetMoleculeBoundsMatrix(confs[0]) pList_0 =EmbedLib.GetAllPharmacophoreMatches(mList_0,bounds_0,pcophore) # ファーマコフォアで設定したフィーチャーのリストを参照(ref)として使用する refFeats = feats # pList[0]で認識されているフィーチャーを #ファーマコフォアのフィーチャーの定義の順番に並べ替えたリストにする probFeats = [pList_0[0][1], pList_0[0][0], pList_0[0][2]] # ref、prob、それぞれのフィーチャーの座標を取得し、リストにする probPts = [list(x.GetPos()) for x in probFeats] refPts = [list(x.GetPos()) for x in refFeats] # 2つの座標をつかって変換行列を取得 ssd,tform = rdAlignment.GetAlignmentTransform(refPts, probPts) # 変換行列を使って、生成した構造の座標を変換 AllChem.TransformMol(confs[0], tform) #変換したmolオブジェクトをmolファイルとして出力 Chem.MolToMolFile(confs[0], 'conf.mol')
出力したmolファイルと、元にした共結晶構造 5NIX.pdb を同時にpymolで表示させました。

もともとのリガンドの色がシアンで、重ね合わせたリガンドが緑色です。 なかなかいい感じで重なっているようにみえます。今回ファーマコフォアとして指定しなかった末端の芳香環についてはズレが大きいように見える一方で、ファーマコフォアマッチングした部分は重なりがよく見えます。うまくいった!!! ・・・のか???
まとめ
以上、今回はファーマコフォアの作成とテスト分子を使った動作検証を行って見ました。ファーマコフォアの基準を満たす3次元構造の取得までが確認できましたので、この取得ができるか否かまでを、ファーマコフォアを満たすことができるか否かという判定基準として活用していけば多分いい感じでスクリーニングになるはず。。。
あまりにもわからないことが多すぎて先生方の記事からひたすらコピペを繰り返しましたが、それでも正しく使えているのか自信がまったくありません。特にBounds Matrixが理解できなかった・・・(3次元に関する条件にしては行列の各要素が3個でもないし・・・)。
色々と間違いがあると思うのでご指摘いただければ幸いです。
~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~
*1:参考: PythonのCounterでリストの各要素の出現個数をカウント
*2:参考: RDKitブログ記事Using Feature Maps
*3:citatition:
- SMARTS viewer https://smartsview.zbh.uni-hamburg.de
RDKitのファーマコフォアの定義を眺める話
前回の記事で複数の共結晶構造に共通する相互作用をPLIPを使って探してみました。この情報をうまく使えばファーマコフォアが作れるはず・・・
ですがそもそもどうやってファーマコフォアを作っていけばわかりません。
ファーマコフォア?
日本薬学会 薬学用語解説の説明によると以下の通り
「ファーマコフォアとは、医薬品のターゲットとの相互作用に必要な特徴を持つ官能基群と、それらの相対的な立体配置も含めた(抽象的な)概念である・・・(略)・・・同じターゲットの同じ部位に結合する医薬品は活性発現に必須である共通の立体的・電子的特徴を持つ官能基群を持っており、これをファーマコフォアという。」
タンパク質の重要そうな残基と相互作用している、リガンド側の構造の(立体的・電子的)特徴を取り出し、
- 官能基の種類
- 相対的立体配置
をもとに抽象化(モデル化)すれば良いということみたいです。
なるほど!!!
しかし概念はわかりましたがどんな特徴を取り出せばよいかわからない・・・
RDKitを使った場合どのようになるのか調べてみました。参考にしたページは記事末尾に記載しています。*1 *2 *3 *4 *5 *6
RDKitにおけるファーマコフォアの特徴
RDKitでファーマコフォアを構成する特徴(特性基、フィーチャー)をみつけるには、
- 特性基の定義をあつめたフィーチャーファクトリーを作成
- それを用いて実際の分子から該当する特性基を探索
という手順をとるようです。どのような構造を特性基として認識するのでしょうか?
RDKitにおける特性基(化学的特徴/ Chemical Featrues)はSMARTSに基づいた言語で定義づけられています。Feature Definition(FDef)ファイルフォーマットという形式のファイルに、特性基の定義づけに必要な情報が記載されています。
基本となる「BaseFeatures.fdef」というファイルが用意されているのでこちらを利用したいと思います。
フィーチャーファクトリーの作成
ファーマコフォアの認識にFDefファイルの情報を利用するには、こちらを読み込んでフィーチャーファクトリーを作成すれば良いということです。
import os from rdkit import RDConfig from rdkit.Chem import AllChem from rdkit import Chem fdef = AllChem.BuildFeatureFactory(os.path.join(RDConfig.RDDataDir,'BaseFeatures.fdef')) print(fdef.GetNumFeatureDefs()) # 27
用意されているフィーチャーの定義は27個あるようです。
フィーチャーファミリー
フィーチャーをより一般化した分類としてフィーチャーファミリーが用意されており、ファーマコフォマ検索はファミリーを用いて行われます。
print(len(fdef.GetFeatureFamilies())) print(fdef.GetFeatureFamilies()) # 8 # ('Donor', 'Acceptor', 'NegIonizable', 'PosIonizable', 'ZnBinder', 'Aromatic', 'Hydrophobe', 'LumpedHydrophobe')
フィーチャーファミリーは全部で8個あり、水素結合ドナー(Donor)やアクセプター(Acceptor)といった名前から構造が推測できるものから、LumpedHydrophobeといった具体的な例がよくわからない(私だけ?)ものまであります。
フィーチャータイプ
より詳細な記述がされたフィーチャータイプを確認したいと思います。
fdef.GetFeatureDefs()) とすると、各フィーチャーの名前をKey、SMARTSによる定義をvalueとする辞書型で情報を書き出すことができます。
とりあえず名前だけ確認します。
print(fdef.GetFeatureDefs().keys()) # dict_keys(['Donor.SingleAtomDonor', 'Acceptor.SingleAtomAcceptor', 'NegIonizable.AcidicGroup', 'PosIonizable.BasicGroup', 'PosIonizable.PosN', 'PosIonizable.Imidazole', 'PosIonizable.Guanidine', 'ZnBinder.ZnBinder1', 'ZnBinder.ZnBinder2', 'ZnBinder.ZnBinder3', 'ZnBinder.ZnBinder4', 'ZnBinder.ZnBinder5', 'ZnBinder.ZnBinder6', 'Aromatic.Arom4', 'Aromatic.Arom5', 'Aromatic.Arom6', 'Aromatic.Arom7', 'Aromatic.Arom8', 'Hydrophobe.ThreeWayAttach', 'Hydrophobe.ChainTwoWayAttach', 'LumpedHydrophobe.Nitro2', 'LumpedHydrophobe.RH6_6', 'LumpedHydrophobe.RH5_5', 'LumpedHydrophobe.RH4_4', 'LumpedHydrophobe.RH3_3', 'LumpedHydrophobe.tButyl', 'LumpedHydrophobe.iPropyl'])
各フィーチャーは「ファミリー名.xxxx」という形式のようなのでファミリー名を紐づけてDataFrame化してみます。
import pandas as pd family_df = pd.DataFrame(columns=['family', 'definition']) family_names = fdef.GetFeatureFamilies() for k,v in fdef.GetFeatureDefs().items(): for fam in family_names: if fam in k: family_df.loc[k] = [fam, v] family_df.head()

SMARTSの定義の文字列がおかしな感じになっています。$などが文字としてそのまま出力されていないみたいです。
各ファミリーに含まれるフィーチャーの数を確認します。
family_df['family'].value_counts() # LumpedHydrophobe 7 # ZnBinder 6 # Aromatic 5 # PosIonizable 4 # Hydrophobe 2 # NegIonizable 1 # Acceptor 1 # Donor 1
名前だけではわかりづらい「LumpedHydrophobe」や「ZnBinder」の種類が多くなっています。まとまった定義づけが難しいのでしょうか?
SMARTSの解読に挑戦
以前の記事でSMARTS記法について、ちょっと勉強しました。折角なのでSMARTSによる定義を理解できるか、書き下してみたいと思います。
書き下した後でSMARTSviewerという便利すぎるサイトを見つけてしまったので、素人の適当解釈は不要!という方はすっ飛ばして下までスクロールしてください。
SMARTSの説明補足
SMARTSを読み始める前に、前回の記事の補足を少々・・・
- Recursive SMARTS
フィーチャーの定義ではRecursive SMARTSの形式が多く用いられていました。Daylight社のSMARTS記法の説明ページによると、「$(SMARTS)」という表記で注目している原子(atom)の置かれている環境を示す、とのことです。
- 「R<数字>」と「r<数字>」の違い
以前の記事では、無理やりSMARTSの説明を日本語化しようとした結果、逆にわかりづらくなっていたので、Rとrについて英語の説明をそのまま貼っておきます。
| Symbol | Symbol name | Atomic property requirements | Default |
|---|---|---|---|
| R |
ring membership | in |
any ring atom |
| r |
ring size | in smallest SSSR ring of size |
any ring atom |
「幾つのSSSR環に含まれているか?」と、「どんなサイズの環に含まれているか?」ということになりそうです。
ドナー
| family | feature | 定義 SMARTS | 説明 | |
|---|---|---|---|---|
| Donor | Donor.SingleAtomDonor | [$([N&!H0&v3, | Hの数が0ではない、結合次数の総数3の脂肪族N | または |
| N&!H0&+1&v4, | Hの数が0ではない、電荷+1で結合次数の総数4の脂肪族N | または | ||
| n&H1&+0, | Hの数が1で、電荷0の芳香族N | または | ||
| $([$([Nv3](-C)(-C)-C)]), | 脂肪族炭素が3つ結合した、結合次数の総数3の脂肪族N | または | ||
| $([$(n[n;H1]), | 芳香族Nと、Hの数が1の芳香族Nが隣接する並び | または | ||
| $(nc[n;H1])])]), | 芳香族N、芳香族Cと、Hの数が1の芳香族Nが隣接する並び | あるいは | ||
| $([O,S;H1;+0])] | Hの数が1で、電荷0の脂肪族OまたはS |
アクセプター
| family | feature | 定義 SMARTS | 説明 | |
|---|---|---|---|---|
| Acceptor | Acceptor.SingleAtomAcceptor | [$([O;H1;v2]), | Hの数が1で、結合次数の総数が2の脂肪族O | あるいは |
| $([O;H0;v2;!$(O=N-*), | Hの数が0で、結合次数の総数が2の脂肪族Oのうち、 O=N-に連結するものではないもの |
または | ||
| $([O;-;!$(*-N=O)]), | 負電荷(-1)を帯びた脂肪族Oのうち、 O=N-に連結するものではないもの |
または | ||
| $([o;+0])]), | 電荷0の芳香族O | あるいは | ||
| $([n;+0;!X3;!$([n;H1](cc)cc), | 電荷0の芳香族Nで、結合の総数3ではなく、また、 芳香族炭素Cが二つ直列した結合2本とHを1つもつ芳香族Nでもないもの |
または | ||
| $([$[N;H0]#[C&v4])]), | Hの数が0の脂肪族Nと、結合次数の総数4の脂肪族Cが、 3重結合で結合したもの(シアノ基) |
または | ||
| $([N&v3;H0;$(Nc)])]), | 結合次数の総数3で結合するHの数が0の脂肪族Nと、かつ、 芳香族Cに結合する脂肪族N |
あるいは | ||
| $([F;$(F-[#6]);!$(FC[F,Cl,Br,I])])] | 炭素と結合するFで、 ハロゲン原子の結合した脂肪族炭素に結合するFではないもの |
イオン化可能なグループ
| family | feature | 定義 SMARTS | 説明 | |
|---|---|---|---|---|
| NegIonizable | NegIonizable.AcidicGroup | [C,S](=[O,S,P])-[O;H1,H0&-1] | 「O、SまたはP」と二重結合する「CまたはS」に結合した、 OH、あるいはO-(H、0個で負電荷) |
|
| PosIonizable | PosIonizable.BasicGroup | [$([$([N;H2&+0][$([C;!$(C=*)])])]), | 他の脂肪族Cと二重結合しているのではない脂肪族C、 およびH2つと結合する電荷0の脂肪族N |
または |
| $([$([N;H1&+0]([$([C;!$(C=*)])])[$([C;!$(C=*)])])]), | 他の脂肪族Cと二重結合しているのではない脂肪族C2つ、 およびH1つと結合する、電荷0の脂肪族N |
または | ||
| $([$([N;H0&+0]([$([C;!$(C=*)])])([$([C;!$(C=*)])])[$([C;!$(C=*)])])]) | 他の脂肪族Cと二重結合しているのではない脂肪族C3つと 結合する、電荷0の脂肪族N |
かつ | ||
| ;!$(N[a])] | 上記3つのNは芳香族原子と結合していないこと | |||
| PosIonizable.PosN | [#7;+;!$([N+]-[O-])] | 「O-」に結合する「N+」ではない、電荷+1のN | ||
| PosIonizable.Imidazole | c1ncnc1 | イミダゾール骨格 | ||
| PosIonizable.Guanidine | NC(=N)N | グアニジン骨格 |
Zinc Binder
| family | feature | 定義 SMARTS | 説明 |
|---|---|---|---|
| ZnBinder | ZnBinder.ZnBinder1 | [S;D1]-[#6] | Cと結合する1級S |
| ZnBinder.ZnBinder2 | [#6]-C(=O)-C-[S;D1] | C-カルボニル-Cに結合する1級S | |
| ZnBinder.ZnBinder3 | [#6]-C(=O)-C-C-[S;D1] | C-カルボニル-C-Cに結合する1級S | |
| ZnBinder.ZnBinder4 | [#6]-C(=O)-N-[O;D1] | C-カルボニル-Nに結合する1級O | |
| ZnBinder.ZnBinder5 | [#6]-C(=O)-[O;D1] | C-カルボニルに結合する1級O | |
| ZnBinder.ZnBinder6 | [#6]-P(=O)(-O)-[C,O,N]-[C,H] | C-PO2に結合する「C or O or N」に「C or H」が結合 |
芳香族
| family | feature | 定義 SMARTS | 説明(原子環境) | 結合の種類 |
|---|---|---|---|---|
| Aromatic | Aromatic.Arom4 | [$([a;r4,!R1&r3])]1: | 4員環(SSSR size4)の芳香族性原子、または 3員環(size3)で、含まれるSSSR ringが1つではないもの |
芳香属性結合(:) |
| [$([a;r4,!R1&r3])]: | 同上 | 同上 | ||
| [$([a;r4,!R1&r3])]: | 同上 | 同上 | ||
| [$([a;r4,!R1&r3])]:1 | 同上 | 同上 | ||
| 1 | 4員環を閉じる | |||
| Aromatic.Arom5 | [$([a;r5,!R1&r4,!R1&r3])]1: | 5員環(SSSR size5)の芳香族性原子、または 4員環(size4)で、含まれるSSSR ringが1つではないもの、または 3員環(size3)で、含まれるSSSR ringが1つではないもの |
芳香属性結合(:) | |
| [$([a;r5,!R1&r4,!R1&r3])]: | 同上 | 同上 | ||
| [$([a;r5,!R1&r4,!R1&r3])]: | 同上 | 同上 | ||
| [$([a;r5,!R1&r4,!R1&r3])]: | 同上 | 同上 | ||
| [$([a;r5,!R1&r4,!R1&r3])]: | 同上 | 同上 | ||
| 1 | 5員環を閉じる | |||
| Aromatic.Arom6 | [$([a;r6,!R1&r5,!R1&r4,!R1&r3])]1: | 6員環(SSSR size6)の芳香族性原子、または 5員環(size5)で、含まれるSSSR ringが1つではないもの、または 4員環(size4)で、含まれるSSSR ringが1つではないもの、または 3員環(size3)で、含まれるSSSR ringが1つではないもの |
芳香属性結合(:) | |
| [$([a;r6,!R1&r5,!R1&r4,!R1&r3])]: | 同上 | 同上 | ||
| [$([a;r6,!R1&r5,!R1&r4,!R1&r3])]: | 同上 | 同上 | ||
| [$([a;r6,!R1&r5,!R1&r4,!R1&r3])]: | 同上 | 同上 | ||
| [$([a;r6,!R1&r5,!R1&r4,!R1&r3])]: | 同上 | 同上 | ||
| [$([a;r6,!R1&r5,!R1&r4,!R1&r3])]: | 同上 | 同上 | ||
| 1 | 6員環を閉じる | |||
| Aromatic.Arom7 | [$([a;r7,!R1&r6,!R1&r5,!R1&r4,!R1&r3])]1: | 7員環(SSSR size7)の芳香族性原子、または 6員環(size6)で、含まれるSSSR ringが1つではないもの、または 5員環(size5)で、含まれるSSSR ringが1つではないもの、または 4員環(size4)で、含まれるSSSR ringが1つではないもの、または 3員環(size3)で、含まれるSSSR ringが1つではないもの |
芳香属性結合(:) | |
| [$([a;r7,!R1&r6,!R1&r5,!R1&r4,!R1&r3])]: | 同上 | 同上 | ||
| [$([a;r7,!R1&r6,!R1&r5,!R1&r4,!R1&r3])]: | 同上 | 同上 | ||
| [$([a;r7,!R1&r6,!R1&r5,!R1&r4,!R1&r3])]: | 同上 | 同上 | ||
| [$([a;r7,!R1&r6,!R1&r5,!R1&r4,!R1&r3])]: | 同上 | 同上 | ||
| [$([a;r7,!R1&r6,!R1&r5,!R1&r4,!R1&r3])]: | 同上 | 同上 | ||
| [$([a;r7,!R1&r6,!R1&r5,!R1&r4,!R1&r3])]: | 同上 | 同上 | ||
| 1 | 7員環を閉じる | |||
| Aromatic.Arom8 | [$([a;r8,!R1&r7,!R1&r6,!R1&r5,!R1&r4,!R1&r3])]1: | 8員環(SSSR size8)の芳香族性原子、または 7員環(size7)で、含まれるSSSR ringが1つではないもの、または 6員環(size6)で、含まれるSSSR ringが1つではないもの、または 5員環(size5)で、含まれるSSSR ringが1つではないもの、または 4員環(size4)で、含まれるSSSR ringが1つではないもの、または 3員環(size3)で、含まれるSSSR ringが1つではないもの |
芳香属性結合(:) | |
| [$([a;r8,!R1&r7,!R1&r6,!R1&r5,!R1&r4,!R1&r3])]: | 同上 | 同上 | ||
| [$([a;r8,!R1&r7,!R1&r6,!R1&r5,!R1&r4,!R1&r3])]: | 同上 | 同上 | ||
| [$([a;r8,!R1&r7,!R1&r6,!R1&r5,!R1&r4,!R1&r3])]: | 同上 | 同上 | ||
| [$([a;r8,!R1&r7,!R1&r6,!R1&r5,!R1&r4,!R1&r3])]: | 同上 | 同上 | ||
| [$([a;r8,!R1&r7,!R1&r6,!R1&r5,!R1&r4,!R1&r3])]: | 同上 | 同上 | ||
| [$([a;r8,!R1&r7,!R1&r6,!R1&r5,!R1&r4,!R1&r3])]: | 同上 | 同上 | ||
| [$([a;r8,!R1&r7,!R1&r6,!R1&r5,!R1&r4,!R1&r3])]: | 同上 | 同上 | ||
| 1 | 8員環を閉じる |
疎水性基
| family | feature | 定義 SMARTS | 説明 |
|---|---|---|---|
| Hydrophobe | Hydrophobe.ThreeWayAttach | [D3,D4; $([#6;+0;!$([#6;$([#6]~[#7,#8,#9])])])] |
3級、または4級の原子で、かつ 電荷0のCのうち 「N,O,F」のいずれかと結合したCではないもの |
| Hydrophobe.ChainTwoWayAttach | [R0;D2;$([#6;+0; !$([#6;$([#6]~[#7,#8,#9])])])] |
環に含まれていない、2級の原子で 電荷0のCのうち 「N,O,F」のいずれかと結合したCではないもの |
疎水性基の塊?
| family | feature | 定義 SMARTS | 説明 | 結合の種類 | |
|---|---|---|---|---|---|
| LumpedHydrophobe | LumpedHydrophobe.Nitro2 | [N;D3;+](=O)[O-] | 3級で電荷+1のNであり、Oと二重結合、電荷-1のOと単結合するもの | (ニトロ基) | |
| LumpedHydrophobe.RH6_6 | [$([r6,!R1&r5,!R1&r4,!R1&r3]); | 6員環(SSSR size6)に含まれる、または 5員環(size5)で、含まれるSSSR ringが1つではないもの、または 4員環(size4)で、含まれるSSSR ringが1つではないもの、または 3員環(size3)で、含まれるSSSR ringが1つではないもの |
かつ | ||
| $([R; $([c,s,S&H0&v2,Br,I, $([#6;+0;!$([#6;$([#6]~[#7,#8,#9])])])])])]1 |
環に含まれる原子で、かつ 「芳香属性C、芳香属性S、結合次数の総数2でHの数0の脂肪族S、Br、I、または 電荷0のCのうち「N,O,F」のいずれかと結合したCではないもの」 |
単結合or芳香属性結合 | |||
| [$([r6,!R1&r5,!R1&r4,!R1&r3]); $([R;$([c,s,S&H0&v2,Br,I, $([#6;+0;!$([#6;$([#6]~[#7,#8,#9])])])])])] |
同上 | 同上 | |||
| [$([r6,!R1&r5,!R1&r4,!R1&r3]); $([R;$([c,s,S&H0&v2,Br,I, $([#6;+0;!$([#6;$([#6]~[#7,#8,#9])])])])])] |
同上 | 同上 | |||
| [$([r6,!R1&r5,!R1&r4,!R1&r3]); $([R;$([c,s,S&H0&v2,Br,I, $([#6;+0;!$([#6;$([#6]~[#7,#8,#9])])])])])] |
同上 | 同上 | |||
| [$([r6,!R1&r5,!R1&r4,!R1&r3]); $([R;$([c,s,S&H0&v2,Br,I, $([#6;+0;!$([#6;$([#6]~[#7,#8,#9])])])])])] |
同上 | 同上 | |||
| [$([r6,!R1&r5,!R1&r4,!R1&r3]); $([R;$([c,s,S&H0&v2,Br,I, $([#6;+0;!$([#6;$([#6]~[#7,#8,#9])])])])])]1 |
同上 | 6員環を閉じる | |||
| LumpedHydrophobe.RH5_5 | [$([r5,!R1&r4,!R1&r3]); | 5員環(SSSR size5)に含まれる、または 4員環(size4)で、含まれるSSSR ringが1つではないもの、または 3員環(size3)で、含まれるSSSR ringが1つではないもの |
かつ | ||
| $([R; $([c,s,S&H0&v2,Br,I, $([#6;+0;!$([#6;$([#6]~[#7,#8,#9])])])])])]1 |
環に含まれる原子で、かつ 「芳香属性C、芳香属性S、結合次数の総数2でHの数0の脂肪族S、Br、I、または 電荷0のCのうち「N,O,F」のいずれかと結合したCではないもの」 |
単結合 or 芳香属性結合 | |||
| [$([r5,!R1&r4,!R1&r3]); $([R;$([c,s,S&H0&v2,Br,I, $([#6;+0;!$([#6;$([#6]~[#7,#8,#9])])])])])] |
同上 | 同上 | |||
| [$([r5,!R1&r4,!R1&r3]); $([R;$([c,s,S&H0&v2,Br,I, $([#6;+0;!$([#6;$([#6]~[#7,#8,#9])])])])])] |
同上 | 同上 | |||
| [$([r5,!R1&r4,!R1&r3]); $([R;$([c,s,S&H0&v2,Br,I, $([#6;+0;!$([#6;$([#6]~[#7,#8,#9])])])])])] |
同上 | 同上 | |||
| [$([r5,!R1&r4,!R1&r3]); $([R;$([c,s,S&H0&v2,Br,I, $([#6;+0;!$([#6;$([#6]~[#7,#8,#9])])])])])] |
同上 | 5員環を閉じる | |||
| LumpedHydrophobe.RH4_4 | [$([r4,!R1&r3]); | 4員環(SSSR size4)に含まれる、または 3員環(size3)で、含まれるSSSR ringが1つではないもの |
かつ | ||
| $([R; $([c,s,S&H0&v2,Br,I, $([#6;+0;!$([#6;$([#6]~[#7,#8,#9])])])])])]1 |
環に含まれる原子で、かつ 「芳香属性C、芳香属性S、結合次数の総数2でHの数0の脂肪族S、Br、I、または 電荷0のCのうち「N,O,F」のいずれかと結合したCではないもの」 |
単結合 or 芳香属性結合 | |||
| [$([r4,!R1&r3]);$([R; $([c,s,S&H0&v2,Br,I, $([#6;+0;!$([#6;$([#6]~[#7,#8,#9])])])])])] |
同上 | 同上 | |||
| [$([r4,!R1&r3]);$([R; $([c,s,S&H0&v2,Br,I, $([#6;+0;!$([#6;$([#6]~[#7,#8,#9])])])])])] |
同上 | 同上 | |||
| [$([r4,!R1&r3]);$([R; $([c,s,S&H0&v2,Br,I, $([#6;+0;!$([#6;$([#6]~[#7,#8,#9])])])])])]1 |
同上 | 4員環を閉じる | |||
| LumpedHydrophobe.RH3_3 | [$([r3]); | 3員環(SSSR size3)に含まれるもの | かつ | ||
| $([R; $([c,s,S&H0&v2,Br,I, $([#6;+0;!$([#6;$([#6]~[#7,#8,#9])])])])])]1 |
環に含まれる原子で、かつ 「芳香属性C、芳香属性S、結合次数の総数2でHの数0の脂肪族S、Br、I、または 電荷0のCのうち「N,O,F」のいずれかと結合したCではないもの」 |
単結合or芳香属性結合 | |||
| [$([r3]);$([R;$([c,s,S&H0&v2,Br,I, $([#6;+0;!$([#6;$([#6]~[#7,#8,#9])])])])])] |
同上 | 同上 | |||
| [$([r3]);$([R;$([c,s,S&H0&v2,Br,I, $([#6;+0;!$([#6;$([#6]~[#7,#8,#9])])])])])]1 |
同上 | 3員環を閉じる | |||
| LumpedHydrophobe.tButyl | [C;!R](-[CH3])(-[CH3])-[CH3] | 環に含まれていないCで、メチル基3つが結合 | |||
| LumpedHydrophobe.iPropyl | [CH;!R](-[CH3])-[CH3] | 環に含まれていないCで、メチル基2つとH1つが結合 |
SMARTSviewer
さて、つらつらと意味不明な日本語を垂れ流してしまいましたが、上に述べたようにSMARTSviewerという便利すぎるサイトを見つけてしまいました。
こちらは ハンブルク大学 バイオインフォマティクスセンター(Universität Hamburg ZBH-Center for Bioinformatics)が開発・公開しているソフトウェアで SMARTSをとてもわかりやすく可視化してくれます。
まずは見た目を・・・

使い方はとても簡単で、「Try your own SMARTS pattern」と書かれた部分にSMARTSを貼り付け、「Go!」をクリックするだけです。「Donwload」をクリックすればpng形式で綺麗な図が手に入ります。*7
それではフィチャーの中で気になったZnBinderで試してみます。
フィーチャー名「ZnBinder.ZnBinder6」 SMARTS「[#6]-P(=O)(-O)-[C,O,N]-[C,H]」の場合・・・

線形表記と比べて分岐構造の把握がしやすく、またカラフルなので原子の違いもわかりやすいです。
Optionで描画方法をデフォルト「Coplete Visualization」から「Element Symbols」にすると、より2次元構造式に近い見た目となります。

とてもいい感じです。インストール版もありアカデミックフリー、ノンアカデミックはライセンス費用が必要みたいです。SMARTSviewerの開発者はResearch Group for Computational Molecular Design (AMD、Prof. Matthias Rarey)グループで、AMD software serverには他にもSMARTSeditorなど面白そうなソフトウェアが多数ありました。*8
記事の最後にSMARTsviwerに投げ込みやすい形式でRDKitのフィーチャーを出力しておきます。文字書き下しのわかりづらさと図の見易さの違いを比較していただければと思います。
まとめ
以上、今回はファーマコフォア作成に向けた手始めとしてRDKitにおける特徴の定義を眺めてみました。・・・というよりSMARTSを読んだだけで力尽きました・・・
FDefファイルの中身(rdkit/Data/BaseFeatures.fdef)をテキストエディタで開いてみたり、RDKitのDocumentation Feature Definition File Formatを確認した方がわかりやすかったかもしれません。
SMARTSviewerを見つけたのが最大の収穫!!
色々と間違いがあると思いますのでご指摘いただければ幸いです。
SMARTS出力
テーブルでは分けたり、エスケープ記号を入れたりしてしまっているので、そのままのSMARTS出力も載せておきます。
for k, v in fdef.GetFeatureDefs().items(): print(k,':',v)
Donor.SingleAtomDonor : [$([N&!H0&v3,N&!H0&+1&v4,n&H1&+0,$([$([Nv3](-C)(-C)-C)]),$([$(n[n;H1]),$(nc[n;H1])])]),$([O,S;H1;+0])] Acceptor.SingleAtomAcceptor : [$([O;H1;v2]),$([O;H0;v2;!$(O=N-*),$([O;-;!$(*-N=O)]),$([o;+0])]),$([n;+0;!X3;!$([n;H1](cc)cc),$([$([N;H0]#[C&v4])]),$([N&v3;H0;$(Nc)])]),$([F;$(F-[#6]);!$(FC[F,Cl,Br,I])])] NegIonizable.AcidicGroup : [C,S](=[O,S,P])-[O;H1,H0&-1] PosIonizable.BasicGroup : [$([$([N;H2&+0][$([C;!$(C=*)])])]),$([$([N;H1&+0]([$([C;!$(C=*)])])[$([C;!$(C=*)])])]),$([$([N;H0&+0]([$([C;!$(C=*)])])([$([C;!$(C=*)])])[$([C;!$(C=*)])])]);!$(N[a])] PosIonizable.PosN : [#7;+;!$([N+]-[O-])] PosIonizable.Imidazole : c1ncnc1 PosIonizable.Guanidine : NC(=N)N ZnBinder.ZnBinder1 : [S;D1]-[#6] ZnBinder.ZnBinder2 : [#6]-C(=O)-C-[S;D1] ZnBinder.ZnBinder3 : [#6]-C(=O)-C-C-[S;D1] ZnBinder.ZnBinder4 : [#6]-C(=O)-N-[O;D1] ZnBinder.ZnBinder5 : [#6]-C(=O)-[O;D1] ZnBinder.ZnBinder6 : [#6]-P(=O)(-O)-[C,O,N]-[C,H] Aromatic.Arom4 : [$([a;r4,!R1&r3])]1:[$([a;r4,!R1&r3])]:[$([a;r4,!R1&r3])]:[$([a;r4,!R1&r3])]:1 Aromatic.Arom5 : [$([a;r5,!R1&r4,!R1&r3])]1:[$([a;r5,!R1&r4,!R1&r3])]:[$([a;r5,!R1&r4,!R1&r3])]:[$([a;r5,!R1&r4,!R1&r3])]:[$([a;r5,!R1&r4,!R1&r3])]:1 Aromatic.Arom6 : [$([a;r6,!R1&r5,!R1&r4,!R1&r3])]1:[$([a;r6,!R1&r5,!R1&r4,!R1&r3])]:[$([a;r6,!R1&r5,!R1&r4,!R1&r3])]:[$([a;r6,!R1&r5,!R1&r4,!R1&r3])]:[$([a;r6,!R1&r5,!R1&r4,!R1&r3])]:[$([a;r6,!R1&r5,!R1&r4,!R1&r3])]:1 Aromatic.Arom7 : [$([a;r7,!R1&r6,!R1&r5,!R1&r4,!R1&r3])]1:[$([a;r7,!R1&r6,!R1&r5,!R1&r4,!R1&r3])]:[$([a;r7,!R1&r6,!R1&r5,!R1&r4,!R1&r3])]:[$([a;r7,!R1&r6,!R1&r5,!R1&r4,!R1&r3])]:[$([a;r7,!R1&r6,!R1&r5,!R1&r4,!R1&r3])]:[$([a;r7,!R1&r6,!R1&r5,!R1&r4,!R1&r3])]:[$([a;r7,!R1&r6,!R1&r5,!R1&r4,!R1&r3])]:1 Aromatic.Arom8 : [$([a;r8,!R1&r7,!R1&r6,!R1&r5,!R1&r4,!R1&r3])]1:[$([a;r8,!R1&r7,!R1&r6,!R1&r5,!R1&r4,!R1&r3])]:[$([a;r8,!R1&r7,!R1&r6,!R1&r5,!R1&r4,!R1&r3])]:[$([a;r8,!R1&r7,!R1&r6,!R1&r5,!R1&r4,!R1&r3])]:[$([a;r8,!R1&r7,!R1&r6,!R1&r5,!R1&r4,!R1&r3])]:[$([a;r8,!R1&r7,!R1&r6,!R1&r5,!R1&r4,!R1&r3])]:[$([a;r8,!R1&r7,!R1&r6,!R1&r5,!R1&r4,!R1&r3])]:[$([a;r8,!R1&r7,!R1&r6,!R1&r5,!R1&r4,!R1&r3])]:1 Hydrophobe.ThreeWayAttach : [D3,D4;$([#6;+0;!$([#6;$([#6]~[#7,#8,#9])])])] Hydrophobe.ChainTwoWayAttach : [R0;D2;$([#6;+0;!$([#6;$([#6]~[#7,#8,#9])])])] LumpedHydrophobe.Nitro2 : [N;D3;+](=O)[O-] LumpedHydrophobe.RH6_6 : [$([r6,!R1&r5,!R1&r4,!R1&r3]);$([R;$([c,s,S&H0&v2,Br,I,$([#6;+0;!$([#6;$([#6]~[#7,#8,#9])])])])])]1[$([r6,!R1&r5,!R1&r4,!R1&r3]);$([R;$([c,s,S&H0&v2,Br,I,$([#6;+0;!$([#6;$([#6]~[#7,#8,#9])])])])])][$([r6,!R1&r5,!R1&r4,!R1&r3]);$([R;$([c,s,S&H0&v2,Br,I,$([#6;+0;!$([#6;$([#6]~[#7,#8,#9])])])])])][$([r6,!R1&r5,!R1&r4,!R1&r3]);$([R;$([c,s,S&H0&v2,Br,I,$([#6;+0;!$([#6;$([#6]~[#7,#8,#9])])])])])][$([r6,!R1&r5,!R1&r4,!R1&r3]);$([R;$([c,s,S&H0&v2,Br,I,$([#6;+0;!$([#6;$([#6]~[#7,#8,#9])])])])])][$([r6,!R1&r5,!R1&r4,!R1&r3]);$([R;$([c,s,S&H0&v2,Br,I,$([#6;+0;!$([#6;$([#6]~[#7,#8,#9])])])])])]1 LumpedHydrophobe.RH5_5 : [$([r5,!R1&r4,!R1&r3]);$([R;$([c,s,S&H0&v2,Br,I,$([#6;+0;!$([#6;$([#6]~[#7,#8,#9])])])])])]1[$([r5,!R1&r4,!R1&r3]);$([R;$([c,s,S&H0&v2,Br,I,$([#6;+0;!$([#6;$([#6]~[#7,#8,#9])])])])])][$([r5,!R1&r4,!R1&r3]);$([R;$([c,s,S&H0&v2,Br,I,$([#6;+0;!$([#6;$([#6]~[#7,#8,#9])])])])])][$([r5,!R1&r4,!R1&r3]);$([R;$([c,s,S&H0&v2,Br,I,$([#6;+0;!$([#6;$([#6]~[#7,#8,#9])])])])])][$([r5,!R1&r4,!R1&r3]);$([R;$([c,s,S&H0&v2,Br,I,$([#6;+0;!$([#6;$([#6]~[#7,#8,#9])])])])])]1 LumpedHydrophobe.RH4_4 : [$([r4,!R1&r3]);$([R;$([c,s,S&H0&v2,Br,I,$([#6;+0;!$([#6;$([#6]~[#7,#8,#9])])])])])]1[$([r4,!R1&r3]);$([R;$([c,s,S&H0&v2,Br,I,$([#6;+0;!$([#6;$([#6]~[#7,#8,#9])])])])])][$([r4,!R1&r3]);$([R;$([c,s,S&H0&v2,Br,I,$([#6;+0;!$([#6;$([#6]~[#7,#8,#9])])])])])][$([r4,!R1&r3]);$([R;$([c,s,S&H0&v2,Br,I,$([#6;+0;!$([#6;$([#6]~[#7,#8,#9])])])])])]1 LumpedHydrophobe.RH3_3 : [$([r3]);$([R;$([c,s,S&H0&v2,Br,I,$([#6;+0;!$([#6;$([#6]~[#7,#8,#9])])])])])]1[$([r3]);$([R;$([c,s,S&H0&v2,Br,I,$([#6;+0;!$([#6;$([#6]~[#7,#8,#9])])])])])][$([r3]);$([R;$([c,s,S&H0&v2,Br,I,$([#6;+0;!$([#6;$([#6]~[#7,#8,#9])])])])])]1 LumpedHydrophobe.tButyl : [C;!R](-[CH3])(-[CH3])-[CH3] LumpedHydrophobe.iPropyl : [CH;!R](-[CH3])-[CH3]
*2:RDKit Documentation
Chemical Featrues and Pharmacophore
Feature Definition File Format
*3:RDKit Python API rdkit.Chem.rdMolChemicalFeatures module
*4: RDKitブログ記事Using Feature Maps
*7:* Web版のHelepページに図は自由に利用してOKと書いてありました。
citatition:
- SMARTS viewer https://smartsview.zbh.uni-hamburg.de
- Schomburg, H.-C. Ehrlich, K. Stierand, M.Rarey; From Structure Diagrams to Visual Chemical Patterns, J. Chem. Inf. Model., 2010, 50 (9), pp 1529-1535
*8: Prof. RareyはBioSolveITという会社のファウンダーでもあるそうです。
共結晶構造でPLIFを作ろうとした話
これまで化合物ライブラリーの①指標での絞り込み、②部分構造での絞り込みを行なって来ました。今まではリガンド側のことしか考えていなかったので、そろそろタンパク質との関係を考慮に入れたいと思います。ですが、いきなりドッキング(?)はハードルが高いので、まずは共結晶構造をもとに重要な相互作用を確認することから始めたいと思います。
以前、RCSB PDBのviewerを使ってリガンド-受容体相互作用を眺めて遊びました。こんな図が見られます。(PDB id: 5J89)*1

こういう情報をなんとか格好いい感じで利用したい!!ということで色々と検索していると似たような相互作用解析方法を見つけました。
Protein-Ligand Interaction Profiler(PLIP)というものです。 文献はこちらNucleic Acids Res. 2015(43)W443-447
いい感じで相互作用を解析してくれるみたいなのでPLIPで遊んでみたいと思います。
PLIP?
まずはページの見た目から・・・こんなページです。

試しに上の複合体(PDB id: 5J89)を投げ込んで見ます。

相互作用を形式に分けてリストアップしてくれるみたいです。結果をダウンロードしてPyMolで眺めることもできるそうです。プロファイラー格好良い!
認識される相互作用
Supplementary Informationによるとデフォルトで以下のような非共有結合性の相互作用を認識してくれるそうです。
| 相互作用 | 基準 | 変数 | |
|---|---|---|---|
| 疎水性相互作用 | Hydrophobic interacrtions | 距離 (4.0Å以下) | HYDROPH_DIST_MAX |
| 水素結合 | Hydrogen Bonds | 距離 (4.1Å以下) 角度 (100°以上) |
HBOND_DIST_MAX HBOND_DON_ANGLE_MIN |
| π-πスタッキング | Aromatic Stacking | 距離 (7.5Å以下) 角度 (T-stacking 90°±30°、 P-stacking 180°±30°) 環の中心同士の距離 (2.0Å以下) |
PISTACK_DIST_MAX PISTACK_ANG_DEV PISTACK_OFFSET_MAX |
| π-カチオン相互作用 | Pi-Cation interactions | 距離 (6.0Å以下) (3級アミンは角度も) |
PICATION_DIST_MAX |
| 塩橋 | Salt Bridges | 電荷の中心間の距離 (5.5Å以下) | SALTBRIDGE_DIST_MAX |
| 水を介した水素結合 | Water-brodged hydrogen bonds | 水分子の位置 (2.5Å~4.0Å) 角度2つ (75°<ω<140°, 100°<θ) ) |
WATER_BRIDGE_MINDIST, WATER_BRIDGE_MAXDIST WATER_BRIDGE_OMEGA_MIN, WATER_BRIDGE_OMEGA_MAX WATER_BRIDGE_THETA_MIN |
| ハロゲン結合 | Halogen bonds | 距離 (4.0Å以下) 角度 (Donor 165°±30°, Acceptor 120°±30°) |
HALOGEN_DIST_MAX, HALOGEN_DON_ANGLE, HALOGEN_ACC_ANGLE, HALOGEN_ANG_DEV |
自分のPCにインストール
コマンドラインからも使えるそうなのでインストールしてみます。
Githubの説明によると原子の属性の判別にOpenBabelを使っているそうで、OpenBabel(>= 2.3.2)のインストールが必要だそうです。他にはオプションですが
- PyMOL(>=1.7.x with Python bindings)
- Imagemagick(>=1.7.x)
- swig
などに依存しているそうです。
pip install plip
とすることでインストールできますが、Python 2.7.x.で実行してくださいとのことで、pip3としてみたらOpenBabelのwheelがどうのこうのというエラーで止まりました。
解析対象の共結晶構造
今回、相互作用解析を行いたい結晶構造はPD-L1と低分子の共結晶構造です。以前、UniProtの情報をもとに作成した記事から、低分子のものを取り出すと以下となります。
| PDB entry | Resolution (Å) | Chain | Positions | リガンド | リガンドID | 文献 |
|---|---|---|---|---|---|---|
| 5J89 | 2.20 | A/B/C/D | 2-134 | BMS-202 | 6GX | Oncotarget 2016(7)30323 |
| 5J8O | 2.30 | A/B | 18-134 | BMS-8 | 6GZ | Oncotarget 2016(7)30323 |
| 5N2D | 2.35 | A/B/C/D | 2-134 | BMS-37 | 8J8 | J. Med. Chem. 2017(60)5857 |
| 5N2F | 1.70 | A/B | 18-134 | BMS-200 | 8HW | J. Med. Chem. 2017(60)5857 |
| 5NIU | 2.01 | A/B/C/D | 18-134 | BMS-1001 | 8YZ | Oncotarget 2017(8)72167 |
| 5NIX | 2.20 | A/B/C/D | 18-134 | BMS-1166 | 8YQ | Oncotarget 2017(8)72167 |
登録されている低分子の構造がおかしいのではないか?といった問題はありますが、今回はそのあたりは無視します。(ご興味のある方は以前の記事をご参照いただければと思います。記事1 記事2 記事3)
解析実行
PLIPはPDB idさえあればPDBのサーバーからデータをとってきて解析してくれるとのことなので、以下のコマンドをターミナルで実行しました。 (Pythonのパス?の通し方がわからなくてJupyter notebookから使えなかった・・・)
alias plip='python ~/pliptool/plip/plipcmd.py' plip -i 5J89 -x
「-i PDBID」で解析したい構造のIDを、「-x」とすることで結果をXMLレポートファイルで出力できます。
このまま実行しようとしたところ、以下のようなエラーが出て来ました。
File "/usr/local/lib/python3.7/site-packages/plip/modules/supplemental.py", line 388, in read_pdb resource.setrlimit(resource.RLIMIT_STACK, (min(2 ** 28, maxsize), maxsize)) ValueError: current limit exceeds maximum limit
よくわからないので該当の箇所を確認して見ました。
def read_pdb(pdbfname, as_string=False): """Reads a given PDB file and returns a Pybel Molecule.""" pybel.ob.obErrorLog.StopLogging() # Suppress all OpenBabel warnings if os.name != 'nt': # Resource module not available for Windows maxsize = resource.getrlimit(resource.RLIMIT_STACK)[-1] resource.setrlimit(resource.RLIMIT_STACK, (min(2 ** 28, maxsize), maxsize)) sys.setrecursionlimit(10 ** 5) # increase Python recoursion limit return readmol(pdbfname, as_string=as_string)
PDBファイルを読み込むための関数のようです。Pythonライブラリのresourceの説明を見るかぎり、エラーとなっている箇所はプログラムによって使用されているシステムリソースを制限する処理のようです。PDBファイルを読み込むリソースの制限なら外してしまっても良さそうなので、コメントアウトして見ました。
def read_pdb(pdbfname, as_string=False): """Reads a given PDB file and returns a Pybel Molecule.""" pybel.ob.obErrorLog.StopLogging() # Suppress all OpenBabel warnings #if os.name != 'nt': # Resource module not available for Windows #maxsize = resource.getrlimit(resource.RLIMIT_STACK)[-1] #resource.setrlimit(resource.RLIMIT_STACK, (min(2 ** 28, maxsize), maxsize)) sys.setrecursionlimit(10 ** 5) # increase Python recoursion limit return readmol(pdbfname, as_string=as_string)
保存して再度実行したらエラーで止まらずに動き、無事XMLファイルが出力されました。こんな雑なことしていいのかよくわかりませんが、とりあえず動いたので良しとします。
RDKitと組み合わせてPLIFを計算
共結晶構造、全6構造に対して実行し、相互作用の情報を持つXMLファイルを手に入れたもののこれをどうしよう???と思っていたところOxford Protein Informatics Group(OPIG)のブログ記事(How to Calculate PLIFs Using RDKit and PLIP )に行き当たりました。PLIPのデータとRDKitを使ってProtein-Ligand interaction fingerprints (PLIFs)というのを計算できるそうです。
PLIF?
株式会社MOLSISのMOEの機能紹介にPLIFの説明がありました。
「リガンド-受容体間の相互作用の種類と強さをフィンガープリントで表現し、複数の結合状態を統計的に解析します。ドッキング結果や複数の複合体構造に含まれる相互作用を解析することで、活性/不活性に関連する相互作用の検出や、活性/不活性を分類する相互作用組み合わせルールの抽出、ルールに適合する活性ポーズに共通するファーマコフォアの検出などが行えます。」
化合物間の類似性評価にフィンガープリントが用いていましたが、タンパク質との相互作用解析にも拡張した、ということのようです。SAR News No.18の記事「実験と連携できる SBDD へ向けて」によるとPLIFの他にもSIFt(JMC2004(47)337)、aPLIF、aPLIED、Pharm-IF(JCIM2010(50)170)といった手法があるようです。*2
OPIGの真似をする
OPIGで紹介されていた方法は、
- PLIPの解析結果を使って結合サイトに含まれるアミノ酸残基と、リガンドとの相互作用に関わる残基を抜き出す
- 1.の情報をもとにRDKitでフィンガープリントにする
- 2.のフィンガープリントで複合体間の相互作用の類似性を評価
といった流れでした。
早速OPIGのコードをコピペしていきます。(日本語の部分は備忘録として私が加えたものです。)
解析ステップ1
PLIPの解析結果(XMLファイル)から情報を取得します。
# XMLを扱う標準ライブラリ(ElementTree)を使う import xml.etree.ElementTree as ET # 結合サイトのアミノ酸残基情報を取り出すための関数を定義 def generate_plif_lists(report_file, residue_list, lig_ident): # uses report.xml from PLIP to return list of interacting residues # and update list of residues in binding site plif_list_all = [] tree = ET.parse(report_file) #ファイルの読み込み root = tree.getroot() #ルート要素を取得する # list of residue keys that form an interaction for binding_site in root.findall('bindingsite'): #findall()で直接の子要素を検索 nest = binding_site.find('identifiers') #find()で最初の子要素にアクセス lig_code = nest.find('hetid') #<hetid>タグに記載されたリガンドIDを取得 if str(lig_code.text) == str(lig_ident): #関数の引数に与えたlig_identとリガンドIDが等しい時だけ情報を取得 #get the plifs stuff here nest_residue = binding_site.find('bs_residues') #結合サイトに含まれるアミノ酸残基のリストを取得 residue_list_tree = nest_residue.findall('bs_residue') for residue in residue_list_tree: res_id = residue.text dict_res_temp = residue.attrib #要素の属性(attribute)を取得 #結合サイトの残基一覧に含まれていない場合は残基を追加する if res_id not in residue_list: residue_list.append(res_id) #リガンドとの相互作用(contact)TRUEのものだけPLIFリストに追加 if dict_res_temp['contact'] == 'True': if res_id not in plif_list_all: plif_list_all.append(res_id) return plif_list_all, residue_list
では定義した関数を使いましょう。まずは結合サイトに含まれるアミノ酸残基(リガンドとの相互作用の有無は関係なし)を格納するための空のリストを作っておきます。
bs_res_list = []
PDBの6つの構造についてPDB IDと、リガンドIDの組み合わせの辞書を作ります。
P_L_dict = {"5J89":"6GX","5J8O":"6GZ","5N2D":"8J8",
"5N2F":"8HW","5NIU":"8YZ","5NIX":"8YQ"}
contact_res_dict = {}
for pdb_id, lig_id in P_L_dict.items():
pl = "conres_" + pdb_id
# xmlファイルは各PDB ID名のフォルダにそれぞれ格納した
xml_path = "PLIP_results/" + pdb_id + "/report.xml"
pl, bs_res_list = generate_plif_lists(xml_path, bs_res_list, lig_id)
contact_res_dict[pdb_id] = pl
得られた辞書(contact_res_dict)はPDB IDをキーとして、リガンドと接触する残基を値としています。
print(contact_res_dict.keys()) # dict_keys(['5J89', '5J8O', '5N2D', '5N2F', '5NIU', '5NIX']) print(contact_res_dict['5J89']) # ['66B', '56B', '121B', '121A', '56A', '115A', '54B', '123A', '115D', '121D', '115C', '121C', '124C', '54D', '56D', '123C', '20C', '56C', '66D']
結合サイトに含まれるアミノ酸残基の全体はbs_res_listに格納されています。
print(len(bs_res_list)) # 136
136個と非常に多いように思います。今回用いた共結晶構造ではPD-L1の2量体に対してリガンドが1つ結合しています。配列の位置としては等しくても、CHAIN名が異なるものを別の残基としてカウントしているため、多くなっているのではないかと思います。
解析ステップ2
残基のリストが得られたのでRDKitのPLIFとします。
以下の関数では結合サイトに含まれるアミノ酸残基全体の長さの次元のビットベクトルを用意し、相互作用に関わっている残基についてビットを立てます。
from rdkit import Chem, DataStructs from rdkit.DataStructs import cDataStructs def generate_rdkit_plif(residue_list, plif_list_all): #generates RDKit plif given list of residues in binding site and list of interacting residues plif_rdkit = DataStructs.ExplicitBitVect(len(residue_list), False) for index, res in enumerate(residue_list): if res in plif_list_all: # print('here') もともとのコードにはあったが面倒なのでコメントアウト plif_rdkit.SetBit(index) else: continue return plif_rdkit rdkit_plif_dict = {} for pdb_id in contact_res_dict.keys(): con_res = contact_res_dict[pdb_id] generated_plif = generate_rdkit_plif(bs_res_list, con_res) rdkit_plif_dict [pdb_id] = generated_plif print(rdkit_plif_dict.keys()) # dict_keys(['5J89', '5J8O', '5N2D', '5N2F', '5NIU', '5NIX'])
6つとも計算されていそうです。一つ取り出して確認してみます。
print(len(rdkit_plif_dict['5J89'])) # 136 print(rdkit_plif_dict['5J89']) # <rdkit.DataStructs.cDataStructs.ExplicitBitVect object at 0x10d246d88>
長さ136のベクトルが生成されています。ExplictiBitVectオブジェクトの扱い方は「化学の新しいカタチ」さんのこちらの記事RDKitでフィンガープリントを使った分子類似性の判定に記載されていました。
どんな情報が含まれているのか見てみます。
print("全体の長さ:", rdkit_plif_dict['5J89'].GetNumBits()) # 全体の長さ: 136 print("ONビット:", list(rdkit_plif_dict['5J89'].GetOnBits())) print("ONビットの数:", rdkit_plif_dict['5J89'].GetNumOnBits()) print("OFFビットの数:",rdkit_plif_dict['5J89'].GetNumOffBits()) print("相互作用残基の数:",len(contact_res_dict['5J89'])) # ONビット: [5, 9, 14, 17, 20, 23, 33, 36, 45, 46, 49, 58, 64, 73, 77, 79, 80, 82, 84] # ONビットの数: 19 # OFFビットの数: 117 # 相互作用残基の数: 19
PDB id 5J89における相互作用に関わる残基の数とRDKitで変換後のONビットの数が一致しています。
DataFrameとheatmapで可視化
ビットベクトルをもう少しわかりやすく眺めたいと思います。目標は先に見た株式会社MOLSISのページのページのように複数の共結晶構造間におけるONビットの位置の違いを並べて可視化することです。
- BitVectをnumpyのarrayに変換
- データフレームに変換
- heatmapで図示
という手順を行ってみます。
import numpy as np # DataFrameのindex としてPDBのIDを使うためにリストを作成 PDB_id_list = [] finger_print_arrays = [] for pdb_id in rdkit_plif_dict.keys(): PDB_id_list.append(pdb_id) # RDKitのBitVectそのままではDataFrameにできなさそうなのでarrayに変換する。 fp = rdkit_plif_dict[pdb_id] arr = np.zeros((1,)) DataStructs.ConvertToNumpyArray(fp, arr) finger_print_arrays.append(arr) import pandas as pd #DataFrameを作成 FP_df = pd.DataFrame(finger_print_arrays, index=PDB_id_list) FP_df

うまくできていそうです。0ばっかり・・・これがスパースという奴か!!!(適当)
可視化します。
import matplotlib as mpl import matplotlib.pyplot as plt import seaborn as sns %matplotlib inline sns.heatmap(FP_df)

おお!それっぽい!y軸は各複合体(PDB id)で、x軸方向にビットベクトルが並んでいます。各ビットが結合サイトに含まれるアミノ酸残基相当し、黒いところがビットベクトルが立っているリガンドと相互作用する残基となっています。
(私の理解が正しければ・・・X軸方向の並びがアミノ酸配列の並びと正確に一致するかはわかりません。)
各複合体で共通してビットが立っているアミノ酸残基が、複数の複合体で相互作用が維持されている重要な残基と解釈できる・・・はず?
図から雑に解釈
上の図をみると5J8O、5N2FがもっともONビットの分散が小さく、5NIU、5NIXがよりベクトル全体に広がってビットが立っています。
先に、今回のPLIFの方法ではCHAIN毎に区別を行っていると述べました。6つの共結晶構造は全てPD-L1の2量体とリガンド1つとの2対1の複合体ですが、より詳しく見ると
- 5J8O、5N2Fは「PD-L1 2: Ligand 1」
- 5J89、5N2D、5NIU、5NIXは「PD-L1 4: Ligand 2」
という違いがあります。タンパク質側のCHAINが4つ含まれている他の複合体と比較して2つしか含まれていない5J8O、5N2Fではとりうるビットベクトルの範囲がそもそも小さいため以上のような結果となっていそうです。
また、5NIU、5NIXでは結合しているリガンドが、他の複合体よりも大きいものとなっています。リガンドが大きい分相互作用する残基が広がっているのかもしれません。
一覧を再掲します。
| PDB entry | Chain | リガンド | リガンドID | リガンド分子量 |
|---|---|---|---|---|
| 5J89 | A/B/C/D | BMS-202 | 6GX | 419.52 |
| 5J8O | A/B | BMS-8 | 6GZ | 494.42 |
| 5N2D | A/B/C/D | BMS-37 | 8J8 | 448.55 |
| 5N2F | A/B | BMS-200 | 8HW | 497.49 |
| 5NIU | A/B/C/D | BMS-1001 | 8YZ | 594.65 |
| 5NIX | A/B/C/D | BMS-1166 | 8YQ | 643.13 |
だいたい分子量とビットの広がりが相関があるような気もします。なんとなく3つのグループ(①5J89と5N2D、②5J8Oと5N2F、③5NIUと5NIX)に分かれそうです。
図をぼんやりと眺めていても定性的な類似性しかわかりませんので、定量的な評価を行いたいと思います。
解析ステップ3
フィンガープリント間の類似性評価をTanimoto係数を計算して比較します。関数をコピペ・・・
def similarity_plifs(plif_1, plif_2): sim = DataStructs.TanimotoSimilarity(plif_1, plif_2) print(sim) return sim
6つの構造から2つ取り出してTanimoto係数を計算しますが、一つずつやるのは大変なのでitertoolsを使ってみます。
import itertools # 重複無しの順列 itertools.permutations(PDB_id_list, 2) # 重複順列 itertools.product(PDB_id_list, repeat = 2) # 今回は可視化のために重複ありの組み合わせを使う tanimoto_list = [] for v in itertools.combinations_with_replacement(PDB_id_list, 2): #6つから2つ取り出しi, j とする。 i, j = v[0], v[1] plif_i = rdkit_plif_dict[i] plif_j = rdkit_plif_dict[j] sim = similarity_plifs(plif_i, plif_j) #用いた2つの構造とTanimoto係数をもつリストを作成 temp_list = [i, j, sim] #リストのリストを作成 tanimoto_list.append(temp_list) #DataFrameとする tanimoto_df = pd.DataFrame() for k in tanimoto_list: tanimoto_df.at[k[0], k[1]] = k[2] #heatmapで可視化 sns.heatmap(tanimoto_df)


色の濃い組み合わせを見ると
- 5J89、5N2D、0.63
- 5J8O、5N2F、0.42
- 5NIU、5NIZ、0.56
となっています。ビットベクトルをそのまま可視化した際の定性的な解釈と似た結果となっており、うまく数値化できていそうです。
残基番号のみで再解析
ところで、共結晶構造を見る限りリガンドを挟んでPD-L1の二量体が対称的に配置されているように見えます。対称的ならば配列の番号が大事で、CHAINを区別していることでむしろ同じ相互作用を別のものと判断しているかもしれません。そこでCHAINの情報を取り除いて同じことをやってみたいと思います。
・・・といってもStep 1で取り出した残基の情報から最後のアルファベットを除くだけですが。。。
# 結合サイトの残基からCHAINの情報を除く bs_res_list_num = [] for i in bs_res_list: #文字列から最後の一文字の手前まで取り出す j = i[:-1] #後でソートしたいので整数型に変換してからリストに加える bs_res_list_num.append(int(j))
重複している情報を取り除き、ソートします。
bs_res_list_num_unique = sorted(set(bs_res_list_num)) print(len(bs_res_list_num_unique)) print(bs_res_list_num_unique) # 40 # [18, 19, 20, 21, 22, 23, 26, 53, 54, 55, 56, 57, 58, 60, 61, 62, 63, 65, 66, 67, 68, 73, 75, 76, 77, 78, 112, 113, 114, 115, 116, 117, 118, 120, 121, 122, 123, 124, 125, 126]
相互作用の残基からCHAINの情報を除きます。今回は重複も先に除きます。
contact_res_dict_num_unique = {}
for k, v in contact_res_dict.items():
tmp_list = []
for i in v:
j = i[:-1]
tmp_list.append(int(j))
tmp_unique = sorted(set(tmp_list))
contact_res_dict_num_unique[k] = tmp_unique
ビットのリストを取得
RDKitのBitVectを介してからnumpyのarrayに戻すと残基番号の情報が失われてしまうので、OPIGの関数を参考に新しくビットのリストを返す関数を作成してみたいと思います。
def PLIF_list_generator(bs_residues, contact_residues): tmp = [] for i in bs_residues: if i in contact_residues: tmp.append(1) else: tmp.append(0) return tmp
PDB idを行、残基番号を列とするDataFrameを作成します。
FP_df_num = pd.DataFrame(index =PDB_id_list, columns = bs_res_list_num_unique) for k, v in contact_res_dict_num_unique.items(): FP_df_num.loc[k] = PLIF_list_generator(bs_res_list_num_unique, v)

複数の共結晶構造で各残基が相互作用に使われているかを確認したいので、残基ごとのビットの足し算も行っておきます。
FP_df_num.loc['bit_sum']=FP_df_num.sum() %matplotlib inline sns.heatmap(FP_df_num)

なんだかいい感じになってきました!
大事そうな残基を探す
先のBitVectを眺めた際の解釈で、6つの共構造は3つのグループに分かれそうだということでした。ということはbit_sumが5以上のものは3つのグループのすべてで少なくとも一度は使われている残基です。(たぶん・・・)(最初4以上にしていましたが5以上の間違いでした)
取り出して見ましょう。
# queryメソッドは行の抽出なので転置してから使う # bitの合計5以上を取り出し、indexをリスト化 Frequent_residues = list(FP_df_num.T.query('bit_sum >= 5').index) print(Frequent_residues) #[54, 56, 115, 121, 123]
これらは具体的にどんな相互作用なのでしょうか?すべての残基を含んでいそうな「PDB id:5NIX(Ligand:8YQ)」をPLIPのサイトで解析してみましょう。(コマンドラインでの解析を諦めた)
「PD-L1 4: Ligand 2」の複合体なのでリガンドが2つ含まれています。結果をテーブルにまとめました。
| 残基番号 | アミノ酸 | 距離 | 相互作用 | 残基番号 | アミノ酸 | 距離 | 相互作用 | ||
|---|---|---|---|---|---|---|---|---|---|
| 8YQ(chain A/B) | 8YQ(chain C/D) | 54C | ILE | 3.89 | Hydrophobic | ||||
| 56B | TYR | 3.82 | Hydrophobic | 56C | TYR | 3.42/3.62 | Hydrophobic | ||
| 115A/B | MET | 3.70/3.80 | Hydrophobic | 115C/D | MET | 3.85/3.82 | Hydrophobic | ||
| 121A/B | ALA | 3.69/3.75 | Hydrophobic | 121C/D | ALA | 3.96/3.57 | Hydrophobic | ||
| 123A | TYR | 3.80/3.52/3.85 | Hydrophobic | 123D | TYR | 3.72 | Hydrophobic |
リガンドの芳香環が目立っていたのでπ-π相互作用ばかりかと思っていましたが、予想外にメチオニンやアラニンとの相互作用が複数見られました。
もう少し広くカウント数4以上の残基として見ます。
# bitの合計4以上の場合 Frequent_residues4 = list(FP_df_num.T.query('bit_sum >= 4').index) print(Frequent_residues4) # [54, 56, 66, 115, 121, 123, 124]
先の結果に66、124が加わりました。アミノ酸残基でいうと以下となります。
| 残基番号 | アミノ酸 | 距離 | 相互作用 | 残基番号 | アミノ酸 | 距離 | 相互作用 | ||
|---|---|---|---|---|---|---|---|---|---|
| 8YQ(chain A/B) | 66B | GLN | 3.72 | Hydrophobic | 8YQ(chain C/D) | ||||
| 124A | LYS | 5.03/3.18 | π-cation/Salt Bridges | 124D | Lys | 3.70 | Water Bridges/Salt Bridges |
疎水性相互作用ばかりだったところに親水性残基のLysが加わりました。こちらの方がバラエティがあって良さそうです。
PLIPのスナップショットを貼っておきます。こんな相互作用です・・・

まとめ
以上、今回はタンパク質とリガンドの相互作用情報を考察するためPLIP、PLIFといった手法を導入して見ました(コピペですが・・・)。この情報をうまく使えばファーマコフォア(?)を考察できるはず・・・?
色々と間違いがあると思うのでご指摘いただければ幸いです。
*1:AS Rose et al. (2018) NGL viewer: web-based molecular graphics for large complexes. Bioinformatics dio:10.1093/bioinformatics/bty419
オルト位置換基で絞り込む話
前回の記事で、SMARTS記法の扱い方がなんとなくわかりました。本題にもどって、ライブラリをオルト位に置換基の入ったビフェニルで絞り込みたいと思います。
その前に、現在残っている化合物数の確認です。
| Enamine_Premium | Enamie_Advenced | Enamine_HTS | UOS_HTS | total | |
|---|---|---|---|---|---|
| 指標でのフィルタリング後 | 4060 | 37431 | 414562 | 106948 | 563001 |
| ビフェニル有 | 12 | 329 | 3182 | 697 | 4220 (0.7%) |
| ビフェニル無 | 4048 | 37102 | 411380 | 106251 | 558781 (99.3%) |
合計4000個程度残っています。このうちオルト位に置換基が入ったものはいくつあるでしょうか?
オルト位置換基による絞り込み
PandasToolsでSDFをよみこむ
4種類のライブラリから抽出した、ビフェニルを部分構造として含む化合物のリストを連結(extend()を使った)し、SDFファイル「biphenyl_library.sdf」として保存しました。こちらをRDKitのPandasToolsをもちいてDataFrameでよみこみます。
PandasToolsの使い方に関しては化学の新しいカタチさんの記事「RDKitのPandasToolsでデータ分析を加速する」を参考にさせていただきました。
from rdkit import rdBase, Chem from rdkit.Chem import AllChem, Draw from rdkit.Chem import PandasTools import pandas as pd biphenyl_df = PandasTools.LoadSDF('./biphenyl_library.sdf') # 数を確認 len(biphenyl_df) # 4220
こんな感じになりました。

Pandas上で部分構造検索
PandasToolsを使うと「>=(ge比較演算子)」で部分構造検索が行えるとのことですので試しに利用して見ます
ortho_biphenyl= Chem.MolFromSmarts('c1ccc(c(*)c1)c1ccccc1') ortho_df = biphenyl_df[biphenyl_df['ROMol'] >= ortho_biphenyl] len(ortho_df) # 2353
オルト位に置換基を持つ化合物は約半数程度の2353個でした。
一致検索後のDataFrameでは、Molオブジェクトを格納したカラムにおいて、一致した部分構造が赤色でハイライトされて表示されています。

忘れずにSDFに書き出しておきます。SDFに残すプロパティを指定するため、DataFrameのカラム名をリストとして取得します。
property_names = list(biphenyl_df.columns) print(property_names) # ['ID', 'MW', 'MolLogP', 'NumHAcceptors', 'NumHDonors', 'NumRotatableBonds', 'ROMol', 'TPSA', 'original_id']
「ROMol」は不要なのでリストから除きます。
property_names.remove('ROMol') print(property_names) # ['ID', 'MW', 'MolLogP', 'NumHAcceptors', 'NumHDonors', 'NumRotatableBonds', 'TPSA', 'original_id']
出力します。
PandasTools.WriteSDF(ortho_df, 'ortho_biphenyl_library.sdf', properties = property_names)
以上で、オルト位に置換基をもつ部分構造での絞り込みが完了しました。
オルト位の置換基の構造的意義
なぜ「オルト位に置換基を持つ」ということを、絞り込みの基準として重要視するのか?ついでなので「オルト位に置換基を持つ」ということの意味について考えたいと思います。
・・・といっても立体障害でビフェニル骨格が捻れるというだけなのですが、、、、
3次元構造を眺める
オルト位の置換基の有無が3次元構造(コンフォメーション)にどのような影響を与えるか?ということを眺めて見ます。3次元構造の生成は「化学の新しいカタチ」さんの記事「RDKitによる3次元構造の生成」を参考にしました。
まずは構造を準備。無置換、オルト位にメチル基を1つもつもの、2つ持つものの3つを作りました。
biPh = Chem.MolFromSmiles('c1ccc(cc1)c1ccccc1') ortho_Me_biPh = Chem.MolFromSmiles('c1ccc(c(C)c1)c1ccccc1') ortho_diMe_biPh = Chem.MolFromSmiles('c1cc(C)c(c(C)c1)c1ccccc1') biPh_list = [biPh, ortho_Me_biPh, ortho_diMe_biPh] Draw.MolsToGridImage(biPh_list)

水素原子を付加し、ETKDG法で3次元構造を生成します。
biPh_3D_list = [] for mol in biPh_list: mol_H = AllChem.AddHs(mol) AllChem.EmbedMolecule(mol_H, AllChem.ETKDG()) biPh_3D_list.append(mol_H)
生成した構造を眺めます。
from rdkit.Chem.Draw import IPythonConsole import py3Dmol v = py3Dmol.view(width=600, height=300, linked=False, viewergrid=(1,3)) for m, i in zip(biPh_3D_list, [(0,0), (0,1), (0,2)]): mb = Chem.MolToMolBlock(m) v.addModel(mb, 'sdf', viewer=i) v.setBackgroundColor('0xeeeeee') v.setStyle({'stick':{}}) v.zoomTo() v.show()
無置換、モノ置換、ジ置換の順にねじれ角度が大きくなっているのがわかります。

もっとわかりやすくするために、重ねて見ます。
アラインメントをとって重ねる
RDKitブログの記事を参考にして3つとも重ねて見ます。
rdMolAlign.AlignMolを使って部分構造のアラインメントを取り3次元構造を重ね合わせます。重ねたい部分構造(template)を明示的に与えるため引数atomMapに無置換のベンゼン環に相当するatom numberを与えます。
こちらや、こちらを参考にエラーに対処していたらやたらとlist()を使うことになってしまった・・・
from rdkit.Chem import rdMolAlign # 無置換フェニルのテンプレートをSmartsから作成 template = Chem.MolFromSmarts('[cH1]1[cH1][cH1][cH1][cH1]c1-c') # テンプレートに一致する無置換ビフェニルの原子を基準にする # タプルのタプルで得られる template_atoms = biPh_3D_list[0].GetSubstructMatches(template) # アラインメントを取る for m in biPh_3D_list: matched_atoms = m.GetSubstructMatches(template) rdMolAlign.AlignMol(m, biPh_3D_list[0], atomMap = list(zip(list(matched_atoms[0]), list(template_atoms[0])))) # 描く v = py3Dmol.view(width=300, height=300) for m in biPh_3D_list: mb = Chem.MolToMolBlock(m) v.addModel(mb, 'sdf') v.setBackgroundColor('0xeeeeee') v.setStyle({'stick':{}}) v.zoomTo() v.show()


静止画ではわかりにくいですが、無置換、モノ置換、ジ置換の順に二面角(Torsion Angle)が大きくなっているのがわかります。
二面角を計算してみる
それぞれ一つずつコンフォマーを取得して二面角を計算して見ます。
まず二面角を計算するために、目的の部分のatom numberを確認します。無置換ビフェニルの場合・・・
Draw.MolToImage(biPh_3D_list[0], includeAtomNumbers = True)

atom numberがわかったので目的の4原子を指定して、2面角を求めます。
from rdkit.Chem import rdMolTransforms conf=biPh_3D_list[0].GetConformer(0) rdMolTransforms.GetDihedralDeg(conf, 2,3,6,7) # 148.88976772600805
モノ置換、ジ置換も同様に計算し、0° - 90°の範囲に値を修正すると以下のようになります。
| 無置換 | モノ置換 | ジ置換 | |
|---|---|---|---|
| 二面角 | 31° | 50° | 81° |
順に二面角が大きくなり、特にジ置換ではほぼ直角近くになっています。
フィンガープリントではどう認識される?
上記の二面角の差異がフィンガープリントとしてはどのように認識できるか、少し見てみたいと思います。Atom PairsとTopological Torsionsでそれぞれ類似性を比較した際にどのようになるか試してみます。
この二つを選んだ理由は名前の雰囲気です・・・(計算方法を理解していません。すみません)。
from rdkit import DataStructs from rdkit.Chem.AtomPairs import Pairs pair_fps = [Pairs.GetAtomPairFingerprint(mol) for mol in biPh_3D_list] from rdkit.Chem.AtomPairs import Torsions tts_fps = [Torsions.GetTopologicalTorsionFingerprintAsIntVect(mol) for mol in biPh_3D_list] # Tanimoto係数、Dice係数をそれぞれの組み合わせで計算 pair_Dice_0_1 = DataStructs.DiceSimilarity(pair_fps[0], pair_fps[1]) pair_Tani_0_1 = DataStructs.TanimotoSimilarity(pair_fps[0], pair_fps[1]) # 残りの組み合わせ、TorsionFingerprintについても同じ計算をしたので省略
結果をまとめると以下になりました。
| 無置換-モノ置換 | 無置換-ジ置換 | モノ置換-ジ置換 | |
|---|---|---|---|
| AtomPair_Tanimoto係数 | 0.76 | 0.59 | 0.77 |
| AtomPair_Dice係数 | 0.86 | 0.75 | 0.87 |
| Torsion_Tanimoto係数 | 0.87 | 0.77 | 0.88 |
| Torsion_Dice係数 | 0.93 | 0.86 | 0.94 |
Torsionの方が差異が大きくなると面白いと思っていたのですが、今回の結果ではAtom Pairの方が置換の数の差を反映しているようにも見えます。いずれにせよ、メチルの数が1つ増えるか2つ増えるかで類似度が下がっていくことに代わりわなさそうです。
オルト位置換を創薬の話に繋げようとする
オルト位に置換基を加えることで分子がねじれ、立体的な構造が変化するということがわかりました。では、このねじれを医薬品に活用した事例は???ということでFasiglifam(TAK-875)の構造を眺めて見たいと思います。2型糖尿病を適応として開発が行われていた武田薬品の化合物で、GPCRの一つGPR40の作動薬です。残念ながら臨床試験第三相で肝臓における安全性の懸念から開発中止となり、上市にはいたらなかったそうです。
Fasiglifam(TAK-875)の構造
なぜこちらを取り上げるのか? 構造を見ていただくとお分かりいただけると思います。 印象的なビフェニル、オルト-ジ置換構造を持っています。
Fasiglifam_mol = Chem.MolFromSmiles('CC1=CC(=CC(=C1C2=CC(=CC=C2)COC3=CC4=C(C=C3)[C@@H](CO4)CC(=O)O)C)OCCCS(=O)(=O)C') from rdkit.Chem.Draw import rdMolDraw2D from IPython.display import SVG highlights = Fasiglifam_mol.GetSubstructMatch(ortho_diMe_biPh) view = rdMolDraw2D.MolDraw2DSVG(500,500) pre_mol = rdMolDraw2D.PrepareMolForDrawing(Fasiglifam_mol) view.DrawMolecule(pre_mol, highlightAtoms=highlights) view.FinishDrawing() svg = view.GetDrawingText() SVG(svg.replace('svg:,',''))

Fasiglifamを見出した構造活性相関の研究に関していくつかの論文が出されています。ずいぶんと以前に読んだ論文のため引用しようにも忘れてしまいました(おそらくJ.Med.ChemやACS Med. Chem. Lett.だったような・・・)。その中でビフェニル構造に置換基を加えることで2面角が変化すること、その構造の変化が活性に大きな影響を与えたことが議論されていたように思います。(いろいろぼんやりとした記憶で不確かですみません・・・)
では立体的にはどのような構造なのか?眺めて見ます。PubChemのID「cid: 24857286」をつかってpy3Dmolで描画して見ます。
化学の新しいカタチさんの記事「py3Dmolを使って化学構造をJupyter上で美しく表示する」を参考にさせていただきました。
view = py3Dmol.view(width= 800, height=300, viewergrid=(1,3), query='cid:24857286') view.setStyle({'cartoon': {}}, viewer=(0,0)) view.setStyle({'stick': {}}, viewer=(0,1)) view.setStyle({'sphere': {}}, viewer=(0,2)) view.show()

ビフェニル構造がほぼ直角にねじれています。
受容体との共結晶構造
FasiglifamとGPR40との共結晶構造が解かれています(PDB id: 4PHU*1)。ついでに眺めて見ます。 *2*3
view = py3Dmol.view(width=680, height=480, query='pdb:4PHU', viewergrid=(1,1)) view.setStyle({'cartoon': {'color': 'spectrum'}}, viewer=(0,0)) view.setStyle({'resn': '2YB'}, {'sphere': {'colorsheme': 'default'}}, viewer=(0,0)) view.addSurface(py3Dmol.SES, {'opacity': 0.6, 'color': '#FADBD8'}, viewer=(0,0)) view.show()

とても興味深いことに、Fasiglifamはその構造の半分程度がGPR40のヘリックスの合間から外に向かって飛び出しています。
上の図では少しわかりにくいのでRCSD PDBのビューワーでリガンドと受容体の相互作用部位を拡大して眺めます。
こんな感じ・・・

ちょうど直角にねじれたビフェニル構造の部分が受容体内-外の境目に来ています。最近ではGPCRの構造が多数解かれるようになってきますが、この構造が公開された当時(2014年)はまだそこまで多くなく、さらにリガンドの結合様式も見たことがないものでこんな結合の仕方あるのか!!!と、とても驚いたのを覚えています。
まとめ
以上、今回はオルト位に置換基を含む化合物のライブラリからの抽出と、その構造的意義についてざっと眺めて見ました。オルト位に置換基を加えることでライブラリ化合物は2400弱にまで絞り込むことができました。後半、古い記憶を辿ってFasiglifamの話などをし始めたのは、ライブラリスクリーニングの方法に行き詰まったからです・・・困難から逃走するタイプ・・・
*1:Crystal structure of Human GPR40 bound to allosteric agonist TAK-875 DOI: 10.2210/pdb4PHU/pdb
*2:High-resolution structure of the human GPR40 receptor bound to allosteric agonist TAK-875. (2014) Nature 513: 124-127
*3:AS Rose et al. (2018) NGL viewer: web-based molecular graphics for large complexes. Bioinformatics dio:10.1093/bioinformatics/bty419